基于DNA序列与基因组信号特征的增强子-启动子相互作用预测方法
本发明属于生物信息学中的基因转录组学领域,具体涉及一种基于dna序列与基因组信号特征的增强子-启动子相互作用预测方法。
背景技术:
1、在机器学习与深度学习蓬勃发展的过去十几年中,生物信息学也经历了一个高速发展的时期,尤其是将生物信息学与深度学习结合在一起的领域取得了十分显著的成就。在生物信息学中的基因转录组学领域,增强子-启动子相互作用一直是一个十分值得研究的领域。虽然现已有许多对增强子-启动子相互作用的研究方法,但是大多都是只基于dna序列输入的研究或是只基于基因组信号特征的研究。目前还不存在结合dna序列和长距离基因组信号特征的双通道输入增强子-启动子相互作用研究。只基于dna序列或基因组信号特征研究的缺点是输入的特征信息不够丰富,无法最大化的利用已知信息来做出最准确的预测。因此如何结合dna序列信息和基因组信号特征,并且合理的进行特征融合,最终提高增强子-启动子相互作用的预测效果,仍然是一个需要解决的技术问题。
技术实现思路
1、为了克服以上技术问题,本发明提供了一种基于dna序列与基因组信号特征的增强子-启动子相互作用预测方法,将dna序列信息和基因组信号特征等不同输入结合起来以预测增强子-启动子相互作用。
2、本发明的目的将通过下面的详细描述来进一步说明。
3、本发明提供一种基于dna序列与基因组信号特征的增强子-启动子相互作用预测方法,包括如下步骤
4、其特征在于,包括以下步骤:
5、s1.数据预处理:构建增强子-启动子相互作用数据集,通过特定程序于相关基因库搜索得到对应dna序列。同时构建基因组表观信号数据集。
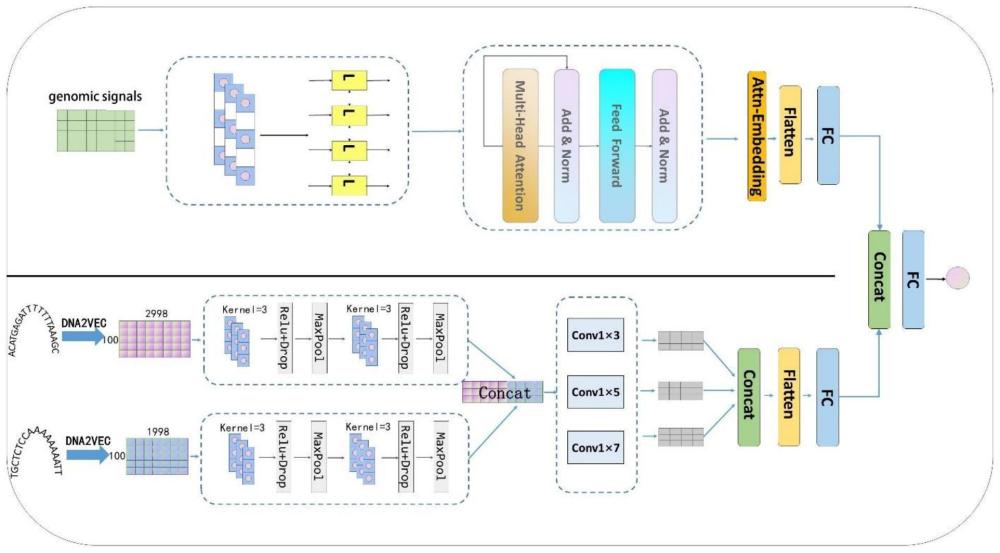
6、s2.构建增强子-启动子相互作用预测方法:分别通过网络架构中的基因组信号特征输入通道与dna序列输入通道处理完对应特征后,将两个通道的输出进行特征融合,最终输出增强子-启动子相互作用的预测值。
7、s3.交叉验证:针对构建出的增强子-启动子相互作用预测网络架构,将数据集根据五折交叉验证的原理进行分类,最终得到了五种训练集与验证集的组合。使用这五种组合分别对增强子-启动子相互作用预测网络架构进行训练与验证,最终可以得到最佳模型。
8、所述步骤s1中的特定程序于相关基因库搜索,包括如下步骤:
9、1)从增强子-启动子相互作用数据集分别取出增强子与启动子的区间,并计算得到增强子与启动子的中心位置。
10、2)在人类参考基因组中找到对应的染色体与染色体中对应的增强子、启动子的中心位置。
11、增强子分别向前和向后各取1500bp与1499bp长度,而启动子则分别向前和向后各取1000bp与999bp。最终分别得到3000bp长度的增强子序列与2000bp长度的启动子序列。进一步的,所述步骤s2中的增强子-启动子相互作用预测方法,包括如下步骤:
12、1)首先获取数据集中增强子序列、启动子序列,以及基因组表观信号数据集中对应区域的信号特征。
13、2)对于获取的增强子序列与启动子序列,通过dna2vec工具进行序列嵌入编码,可以得到能更好的表示短序列的向量。同时通过该方法得到的嵌入矩阵,可以更好的捕捉到dna序列中不同氨基酸之间的联系,更好的通过向量表达出来不同氨基酸之间的依赖关系。
14、3)将增强子与启动子的嵌入矩阵输入到深度学习网络架构中的dna序列输入通道;将基因组信号特征输入到网络架构中的基因组信号特征输入通道;通过深度学习网络提取出浅层和深层特征,最终将两个通道的不同输出特征矩阵合并到一起,通过全连接层后,
15、最终通过softmax函数输出最终的增强子-启动子相互作用预测结果。
16、进一步的,所述步骤s2中的深度学习网络架构,包括如下部分:
17、1)基因组信号特征通道:基因组信号特征通道首先由空洞卷积与双向长短期记忆网络组成浅层特征提取部分。然后通过transformer架构,提取长距离上的依赖特征,最终将提取后的特征输入到特征融合模块。
18、2)dna序列通道:首先通过卷积模块(一维卷积、relu激活函数、dropout函数、最大池化层)提取相应特征。然后通过多尺度卷积层分别提取不同尺度上的特征,最终将多个特征矩阵通过concat函数后输入到特征融合模块。
19、3)特征融合模块:该模块的输入分别来自于基因组信号特征通道与dna序列通道。通过将来自两个通道的一维特征进行concat连接后,通过多层全连接层后,最终通过softmax函数得到增强子-启动子相互作用预测概率。
20、通过以上这三个模块构建,就得到了深度学习网络架构。
21、进一步的,所述步骤s3中交叉验证的步骤包括:
22、1)数据集划分:将训练集进一步划分为5折。每个折都具有相同数量的样本,且尽可能保持样本之间的分布致性。5折交叉验证中的每一折都会被轮流作为验证集,而其他4个折则作为训练集。
23、2)模型训练与验证:对于每一次交叉验证,使用4个折的数据作为训练集,使用剩下的一个折作为验证集。在训练集上,根据增强子-启动子相互作用模型的算法和超参数进行模型训练,得到训练好的增强子-启动子相互作用模型。然后,在验证集上使用训练好的预测模型进行预测,并进行相关的性能评估。
24、与现有技术相比,本发明的有益效果包括:
25、1)本发明采用了dna序列数据集与基因组信号特征数据集,这两个不同的数据集能够从不同方面更好的表达出基因转录方面的信息,更好的完成预测。
26、2)本发明设计了双通道深度学习网络架构,网络架构中的两个通道采取了不同的深度学习网络,能更适合两个通道中不同的输入特征,使得dna序列信息的上下文信息与基因组信号特征的上下文信息能够更好的被提取,这为生物信息学中更好的研究增强子-启动子相互作用以及基因转录机制提供了新的见解与技术。
技术特征:
1.基于dna序列与基因组信号特征的增强子-启动子相互作用预测方法,其特征在于:包括如下步骤:
2.根据权利要求1所述的基于dna序列与基因组信号特征的增强子-启动子相互作用预测方法,其特征在于:所述步骤s1中的特定程序于相关基因库搜索,包括如下步骤:
3.根据权利要求1所述的基于dna序列与基因组信号特征的增强子-启动子相互作用预测方法,其特征在于:所述步骤s2中的增强子-启动子相互作用预测方法,包括如下步骤:
4.根据权利要求3所述的基于dna序列与基因组信号特征的增强子-启动子相互作用预测方法,其特征在于:所述步骤3中的深度学习网络架构,包括如下部分:
5.根据权利要求1中所述的基于dna序列与基因组信号特征的增强子-启动子相互作用预测方法,其特征在于:所述步骤s3中,交叉验证的步骤包括:
技术总结
本发明提供一种基于DNA序列与基因组信号特征的增强子‑启动子相互作用预测方法,包括如下步骤:1)数据预处理2)构建增强子‑启动子相互作用预测方法3)交叉验证。本发明属于生物信息学领域,本发明提供的基于DNA序列与基因组信号特征的增强子‑启动子相互作用预测方法,为基因转录组学领域由于输入不够丰富导致预测效果差、泛化性差的问题提供了解决方案。它以DNA序列与基因组信号特征同时输入到不同的通道中分别处理,最终完成预测任务。同时使用了交叉验证方法与独立测试集进行模型评估。这为增强子‑启动子相互作用研究领域提供了新的见解。
技术研发人员:宋弢,宋浩楠,王珣
受保护的技术使用者:中国石油大学(华东)
技术研发日:
技术公布日:2024/2/19
- 还没有人留言评论。精彩留言会获得点赞!