编码丙谷二肽生物合成酶的基因及其应用的制作方法
本发明涉及一种丙谷二肽的生物合成方法,尤其是直接采用重组大肠杆菌菌体作为全细胞催化剂高效合成丙谷二肽,属于生物技术领域。
背景技术:
丙谷二肽(L-Ala-Gln),又称为N(2)-L-丙氨酰-L-谷氨酰胺,具有溶解度高,水溶性和热稳定性强的优点已逐渐替代谷氨酰胺(L-Gln)成为主要的肠外营养用药。
目前,丙谷二肽的合成主要采用化学合成法和生物酶催化法。化学合成法生产丙谷二肽反应步骤多、副产物多、试剂毒性大且非环境友好(唐果.N(2)-L-丙氨酰-L-谷氨酰胺二肽的合成与反应研究.厦门大学,2004;中国专利,丙-谷二肽合成方法,CN1392156A)。
而生物酶催化法也存在用酶量大,肽生产率低且仅能催化合成部分较高疏水性氨基酸等弊端。近年来,国外研究人员从枯草芽胞杆菌中获得了一种氨基酸连接酶(Lal),开发出发酵生产二肽的专利技术,但这种催化合成丙谷二肽的过程需要外源提供ATP,导致二肽的生产成本仍然较高,无法在市场大范围推广使用(Tabata K,Hashimoto S.Fermentative production of L-alanyl-L-glutamine by a metabolically engineered Escherichia coli strain expressing L-amino acidα-ligase.Applied and environmental microbiology,2007,73(20):6378-6385.)。
CN104480075A和CN105274174A公开了两种生物酶催化合成丙谷二肽的方法,其采用生物酶冻干粉或生物酶缓冲液进行催化反应,但由于酶分离纯化成本高,且反应后难于回收再利用,不适用于实际工业化生产。
技术实现要素:
本发明目的在于提供一套完整的生物转化生产丙谷二肽的技术方案。从寻找优异的编码丙谷二肽合成酶的外源性基因开始,并期望通过基因工程的方法构建重组工程菌,并据其设计合理的生物发酵工程方案,以实现丙谷二肽绿色、高效、低成本的工业化生产。
为实现上述目的,本发明首先提供一种编码丙谷二肽生物合成酶的基因,其核苷酸序列如SEQ ID NO.1所示。
其二,本发明提供一种丙谷二肽生物合成酶,由上述核苷酸序列为SEQ ID NO.1的基因编码,该酶的氨基酸序列如SEQ ID NO.2所示。
其三,本发明提供一种重组载体,该重组载体含有上述核苷酸序列为SEQ ID NO.1的编码丙谷二肽生物合成酶的基因。
再者,本发明提供一种重组大肠杆菌,其特征在于,是将核苷酸序列如SEQ ID NO.1所示的基因转入大肠杆菌获得的。
最后,本发明提供一种丙谷二肽的生物合成方法,其包括使用上述本发明的重组大肠杆菌对底物的转化反应的步骤。
本发明所述的编码丙谷二肽生物合成酶的基因具有优异的促底物转化生成丙谷二肽的能力。本发明进一步,利用该基因构建重组质粒和重组工程菌,并进一步利用重组的大肠杆菌在发酵体系中直接催化合成丙谷二肽,提高了丙谷二肽的转化效率,同时解决了生物转化工程中酶分离纯化难、酶活不稳定、成本高的问题。所述的方法与现有技术相比具有如下技术效果:
1)实验原料经济易得,设备操作和控制简便,全细胞催化合成路径简单环保,同时省去了酶分离纯化的步骤,降低了能耗及生产成本;
2)反应速率快,反应时间短,摩尔转化率最高可达83.3%,离心反应液即能终止反应,无需加热使酶失活,避免高温引起谷氨酰胺的毒效应;
3)菌体细胞的循环利用,减少了菌体培养次数,节省了生物酶制备及加热灭活的经济成本,缩短了丙谷二肽的反应周期,提高了生产强度,具有明显的市场竞争力,更适合于丙谷二肽的工业化生产。
附图说明
本发明附图3幅,分别是:
图1为重组表达载体结构示意图。
图2为全细胞催化合成丙谷二肽的相对酶活性随反应温度变化图。
图3为全细胞催化合成丙谷二肽的相对酶活性随细胞循环利用次数变化图。
具体实施方式
本发明首先确认获得了一段编码丙谷二肽生物合成酶的基因,其核苷酸序列如SEQ ID NO.1所示。该基因的供体为鞘氨醇杆菌属微生物,更具体地,是泗阳鞘氨醇杆菌(Sphingobacterium siyangensis)。
SEQ ID NO.1的基因序列编码的一段氨基酸序列如SEQ ID NO.2所示。该氨基酸序列由619个氨基酸组成。与据所知最为接近的现有技术(ZL03817916.4)相比,有3个氨基酸发生改变:188位氨基酸由Y(酪氨酸)变为F(苯丙氨酸),两者均属于芳香族氨基酸,由不带电荷的极性R基氨基酸转变为了非极性R基氨基酸;546/547位氨基酸:由AP(丙氨酸/脯氨酸)变为PS(脯氨酸/丝氨酸),是由中性氨基酸/杂环氨基酸向杂环氨基酸/含羟基氨基酸的转变,同时也由非极性R基氨基酸变为了不带电荷的极性R基氨基酸。进一步酶功能域分析结果显示:转酯酶主要有两个功能域,分别为:Peptidase S15(44-331)和PepX_C(382-613)。其中,Peptidase S15(44-331)域,属于X-Pro dipeptidyl-peptidase(S15family),是潜在的转酯酶关键催化中心域;而PepX_C(382-613)域,则属于C端非催化域,但与二肽基肽酶(S9B蛋白家族的丝氨酸肽链外切酶)存在许多相似的结构。虽然对其生理功能的研究需进一步深入,但从现有技术中可以获得两个信息:蛋白质二聚化是酶具有催化活性所必需的;酶的糖基化可影响其生理功能。本发明中SEQ ID NO.2氨基酸序列中188位氨基酸突变可能改变了酶的催化功能域;而546/547位氨基酸突变可能对蛋白质二聚化产生改变,并且通过以上两点,大大改变了所述酶的催化活性。
在确定上述生物酶基因的信息之后,本发明进一步扩增和保存质粒。具体地,使用大肠杆菌E.coli DH5α扩增和保存质粒。
在此基础上,将目的基因和表达载体酶切后,经重组构建重组载体,优选的载体是pET29a。
所构建的重组载体可以采用现有生物技术中的方法转化至表达宿主细胞中以构建携带目标基因的生物工程菌,这样的方法可举例但不限于热击法。表达宿主在本发明中的具体选择是大肠杆菌,包括所有根据现有技术可作为转化宿主的大肠杆菌,可列举例优选但不限于E.coli BL21(DE3)。
接下来,本发明提供一种基于应用上述重组大肠杆菌进行丙谷二肽合成的生物合成方法。在该方法中,至少包括使用上述方法获得的重组大肠杆菌按照现有技术进行发酵生产的过程,其中涉及所述的重组大肠杆菌对底物的转化反应的步骤。
丙谷二肽的生物合成是一项相对成熟的技术,其底物按照现有技术可描述为包括羧基组分和胺组分的底物,本发明中,所述的羧基组分选自氨基酸酯和氨基酸酰胺,最为优选L-丙氨酸甲酯氢氯化物;所述的胺组分选自氨基酸、C-保护的氨基酸和胺,最为优选L-谷氨酰胺。反应体系中的底物浓度按照反应物的比例反应关系设置最有利于生产。反应底物浓度无上下限设置。但最高反应物浓度在一定程度上决定于反应底物在体系中的溶解度,并且随着反应底物浓度的增加,对转化存在一定程度的抑制。本发明的具体实施方式中,谷氨酰胺的溶解度最高约250mM,但我们在生产中可以按照高于此的标准设定反应物投料量,比如用到的600mM,此时原始的反应体系中已经有部分是不溶的固体状态,但随反应进行,可逐步溶解,并不影响转化反应的进行。因此,该投料方法一方面保证反应的顺利进行,另一方面降低操作成本。作为可实施的操作方式,本发明中,所述的羧基组分和胺组分在初始反应体系中的浓度可设置为50~600mM。
另一方面,本发明所述的生物转化合成丙谷二肽的方法中,转化反应体系pH是8.0~10.0,优选8.0~9.0。
再一方面,本发明所述的生物转化合成丙谷二肽的方法中,所述的转化反应体系中重组大肠杆菌用量是(体系)OD600=0.5~5.0。
根据优化的结果,上述本发明的丙谷二肽的生物合成方法的较为具体的实施方式之一,可以描述为包括下述操作步骤的方法:
(1)将底物L-丙氨酸甲酯盐酸盐(L-Ala-OMe)和L-谷氨酰胺(L-Gln)溶于缓冲溶液中,并调节pH为8.0~10.0;
(2)将重组大肠杆菌加入步骤(1)所得的体系中进行反应,反应温度15~40℃,反应体系pH值为8.0~10.0;
(3)以pH值不再降低作为反应达到终点的标志,收集反应液离心分离菌体以终止反应。
该具体实施方式中,本发明所述的丙谷二肽的生物合成路径可简单描述为:
上述方法描述中,一方面,步骤(1)中所述的缓冲溶液可以由本领域技术人员根据现有技术确定,优选但不仅限于硼酸盐缓冲液;且步骤(1)中优选使用碱液调节体系pH为8.0~9.0。另一方面,步骤(2)中所述及的反应温度优选25℃,当然,允许的测量或控制误差所导致的一定范围内的温度波动也是可以接受的;该步骤反应体系的pH值仍然优选8.0~9.0。
基于上文所描述的丙谷二肽的生物合成方法,由于在催化反应合成的体系中使用的全细胞进行生产,反应进行到适当程度可以通过物理分离重组大肠杆菌终止反应,并且分离出来的重组大肠杆菌可以作为工程菌继续添加到生物转化的反应体系中使用,实现循环利用,因此,该方法最为重要的一个实施方式中,还包括菌体循环利用的步骤。亦即将上述步骤(3)反应终止后分离所得的菌体加入至新的反应体系中,循环使用。
下述实施方式,将更加完整具体地阐述本发明的内容及优选模式,该具体实施方式中,所述的丙谷二肽的生物合成方法包括如下步骤:
(1)构建重组大肠杆菌;
克隆核苷酸序列如SEQ ID NO.1的目标基因,使用表达载体pET29a构建包括该基因的重组载体,并采用热击法将重组载体转化至表达宿主E.coli BL21(DE3)中;
(2)利用抗性基因筛选、PCR及酶切验证步骤(1)所获得的阳性转化子,保存于斜面培养基;
所述的斜面培养基:酵母浸粉5g/L,胰蛋白胨10g/L,NaCl 10g/L,琼脂粉20g/L,121℃灭菌15min,冷却后加入终浓度为10~100μg/mL的卡那霉素;
(3)活化后的重组大肠杆菌转接至种子培养基,于37℃,200rpm的条件下过夜,以一定的接种量接种到发酵培养基中,进行扩大培养,待细胞OD600=0.4~1.0后,加入终浓度为0.2~2.0mM的异丙基硫代半乳糖苷(IPTG)过夜低温诱导培养;
所述的种子培养基和发酵培养基:酵母浸粉5g/L,胰蛋白胨10g/L,NaCl 10g/L,121℃灭菌15min,冷却后加入终浓度为10~100μg/mL的卡那霉素;
(4)丙谷二肽的生物合成:将底物L-丙氨酸甲酯盐酸盐(L-Ala-OMe)和L-谷氨酰胺(L-Gln)溶于缓冲溶液(根据权利要求5所述的底物浓度和缓冲液种类),并加入步骤(3)的培养物,调节pH为8.0~9.0,体系在25±1℃条件下反应,至pH值不再降低作为反应终点,收集反应液离心分离菌体;
(5)将步骤(4)分离所得菌体加入到新的反应体系中,循环生产丙谷二肽。
下述非限制性实施例用于对本发明的内容作进一步说明,不应当理解为对本发明任何形式的限定。
实施例1
生物酶表达载体及重组大肠杆菌的构建:
根据已知的相关生物酶核苷酸序列(SEQ ID NO.5),设计引物(SEQ ID NO.3/4),以来源于中国普通微生物菌种保藏管理中心的泗阳鞘氨醇杆菌(Sphingobacterium siyangensis,菌株编号:1.6855)基因组为模板扩增该生物酶基因SEQ ID NO.1。
其中,PCR反应体系(50μL):
PCR反应条件:
PCR产物经胶回收纯化后,与质粒pMDTM19-T连接,阳性克隆提取质粒后测序。酶切获得阳性克隆中的生物酶基因与经过同样处理的表达载体pET29a进行连接,连接产物转化至E.coli DH5α感受态细胞,在LB抗性培养基上培养,挑取单菌落,小量提取质粒酶切鉴定并进行DNA序列测定,得到生物酶表达载体。再利用热击法转化至表达宿主感受态细胞中,筛选获得能催化合成丙谷二肽的重组大肠杆菌。
实施例2
重组大肠杆菌的发酵培养:
将重组大肠杆菌接种至LB液体培养基:酵母浸粉5g/L,胰蛋白胨10g/L,NaCl 10g/L)中,37℃,200rpm的条件下活化菌体。之后转接至种子培养基中,37℃,200rpm过夜振荡培养。以1%的接种量接种到1.5L发酵培养基中,37℃,200rpm通气培养细胞至0D620=0.6~0.8后,加入诱导剂IPTG诱导过夜后,离心收集菌体。作为本发明中下述实施例所使用的重组大肠杆菌。
实施例3
50mM底物浓度下全细胞催化合成丙谷二肽:
称取0.698g L-丙氨酸甲酯盐酸盐(50mM L-Ala-OMe)和1.462g L-谷氨酰胺(100mM L-Gln),溶解于90mL 0.2M pH8.7硼酸盐缓冲溶液中,控制温度为25℃,并用6M NaOH水溶液调节pH至8.5。加入重组大肠杆菌至反应体系中OD600=0.75,用6M NaOH溶液维持pH稳定,10min后离心去除细胞以终止反应。利用高效液相色谱法(中国药典2010年版二部附录VD)测得丙谷二肽的最高浓度为9.04g/L,摩尔转化率83.3%。
实施例4
100mM底物浓度下全细胞催化合成丙谷二肽:
称取1.396g L-丙氨酸甲酯盐酸盐(100mM L-Ala-OMe)和1.462g L-谷氨酰胺(100mM L-Gln),溶解于90mL0.2M pH8.7硼酸盐缓冲溶液中,控制温度为25℃,并用6M NaOH水溶液调节pH至8.5。加入重组大肠杆菌至反应体系中OD600=2.0,用6M NaOH溶液维持pH稳定,10min后离心去除细胞以终止反应。利用高效液相色谱法(中国药典2010年版二部附录V D)测得丙谷二肽的最高浓度为15.92g/L
实施例5
200mM底物浓度下全细胞催化合成丙谷二肽:
称取2.792g L-丙氨酸甲酯盐酸盐(200mM L-Ala-OMe)和2.924g L-谷氨酰胺(200mM L-Gln),溶解于90mL0.2M pH8.7硼酸盐缓冲溶液中,控制温度为25℃,并用6M NaOH水溶液调节pH至8.5。加入重组大肠杆菌至反应体系中OD600=2.0,用6M NaOH溶液维持pH稳定,10min后离心去除细胞以终止反应。利用高效液相色谱法测得丙谷二肽的最高浓度为31.34g/L
实施例6
400mM底物浓度下全细胞催化合成丙谷二肽:
称取5.584g L-丙氨酸甲酯盐酸盐(400mM L-Ala-OMe)和5.848g L-谷氨酰胺(400mM L-Gln),溶解于90mL0.2M pH8.7硼酸盐缓冲溶液中,控制温度为25℃,并用6M NaOH溶液调节pH至8.5。加入重组大肠杆菌至反应体系中OD600=2.0,用6M NaOH溶液维持pH稳定,20min后离心去除细胞以终止反应。利用高效液相色谱法测得丙谷二肽的最高浓度为61.53g/L。
实施例7
600mM底物浓度下全细胞催化合成丙谷二肽:
称取8.376g L-丙氨酸甲酯盐酸盐(600mM L-Ala-OMe)和8.772g L-谷氨酰胺(600mM L-Gln),溶解于90mL 0.2M pH8.7硼酸盐缓冲溶液中,控制温度为25℃,并用6M NaOH水溶液调节pH至8.5。加入重组大肠杆菌至反应体系中OD600=2.0,用6M NaOH溶液维持pH稳定,20min后离心去除细胞以终止反应。利用高效液相色谱法(中国药典2010年版二部附录V D)测得丙谷二肽的最高浓度为79.91g/L。
实施例8
不同反应温度对全细胞催化合成丙谷二肽的影响:
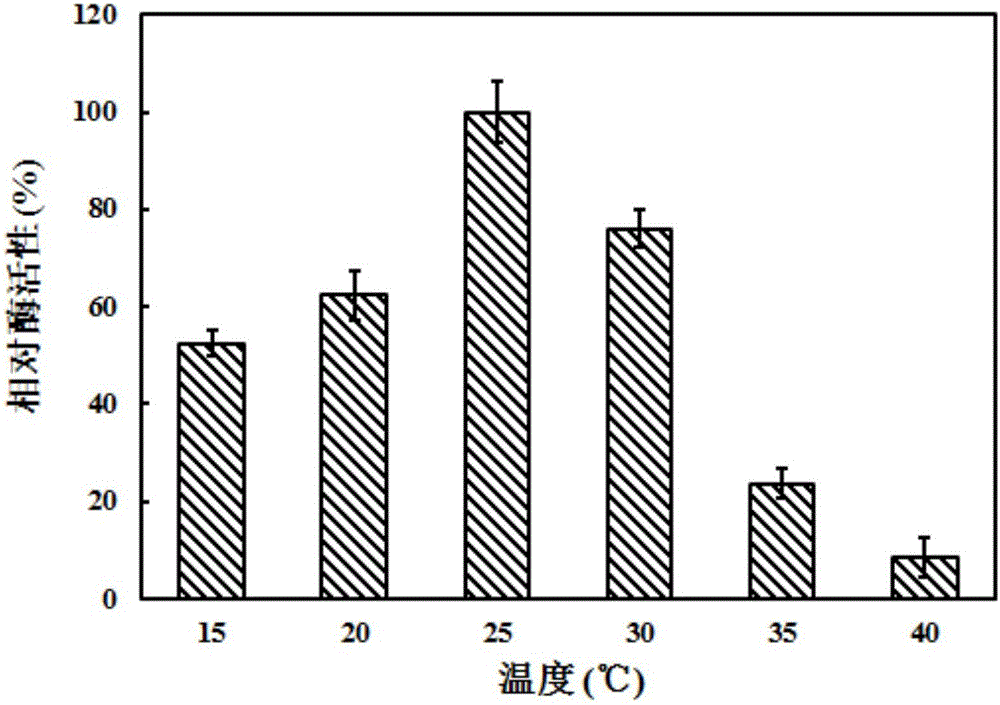
以1%的接种量接种重组大肠杆菌至LB发酵培养基中,37℃下200rpm通气培养细胞OD620=0.73。然后加入诱导剂IPTG诱导生物酶表达。离心收集细胞进行丙谷二肽催化反应。称取5.584g L-丙氨酸甲酯盐酸盐(400mM L-Ala-OMe)和5.848g L-谷氨酰胺(400mM L-Gln),溶解于90mL0.2M pH8.7硼酸盐缓冲溶液中,用6M NaOH溶液调节pH至8.5,分别在不同的反应温度下进行催化合成反应。利用高效液相色谱法(中国药典2010年版二部附录V D)测定丙谷二肽的浓度。重组大肠杆菌全细胞可在15~40℃范围内催化合成丙谷二肽,25℃下全细胞催化合成丙谷二肽的产量最高,以其酶活性为100%,绘制出不同反应温度下的相对酶活性,如图2所示。
实施例9
菌体循环使用高效合成丙谷二肽:
重组大肠杆菌的培养与实施例2所述的方法相同,将收集的菌体细胞按照实施例3进行催化合成反应,当反应终止后,利用高效液相色谱法(中国药典2010年版二部附录V D)测得丙谷二肽的浓度,以其对应的酶活力为100%。
离心收集菌体细胞作为下一轮培养工程菌,重复上述反应过程,分别求出第一次循环利用菌体的相对酶活性为99.8%,第二次循环利用菌体的相对酶活性为97.8%,第三次循环利用菌体的相对酶活性为94.4%,如图3所示。由于离心回收菌体的过程中,细胞会有少量损失,因此多次循环利用重组大肠杆菌细胞催化合成丙谷二肽的过程中酶活性是稳定的,适合于应用到工业化生产中。
实施例10
与现有技术的比较:
日本味之素公司(专利ZL03817916.4)在丙谷二肽酶法合成领域中使用的转酯酶在生物安全性,转化效率以及生产效率等方面在国际上处于领先水平。(Biosci Biotechnol Biochem.2013;77(3):618-23;2011;75(11):2087-2092)在文献中报道的最优的结果为反应体系中细胞OD610约为4.6的情况下,两种底物的摩尔转化率为67%,但催化反应时间较长,为40min。与之相比,本专利构建的转酯酶表达体系,在细胞量OD600为0.75的反应条件下,10min内摩尔转化率可以达到83.3%,具有更快的生产效率和更高的摩尔转化率。
表1中列出了更为具体的对比数据(日本味之素数据来源:Enzymatic production of L-Alany-L-glutamine by recombinant E.coli expressingα-Amino acid ester Acyltransferase from Sphingobacterium siyangensis.Bioscience,biotechnology,and biochemistry,2013,77(3):618-623.),从该结果可以看出,在相似的底物条件下,本发明的方法中,也可以在远远低于现有技术ZL03817916.4的反应时间条件下实现更优异的转化率。可见:相似的反应体系中,本发明的细胞酶催化活力更强。
表1
SEQUENCE LISTING
<110> 大连医诺生物有限公司
<120> 编码丙谷二肽生物合成酶的基因及其应用
<130> N/A
<160> 5
<170> PatentIn version 3.3
<210> 1
<211> 1860
<212> DNA
<213> Sphingobacterium siyangensis
<400> 1
atgaaaaata caatttcgtg cctaacttta gcgcttttaa gcgcaagcca gttacatgct 60
caaacagctg ccgactcggc ttatgttaga gatcattatg aaaagaccga agtagcaatc 120
cccatgcgag atgggaagaa attatttact gcgatctaca gtccaaaaga caaatccaag 180
aaatatccag ttttactcaa tagaacaccc tacacggttt ctccttatgg gcagaacgaa 240
tacaaaaaaa gcttgggaaa ctttccccaa atgatgcgtg aaggttatat tttcgtttat 300
caggatgtcc gtggcaagtg gatgagcgaa ggtgattttg aagatattcg tccgaccacc 360
tacagcaaag ataaaaaagc aatcgatgaa agtacggata cctatgatgc gcttgaatgg 420
ttacagaaaa atctcaaaaa ctataatgac aaagccgggc tctatgggat ttcctatcca 480
ggcttctatt ctaccgtcgg attggtcaaa acacacccga gcttgaaggc agtctcccca 540
caggctcccg taacagactg gtttatcggc gacgacttcc accataatgg cgtattgttt 600
cttcaggatg catttacatt catgtcaacc tttggtgtcc cacgtccaaa acccattaca 660
ccggatcaat ttaagggcaa aattcaaatc aaagaagccg ataaatataa cttttttgca 720
gaagcaggaa cagcgcggga actcaaagaa aagtattttg gtgactccgt acaattttgg 780
aatggcctgt ttaaacatcc cgactatgat gatttttgga aatcgcgtgt gatcaccaat 840
tctttacagg aggtaaaacc agctgtgatg gtggttggtg gtttctttga cgcggaagat 900
gcttatggaa catttaagac ttaccaatcg attgaggata aaagcaaaaa aaacaactcg 960
attttagtcg cgggaccttg gtatcatggc ggctgggttc gtgcagaagg aaactattta 1020
ggtgatatcc aatttgagaa aaaaaccagt attacttatc aggagcaatt tgaacaaccg 1080
tttttcaaat attacctaaa agatgaagga aacttcgccc cttccgaagc taacattttt 1140
gtctcaggca gcaacgaatg gaaacatttc gaacaatggc cgccaaaaaa tgtagagaca 1200
aaaaaactat acttccaacc tcaggggaaa cttggatttg acaaagttca acgtacagat 1260
tcctgggatg aatatgtaac agaccctaat aaacttgttc cgcatcaagg tgggttaatt 1320
caaaaccgaa cacgggagta tatggtagat gatcagcgtt tcgcagctag tcgccctgat 1380
gtcatggttt atcaaacgga accgttgacg gaggatctga cgatagtagg cccaatcaaa 1440
aacttcctca aagtctcctc aacaggaaca gacgcggact atgttgtcaa actgattgat 1500
gtatacccga acgatgctgc aagttatcaa ggaaaaacaa tggctggata tcaaatgatg 1560
gtacgtggtg agatcatggc ggggaaatac cgaaatggtt ttgataaagc acaggccttg 1620
actccaggta tggtcgaaaa ggttaatttt gaaatgccag acgttgcgca taccttcaaa 1680
aaaggacatc gcattatggt tcaggtacaa aactcatggt ttccgttagc agaacgaaat 1740
ccacaggtat tcttaccgtc ttatacagcc accaaagctg acttccgtaa ggctacccaa 1800
cgtatttttc acgatgtgaa caatgccaca tacatcgaat tttctgtcct caaagattag 1860
<210> 2
<211> 619
<212> PRT
<213> Sphingobacterium siyangensis
<400> 2
Met Lys Asn Thr Ile Ser Cys Leu Thr Leu Ala Leu Leu Ser Ala Ser
1 5 10 15
Gln Leu His Ala Gln Thr Ala Ala Asp Ser Ala Tyr Val Arg Asp His
20 25 30
Tyr Glu Lys Thr Glu Val Ala Ile Pro Met Arg Asp Gly Lys Lys Leu
35 40 45
Phe Thr Ala Ile Tyr Ser Pro Lys Asp Lys Ser Lys Lys Tyr Pro Val
50 55 60
Leu Leu Asn Arg Thr Pro Tyr Thr Val Ser Pro Tyr Gly Gln Asn Glu
65 70 75 80
Tyr Lys Lys Ser Leu Gly Asn Phe Pro Gln Met Met Arg Glu Gly Tyr
85 90 95
Ile Phe Val Tyr Gln Asp Val Arg Gly Lys Trp Met Ser Glu Gly Asp
100 105 110
Phe Glu Asp Ile Arg Pro Thr Thr Tyr Ser Lys Asp Lys Lys Ala Ile
115 120 125
Asp Glu Ser Thr Asp Thr Tyr Asp Ala Leu Glu Trp Leu Gln Lys Asn
130 135 140
Leu Lys Asn Tyr Asn Gly Lys Ala Gly Leu Tyr Gly Ile Ser Tyr Pro
145 150 155 160
Gly Phe Tyr Ser Thr Val Gly Leu Val Lys Thr His Pro Ser Leu Lys
165 170 175
Ala Val Ser Pro Gln Ala Pro Val Thr Asp Trp Phe Ile Gly Asp Asp
180 185 190
Phe His His Asn Gly Val Leu Phe Leu Gln Asp Ala Phe Thr Phe Met
195 200 205
Ser Thr Phe Gly Val Pro Arg Pro Lys Pro Ile Thr Pro Asp Gln Phe
210 215 220
Lys Gly Lys Ile Gln Ile Lys Glu Ala Asp Lys Tyr Asn Phe Phe Ala
225 230 235 240
Glu Ala Gly Thr Ala Arg Glu Leu Lys Glu Lys Tyr Phe Gly Asp Ser
245 250 255
Val Gln Phe Trp Asn Asp Leu Phe Lys His Pro Asp Tyr Asp Asp Phe
260 265 270
Trp Lys Ser Arg Val Ile Thr Asn Ser Leu Gln Glu Val Lys Pro Ala
275 280 285
Val Met Val Val Gly Gly Phe Phe Asp Ala Glu Asp Ala Tyr Gly Thr
290 295 300
Phe Lys Thr Tyr Gln Ser Ile Glu Asp Lys Ser Lys Lys Asn Asn Ser
305 310 315 320
Ile Leu Val Ala Gly Pro Trp Tyr His Gly Gly Trp Val Arg Ala Glu
325 330 335
Gly Asn Tyr Leu Gly Asp Ile Gln Phe Glu Lys Lys Thr Ser Ile Thr
340 345 350
Tyr Gln Glu Gln Phe Glu Gln Pro Phe Phe Lys Tyr Tyr Leu Lys Asp
355 360 365
Glu Gly Asn Phe Ala Pro Ser Glu Ala Asn Ile Phe Val Ser Gly Ser
370 375 380
Asn Glu Trp Lys His Phe Glu Gln Trp Pro Pro Lys Asn Val Glu Thr
385 390 395 400
Lys Lys Leu Tyr Phe Gln Pro Gln Gly Lys Leu Gly Phe Asp Lys Val
405 410 415
Gln Arg Thr Asp Ser Trp Asp Glu Tyr Val Thr Asp Pro Asn Lys Pro
420 425 430
Val Pro His Gln Gly Gly Leu Ile Gln Asn Arg Thr Arg Glu Tyr Met
435 440 445
Val Asp Asp Gln Arg Phe Ala Ala Ser Arg Pro Asp Val Met Val Tyr
450 455 460
Gln Thr Glu Pro Leu Thr Glu Asp Leu Thr Ile Val Gly Pro Ile Lys
465 470 475 480
Asn Phe Leu Lys Val Ser Ser Thr Gly Thr Asp Ala Asp Tyr Val Val
485 490 495
Lys Leu Ile Asp Val Tyr Pro Asn Asp Ala Ala Ser Tyr Gln Gly Lys
500 505 510
Thr Met Ala Gly Tyr Gln Met Met Val Arg Gly Glu Ile Met Ala Gly
515 520 525
Lys Tyr Arg Asn Gly Phe Asp Lys Ala Gln Ala Leu Thr Pro Gly Met
530 535 540
Val Glu Lys Val Asn Phe Glu Met Pro Asp Val Ala His Thr Phe Lys
545 550 555 560
Lys Gly His Arg Ile Met Val Gln Val Gln Asn Ser Trp Phe Pro Leu
565 570 575
Ala Glu Arg Asn Pro Gln Val Phe Leu Pro Ser Tyr Thr Ala Thr Lys
580 585 590
Ala Asp Phe Arg Lys Ala Thr Gln Arg Ile Phe His Asp Val Asn Asn
595 600 605
Ala Thr Tyr Ile Glu Phe Ser Val Leu Lys Asp
610 615
<210> 3
<211> 31
<212> DNA
<213> 人工序列
<400> 3
cgcggatcca tgaaaaatac aatttcgtgc c 31
<210> 4
<211> 31
<212> DNA
<213> 人工序列
<400> 4
ccgctcgagc taatctttga ggacagaaaa t 31
<210> 5
<211> 1860
<212> DNA
<213> Sphingobacterium sp.
<400> 5
atgaaaaata caatttcgtg cctaacttta gcgcttttaa gcgcaagcca gttacatgct 60
caaacagctg ccgactcggc ttatgttaga gatcattatg aaaagaccga agtagcaatt 120
cccatgcgag atgggaaaaa attatttact gcgatctaca gtccaaaaga caaatccaag 180
aaatatccag ttttgctcaa tagaacgccc tacacggttt ctccttatgg gcagaacgaa 240
tacaaaaaaa gtttgggaaa ctttccccaa atgatgcgtg aaggctatat tttcgtttac 300
caggatgtcc gtggcaagtg gatgagcgaa ggtgattttg aagatatacg tccgaccacg 360
tacagcaaag ataaaaaagc aatcgatgaa agtacggata cctatgatgc gcttgaatgg 420
ttacagaaaa atctcaaaaa ctataatggc aaagccgggc tctatgggat ttcctatcca 480
ggcttctatt ctaccgtcgg attggtcaaa acacacccga gcttgaaggc agtctcccca 540
caggctcccg taacagactg gtatatcggc gacgacttcc accataatgg cgtattgttt 600
cttcaggatg catttacatt catgtcaacc tttggtgtcc ctcgtccaaa acccattaca 660
ccggatcaat ttaagggcaa aattcagatc aaagaagccg ataaatataa cttttttgca 720
gaagcaggaa cagcgcggga actcaaagaa aaatactttg gtgactccgt acaattttgg 780
aatgacctgt ttaagcatcc cgactatgat gatttttgga aatcgcgtgt gatcaccaat 840
tctttacagg aggtaaaacc agctgtgatg gtggttggtg gtttctttga cgcggaagat 900
gcttatggaa catttaagac ctaccaatcg attgaggata aaagcaaaaa aaacaactcg 960
attttagtcg cgggaccttg gtatcatggc ggctgggttc gtgcagaagg aaactattta 1020
ggtgatatcc aatttgagaa aaaaaccagt attacttatc aggaacaatt tgaacaaccg 1080
tttttcaaat attacctaaa agatgaagga aacttcgccc cttccgaagc caacattttt 1140
gtttcaggca gcaacgaatg gaaacatttc gaacaatggc caccaaaaaa tgtagagaca 1200
aaaaaactat acttccaacc tcaggggaaa cttggatttg acaaagttca acgtacagat 1260
tcctgggatg aatatgtaac agacccgaat aaacctgttc cgcatcaagg tgggttaatt 1320
caaaaccgaa cacgggagta tatggtagat gatcaacgtt tcgcggctag tcgccctgat 1380
gtcatggttt atcaaacgga accgttgacg gaggacctga cgatagtagg cccaatcaaa 1440
aactttctca aagtttcttc aacaggaaca gacgcggact atgttgtcaa actgattgac 1500
gtttatccga atgatgcagc aagttatcaa ggaaaaacaa tggctggata tcaaatgatg 1560
gtacgtggtg agatcatggc ggggaaatac cgaaatggtt tcgataaagc gcaggccttg 1620
actccaggta tggtcgaaaa ggtgaatttt gaaatgccag acgttgcgca taccttcaaa 1680
aaaggacatc gcattatggt tcaggtacaa aactcatggt ttccgctggc agaacgaaat 1740
ccacaggtgt ttttagcacc ttatacagct accaaagctg atttccgcaa agctacccaa 1800
cgtatttttc acgatgtgaa caatgccaca tacatcgaat tttctgtcct caaagattag 1860
- 还没有人留言评论。精彩留言会获得点赞!