耐酸1,3-1,4-β-葡聚糖酶突变体
本发明涉及耐酸1,3-1,4-β-葡聚糖酶突变体,属于基因工程和酶工程技术领域。
背景技术:
β-葡聚糖是许多谷类作物(大麦、高粱、水稻等)胚乳细胞壁的主要成分,溶于水后会使溶液黏度加大,给工业生产带来许多问题。在啤酒酿造过程中,麦芽中的β-葡聚糖大量溶解于麦汁,导致麦汁黏度增加,过滤速度降低且易堵塞麦糟结构,导致麦汁过滤速度减慢,冷却过程中冷凝物析出增加,降低麦汁收率,同时会降低成品啤酒的非生物稳定性。在禽畜养殖过程中,由于动物胃肠道缺乏分解β-葡聚糖的酶,无法有效消化和吸收谷物中的β-葡聚糖,降低了饲料利用率,β-葡聚糖还会增加肠道液体粘度,干扰消化酶与底物之间的作用,降低营养物质的吸收效率,同时为肠道有害提供了营养条件,有害菌增殖导致肠道菌群失调,禽畜肠道疾病增加。
1,3-1,4-β-葡聚糖酶,简称β-葡聚糖酶,属于糖苷水解酶家族16,它通过水解作用将β-葡聚糖中与β-1,3-糖苷键紧密相邻的β-1,4-糖苷键打断,将β-葡聚糖分解为小分子量的低聚葡萄糖,能够有效地解决β-葡聚糖在上述行业中的问题。然而,β-葡聚糖酶的工业应用环境通常具有高温、低ph的特点,例如麦汁糖化温度(65-70℃)和酸性ph(ph5.0左右)、禽畜消化系统的酸性环境(ph3.0-4.0),严重限制了β-葡聚糖酶的高效工业应用。
天然来源的β-葡聚糖酶最适ph集中在ph6.0-7.5,最适温度集中在40-60℃,难以满足工业需求,因此提高野生型β-葡聚糖酶的催化活性、热稳定性和耐酸性是其高效工业应用的重要前提。目前对β-葡聚糖的催化活性和热稳定性的研究已相对透彻,而对β-葡聚糖酶耐酸性的研究相对缺乏。
因此,若能获得耐酸性的β-葡聚糖酶,将对于β-葡聚糖酶的工业化应用具有重要意义。
技术实现要素:
为了解决目前β-葡聚糖酶在酸性条件下酶活较低、耐受性差的问题,本发明通过对来源于芽孢杆菌属的1,3-1,4-β-葡聚糖酶进行结构分析,找到了潜在的可能影响酶耐酸性的关键位点并加以改造,同时对酶表面的酸性残基也做了相应改造。从诸多改造后的突变体中筛选得到了耐酸性提升的突变体,使其能在ph4.5-5.5之间具备更高的耐受性。
本发明提供了1,3-1,4-β-葡聚糖酶突变体,所述突变体以seqidno.5所示1,3-1,4-β-葡聚糖酶为亲本酶,将亲本酶的第1位、第133位、第134位中的一个或多个位点的氨基酸突变。
在一种实施方式中,将亲本酶第1位突变为谷氨酸,得到的突变体命名为q1e,氨基酸序列如seqidno.1所示。
在一种实施方式中,将亲本酶第133位突变为亮氨酸,得到的突变体命名为i133l,氨基酸序列如seqidno.2所示。
在一种实施方式中,将亲本酶第134位突变为丙氨酸,得到的突变体命名为v134a,氨基酸序列如seqidno.3所示。
在一种实施方式中,将亲本酶第1位突变为谷氨酸,并将第133位突变为亮氨酸,得到的突变体命名为q1e/i133l,氨基酸序列如seqidno.4所示。
本发明提供了编码所述1,3-1,4-β-葡聚糖酶突变体的基因。
本发明提供了携带权利要求3所述基因的重组质粒。
在一种实施方式中,所述表达载体为pet系列、duet系列、pgex系列、phy300、phy300plk、ppic系列中的任意一种。
在一种实施方式中,所述表达载体为pet28a(+)质粒。
本发明提供了表达所述突变体,或携带所述基因的微生物细胞。
在一种实施方式中,所述微生物细胞以大肠杆菌为出发菌株。
在一种实施方式中,所述大肠杆菌为大肠杆菌bl21(de3)。
本发明的另一个目的是提供一种构建所述1,3-1,4-β-葡聚糖酶突变体的方法,是以bacillusterquilensiscgx5-1来源1,3-1,4-β-葡聚糖酶基因为模板,采用重叠延伸pcr的方法进行定点突变获得;得到的β-葡聚糖酶突变体基因的核苷酸序列如seqidno.6、seqidno.7、seqidno.8或seqidno.9所示;经基因测序鉴定所有突变位点均已成功按照预设目标突变,随后将编码突变体的基因片段连接至pet28a(+)质粒并转化大肠杆菌bl21(de3)。
本发明提供了生产所述突变体的方法,所述方法是以表达所述突变体、或携带所述基因的微生物细胞为发酵菌株,发酵生产突变体。
在一种实施方式中,将微生物细胞接种至tb培养基中培养至生长对数期,加入iptg和α-乳糖诱导表达,表达结束后离心收集上清液,即为含有所述突变体的粗酶液。
本发明提供了所述突变体,或所述基因,或所述微生物细胞在水解β-葡聚糖中的应用。
本发明的有益效果:
本发明提供的四株β-葡聚糖酶突变体与野生酶相比,具有更高的耐酸性、催化活性和催化效率。四株突变酶的比酶活值分别提高了30.53%、7.91%、44.63%和16.92%,同时催化效率kcat/km值分别提高了60.94%、14.69%、91.20和33.57%,耐酸性也得到了一定的提高,特别是突变体q1e/i133l在ph4.0-8.0条件下的相对酶活均高于野生酶,在ph4.5时具有119.86%的相对酶活,而野生酶仅为32.23%。由此可见四株突变酶的耐酸性和催化活性与野生酶相比均有所提高,在食品、饲料、益生元和生物燃料领域具有极高的应用前景。
附图说明
说明各附图所表示的含义
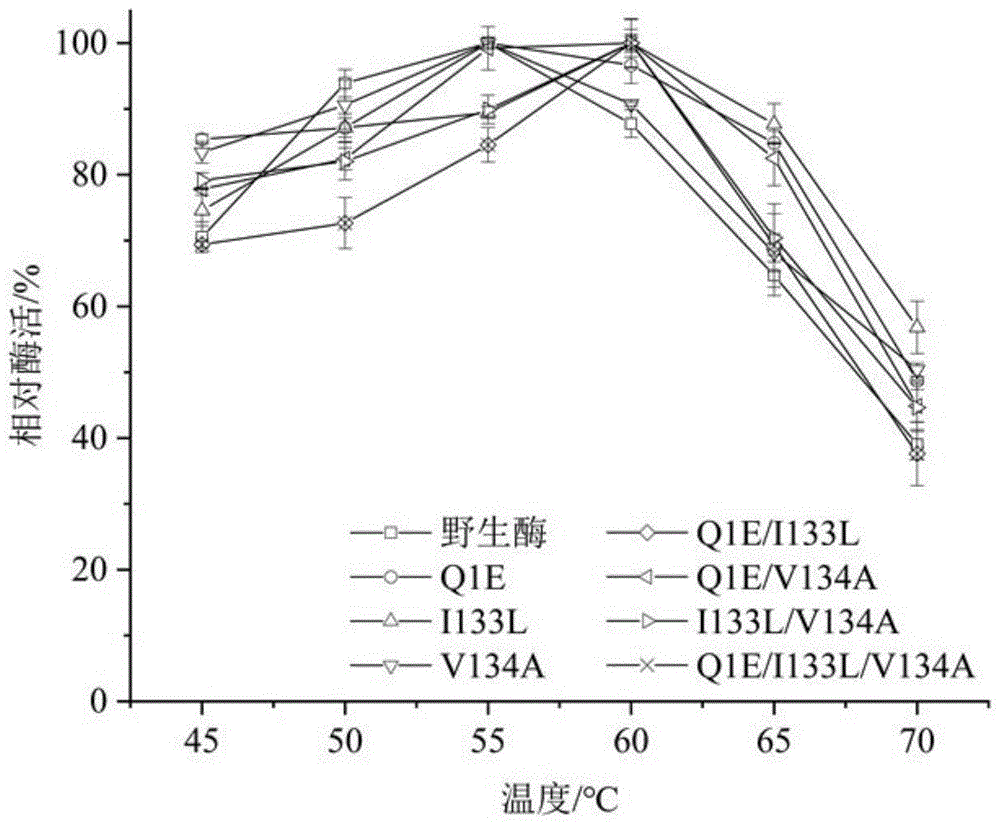
图1为野生酶与四株β-葡聚糖酶突变体的最适温度比较图;□:野生酶,○:q1e,δ:i133l,▽:v134a,◇:q1e/i133l,
图2为野生酶与四株β-葡聚糖酶突变体的最适ph比较图;□:野生酶,○:q1e,δ:i133l,▽:v134a,◇:q1e/i133l,
图3为野生酶与四株β-葡聚糖酶突变体的ph稳定性比较图;□:野生酶,○:q1e,δ:i133l,▽:v134a,◇:q1e/i133l,
具体实施方式
下述实施中涉及的培养基和溶液如下:
luria-bertani(lb)培养基:氯化钠10g·l-1,蛋白胨10g·l-1,酵母粉5g·l-1;配制固体培养基时需加入20g·l-1琼脂粉;灭菌条件:121℃、20min。
terrific-broth(tb)培养基:甘油14.1g·l-1,磷酸二氢钾2.17g·l-1,磷酸氢二钾2.74g·l-1,胰蛋白胨12.5g·l-1,酵母粉20g·l-1;灭菌条件:121℃、20min。
反应终止液:乙酸钾32.5g·l-1,硫酸锌4.0g·l-1,乙二醇甲醚80%(v/v);采用浓盐酸调节溶液ph至8.0;室温避光保存,用时取上清液。
β-葡聚糖酶比酶活的测定方法如下:
采用改良azo法测定β-葡聚糖酶的催化活性。单位酶活定义:在60℃、ph6.5的条件下,每分钟降解大麦β-葡聚糖生成1μmol还原糖所需的酶量。酶活测定步骤:将200μlazo蓝色大麦β-葡聚糖底物与20mmol·l-1、ph6.5磷酸钠缓冲液等体积混合,于水浴锅中预热,温度设置为60℃,随后加入100μlβ-葡聚糖酶,轻轻吹吸混匀,酶与底物进行反应。待精确反应10min后,轻轻向反应体系中加入3ml反应终止液,静置5-10min,7000×g离心5min,测定上清液在590nm的吸光值,对照组为以100μl去离子水代替酶液处理所得样品。酶活计算公式如下:β-葡聚糖酶酶活(u·ml-1)=(od590+0.0558)/(0.0012×180);其中n为酶液稀释倍数,od590为上清液在590nm的吸光值。
实施例1:β-葡聚糖酶耐酸性位点及突变体的筛选
从ncbi数据库和文献检索八株芽孢杆菌来源的中温1,3-1,4-β-葡聚糖酶,分别为vel(bacillusvelezensis来源,最适ph为5.0)、amy(bacillusamyloliquefaciens来源,最适ph为5.0)、lic(bacilluslicheniformis来源,最适ph为5.6)、3o5s(bacillussubtilis168来源,最适ph为6.0)、amy2(bacillusamyloliquefacienstb2来源,最适ph为6.2)、sub(bacillussubtilisma139来源,最适ph为6.4)、wt(bacillusterquilensiscgx5-1来源,最适ph为6.5)、1gbg(bacilluslicheniformis来源,最适ph为7.0)和met(bacillusmethylotrophicuss2来源,最适ph为7.5);将其按照最适ph值从低到高排列,进行氨基酸序列比对,筛选非保守残基中在最适ph低于ph6.0序列中高度保守的残基进行突变,在特基拉芽孢杆菌cgx5-1来源β-葡聚糖酶基础上得到d8e、g12s、n28d、l44s、i133l、i133v、v134a和e191d共8株单点突变体。
蛋白质表面对于酶吸引底物靠近和在极端环境下维持其构象稳定性具有重要作用,此外,最适ph值较低的酶表面通常具有更多的酸性残基,推测酶表面酸性残基对于维持酶在酸性环境下的结构稳定性和催化活性具有重要作用。通过getarea在线服务器计算了野生酶残基暴露程度,从中筛选出完全暴露(暴露程度>30%)的谷氨酰胺和天冬酰胺,将其分别突变为酸性较强的谷氨酸和天冬氨酸,得到q1e、q171e、n11d、n26d、n37d、n129d、n142d、n154d、n170d、n197d和n200d共11株单点突变体。
将上述得到的19株单点突变酶,在40℃和四个ph(ph4.5、ph5.5、ph6.5和ph7.5)下温浴1h后,分别测定其相对酶活,以未处理酶活为100%相对酶活,结果如表1所示:在ph4.5下,q1e、q171e和n26d的相对酶活较野生型得到了极大的提升;当ph达到ph5.5时,i133l、v134a、e191d、q1e、q171e、n26d和n200d的相对酶活较野生酶有所提高;而在ph6.5和ph7.5条件下温浴处理后,i133l、v134a、e191d、q1e、q171e、n26d和n200d这7株单突变酶的相对酶活均高于50%,说明这7株突变酶在酸性环境下的稳定性有所增强。
为了确保突变体的催化活性,比较了7株单突变酶的比酶活,结果显示i133l、v134a、q1e和n200d突变体的比酶活分别为5372.22±126.54u/mg、4441.36±157.49u/mg、5952.72±541.36u/mg和5248.13±296.48u/mg,和野生酶相比分别提高了30.53%、7.91%、44.63%和27.51%,而其他4株单突变体的比活力值低于野生酶。
为了确保突变体的温度稳定性,测定了i133l、v134a、q1e和n200d突变体的在ph6.5和六个不同温度下(45℃、50℃、55℃、60℃、55℃和70℃)的相对酶活,以最大酶活为100%相对酶活。最适温度分析结果发现i133l突变体的最适温度为60℃,较野生酶提高了5℃,在65℃时仍具有87.71%相对酶活,此时野生酶仅为64.70%相对酶活。v134a、q1e突变体的最适温度未较野生型发生显著差异,而n200d突变体的最适温度降低了5℃,在55℃-70℃下的相对酶活均低于野生酶,65℃时的相对酶活降低了30.52%,说明其热稳定性较差。
表1野生酶和单点突变酶的酶活
实施例2:耐酸性β-葡聚糖酶多突变体的构建
在实施例1获得的三个单点突变体i133l、v134a和q1e的基础上,构建3个双突变体q1e/i133l、q1e/v134a、i133l/v134a和1个三突变体q1e/i133l/v134a并表征了其酶学性质。
以含特基拉芽孢杆菌cgx5-1分泌β-葡聚糖酶基因的重组质粒pet28a(+)-bglt(jinjingwang,chengtuoniu,xiaolingliu,xichen,qili.characterizationofanew1,3-1,4-β-glucanasegenefrombacillustequilensiscgx5-1.appliedbiochemistryandbiotechnology,2014,173(3))为模板,通过重叠延伸pcr的方法扩增得到含相应位点发生突变的重组质粒。
用于引入q1e突变的引物(小写字母为突变碱基):
正向引物:5’-tcacttctactgcctcggctgaaacaggtg-3’,seqidno.10,
反向引物:5’-tcaaaaaacgatccacctgtttcagccgaggc-3’,seqidno.11;
用于引入i133l突变的引物(小写字母为突变碱基):
正向引物:5’-aaccatgagaagctggttgatctcgggtttgatgca-3’,seqidno.12,反向引物:5’-cgagatcaaccagcttctcatggtttcctgcacc-3’,seqidno.13;
用于引入v134a突变的引物(小写字母为突变碱基):
正向引物:5’-tgagaagattgcagatctcgggtttgatgcagcc-3’,seqidno.14,反向引物:5’-acccgagatctgcaatcttctcatggtttcctgc-3’,seqidno.15;
pcr反应体系为:2×phantamaxmastermix25μl,10μm正向引物1μl,10μm反向引物1μl,模板dna1μl,双蒸水补齐至50μl;pcr反应扩增条件为:95℃预变性5min;随后95℃变性1min,55℃30s,72℃5min共30个循环;保存温度为4℃。
采用重叠延伸pcr获得单个位点突变的重组质粒后,用dpnⅰ酶水解上述pcr产物,以除去模板链。酶切体系为:pcr产物46ul,dpnⅰ1ul,10×buffer5ul。酶切条件为:37℃酶切1h。
以单突变体为模板进行pcr,最终获得对应两个位点突变的重组质粒。将上述pcr产物用dpnⅰ酶水解上述pcr产物,以除去模板链。
以双突变体为模板进行pcr,最终获得对应三个位点突变的重组质粒。将上述pcr产物用dpnⅰ酶水解上述pcr产物,以除去模板链。
将酶切产物直接转化大肠杆菌bl21(de3)感受态细胞;涂布卡那霉素抗性平板,筛选阳性克隆;提取质粒测序,分别为含有突变体q1e/133l、q1e/v134a、i133l/v134a、q1e/i133l/v134a的重组菌。
实施例3:β-葡聚糖酶突变体的表达及酶学性质测定
1、β-葡聚糖酶突变体的表达及纯化
挑取实施例1中获得的重组菌株的单菌落接种含50μg/ml卡那霉素的lb液体培养基,37℃180rpm培养10-12h,按4%(v/v)接种量转接至含50μg/ml卡那霉素的tb液体培养基,37℃200rpm培养至od600为0.8-1.0,加入终浓度0.0336mm的iptg和终浓度10mm的α-乳糖,24℃200rpm诱导表达12h,得到发酵液。将发酵液于4℃、8000rpm离心20min,弃菌体收集上清。
采用ni-nta亲和层析柱纯化目的蛋白。蛋白纯化过程中使用到的1×bindingbuffer和不同浓度的咪唑溶液使用前应经0.45μm滤膜真空抽滤,并超声除气20min。
首先用1×bindingbuffer平衡ni-nta亲和层析柱,将得到的上清液装载上柱,随后采用1×bindingbuffer洗脱,直至od280平稳,分别采用50mm、100mm及250mm终浓度的咪唑溶液洗脱目的蛋白,收集洗脱液。
对洗脱液进行酶活测定和sds-page分析,发现突变酶主要在咪唑浓度100mm时被洗脱,且条带单一。将含有目的蛋白的洗脱液经截留分子量3000da的超滤管浓缩后,通过gepd-10脱盐柱将目的蛋白置换至20mm磷酸盐缓冲液(ph6.5),得到纯化的突变酶样品。
2、酶学性质分析
(1)蛋白浓度测定
采用bradford法测定蛋白浓度,具体步骤如下:取200μlbradford工作液与20μl待测样品,迅速混匀;室温25-30℃反应10min,于595nm下测定吸光值,每个样品三个平行,空白样品为ph6.5磷酸盐缓冲液。将所得吸光值带入标准曲线即可得样品的蛋白浓度。
(2)最适温度测定
取适量酶液于不同反应温度(45、50、55、60、65、70℃)测定实施例3中获得的纯化突变酶的酶活,以最高酶活为100%相对酶活,考察酶的最适作用温度(结果见图1)。单突变体q1e和v134a的最适温度与野生酶相同,而i133l、q1e/i133l、q1e/i133l、i133l/v134a和q1e/i133l/v134a的最适温度为60℃,与野生酶相比提高了5℃,说明突变体i133l、q1e/i133l、q1e/i133l、i133l/v134a和q1e/i133l/v134a温度耐受性有所增强。
(3)最适ph值测定
使用20mm醋酸/醋酸钾缓冲液(ph4.0、ph4.5、ph5.0和ph5.5)和20mm磷酸钠缓冲液(ph6.0、ph6.5、ph7.0、ph7.5和ph8.0)与大麦β-葡聚糖底物等体积混合,配置底物反应缓冲液,测定获得的纯化突变酶的酶活,以最高酶活为100%相对酶活,考察酶的最适作用ph(结果见图2)。v134a、q1e/i133l、q1e/i133l、i133l/v134a和q1e/i133l/v134a的最适ph值为ph6.0,与野生酶相同。而单突变体i133l的最适ph值降低了0.5,在ph4.5时相对酶活达到51.17%,而此时野生酶仅有24.51%,说明i133l的最适ph值向酸性环境移动且酸性ph下的活性有所增强。
(4)ph稳定性测定
将获得的纯化突变酶于20mm醋酸/醋酸钾缓冲液(ph4.0、ph4.5、ph5.0和ph5.5)和20mm磷酸钠缓冲液(ph6.0、ph6.5、ph7.0、ph7.5和ph8.0)中稀释,于40℃保存1h后测定其酶活,以未处理样品测定酶活为100%相对酶活,考察酶的ph稳定性(结果见图3)。i133l、q1e/v134a和q1e/i133l/v134a在ph4.0-8.0范围内的相对酶活与野生酶相比无明显提高,v134a和i133l/v134a在ph4.5-6.0范围内的相对酶活略高于野生酶,而突变体q1e和q1e/i133l在ph4.0-8.0范围内的相对酶活均高于野生酶,特别是在ph4.0-5.5范围内的相对酶活明显高于野生酶,在ph4.0条件下水浴处理后相对酶活分别达到76.34%和81.28%,而野生酶仅为27.25%,说明q1e和q1e/i133l的耐酸性得到极大的提高。
实施例4:β-葡聚糖酶突变体动力学参数分析
表2野生酶与β-葡聚糖酶突变体的动力学参数比较
将大麦β-葡聚糖底物与20mm、ph6.5的磷酸钠缓冲液配制成不同浓度的底物溶液(0.5、1.0、1.25、2.5、4.5和10mg·ml-1),于最适条件下与适当浓度的β-葡聚糖酶反应,测定酶活。以单位时间内生成的还原糖的量作为酶反应的初始速度v,由米氏常数方程v=-km·v/[s]+vmax,以测得数据绘制eadi-hofstee曲线,通过线性拟合得到km和vmax值,由vmax和对应的底物浓度可求得酶催化常数kcat值,进一步计算可得催化效率常数kcat/km值,实验结果列于表2。与野生酶相比,i133l、v134a、q1e、q1e/i133l和q1e/i133l/v134a的km值均有所下降,表明酶与底物的亲和力提高;i133l、v134a、q1e和q1e/i133l的比酶活分别提高了30.53%、7.91%、44.63%和16.92%,同时催化效率分别提高了60.94%、14.69%、91.20和33.57%,说明i133l、v134a、q1e和q1e/i133l的催化活性得到了提高。i133l、v134a、q1e和q1e/i133l突变体的kcat值不低于野生酶,而q1e/v134a、i133l/v134a和q1e/i133l/v134a突变体的kcat值显著下降,不利于酶在工业上的应用。
虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
sequencelisting
<110>江南大学
<120>耐酸1,3-1,4-β-葡聚糖酶突变体
<130>baa210358a
<160>15
<170>patentinversion3.3
<210>1
<211>214
<212>prt
<213>人工序列
<400>1
gluthrglyglyserphepheasppropheasnglytyrasnsergly
151015
phetrpglnlysalaaspglytyrserasnglyasnmetpheasncys
202530
thrtrpargalaasnasnvalsermetthrserleuglyglumetarg
354045
leualaleuthrserprosertyrasnlyspheaspcysglygluasn
505560
argservalglnthrtyrglytyrglyleutyrgluvalargmetlys
65707580
proalalysasnthrglyilevalserserphephethrtyrthrgly
859095
prothraspglythrprotrpaspgluileaspileglupheleugly
100105110
lysaspthrthrlysvalglnpheasntyrtyrthrasnglyalagly
115120125
asnhisglulysilevalaspleuglypheaspalaalaasnalatyr
130135140
histhrtyralapheasptrpglnproasnserilelystrptyrval
145150155160
aspglyglnleuserhisthralathrasnglnileprothrthrpro
165170175
glylysilemetmetasnleutrpasnglythrglyvalaspglutrp
180185190
leuglysertyrasnglyvalasnproleutyralahistyrasptrp
195200205
valargtyrthrlyslys
210
<210>2
<211>214
<212>prt
<213>人工序列
<400>2
glnthrglyglyserphepheasppropheasnglytyrasnsergly
151015
phetrpglnlysalaaspglytyrserasnglyasnmetpheasncys
202530
thrtrpargalaasnasnvalsermetthrserleuglyglumetarg
354045
leualaleuthrserprosertyrasnlyspheaspcysglygluasn
505560
argservalglnthrtyrglytyrglyleutyrgluvalargmetlys
65707580
proalalysasnthrglyilevalserserphephethrtyrthrgly
859095
prothraspglythrprotrpaspgluileaspileglupheleugly
100105110
lysaspthrthrlysvalglnpheasntyrtyrthrasnglyalagly
115120125
asnhisglulysleuvalaspleuglypheaspalaalaasnalatyr
130135140
histhrtyralapheasptrpglnproasnserilelystrptyrval
145150155160
aspglyglnleuserhisthralathrasnglnileprothrthrpro
165170175
glylysilemetmetasnleutrpasnglythrglyvalaspglutrp
180185190
leuglysertyrasnglyvalasnproleutyralahistyrasptrp
195200205
valargtyrthrlyslys
210
<210>3
<211>214
<212>prt
<213>人工序列
<400>3
glnthrglyglyserphepheasppropheasnglytyrasnsergly
151015
phetrpglnlysalaaspglytyrserasnglyasnmetpheasncys
202530
thrtrpargalaasnasnvalsermetthrserleuglyglumetarg
354045
leualaleuthrserprosertyrasnlyspheaspcysglygluasn
505560
argservalglnthrtyrglytyrglyleutyrgluvalargmetlys
65707580
proalalysasnthrglyilevalserserphephethrtyrthrgly
859095
prothraspglythrprotrpaspgluileaspileglupheleugly
100105110
lysaspthrthrlysvalglnpheasntyrtyrthrasnglyalagly
115120125
asnhisglulysilealaaspleuglypheaspalaalaasnalatyr
130135140
histhrtyralapheasptrpglnproasnserilelystrptyrval
145150155160
aspglyglnleuserhisthralathrasnglnileprothrthrpro
165170175
glylysilemetmetasnleutrpasnglythrglyvalaspglutrp
180185190
leuglysertyrasnglyvalasnproleutyralahistyrasptrp
195200205
valargtyrthrlyslys
210
<210>4
<211>214
<212>prt
<213>人工序列
<400>4
gluthrglyglyserphepheasppropheasnglytyrasnsergly
151015
phetrpglnlysalaaspglytyrserasnglyasnmetpheasncys
202530
thrtrpargalaasnasnvalsermetthrserleuglyglumetarg
354045
leualaleuthrserprosertyrasnlyspheaspcysglygluasn
505560
argservalglnthrtyrglytyrglyleutyrgluvalargmetlys
65707580
proalalysasnthrglyilevalserserphephethrtyrthrgly
859095
prothraspglythrprotrpaspgluileaspileglupheleugly
100105110
lysaspthrthrlysvalglnpheasntyrtyrthrasnglyalagly
115120125
asnhisglulysleuvalaspleuglypheaspalaalaasnalatyr
130135140
histhrtyralapheasptrpglnproasnserilelystrptyrval
145150155160
aspglyglnleuserhisthralathrasnglnileprothrthrpro
165170175
glylysilemetmetasnleutrpasnglythrglyvalaspglutrp
180185190
leuglysertyrasnglyvalasnproleutyralahistyrasptrp
195200205
valargtyrthrlyslys
210
<210>5
<211>214
<212>prt
<213>bacillustequilensis
<400>5
glnthrglyglyserphepheasppropheasnglytyrasnsergly
151015
phetrpglnlysalaaspglytyrserasnglyasnmetpheasncys
202530
thrtrpargalaasnasnvalsermetthrserleuglyglumetarg
354045
leualaleuthrserprosertyrasnlyspheaspcysglygluasn
505560
argservalglnthrtyrglytyrglyleutyrgluvalargmetlys
65707580
proalalysasnthrglyilevalserserphephethrtyrthrgly
859095
prothraspglythrprotrpaspgluileaspileglupheleugly
100105110
lysaspthrthrlysvalglnpheasntyrtyrthrasnglyalagly
115120125
asnhisglulysilevalaspleuglypheaspalaalaasnalatyr
130135140
histhrtyralapheasptrpglnproasnserilelystrptyrval
145150155160
aspglyglnleuserhisthralathrasnglnileprothrthrpro
165170175
glylysilemetmetasnleutrpasnglythrglyvalaspglutrp
180185190
leuglysertyrasnglyvalasnproleutyralahistyrasptrp
195200205
valargtyrthrlyslys
210
<210>6
<211>645
<212>dna
<213>人工序列
<400>6
gaaacaggtggatcgttttttgacccttttaacggctataactccggtttttggcaaaaa60
gcagatggttattcgaatggaaatatgttcaactgcacgtggcgggctaataacgtatca120
atgacgtcattgggtgaaatgcgtttagcgctaacaagcccatcttataacaagtttgac180
tgcggggaaaaccgttctgttcaaacatatggctatggactttatgaagtcagaatgaaa240
ccagctaaaaacacagggatcgtttcatcgttcttcacttacacaggtccaacagatgga300
actccttgggatgagattgatatcgaatttttaggaaaagacacaacgaaggttcaattt360
aactattatacaaatggtgcaggaaaccatgagaagattgttgatctcgggtttgatgca420
gccaatgcctatcatacttatgcattcgattggcagccaaactctattaaatggtatgtc480
gacgggcaattaaaacatactgcaacaaaccaaattccgacaacacctggaaagatcatg540
atgaacttgtggaatggcacgggtgtcgatgaatggcttggctcctacaatggtgtaaat600
ccgctatacgctcattatgactgggtgcgctatacaaaaaaataa645
<210>7
<211>645
<212>dna
<213>人工序列
<400>7
caaacaggtggatcgttttttgacccttttaacggctataactccggtttttggcaaaaa60
gcagatggttattcgaatggaaatatgttcaactgcacgtggcgggctaataacgtatca120
atgacgtcattgggtgaaatgcgtttagcgctaacaagcccatcttataacaagtttgac180
tgcggggaaaaccgttctgttcaaacatatggctatggactttatgaagtcagaatgaaa240
ccagctaaaaacacagggatcgtttcatcgttcttcacttacacaggtccaacagatgga300
actccttgggatgagattgatatcgaatttttaggaaaagacacaacgaaggttcaattt360
aactattatacaaatggtgcaggaaaccatgagaagctggttgatctcgggtttgatgca420
gccaatgcctatcatacttatgcattcgattggcagccaaactctattaaatggtatgtc480
gacgggcaattaaaacatactgcaacaaaccaaattccgacaacacctggaaagatcatg540
atgaacttgtggaatggcacgggtgtcgatgaatggcttggctcctacaatggtgtaaat600
ccgctatacgctcattatgactgggtgcgctatacaaaaaaataa645
<210>8
<211>645
<212>dna
<213>人工序列
<400>8
caaacaggtggatcgttttttgacccttttaacggctataactccggtttttggcaaaaa60
gcagatggttattcgaatggaaatatgttcaactgcacgtggcgggctaataacgtatca120
atgacgtcattgggtgaaatgcgtttagcgctaacaagcccatcttataacaagtttgac180
tgcggggaaaaccgttctgttcaaacatatggctatggactttatgaagtcagaatgaaa240
ccagctaaaaacacagggatcgtttcatcgttcttcacttacacaggtccaacagatgga300
actccttgggatgagattgatatcgaatttttaggaaaagacacaacgaaggttcaattt360
aactattatacaaatggtgcaggaaaccatgagaagattgcagatctcgggtttgatgca420
gccaatgcctatcatacttatgcattcgattggcagccaaactctattaaatggtatgtc480
gacgggcaattaaaacatactgcaacaaaccaaattccgacaacacctggaaagatcatg540
atgaacttgtggaatggcacgggtgtcgatgaatggcttggctcctacaatggtgtaaat600
ccgctatacgctcattatgactgggtgcgctatacaaaaaaataa645
<210>9
<211>645
<212>dna
<213>人工序列
<400>9
gaaacaggtggatcgttttttgacccttttaacggctataactccggtttttggcaaaaa60
gcagatggttattcgaatggaaatatgttcaactgcacgtggcgggctaataacgtatca120
atgacgtcattgggtgaaatgcgtttagcgctaacaagcccatcttataacaagtttgac180
tgcggggaaaaccgttctgttcaaacatatggctatggactttatgaagtcagaatgaaa240
ccagctaaaaacacagggatcgtttcatcgttcttcacttacacaggtccaacagatgga300
actccttgggatgagattgatatcgaatttttaggaaaagacacaacgaaggttcaattt360
aactattatacaaatggtgcaggaaaccatgagaagctggttgatctcgggtttgatgca420
gccaatgcctatcatacttatgcattcgattggcagccaaactctattaaatggtatgtc480
gacgggcaattaaaacatactgcaacaaaccaaattccgacaacacctggaaagatcatg540
atgaacttgtggaatggcacgggtgtcgatgaatggcttggctcctacaatggtgtaaat600
ccgctatacgctcattatgactgggtgcgctatacaaaaaaataa645
<210>10
<211>30
<212>dna
<213>人工序列
<400>10
tcacttctactgcctcggctgaaacaggtg30
<210>11
<211>32
<212>dna
<213>人工序列
<400>11
tcaaaaaacgatccacctgtttcagccgaggc32
<210>12
<211>36
<212>dna
<213>人工序列
<400>12
aaccatgagaagctggttgatctcgggtttgatgca36
<210>13
<211>34
<212>dna
<213>人工序列
<400>13
cgagatcaaccagcttctcatggtttcctgcacc34
<210>14
<211>34
<212>dna
<213>人工序列
<400>14
tgagaagattgcagatctcgggtttgatgcagcc34
<210>15
<211>34
<212>dna
<213>人工序列
<400>15
acccgagatctgcaatcttctcatggtttcctgc34
- 还没有人留言评论。精彩留言会获得点赞!