本发明涉及核酸测序领域,具体涉及一种基于pacbio三代测序平台的mrna 3’末端文库构建方法。
背景技术:
1、dna测序技术的出现,极大地推动了生物学的发展。随着测序技术的进步,下一代测序(ngs)技术已广泛应用于生命科学,其制备核酸测序和构建ngs文库的方法也在不断发展。ngs文库构建的基础是将靶标核酸rna或dna制备成与所用测序系统兼容的形式。目前已成功构建了多种ngs文库,包括基因组dna文库、mrna文库、srna文库等。为了解转录本poly(a)尾末端核苷酸修饰类型,多种rna3′末端ngs文库被成功构建用于poly(a)尾末端核苷酸修饰。
2、为了直接测定转录组的3'端序列,最初开发出来的ngs文库为tail-seq,用于二代测序平台(illumina hiseq)。基于5'末端产生51nt的序列(读取1,用于基因组定位以识别转录物)和从3'末端产生231nt的序列(读取2,用于3'末端序列测定)而实现转录组的3'端的检测,并成功鉴定到mrna3'末端广泛存在的尿苷酸和鸟苷酸修饰。由于二代测序平台(illumina hiseq)最大测序长度为250nt,因此tail-seq文库构建过程中需要rnase t1部分消化mrna,然后切胶回收500-1000nt长度的mrna,这种处理方法会漏掉部分不在500-1000nt长度范围的mrna,造成数据失真。
3、tail-seq技术限于测序长度的原因,无法真正还原mrna和poly(a)尾在体内的真实状态,因此,研究人员又开发了一种互补的dna文库制备方法,即flam-seq技术。flam-seq是一种快速简便的方法,使用pacbio三代测序平台可对整个mrna进行高质量测序。该方法与单分子测序相结合,实现了转录组全长mrna和精准的poly(a)尾序列测定,展现了非a核苷酸可以掺入poly(a)尾中。但缺点是它无法检测poly(a)尾3′最末端的序列。
4、此外,另一种三代测序纳米孔测序平台rna直接测序技术(nanopore direct rnasequencing)也能检测全长mrna和poly(a)尾序列(workman re et al.,2019),这种测序只需要在mrna3′末端先后加上poly(t)接头和测序接头,不经反转、无须扩增就可以对mrna直接测序。这种方法能够直接读取mrna序列和mrna3′末端不同的修饰类型,缺点在于错误率高而无法确认mrna3′末端的修饰是由测序错误造成,还是非模板添加修饰。
技术实现思路
1、基于以上不足之处,本发明提供一种基于pacbio三代测序平台的mrna 3’末端文库构建方法,更好的解决mrna的3′末端修饰问题。
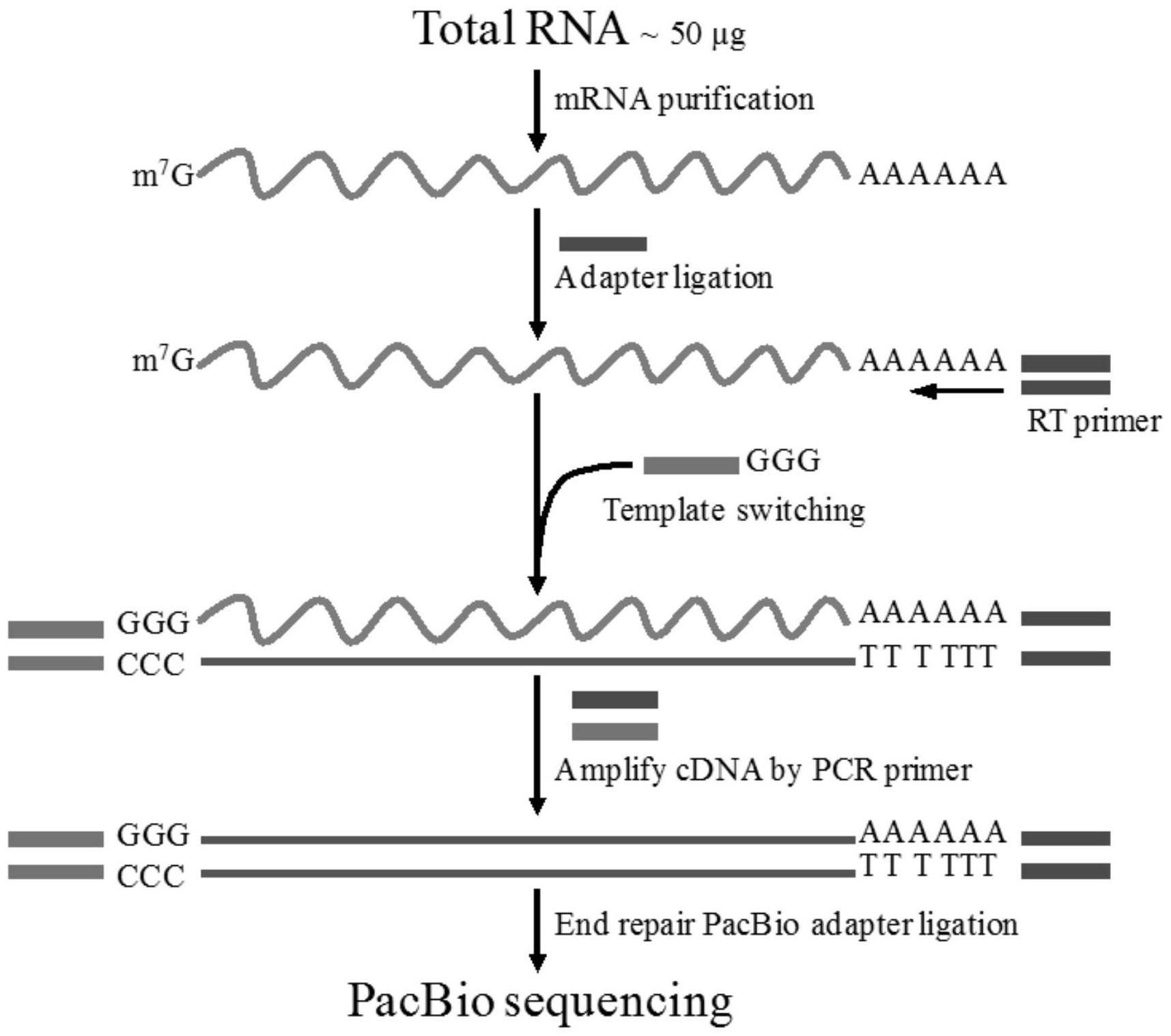
2、本发明所采用的技术方案如下:一种基于pacbio三代测序平台的mrna 3’末端文库构建方法,方法如下:通过sera-mag oligo(dt)bead方法纯化mrna,并在纯化后的mrna3’末端添加接头序列,以及利用template switch oligo的方法得到包含转录物的完整5'端和3’端的反转录cdna,并且选择3’通用引物和5'通用引物进行pcr扩增,从而获得完整的双链cdna,t-a克隆并测序发现能够检测到poly(a)尾端;最后,在3’通用引物和5'通用引物的5’端加上index序列并进行pcr扩增,将pcr扩增产物进行pacbio sequelⅱccs测序,产出符合要求且稳定的测序数据,表明已成功构建适用于检测mrna3’末端的三代测序pacbio文库。
3、进一步的,纯化后的mrna 3’末端添加接头序列如下:5′-rappagatcggaagagcacacgtct-nh2-3′,其中,3’接头序列的5’端预腺苷化处理,3’端氨基封闭处理。
4、进一步的,以3’接头为rt引物:5′-agacgtgtgctcttccgatct-3′,并利用templateswitch oligo方法其所采用的tso序列如下:5'–aagcagtggtatcaacgcagagtacgcggg-3',得到反转录cdna。
5、本发明具有如下优点及效果:本发明的方法构建的文库长读长,可实现mrna和poly(a)尾检测;可以检测poly(a)尾3′最末端的序列,即mrna3′末端非模板添加序列;测序准确率高。
技术特征:1.一种基于pacbio三代测序平台的mrna 3’末端文库构建方法,其特征在于,方法如下:通过sera-mag oligo(dt)bead方法纯化mrna,并在纯化后的mrna 3’末端添加接头序列,以及利用template switch oligo的方法得到包含转录物的完整5'端和3’端的反转录cdna,并且选择3’通用引物和5'通用引物进行pcr扩增,从而获得完整的双链cdna,t-a克隆并测序发现能够检测到poly(a)尾端;最后,在3’通用引物和5'通用引物的5’端加上index序列并进行pcr扩增,将pcr扩增产物进行pacbio sequelⅱccs测序,产出符合要求且稳定的测序数据。
2.根据权利要求1所述的一种基于pacbio三代测序平台的mrna 3’末端文库构建方法,其特征在于,纯化后的mrna 3’末端添加接头序列如下:5′-rappagatcggaagagcacacgtct-nh2-3′,其中,3’接头序列的5’端预腺苷化处理,3’端氨基封闭处理。
3.根据权利要求2所述的一种基于pacbio三代测序平台的mrna 3’末端文库构建方法,其特征在于,以3’接头为rt引物:
技术总结本发明涉及核酸测序领域,具体涉及一种基于PacBio三代测序平台的mRNA3’末端文库构建方法,解决了mRNA的3′末端修饰问题。通过Sera‑Mag Oligo(dT)Bead方法纯化mRNA,并在纯化后的mRNA 3’末端添加接头序列,以及利用Template switch oligo的方法得到包含转录物的完整5'端和3’端的反转录cDNA,并且选择3’通用引物和5'通用引物进行PCR扩增,从而获得完整的双链cDNA,T‑A克隆并测序发现能够检测到poly(A)尾端;最后,在3’通用引物和5'通用引物的5’端加上Index序列并进行PCR扩增,将PCR扩增产物进行PacBio SequelⅡCCS测序,产出符合要求且稳定的测序数据。本发明可以检测poly(A)尾3′最末端的序列,测序准确率高。
技术研发人员:莫小为,于宇,孔稳稳,王铭亮,莫蓓莘
受保护的技术使用者:深圳大学
技术研发日:技术公布日:2024/1/13
