一种体内DNA存储、随机读取和改写的方法
本发明属于数据存储,具体涉及一种体内dna存储、随机读取和改写的方法。
背景技术:
1、随着网络技术的进步,信息的交流以及产生都呈爆炸式的增长。对于如此海量的信息,如何存储将成为亟需面对的问题。现有的硅基存储技术已经无法满足需求的增长,研究者已经将目光聚焦在其它物质上,其中脱氧核糖核酸(dna)作为存储介质的研究更是研究的热点。dna作为信息存储介质,具有存储密度高、存储时间长且稳定、能耗低的优势。然而,dna信息存储数据库中信息的随机读取和重写仍然是一个巨大挑战。
2、考虑到效率和成本,通过测序或重写数据库中所有dna来读取或更新部分数据是不切实际的,因此在过去几年中,设计了各种方法从dna数据库中选择性读取数据。最初,采用聚合酶链式反应(pcr)选择性地读取数据,通过使用序列上随机访问适配器对应的引物从dna库中扩增目标dna序列。这种方法成功地从200mb的dna数据库中检索数据。然而,编码随机访问适配器产生的存储空间成本过高,随机访问对dna数据库不可逆的消耗,以及不能扩增长dna数据链(pcr通常只能扩增小于10kb的dna数据链)。最近,出现了一些新方法用于随机访问dna数据库,例如,使用生物素标记的引物进行磁珠提取,或使用表面标记的单链dna条形码访问二氧化硅包被的dna数据。然而,这些方法在数据可重用性、实验复杂性以及实用性方面仍然具有相当大的局限性。此外,目前基于dna的存储系统普遍缺乏数据重写能力,限制了其应用仅限于只读数据存储。受到细胞复制信息能力的启发,设计了各种用于体内数据存储的方法,为体内dna数据存储打开了大门。最近的一项研究描述了一种细胞内基因组编辑方法,该方法成功地从dna存储的数据中重写了一些“单词”,然而,用这种方法操作大型数据文件可能具有挑战性,而且它没有考虑到数据的随机读取。
3、总的来说,目前的dna数据存储系统存在大数据文件随机读取或重写困难的问题,一般不能在单个系统内实现随机读取和重写,严重制约了其实际应用。
技术实现思路
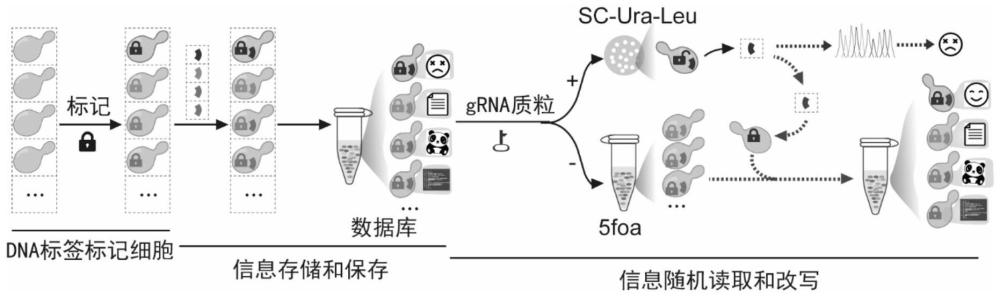
1、有鉴于此,本发明的目的在于针对现有技术dna存储信息库无法删除和重复改写的问题,提供了一种体内dna存储、随机读取和改写的方法,能够精准的删除后和改写体内存储的dna信息,且dna数据库可重复使用。
2、为实现上述目的,本发明采用如下技术方案:
3、一种体内dna存储、随机读取和改写的方法,所述dna存储包括以下步骤:
4、生成用于标记不同菌株的标签tag;
5、构建元件顺序为酿酒酵母ix染色体左臂、ura3-n、标签tag、ura3-c、nat、酿酒酵母ix染色体右臂的ura3标签;
6、构建元件顺序为酿酒酵母iii染色体左臂、leu2-n、标签tag、leu2-c、hygb、酿酒酵母iii染色体右臂的leu2标签;
7、将所述ura3标签和leu2标签依次插入表达cas9蛋白的酿酒酵母的ix和iii染色体,筛选成功插入dna标签的菌株;
8、向成功插入dna标签的菌株中转入存有目的信息的dna,标签tag序列与存储信息一一对应,实现信息特异性标记,得到有dna标签标记的信息菌株。
9、进一步的,按照同样的方法构建存储其他信息的信息菌株,将所有的信息菌株一起保存,构成dna数据库。
10、进一步的,所述标签tag需满足以下条件:
11、序列表达的grna不靶向基因组序列;cg含量在40-60%,均聚物长度小于4,无发夹结构;序列表达的grna不靶向其它tag序列。
12、更进一步的,标签tag序列前12bp与基因组序列至少两个错配,以使序列表达的grna不靶向基因组序列,标签tag序列相互之间前12bp至少两个错配,以使序列表达的grna不靶向其它标签tag序列。
13、更进一步的,所述ura3-n和ura3-c带有100bp同源序列,leu2-n和leu2-c带有100bp同源序列。
14、更进一步的,所述随机读取包括以下步骤:
15、构建靶向标签tag且含有质粒消除元件的grna表达质粒;
16、将目的信息对应标签tag的grna表达质粒转入dna数据库,使用营养缺陷型培养基筛选出带有目的信息的菌株,对目的信息进行读取和编辑。
17、更进一步的,所述grna表达质粒由prs421作为载体骨架,插入靶向标签tag的通用grna表达框和靶向2u ori的pgals启动子的grna表达框,靶向2μori的pgals启动子的grna表达框作为质粒消除元件。
18、更进一步的,所述营养缺陷型培养基为固体培养基cm-met-his-leu-ura。
19、进一步的,所述改写包括以下步骤:将目的信息对应标签tag的grna表达质粒转入dna数据库,使用添加5-氟乳清酸的液体培养基培养,删除带有目的信息的菌株,进一步将编辑后的dna信息放回dna数据库,实现数据库信息改写和更新。
20、更进一步的,删除带有目的信息的菌株后,使用半乳糖液体培养基培养实现grna表达质粒的消除。
21、本发明具有如下有益效果:
22、本发明利用crispr-cas9系统靶向特异性序列的特点,设计了可用于标记不同菌株的dna标签,使用dna标签标记携带不同存储信息的菌株。通过dna标签正筛选可筛选出含有目的存储信息的菌株,实现随机获取dna数据库的信息;通过dna标签可负筛选掉含有目的存储信息的菌株,实现随机删除dna数据库的信息;将删除信息更改后,放回dna数据库,可实现体内dna信息存储的改写功能。经过删除和更改的dna数据库可进行下一步的读取、删除和改写。
技术特征:
1.一种体内dna存储、随机读取和改写的方法,其特征在于,所述dna存储包括以下步骤:
2.根据权利要求1所述的方法,其特征在于,按照同样的方法构建存储其他信息的信息菌株,将所有的信息菌株一起保存,构成dna数据库。
3.根据权利要求1所述的方法,其特征在于,所述标签tag需满足以下条件:
4.根据权利要求3所述的方法,其特征在于,标签tag序列前12bp与基因组序列至少两个错配,以使序列表达的grna不靶向基因组序列,标签tag序列相互之间前12bp至少两个错配,以使序列表达的grna不靶向其它标签tag序列。
5.根据权利要求1所述的方法,其特征在于,所述ura3-n和ura3-c带有100bp同源序列,leu2-n和leu2-c带有100bp同源序列。
6.根据权利要求2所述的方法,其特征在于,所述随机读取包括以下步骤:
7.根据权利要求6所述的方法,其特征在于,所述grna表达质粒由prs421作为载体骨架,插入靶向标签tag的通用grna表达框和靶向2u ori的pgals启动子的grna表达框,靶向2μori的pgals启动子的grna表达框作为质粒消除元件。
8.根据权利要求7所述的方法,其特征在于,所述营养缺陷型培养基为固体培养基cm-met-his-leu-ura。
9.根据权利要求6所述的方法,其特征在于,所述改写包括以下步骤:将目的信息对应标签tag的grna表达质粒转入dna数据库,使用添加5-氟乳清酸的液体培养基培养,删除带有目的信息的菌株,进一步将编辑后的dna信息放回dna数据库,实现数据库信息改写和更新。
10.根据权利要求9所述的方法,其特征在于,删除带有目的信息的菌株后,使用半乳糖液体培养基培养实现grna表达质粒的消除。
技术总结
本发明属于数据存储技术领域,具体涉及一种体内DNA存储、随机读取和改写的方法。本发明利用CRISPR‑Cas9系统靶向特异性序列的特点,设计了可用于标记不同菌株的DNA标签,使用DNA标签标记携带不同存储信息的菌株。通过DNA标签正筛选可筛选出含有目的存储信息的菌株,实现DNA数据库信息的随机读取功能;通过DNA标签可负筛选掉含有目的存储信息的菌株,实现DNA数据库信息的随机删除功能;将删除信息更改后,放回DNA数据库,可实现体内DNA信息存储的改写功能。经过删除和更改的DNA数据库可进行下一步的读取、删除和改写。
技术研发人员:赵广厚,侯照华,王翔翔
受保护的技术使用者:西北工业大学
技术研发日:
技术公布日:2024/4/29
- 还没有人留言评论。精彩留言会获得点赞!