乳腺癌诊断标志物及其应用的制作方法
1.本发明涉及肿瘤诊断领域,具体涉及乳腺癌诊断标志物及其应用。
背景技术:
2.乳腺癌是女性常见的恶性肿瘤之一,全球肿瘤流行病学统计数据显示,2012年全球乳腺癌新发病例约为170万人次,死亡病例约占52.2万人次。在中国乳腺癌已成为女性高发肿瘤中的第一位,美国等西方国家乳腺癌的发病率和死亡率占所有肿瘤的第二位。随着当代医学治疗水平的不断进步,乳腺癌的治愈率及生存率都有显著的提高,但是乳腺癌的转移和复发仍然是威胁着乳腺癌患者的严重的危险因素,转移和复发的出现不仅会增加治疗的难度,更会降低患者的生存时间。有文献报道,首发乳腺癌患者的5年生存率可达到60%-70%,而乳腺癌局部复发后患者的5年生存率仅为42%-49%。因此,深入研究乳腺癌复发转移的机制,寻找新的影响乳腺癌预后的关键因素,并以此为基础开展研发防治乳腺癌复发转移的有效措施是目前乳腺癌研究中的重要的科学问题,对进一步提高乳腺癌的整体治疗水平具有十分重要的临床意义。
3.目前治疗实体瘤的手段包括直接攻击,如化疗、放疗和手术。然而,这些手段会对正常组织产生一定损害。与标准化疗不同,靶向治疗策略旨在通过更好地靶向肿瘤,避免损伤正常组织。靶向药物通过识别肿瘤细胞的致癌部位(细胞表面抗原、膜蛋白分子或基因片段)来诱导肿瘤细胞坏死和凋亡。食品和药物管理局批准了许多靶向治疗方法来治疗某些类型的癌症。内分泌治疗(et)彻底改变了乳腺癌的治疗方法,尤其是在管腔肿瘤患者中,et占辅助全身治疗的大部分收益。雌激素受体(er)和her2是治疗的主要靶点和药物开发的重点。尽管目前可用的et药物,如他莫昔芬和曲妥珠单抗,通常有效且耐受性良好,但并非所有患者都能平等获益。因此,对于乳腺癌患者,需要确定新型的基因作为乳腺癌生物标志物和治疗靶点,以改善早期诊断和治疗结果。
4.adrm1被鉴定为转移性肿瘤细胞中的粘附调节分子-1,编码一种完整的质膜蛋白,参与发育、细胞粘附、去泛素化和蛋白质水解。作为26s蛋白酶体上的两种主要泛素受体之一,adrm1可以选择性地介导关键蛋白质的降解,并参与多种细胞过程。研究表明,人类adrm1在肿瘤发生中的潜在作用,也被确定为癌症中的扩增靶点,其过度表达与早期癌症复发和低生存率显著相关。靶向adrm1的药物可能是癌症治疗的良好策略。tp53肿瘤抑制蛋白作为细胞应激传感器,在诸如dna损伤和癌基因激活(致癌应激)等应激条件下被激活。这可以通过下游靶基因的反式激活抑制细胞分裂或存活。此外,tp53还参与多种其他细胞过程,包括dna修复、分化和干细胞更新。tp53的种系突变可能导致更高的乳腺癌风险。突变的tp53作为乳腺癌治疗的靶点有几个吸引人的特点。首先,tp53在大约30%的所有病例和大约80%的三阴性(tn)肿瘤(即er、pr和her2阴性的肿瘤)病例中发生突变,表明tp53是乳腺癌中最常见的突变基因。其次,该基因在几乎90%的有脑转移的乳腺癌患者中发生突变。最后,tp53突变是克隆性的,应该存在于乳腺癌的所有恶性细胞中。
技术实现要素:
5.本发明的目的在于提供一种乳腺癌诊断标志物及其应用,adrm1和tp53蛋白诊断标志物均与乳腺癌发病率呈正相关,在乳腺癌组织中呈高表达,通过adrm1蛋白和tp53蛋白联合检测肿瘤患者组织中adrm1和tp53蛋白的表达,准确率高。
6.本发明解决其技术问题所采用的技术方案是:
7.用于乳腺癌诊断的adrm1和tp53蛋白生物标志物,所述adrm1和tp53蛋白生物标志物可用于检测肿瘤患者组织中的adrm1和tp53蛋白的表达。
8.adrm1和tp53蛋白在制备联合诊断乳腺癌产品中的应用,该乳腺癌诊断的adrm1和tp53蛋白生物标志物可用于乳腺癌辅助诊断。
9.优选的,乳腺癌诊断产品包括免疫组化方法检测生物学样品中adrm1和tp53蛋白表达水平。
10.优选的,免疫组化检测试剂包括二甲苯、乙醇、磷酸盐缓冲液、dba显色试剂、苏木素和树胶。
11.优选的,检测蛋白表达水平的方法包括:(1)检测adrm1和tp53蛋白在生物学样品中的表达水平;(2)adrm1和tp53蛋白表达水平的升高与乳腺癌相关。
12.本发明的有益效果:
13.本发明首次公开了可用于乳腺癌诊断的adrm1蛋白生物标志物及其应用,adrm1蛋白生物标志物在乳腺癌组织中呈高表达,与乳腺癌的发生呈正相关,是潜在的乳腺癌诊断的生物标志物。adrm1蛋白生物标志物的开发,有望成为乳腺癌辅助诊断和靶向治疗的一种新手段。
14.本发明首次发现tp53突变型和非突变型乳腺癌中adrm1的表达水平显著高于正常乳腺癌中的表达水平,表明adrm1的表达与tp53在乳腺癌中的表达密切相关。通过adrm1和tp53蛋白联合检测肿瘤患者组织中adrm1和tp53蛋白的表达,检测结果准确率高。
附图说明
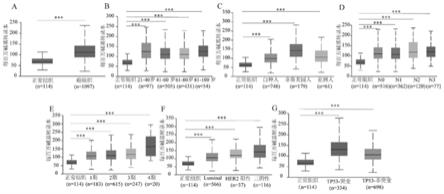
15.图1使用ualcan数据库根据乳腺癌患者临床参数评估不同分组时adrm1表达情况,(a)样本类型,(b)年龄,(c)种族,(d)淋巴结状态,(e)组织学分级,(f)乳腺癌分类,(g)tp53突变状态,*p《0.05,**p《0.01,***p《0.001。
16.图2使用ualcan数据库根据乳腺癌患者临床参数评估不同分组时tp53的表达情况,(a)样本类型,(b)年龄,(c)种族,(d)淋巴结状态,(e)组织学分级,(f)乳腺癌分类,*p《0.05,**p《0.01,***p《0.001。
17.图3为利用cytoscape构建adrm1和tp53的基因-基因相互作用网络图。
18.图4为adrm1在乳腺癌患者中的生存分析图。
19.图5为hsa-let-7b在乳腺癌患者中的生存分析图。
20.图6为hsa-mir-125b在乳腺癌患者中的生存分析图。
21.图7为hsa-let-7b、hsa-mir-125b与adrm1、tp53匹配的碱基序列图。
22.图8为hsa-let-7b-5p和hsa-mir-125b-5p在乳腺癌组织和正常组织中的表达图。
23.图9为mirna的靶基因go和kegg富集分析图。
具体实施方式
24.下面对本发明涉及的结构或这些所使用的技术术语做进一步的说明。这些说明仅仅是采用举例的方式进行说明本发明的方式是如何实现的,并不能对本发明构成任何的限制。
25.实施例1adrm1蛋白在乳腺癌组织中的表达水平测定
26.(1)样本收集
27.收集本院经病理诊断的乳腺癌患者手术切除组织样本148例,患者入选标准为:手术切除病理证实为乳腺癌;术前未行放疗、化疗、激素及生物靶向治疗。
28.(2)免疫组织化学方法检测
29.将乳腺癌巢组织石蜡标本连续4μm切片,常规脱蜡处理,pbs清洗,按照免疫组化试剂盒说明书操作,小鼠抗人adrm1稀释度为l:500,小鼠抗人tp53稀释度为l:100,以磷酸盐缓冲液(pbs)代替一抗作为阴性对照。dab室温显色5-l0min,自来水冲洗,苏木素复染,梯度乙醇脱水,二甲苯透明,树胶封片,光学显微镜观察。
30.(3)半定量法对免疫组化结果分析
31.以肿瘤细胞染色强度结合阳性细胞比例进行评分。染色强度评分以大多数细胞呈现的染色状态为依据,计分标准:无着色计为0分,淡黄色计为1分,棕黄色计为2分,棕褐色计为3分。阳性细胞比例评分是在光学显微镜下随机选择5个高倍镜视野(400
×
),计算呈现染色阳性的肿瘤细胞占视野内所有肿瘤细胞的百分比,以各视野的均值作为阳性细胞百分比,计分标准:无肿瘤细胞着色计为0分,1%-30%肿瘤细胞着色计为1分,30%-60%肿瘤细胞着色计为2分,﹥60%肿瘤细胞着色计为3分。以染色强度评分与阳性细胞比例评分的乘积为最终分值。评分时避开肿瘤坏死区域和肿瘤边缘部分,评分只包括肿瘤细胞,肿瘤间质细胞不作计数。最终分值0-3分定义为低表达,4-9分定义为高表达。
32.(4)免疫组化结果判定
33.由两名高年资临床病理科医师使用同一病理显微镜各自独立进行,对于结果不一致的切片,邀请另一位病理科医师会同原来两位医师集体判定。
34.(5)乳腺癌组织中adrm1表达与临床特征的相关性分析
35.计量资料采用x
±
s进行描述,采用两独立样本t检验;计数资料采用率(或百分比)进行统计学描述,采用两独立样本卡方检验;数据整理及分析过程均在spss 22.0软件中进行,p<0.05(双侧)数据有统计学意义,结果见表1。
36.表1adrm1和tp53表达与乳腺癌临床病理参数的关系
37.[0038][0039]
结果显示,148例乳腺癌患者中,adrm1高表达83例,低表达65例,adrm1蛋白的表达与乳腺癌患者的临床分期具有显著的相关性,adrm1蛋白的表达量越高其临床分期越高,即adrm1蛋白的表达越高表明乳腺癌的恶性程度越高。
[0040]
实施例2乳腺癌组织和正常组织中adrm1和tp53基因的表达差异
[0041]
利用ualcan数据库(http://ualcan.path.uab.edu/index.html)分析乳腺癌中adrm1的表达水平。筛选条件如下:gene symbol=adrm1;tcga dataset=breast invasive carcinoma。满足条件的乳腺癌样本有1097例,正常组织样本有114例。并利用ualcan数据库进一步分析adrm1与年龄、种族、淋巴结转移分期、组织学分级和乳腺癌分类的关系以及在突变型和非突变型tp53乳腺癌样本中的表达情况,结果见图1。
[0042]
与正常对照组相比,乳腺癌患者的adrm1表达水平升高(p《0.001,图1a),并且adrm1的表达水平与年龄、种族、淋巴结转移分期、组织学分级和乳腺癌分类显著相关(图1b-f)。此外,tp53突变型和非突变型乳腺癌中adrm1的表达水平显著高于正常乳腺癌中的表达水平(图1g),表明adrm1的表达与tp53在乳腺癌中的表达密切相关。
[0043]
利用ualcan数据库(http://ualcan.path.uab.edu/index.html)分析乳腺癌中tp53的表达水平。筛选条件如下:gene symbol=adrm1;tcga dataset=breast invasive carcinoma。满足条件的乳腺癌样本1097例,正常组织样本114例。并利用ualcan数据库进一步分析tp53与年龄、种族、淋巴结转移分期、组织学分级和乳腺癌分类的关系,结果见图2。与正常对照组相比,乳腺癌患者的tp53表达水平升高(p《0.001,图2a),并且tp53的表达水平与年龄、种族、淋巴结转移分期、组织学分级和乳腺癌分类显著相关(图2b-f)。
[0044]
实施例3adrm1和tp53的mirna的筛选方法
[0045]
(1)筛选共同作用于adrm1和tp53的mirna
[0046]
通过在线数据库mirtarbase(http://www.mirbase.org/)、mirtarbase(http://mirtarbase.cuhk.edu.cn/php/index.php)、targetscan(http://www.targetscan.org/mamm_31/)和transmir(http://www.cuilab.cn/transmir)预测adrm1和tp53与mirna的互作关系。采用cytoscape软件(www.cytoscape.org/)构建和可视化mirna对adrm1和tp53的调控网络。结果发现,包括hsa-let-7b-5p,hsa-mir-125b-5p,hsa-mir-24-3p,hsa-mir-1233-5p,hsa-mir-5693,hsa-mir-149-3p,hsa-mir-6766-5p,hsa-mir-4728-5p,hsa-mir-6756-5p,hsa-mir-19b-3,hsa-mir-6778-5p,hsa-mir-194-3p,hsa-mir-6785-5p,hsa-mir-19a-3p和hsa-mir-6883-5在内的15个mirna共同靶向adrm1和tp53(图3)。
[0047]
(2)进一步筛选共同作用于adrm1和tp53的mirna
[0048]
使用kaplan-meier(www.kmplot.com)评估乳腺癌中adrm1、tp53及互作mirna表达水平的预后意义。根据基因表达水平将数据分为高表达组和低表达组,并分析不同her、er和淋巴结状态下的总生存率(os)。计算具有95%置信区间和对数秩p值的危险比,显著性设置为p《0.05。基于mirna与靶基因表达趋势相反的特点,进一步筛选通过调控adrm1和tp53的表达影响乳腺癌进展的mirna。
[0049]
kaplan-meier生存曲线显示adrm1 mirna的高表达与较差的os相关(hr=1.41,95%ci:1.26-1.57,p《0.001)。her2、er和淋巴结状态的分组分析表明,adrm1的高表达显著降低er阳性(hr=1.52,95%ci:1.09-2.11,p=0.012)、her2阴性(hr=1.37,95%ci:1.1-1.71,p=0.0047)淋巴结阴性(hr=1.44,95%ci:1.02-2.03,p=0.038)患者的总生存率(图4)。
[0050]
本发明还进行了mirna的生存分析。分析显示,hsa-let-7b和hsa-mir-125b的表达与总生存率呈正相关。也就是说,hsa-let-7b和hsa-mir-125b的表达越高,总生存率越好(hsa-let-7b:hr=0.66,95%ci:0.54-0.8,p=4.1e-05;hsa-mir-125b:hr=0.72,95%ci:0.58-0.88,p=0.0015)。随后的分组分析也显示hsa-let-7b的低表达显著降低er阳性(hr=0.72,95%ci:0.57-0.92,p=0.0089)、her2阳性(hr=0.65,95%ci:0.49-0.86,p=0.0026)、淋巴结阳性(hr=0.63 95%ci:0.47-0.83,p=0.0012)患者的总生存率(图5)。图6所示,hsa-mir-125b的低表达显著降低er阳性(hr=0.62,95%ci:0.47-0.820,p=0.00071)、er阴性(hr=0.63,95%ci:0.48-0.83,p=0.00078)、her2阳性(hr=0.71,95%ci:0.53-0.94,p=0.018)、淋巴结阳性(hr=0.78,95%ci:0.6-1.01,p=0.061)、淋巴结阴性(hr=0.55,95%ci:0.39-0.79,p=0.00078)患者的总生存率。
[0051]
(3)在(2)中获得的mirna上寻找与adrm1和tp53匹配的碱基序列,验证(2)中筛选的mirna是否是adrm1和tp53的共同调控基因
[0052]
在ncbi(https://www.ncbi.nlm.nih.gov/)主页上查找最后筛选到的mirna序列,采用序列比对的方法寻找与adrm1和tp53匹配的区域。通过比对序列,发现hsa-let-7b-5p和hsa-mir-125b-5p两个mirna与tp53和adrm1具有匹配的结合位点(图7)。
[0053]
(4)分析mirna在乳腺癌和正常组织中的表达水平,进一步验证筛选出的mirna是否是adrm1和tp53的共同调控基因
[0054]
利用在线数据库encori(http://starbase.sysu.edu.cn/panmirdiffexp.php)分
析筛选到的mirna在乳腺癌和正常组织中的表达水平。筛选条件如下:mirna=hsa-let-7b-5p,cancer=breast invasive carcinoma;mirna=hsa-mir-125b-5p,cancer=breast invasive carcinoma。满足条件的乳腺癌样本1085例,正常组织样本104例。结果表明,与正常组织相比,乳腺癌组织中的hsa-let-7b-5p和hsa-mir-125b-5p显著减少(图8)。这些数据表明,hsa-let-7b-5p和hsa-mir-125b-5p是与乳腺癌进展相关的adrm1和tp53的可能调控基因。
[0055]
(5)对筛选的mirna的靶基因进行富集分析
[0056]
利用david(https://david.ncifcrf.gov/)综合分析工具进行go(go:http://geneontology.org/)功能富集分析及kegg(kegg:http://http://www.kegg.jp/kegg/)信号通路分析,挖掘mirna的靶基因主要发挥的生物学功能,p《0.05为有统计学意义。go分析中,最重要的富集通路有应激激活mapk级联的调节,应激激活mapk级联的正调节,tp53类介体的信号转导,map激酶活性的正调节,map激酶活性的调节,mirna代谢过程,rna沉默基因中小rna的产生,以及产生参与mirna基因沉默的mirna。在kegg分析中,靶基因在癌症途径、癌症中的microrna、wnt信号途径、粘附蛋白结合蛋白、mapk信号途径和tgf-β信号途径中显著富集(图9)。这些结果表明这些靶基因可能参与了癌症的发生发展。
[0057]
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1