一种病理学图像的口腔癌上皮组织区域自动分割方法与流程
1.本公开属于医学图像处理、机器学习技术领域,特别涉及一种病理学图像的口腔癌上皮组织区域自动分割方法。
背景技术:
2.口腔鳞状细胞癌(0c
‑
scc)是世界范围内最常见的头颈部恶性肿瘤,它具有对邻近组织的高侵袭能力和对远处器官的转移潜力。因此需要更好的诊断和风险分层工具,用于oc
‑
scc患者的个性化治疗。
3.病理学家诊断和危险因素分层的方法始于确定肿瘤区域。这些区域通常由病理学家通过光学显微镜使用常规的染色组织进行分析。整个肿瘤区域由癌细胞及其支持组织基质组成。就癌而言,上皮肿瘤区域是评估最多的部分。这些隔室的组织形态学分析已经进行了数十年,通常可以可靠地鉴定出肿瘤的类型和程度。此外,已发现oc
‑
scc的潜在形态特征与患者预后相关。但是,由于缺乏有效的标记工具,并且标记容易遭受观察者内部和观察者之间的可变性的影响,因为它耗时且依赖于专家,病理学家无法有效地再现性地识别或量化这些组织学标志。
4.在过去的几年中,使用机器学习(ml),尤其是深度学习(dl)进行的各种定量数字病理图像分析已显示出能够从数字化苏木精
‑
伊红(h&e)染色的组织切片中“解锁”肿瘤原始的亚视觉属性。在这项工作中,已经能够通过相同的dl网络体系结构alexnet从h&e染色的图像中分类,检测或分割不同的组织学原语,包括上皮,肾小管,淋巴细胞,有丝分裂和癌。但是,当将ml/dl应用于更特定的区域时,在wsi的上皮分割中,可能会遇到诸如ground truth标注的耗时枯燥,染色的多变性以及基于像素块(patch)处理图像的信息难以聚集的挑战。
5.使用计算机辅助模式识别工具进行的先进的数字病理分析已显示出可以“解锁”亚视觉属性,并提供了肿瘤的定量特征。上皮和其他组织之间的自动区分是开发用于检测和客观表征oc
‑
scc的自动化方法的重要前提。
技术实现要素:
6.为了解决上述问题,本公开提供了一种病理学图像的口腔癌上皮组织区域自动分割方法,其包括如下步骤:
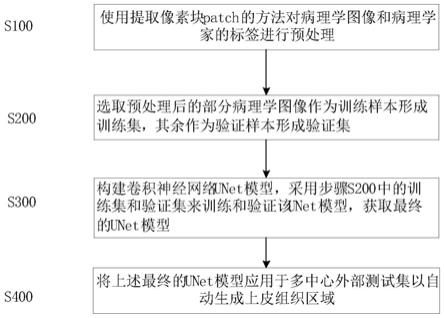
7.s100:使用提取像素块patch的方法对病理学图像和病理学家的标签进行预处理;
8.s200:选取预处理后的部分病理学图像作为训练样本形成训练集,其余作为验证样本形成验证集;
9.s300:构建卷积神经网络unet模型,采用步骤s200中的训练集和验证集来训练和验证该unet模型,获取最终的unet模型;
10.s400:将上述最终的unet模型应用于多中心外部测试集以自动生成上皮组织区域。
11.通过上述技术方案,提出了一个基于unet的深度学习框架,用于从h&e染色的组织图像中自动检测上皮组织区域。首先使用提取patch的方法对图像和标签进行预处理,对dl模型进行了训练和验证,并在不同放大倍数下利用了两类组织微阵列(tma)图像以及详尽的人类专家标注。然后在来自不同机构的数据集中独立测试了这个锁定的模型。这些独立的数据集由全幅玻璃图像(wsi)组成,并与训练和验证阶段无关。
12.该方法并没有对每个数据集中分别进行训练、验证和测试。因为,如果对每个数据集都进行训练验证和测试,那么就相当于在已知的这些数据集的信息的基础上做的测试,这样的测试的结果,就对“未知的”数据的说服力差。该方法仅使用了2个中心(机构)的tma图像,对外部3个中心(机构)的wsi图像进行测试,该方法在训练和测试使用了不同的图像类型(tma和wsi),并在外部测试集上取得可观的效果,对未知图像更具有说服力。
附图说明
13.图1是本公开一个实施例中所提供的一种病理学图像的口腔癌上皮组织区域自动分割方法的流程图;
14.图2是本公开一个实施例中代表性的tma和其上皮组织标注图;
15.图3是本公开一个实施例中代表性的wsi和其对应多边形框的标注图;
16.图4是本公开一个实施例中用于口腔癌的上皮组织分割的unet模型结构图;
17.图5是本公开一个实施例中六个代表性的验证tma图像结果对照图;
18.图6是本公开一个实施例中四个代表性的测试wsi图像结果对照图。
具体实施方式
19.在一个实施例中,如图1所示,公开了提供了一种病理学图像的口腔癌上皮组织区域自动分割方法,其包括如下步骤:
20.s100:使用提取像素块patch的方法对病理学图像和病理学家的标签进行预处理;
21.s200:选取预处理后的部分病理学图像作为训练样本形成训练集,其余作为验证样本形成验证集;
22.s300:构建卷积神经网络unet模型,采用步骤s200中的训练集和验证集来训练和验证该unet模型,获取最终的unet模型;
23.s400:将上述最终的unet模型应用于多中心外部测试集以自动生成上皮组织区域。
24.就该实施例而言,使用基于unet的深度学习框架从两种类型的组织病理学图像中分割上皮区域:组织微阵列(tma)和全幅玻璃图像(wsi)。在训练阶段,总共使用了来自两个不同机构的190名患者的212个带标记的tma进行模型训练。在测试阶段,使用了来自三个不同机构的477名oc
‑
scc患者的478个wsi进行测试。最后,将结果与病理学家标注的wsi进行比较。
25.tma类型图像的标签直接是二值图像,wsi类型图像的标签是病理学专家通过标注软件(如qupath)对wsi图像进行标注,首先得出的是包含标注区域多边形的坐标的集合的xml文件,然后将xml文件转化为二值图像。
26.训练过程中的预处理,就是对tma图像的处理,主要是大图裁成小图的过程。我们
拿到的tma图像是40x的。(40x,40乘,是在显微镜下40x放大倍率下得到的图片。)步骤如下:1.将40x的原图,下采样至10x,如原本是8000x8000像素的,下采样至2000x2000像素,(4倍关系)。2.然后在10x的图像取patch(256x256),不重合地取(non
‑
overlapping);标签(二值图像),也是这么处理。3.这样每一个tma图像都得到了一些patch,在划分训练和验证集的时候,是按照tma级别划分的,也就是属于同一个tma的patch们,不会同时出现在训练集和验证集中。
27.在另一个实施例中,病人选择如下:经华盛顿大学(wu)在圣路易斯人类研究保护办公室,俄亥俄州立大学(osu),旧金山退伍军人管理局医学中心(sfva),加利福尼亚大学旧金山分校(ucsf),范德比尔特大学医学中心(vumc)。口腔鳞状细胞癌的患者是从人类研究保护办公室批准的放射肿瘤学和耳鼻咽喉头颈外科临床医生数据库中鉴定出来的。放射肿瘤学数据库是被许可的由一名放射肿瘤学家治疗的患者的集合。
28.本方法包括由五个独立且特征明确的福尔马林固定石蜡包埋(ffpe)和h&e染色的全幅玻璃图像(wsi)和组织微阵列(tma)所组成的数据集,代表总共n=667例患者。所有wsi和tma切片均使用aperio scanscope xt数字扫描仪以40倍放大率进行数字扫描,每个像素的分辨率为0.25μm。为tma上的每个肿瘤分配了一个编码编号,以便共享,并带有仅研究病理学家已知的实际患者数据的链接。对于tma的产生,研究病理学家选择了一种2mm中央肿瘤穿孔使用(即载玻片上代表肿瘤最多,代表最好的那些穿孔)。
29.这五个数据集分别由d1(来自osu的75例患者的97个tma),d2(来自wu的115例患者的115个tma),d3(来自sfva的94例患者的95个wsi),d4(来自vumc的182例患者的182wsi)和d5(来自ucsf的201名患者的201个wsi)。来自d1,d2和d4的患者的相应临床病理和结果信息是从irb批准的回顾性图表审查(收集数据集的各机构)获得的。数据集d1和d2用于自动上皮组织分割模型的训练,而数据集d3,d4和d5用于独立测试训练后的模型。表1总结了所有五个数据集的患者的临床和病理数据。
[0030][0031]
表1
[0032]
在另一个实施例中,标注如下:数据集d1和d2由212个tma组成,所有上皮区域均由
一名研究病理学家使用内部开发的标注工具进行标注。图2显示了代表性的tma(a,b)d1和(c,d)d2和带标注的上皮组织区域的二进制图像。由于数据集d1和d2用于模型训练,因此所有上皮区域都进行了详尽标注,我们尝试使标注尽可能精确。其中,图2中每个图像的宽度或高度为5k
‑
7k像素;(a,b)是来自d1的tma;(c,d)是来自d2的tma;第三列和第四列图像是第一列和第二列中这些图像的详细缩放部分。
[0033]
为了提供训练模型的准确评估,对d3,d4和d5中的n=478个wsi的所有上皮区域进行了标注了。图3显示了(a,b,c)d3和(d,e,f)d4和(g,h,i)d5的代表性wsi和带标注的上皮组织区域(多边形线段框)。由于注释的实质性工作量,某些wsi标注可能并不详尽,但是,我们尝试使标注尽可能精确。
[0034]
在另一个实施例中,所述训练集是由组织微阵列tma以及病理学家制作的标签构成。
[0035]
就该实施例而言,一组212个tma以及由病理学家制作的标注被用来训练和验证模型以识别上皮组织区域。tma的平均大小为6000x6000像素。我们将训练数据集d1和d2分为9∶1的训练和验证集。来自同一tma的patch将不会同时放在训练和验证集中。对模型进行了50个epoch的训练,并在每个epoch的末尾采用了验证集来跟踪模型收敛的程度。通过最小化验证集上的误差来选择最终模型,然后将其锁定。
[0036]
在另一个实施例中,所述测试集是由全幅玻璃图像wsi构成。
[0037]
就该实施例而言,将该模型应用于由wsi数据集d3,d4和d5组成的独立测试集,以自动生成上皮区域。
[0038]
在另一个实施例中,评估模型在独立测试集中的性能。
[0039]
就该实施例而言,在评估中,我们使用了以下指标:
[0040]
像素精度:只需找到正确分类的像素数量除以像素总数即可。对于2个类别(0:负类,1:正类)。
[0041][0042]
其中p
ij
是预测为类i并实属于类j的数量。
[0043]
召回率(recall):
[0044][0045]
阳性预测值(positive predictive value):
[0046][0047]
dice系数(dice coefficient):
[0048][0049]
其中tp表示真阳性数量,fp表示假阳性数量,fn表示假阴性数量。
[0050]
在另一个实施例中,所述卷积神经网络unet模型由28层组成,每个卷积层都有一个填充操作。
[0051]
就该实施例而言,用于口腔癌的上皮组织分割的unet模型结构如图4所示。该网络由28层组成,其中包含14788929个参数。每个卷积层都有一个填充操作(padding),它将使输出的高度和宽度大小与输入的相同。
[0052]
所有图像均以40倍放大倍率(0.25μm/像素)进行扫描。我们分别使用5倍,10倍和20倍放大率的图像训练和验证了模型。在准备训练样本时,基本的水平和垂直翻转操作用于数据增强。为了使我们的模型专注于学习组织形态并减少由颜色变化引起的影响,我们还通过调整图像的亮度(1,1.4),对比度(1.1.4),饱和度(1,1.4),色相(
‑
0.5、0.5)和高斯模糊{1、2}。
[0053]
在另一个实施例中,所述卷积神经网络unet模型由卷积块,反卷积块,池化层和输出层组成。
[0054]
在另一个实施例中,所述卷积块共有5组,每组卷积块均有2层卷积层,每层卷积层之后都跟有一层批归一化层、一层relu激活层,除第一组卷积块外,其他卷积块都有一层池化层在其头部;所述反卷积块共有4组,每一组反卷积块均有一层反卷积层和2层卷积层,并且每层卷积层之后都跟有一层批归一化层、一层relu激活层;所述输出层仅包含一层卷积层。
[0055]
在另一个实施例中,所述卷积块及反卷积块中的卷积层的卷积核大小为3,步长为1,填充为1;所述卷积块中池化层的卷积核大小为2,步长为2,填充为0;所述反卷积块中的反卷积层的卷积核大小为2,步长为2,填充为0;所述输出层的卷积层的卷积核大小为1,步长为1,填充为0。
[0056]
在另一个实施例中,所述卷积神经网络unet模型的输出布置了一个通道,该通道的输出将输入进bcewithlogitsloss损失函数,得到上皮组织概率图,该概率图以0.5为分界转化为二值图,用于与专家标记进行比较。
[0057]
就该实施例而言,bcewithlogitsloss损失函数先将网络的输出传入sigmoid函数获得归一化的概率图,即sigmoid函数会将网络的输出约束在0到1的范围内以表示概率,再通过交叉熵函数与专家标记的二值图进行对比。
[0058]
在另一个实施例中,所述卷积神经网络unet模型训练使用了0.01学习率和1e
‑
8的权重衰减的rmsprop优化算法。
[0059]
在另一个实施例中,在训练期间,将组织微阵列tma和其二值标签图对应地下采样和训练。
[0060]
就该实施例而言,由于我们试图比较具有不同放大倍率(即5倍,10倍和20倍)训练集的模型的性能,因此将所有tma和其二值标签图对应地下采样和训练。
[0061]
在另一个实施例中,在训练期间,将组织微阵列tma和相关的标注裁剪为不重叠的256x256像素块。
[0062]
就该实施例而言,裁剪tma,然后将其拆分为256x256像素的非重叠图像patch,从而产生8802个训练patch(191个tma)和954个验证patch(21个tma)。
[0063]
在另一个实施例中,所有像素块patch从带标注的上皮组织和非上皮组织区域中提取。
[0064]
就该实施例而言,所有patch从带标注的上皮组织和非上皮组织区域中提取,大小为256x256像素。
[0065]
在另一个实施例中,表2表示不同放大倍率时验证集中的模型性能。表3表示不同放大倍率时测试集中的模型性能。其中,在下表结果对比中hea被表示为是一种智能的h&e图像增色,在爪stomicstk中实现,可以添加到我们的色彩增强中。se则表示为通过在向下采样中向每个卷积层添加se块,是注意力加强的基本的unet网络结构模型。在验证集上,上皮组织分割10x放大倍率下unet模型与病理学家对地面事实的标注相比,像素精度(pa)为88.05%,召回率(r)为82.74%,阳性预测值(ppa)为86.41%,dice系数(d)为84.53%。根据我们的其他指标,我们测试集中注释的冲突或不精确部分可能导致召回率低。
[0066]
放大倍率模型losspa%r%ppv%d%5unet0.3482.8488.1270.2378.1710unet0.2788.0582.7486.4184.5310unet+hea0.2887.3186.2882.4184.3010unet+se0.3086.2891.7977.5784.0820unet0.2986.7181.4086.2483.75
[0067]
表2
[0068]
数据集放大倍率模型pa%r%ppv%d%d35unet82.5035.7059.0239.42d310unet86.0440.4377.9752.82d310unet+hea85.5750.5267.7257.23d310unet+se81.4053.4752.5554.57d320unet85.1931.9379.7746.35d45unet80.0964.5046.0348.71d410unet88.7751.9678.7258.82d410unet+hea88.4346.7181.3955.57d410unet+se85.3859.7359.5654.67d420unet88.6049.3178.7456.17d55unet86.6657.5585.1763.55d510unet90.6773.0888.8477.29d510unet+hea89.0265.1289.5473.60d510unet+se90.2878.4083.0275.93d520unet90.4771.6388.4975.20
[0069]
表3
[0070]
图5中显示了六个代表性的验证tma图像,其中第一列显示原始tma图像,第二列显示专家制作的标注的二值图,最后一列显示的是我们的模型得出的结果。专家标注和预测结果在总体形状上非常接近,但是预测结果在小块和边缘处更容易区分。图6显示了d3
‑
d5上wsi的四个代表性测试结果,其中第一列显示了原始wsi及其病理学家(顶部)和机器(底部)所做标注的二值图。第二列显示由上一列中的矩形框注释的原始wsi的放大细节。第三和第四列显示专家和机器制作的放大部分的二值图。最后一列显示了由我们的框架制作的放大部分的热度图。很容易在边界及内部细节上注意到机器的结果比专家的结果更精确。
[0071]
尽管以上结合附图对本发明的实施方案进行了描述,但本发明并不局限于上述的
具体实施方案和应用领域,上述的具体实施方案仅仅是示意性的、指导性的,而不是限制性的。本领域的普通技术人员在本说明书的启示下和在不脱离本发明权利要求所保护的范围的情况下,还可以做出很多种的形式,这些均属于本发明保护之列。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1