基于Squared-chord距离的生物信息标签分布学习方法
本发明属于数据挖掘方法,涉及一种基于squared-chord距离的生物信息标签分布学习方法。
背景技术:
1、单标签学习(sll)与多标签学习(mll)是解决分类问题的两个成熟范例,两者都可以看作建立从样本空间到标签空间的映射关系。单标签学习假设每个样本只有一个确定的标签或类别,这种情况在许多分类任务中都很常见,例如图像分类、文本分类等。然而,在实际处理问题的过程中,标签与标签之间往往并不冲突,甚至许多标签是存在关联性。如果在这种分类情况下只考虑单标签学习,那么就会丢失许多重要的样本标签,导致分类的结果失去精确性和完整性。为了更好地解决单标签学习无法解决的这些涉及多个标签的问题,有关研究人员提出了多标签学习这一概念和相关算法,目的是有效解决多标签分类问题,提高分类的精确性和完整性。多标签学习假设样本可能与一组标签或类别相关,最初采用基于规则的方法或启发式算法。大量研究表明,多标签学习是一种有效且广泛使用的学习范式,在文本分类、图像标注等方面得到了应用,能够解决更多的分类问题。
2、然而多标签学习也存在局限性,通过多标签学习能够获得一组与样本相关的标签,但是各个标签在多大程度上描述了样本,也就是标签的重要性不得而知。为了解决多标签学习的局限性,标签分布学习(ldl)这一概念被提出,将标签输出改用分布的形式替代。现有的标签分布学习算法主要分为三类:问题转化、算法自适应和专门算法。基于问题转化策略的方法主要将标签分布学习问题转化为单标签学习问题,然后用现有的学习方法去解决。基于算法自适应策略的方法主要将一些现有的学习方法自适应到标签分布学习问题中。最后,基于专门算法策略的标签分布学习方法是专门设计用以解决标签分布学习问题的,这类算法通常由三部分组成,分别是目标函数,输出模型和优化方法。目前标签分布学习能够应用于人脸年龄估计、面部情感识别和生物信息学等领域。其中针对生物信息领域,标签分布学习能够帮助预测标签生物信息表达水平。例如,研究发现,一种蛋白质可能与多种癌症相关,并且该蛋白质在不同相关癌症细胞中的表达水平不同。因此,利用标签分布学习,可以帮助获得不同癌症对蛋白质的描述程度。
3、现有的能够预测生物信息标签分布的算法中,基于专门算法策略的一些方法往往能取得更好的预测性能。然而,这些方法通常利用kl散度构造目标函数,方法之间性能差异不大,急需开发更好的生物信息标签分布预测方法,提高生物信息相关标签分布预测性能。
技术实现思路
1、本发明的目的是提供一种基于squared-chord距离的生物信息标签分布学习算法,来解决现有技术存在的不足,提高生物信息相关标签分布预测性能。
2、本发明的具体步骤如下:
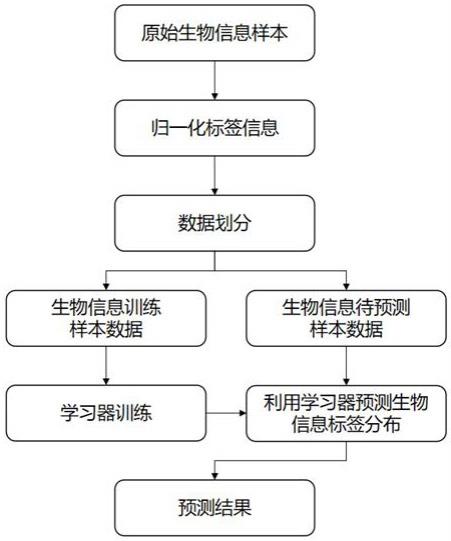
3、步骤1:获取生物信息样本数据,包含生物信息特征和相对应的通过自然衡量标准获取的一组生物信息表达水平;
4、对生物信息表达水平归一化后得到与生物信息相关的不同描述程度,作为标签分布。
5、步骤2:对获取的生物信息数据进行划分,将原始生物信息样本数据分为训练样本数据和待预测样本数据;
6、步骤3:初始化参数矩阵和迭代更新矩阵,利用squared-chord距离和最大熵模型构造目标函数;所述的迭代更新矩阵用于优化所述参数矩阵;
7、步骤4:根据步骤3中初始化的参数矩阵和迭代更新矩阵,以及目标函数的一阶梯度,利用bfgs优化方法优化参数矩阵,直到目标函数的一阶梯度小于预先设定的收敛条件值,得到最佳参数矩阵。
8、步骤5:通过优化后获得生物信息标签分布学习模型,将步骤2中划分得到的待预测生物样本数据输入至生物信息标签分布学习模型中,由模型预测生物信息样本相关的标签分布。
9、本发明的有益效果是:本发明利用squared-chord距离构造了能够预测生物信息标签分布的算法,通过最小化预测分布与真实分布的距离,使得预测分布与真实分布尽可能相近。同时利用l1正则化进行参数矩阵稀疏化,利用frobenius范数作为防止过拟合的正则化项,提高预测准确性。
技术特征:
1.基于squared-chord距离的生物信息标签分布学习方法,其特征在于该方法包括以下步骤:
2.根据权利要求1所述的基于squared-chord距离的生物信息标签分布学习方法,其特征在于:步骤3中初始化参数矩阵θ为n×m的对角矩阵,迭代更新矩阵为单位矩阵。
3.根据权利要求2所述的基于squared-chord距离的生物信息标签分布学习方法,其特征在于:所述的目标函数t(θ)表达如下:
技术总结
本发明公开了一种基于Squared‑chord距离的生物信息标签分布学习方法。本发首先获取生物信息样本数据;其次对获取的生物信息数据划分为训练样本数据和待预测样本数据;然后初始化参数矩阵和迭代更新矩阵,利用Squared‑chord距离和最大熵模型构造目标函数;利用BFGS优化方法优化参数矩阵,直到目标函数的一阶梯度小于预先设定的收敛条件值,得到最佳参数矩阵。最后将待预测生物样本数据输入至优化后的生物信息标签分布学习模型中,由模型预测生物信息样本相关的标签分布。本发明利用Squared‑chord距离最小化预测标签分布与真实标签分布距离的同时,考虑模型的稀疏化,能够有效预测生物信息标签分布。
技术研发人员:杨柏林,纪国强,傅晨浩,马希骜
受保护的技术使用者:浙江工商大学
技术研发日:
技术公布日:2024/1/15
- 还没有人留言评论。精彩留言会获得点赞!