MIF抑制剂及其使用方法与流程
本申请根据35u.s.c.§119(e)要求属于2016年9月2日提交的美国临时申请第62/383,209号的权益,其在此被通过引用以其整体并入本文。
拨款信息:本发明是在国立卫生研究院拨款k99/r00ns078049,da000266,r01ns067525,r37ns067525和ns38377的政府支持下完成的。政府在本发明中拥有某些权利。
发明背景
发明领域
本发明整体上涉及巨噬细胞移动抑制因子(mif),并且更具体地涉及mif抑制剂在治疗疾病中的用途。
背景
聚(adp-核糖)(par)聚合酶-1(parp-1)是被dna损伤激活的重要的细胞核酶,其在dna损伤处促进dna修复(1)。parp-1的过度活化会导致被称为依赖性细胞死亡(parthanatos)的不依赖胱天蛋白酶(caspase)的固有细胞死亡程序(2,3),该程序在许多器官系统受到大量毒性损伤后发挥突出的作用(4,5),所述毒性损伤包括中风和心肌梗死后的缺血再灌注损伤、炎性损伤、活性氧物质诱导的损伤、谷氨酸兴奋毒性和神经退行性疾病例如帕金森病和阿尔茨海默病(2,4,6)。与parp-1是关键的细胞死亡介导因子的观点一致,parp抑制剂或parp-1的基因缺失针对这些和其他细胞损伤范例和人类疾病模型有着意义深远的保护作用(2,4,5,7)。
奠定依赖性细胞死亡的分子机制涉及par-依赖性凋亡诱导因子(aif)从线粒体释放并转位到细胞核,导致dna片段化为20-50kb的片段(2,8-11)。aif本身没有明显的核酸酶活性(2)。虽然已经有研究表明ced-3蛋白酶抑制剂(cps)-6,一种秀丽隐杆线虫(caenorhabditiselegans(c.elegans))中的核酸内切酶g(endog)同源物,与蠕虫aif同源物(wah-1)协作促进dna降解(12),但是endog似乎在哺乳动物中的短暂局灶性脑缺血后的parp-依赖性染色质溶解和细胞死亡中不发挥重要作用(13)。在依赖性细胞死亡过程中负责染色质溶解的核酸酶尚不被知晓。
发明概述
本发明基于将巨噬细胞移动抑制因子(mif)鉴定为parp-1依赖性的aif-相关核酸酶(paan)。
在一个实施方案中,本发明提供了治疗受试者中的与增强的聚[adp-核糖]聚合酶1(parp-1)活化相关的疾病的方法。该方法包括向受试者施用治疗有效量的巨噬细胞移动抑制因子(mif)的核酸酶活性抑制剂,从而治疗或减轻该疾病的症状。
在一个方面,疾病是炎症性疾病。另一方面,炎症性疾病是阿尔茨海默病、强直性脊柱炎、关节炎、骨关节炎、类风湿性关节炎、银屑病关节炎、哮喘动脉粥样硬化、克罗恩病、结肠炎、皮炎憩室炎、纤维肌痛、肝炎、肠易激综合征、系统性红斑狼疮、肾炎、溃疡性结肠炎或帕金森病。
在一个实施方案中,该抑制剂是例如来自混合(hybrid)大环rapafucin文库的大环rapafucin化合物。
本发明还提供了筛选巨噬细胞移动抑制因子(mif)抑制剂的方法,包括例如以下的步骤:固定单链的胺修饰的mif靶dna,随后在有和没有来自大环rapafucin文库的化合物的情况下孵育mif;将单链的胺修饰的mif靶dna与生物素化的dna杂交,该生物素化的dna与单链的胺修饰的mif靶dna互补,接下来与链霉亲和素酶缀合物一起孵育,接下来与底物孵育,其中该链霉亲和素酶缀合物作用于该底物。将在有文库化合物情况下的mif的吸光度和没有文库化合物情况下的mif的吸光度进行比较,以便基于吸光度的变化来确定化合物是否为抑制剂。
附图简述
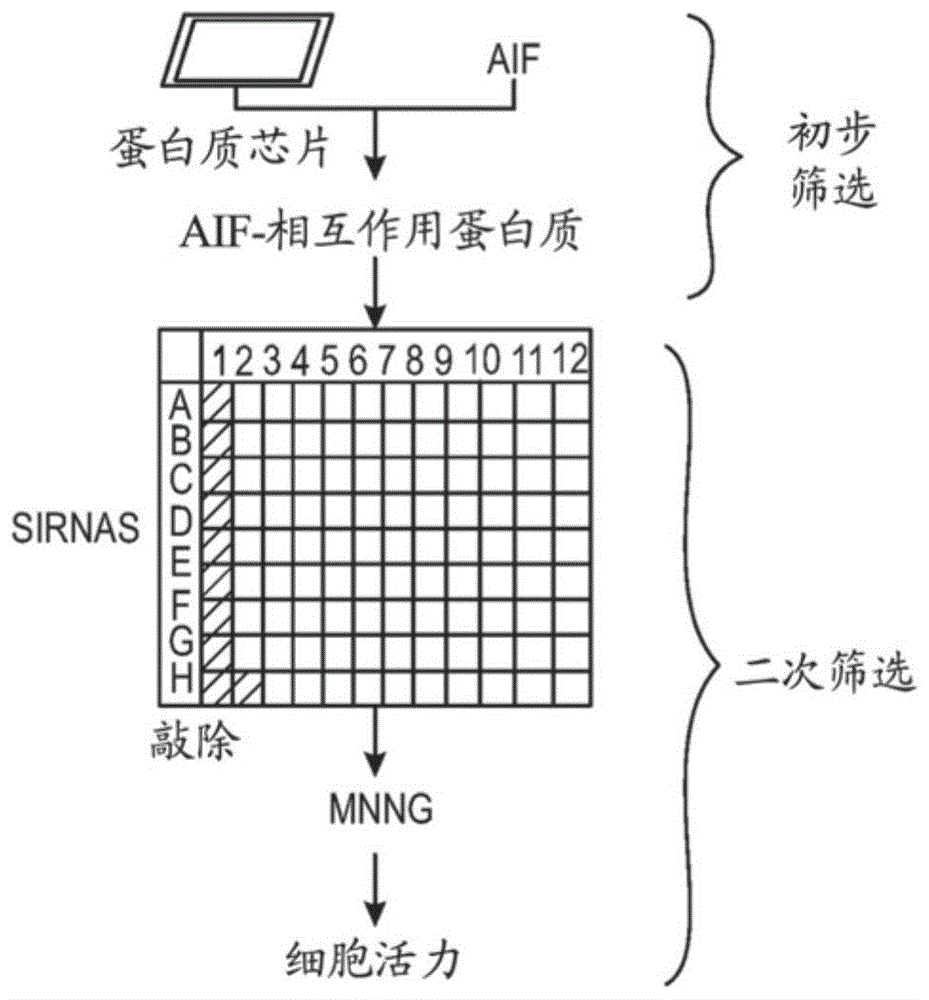
图1.mif作为介导parp-1依赖性细胞死亡的关键性细胞死亡效应因子的鉴定。(a)鉴定参与parp-1依赖性细胞死亡的aif-相关蛋白的策略。(b)在mnng处理(50μm,15分钟)后24小时,hela细胞中的基于sirna的parp-1依赖性细胞活力高通量筛选。n=8。实验在4次独立测试中重复。(c)mif的pd-d/e(x)k结构域的示意图。(d)人mif和其他核酸酶的核酸酶结构域的比对。序列上方的箭头指示β-链,且矩形表示α-螺旋。突变的氨基酸残基用箭头和数字指示(参见结果)。核酸酶和cxxcxxhx(n)c结构域分别以绿色和粉红色突出显示。(e)mif三聚体(pdb:1gd0)(左)和三聚体形式的mifpd-d/e(x)k基序(右)的晶体结构。
图2.mif是切割基因组dna的新型核酸酶。(a)使用pcdna为底物的体外mif核酸酶测定。(b)在含有mg2+(10mm)、含有或不含有edta(50mm)或含有ca2+(2mm)、含有或不含有edta(25mm)的缓冲液中的体外脉冲场凝胶电泳-mif核酸酶测定,使用人类基因组dna作为底物。(c)对于用或未用dpq(30μm)或iso-1(100μm)处理的mif缺陷的hela细胞和野生型hela细胞中的mnng诱导的dna损伤的脉冲场凝胶电泳测定。(d)使用人类基因组dna作为底物,对mifwt和mif突变体的核酸酶测定。
图3.mif结合并切割单链dna。(a)由chip-seq确定的mifdna结合基序。(b)mif具有结构特异性地与ssdna结合,但不与双链dna结合。5’生物素标记的具有不同结构或不同序列的小dna底物被用在emsa测定中(对底物的说明参见图19,而序列参见表1)。(c)mif具有结构特异性地切割在茎环ssdna的3’端的未配对碱基。5’或3’生物素标记的具有不同结构或不同序列的小dna底物被用于核酸酶测定(对底物的说明参见图19,而序列参见表1)。使用纯化自3次独立制备的mif蛋白重复实验4次。(d)mif从未标记的ps30和3f1底物上切割掉3’未配对的碱基。梯形物1和2是使用ps30及其切割产物,通过一个接一个地去除其3’核苷酸而定制的。梯形物1使用ps30、ps28、ps26、ps24、ps22和ps20制备。梯形物2使用ps29、ps27、ps25、ps23和ps21制备。(e)未标记的ps30和3f1底物上的mif切割位点。
图4.在nmda处理后,需要aif来招募mif进入细胞核。(a)结合aif的固定的gst-mifwt和gst-mif变体的gst拉下(gst-pulldown)测定。(b)mifwt和mif变体的核酸酶活性和aif结合活性。(c-d)在生理条件和nmda处理的条件下,mif和aif在皮层神经元中的免疫共沉淀。星形指示igg。(e-g)在nmda处理后,野生型、aif敲除型和mif敲除型皮层神经元的aif和mif核转位。核后(postnuclear)部分(pn)和核部分(n)中aif和mif信号的强度在g中示出。(h)mif在wt和ko神经元中的表达。(i)在nmda处理后,皮层神经元中flag标记的mif变体和aif的免疫共沉淀。(j-l),在nmda处理后,aif和外源性mifwt和mif变体在mifko皮层神经元中的核转位。比例尺,20μm。在核后部分(pn)和核部分(n)中aif和mif信号的强度在l中示出。示出了平均值±sem。实验重复至少3次。*p<0.05,**p<0.01,***p<0.001,学生t检验(student’sttest)(d)和单向anova(g,l)。
图5.在体外和体内,mif核酸酶活性对dna损伤和缺血性神经元细胞死亡至关重要。(a)在表达mifwt、e22q或e22a的mifwt、ko和ko皮层神经元中nmda(500μm,5min)诱导的细胞毒性。(b),通过彗星测定确定的表达mifwt、e22q或e22a的mifwt、ko和ko神经元在处理后6小时的nmda引起的dna损伤的代表性图像。虚线表示头部和尾部的中心。比例尺,20μm。(c)mifwt和ko神经元以及表达mifwt、e22q和e22a的ko神经元在处理后6h的nmda诱导的dna损伤的脉冲场凝胶电泳测定。(d)在45分钟mcao后24小时,注射aav2-mifwt、e22q或e22a的mifwt、ko和ko小鼠的ttc染色的代表性图像。(e)在45分钟mcao后1天或7天,皮层、纹状体和半球梗塞体积的定量。(f-g)在mcao手术后1天、3天或7天,通过0-5量表的开放场评估神经性缺陷。wtmcao(n=29),komcao(n=20),ko-wtmcao(n=23)。ko-e22qmcao(n=22),ko-e22amcao(n=19)。平均值±sem在a,e,f,g中示出。*p<0.05(e,f),***p<0.001(a,e),单向avova。***p<0.001(g),wt对ko,ko-wt对ko-e22q/ko-e22a在不同时间点的双向anova。
图6.使用大环化合物文库的mif抑制剂筛选的鉴定。基于切割测定筛选用于mif抑制剂的大环物质的示意图。将单链的胺修饰的寡核苷酸(mif靶dna)固定在dna-bind平板上,并在有或没有抑制剂的情况下在mif蛋白中孵育。在mif切割后,使片段与生物素标记的互补寡核苷酸杂交,并通过监测450nm处的吸光度来检测所述片段。
图7.大环rapafucin文库的示意图。
图8.mif抑制剂的筛选结果。来自大环文库的38个平板的mif切割的抑制百分比的散点图。蓝线是没有与mif一起孵育的阳性对照,且绿线是与mif一起孵育的阴性对照。右图代表所测试的化合物的直方图。
图9.用于mif抑制剂的筛选的各个化合物的结果。mif切割的抑制百分比(x轴)和mnng诱导的细胞死亡的抑制(y轴)的散点图。
图10.4个击中物(hit)的剂量依赖性确认。(a)评估了4个候选物在用mnng处理的hela细胞中的细胞保护作用。候选物提供剂量依赖性细胞保护。(b)对4个候选物在tbe凝胶中进行切割测定。候选物可以防止mif切割底物。
图11.在有或没有与2个命中物的情况下用pff处理初级皮层神经元,持续14天。图像显示经由2个命中物的pff诱导的细胞死亡(左)。比例尺,50μm。定量经由2个命中物的pff诱导的细胞死亡。条形图(bar)反映了来自三次实验的平均值±s.d.。**p<0.005,***p<0.001(双尾非配对t-检验)。
图12.parp-1依赖性细胞死亡不需要endog。(a)使用crispr-cas9系统敲除sh-sy5y细胞中的endog。ev,空载体。(b)敲除endog对mnng诱导的细胞死亡没有影响。(c)敲除endog对mnng引起的dna损伤没有影响。
图13.mif的敲除保护细胞免于mnng和nmda诱导的细胞死亡。(a)用人的mifshrna1-3ires-gfp慢病毒或非靶向(nt)shrnaires-gfp慢病毒转导的hela细胞的代表性图像。(b)在shrna转导后hela细胞中的mif蛋白水平。hmifshrna1、2和3在hela细胞中引起mif蛋白减少83.3±7.1%、71.6±3.2%和82.7±6.3%。(c)mnng(50μm,15分钟)诱导的hela细胞死亡的定量。示出了平均值±sem。***与dmso对照相比,p<0.001。###与mnng处理的wt相比,p<0.001。(d)用小鼠mifshrna1-3ires-gfp或非靶向(nt)shrnaires-gfp慢病毒转导的皮层神经元的代表性图像。(e)shrna转导后皮层神经元中的mif蛋白水平。(f)mif敲除神经元中nmda(500μm,5分钟)诱导的神经元细胞死亡的定量。mmifshrna1、2和3导致皮层神经元中mif蛋白减少84.5±8.2%、90.1±7.1%和92.2±3.3%。示出了平均值±sem。***与css对照相比,p<0.001。###与nmda处理的wt相比,p<0.001。(g)mif敲除的和mif过表达突变体的代表性免疫印迹,所述突变体能在皮层神经元中抵抗shrna1和shrna3。(h)对mif敲除的皮层神经元和抵抗shrna1和shrna3的过表达mif突变体的细胞中nmda诱导的神经元细胞死亡的定量。示出了平均值±sem已经示出。***与css对照相比,p<0.001。###与nmda处理的wt相比,p<0.001,单向anova。比例尺,100μm。mif信号的强度在c、f和h中示出。实验在三个独立的试验中重复。
图14.mif含有pd-d/e(x)k核酸酶基序。(a)来自人、小鼠、大鼠、猴子、猪、牛、绵羊、兔和鼩鼱(sorex)的mif的核酸酶结构域的比对。(b)来自人、小鼠、大鼠、猴子、猪、牛、绵羊、兔子和鼩鼱的mif的cxxcxxhx(n)c结构域的比对。(c)pd-d/e(x)k核酸酶中活性位点的保守性拓扑结构。图像由kosinski等人(18)修改。α螺旋显示为圆形,而β链显示为三角形。β链的方向指示平行或反向平行。(d)mif三聚体的晶体结构(pdb:1gd0)。每个单体用一种不同的颜色表示。(e)mif三聚体的拓扑结构说明各个结构域的方向与pd-d/e(x)k基序相似。(f)含有pd-d/e(x)k结构域的mif单体的晶体结构,该结构通过从三聚体中隐藏两种单体(d中的红色虚线)而从三聚体而获得。(g)mif单体在mif三聚体中的拓扑结构。(h)显示了每种单体都有pd-d/e(x)k结构域。pd-d/e(x)k基序由来自一个单体的两条平行β链(β4和β5)和来自相邻单体的两条反向平行的链(β6和β7)组成。(i)mif单体在mif三聚体中的拓扑结构和ecorv的相似性的示意图,说明了各种结构域在其核酸酶结构域中的相似取向。α螺旋显示为圆形,β链显示为三角形。(j)ecorv单体的拓扑结构。(k)mif三聚体中的mif单体和ecorv单体(红色)的比对。(l-o)mif中的pd-d/e(x)k基序和包括ecori(品红色,pdb:1qc9)、ecorv(浅蓝色,pdb:1sx8)、exoiii(红色,pdb:1ak0)和pvuii(橙色,pdb:1pvu)在内的其他熟知的核酸酶的pd-d/e(x)k基序的比对。所有五个基序在β-片层对α螺旋中显示了四条β链的相似取向,如在典型的pd-d/e(x)k基序活性位点中观察到的。
图15.mif是新型核酸酶。(a)mif与人类基因组dna(hgdna,200ng)在含有10mmmgcl2的ph7.0的tris-hcl缓冲液中于37℃孵育4小时的浓度依赖性。(b)mif(4μm)与hgdna在含有10mmmgcl2的ph7.0的tris-hcl缓冲液中于37℃孵育的时间进程。(c)mif(8μm)与hgdna在具有如所示的不同离子的ph7.0的tris-hcl缓冲液中于37℃用孵育4小时。(d)使用人类基因组dna为底物,用纯化的蛋白质(4μm)进行体外脉冲场凝胶电泳-核酸酶测定。(e)将不同的纯化的mif突变体(对mif的氨基酸序列的说明参见图1d)与hgdna在含有10mmmgcl2的ph7.0的tris-hcl缓冲液中于37℃孵育4小时。示出了纯化的mifwt蛋白和mif突变体的考马斯蓝染色(下方的图块)。(f)谷氨酸残基突变为谷氨酰胺、天冬氨酸和丙氨酸。(g)纯化的mifwt蛋白和mif突变体的考马斯蓝染色。(h)将不同的纯化的mif突变体(对突变的说明参见图1d)与hgdna在含有10mmmgcl2的ph7.0的tris-hcl缓冲液中于37℃孵育4小时。示出了纯化的mifwt蛋白和mif突变体的考马斯蓝染色(下方的图块)。使用在三次独立制备中纯化的mif蛋白重复实验。
图16.mif突变对蛋白质折叠和酶活性的影响。(a)mif蛋白的氧化还原酶活性。(b)mif蛋白的互变异构酶活性。平均值±sem在b和c中示出.**p<0.01,单向avona。(c)mif蛋白(野生型、e22q和e22a)(实线)和蛋白标准品(虚线)的fplc图谱。(d)来自fplc的mif级分的考马斯蓝染色。(e-m)在存在和不存在氯化镁(mg)和/或氯化锌(zn)的情况下,纯化的mif重组蛋白的uv-cd分析。使用从三次独立制备中纯化的mif将实验重复三次。
图17.通过chip-seq表征mif-dna结合。(a)对于dmso和mnng处理的细胞的chip-seq,染色质的超声波处理片段在100-200bp的范围内。(b)mifchip的代表性免疫印迹图像。(c)来自四个不同的文库的读段(read)的数量和覆盖范围,四个不同文库包括dna输入和从dmso或mnng(50μm)处理的细胞制备的mifchip样品。(d)mnng处理的细胞中跨不同基因组区域的mifchip-峰分布。饼图显示,mif倾向于与基因的启动子结合(约36%的chip区域位于启动子中)。(e-f)以两种不同染色体窗口尺寸显示的对于mif在基因组上的富集的代表性igv可视化图。前两行显示了来自dmso和mnng处理的细胞的chip-seq数据的tdf文件。第三和第四行显示了dmso和mnng处理的样品的bed文件(bedfile)。仅在mnng处理的样品中观察到峰,但在dmso处理的样品中没有观察到峰。最后一行表示hg19参考基因。(g)通过qpcr证实了dmso和mnng处理的细胞中的mif染色质富集具有非p(非峰区)、p55101、p66005、p65892、p36229、p46426和p62750(峰区)。
图18.mif与单链的dna结合。(a)mifdna结合基序的比对。(b)显示了沟(groove)/结合袋(bindingpocket)(箭头)的mif三聚体(pdb登录号1fim)表面的图像(上方图块)。在沟中含有dsdna的mif三聚体模型(中间图块)。中间图块中的右图像显示了mif-dsdna(pdb登录号1bna)与mif-ssdna(pdb登录号2rpd)的叠加(overlay)的模型的侧视图。i-iii,卡通图像显示p16和d17残基靠近dsdna和ssdna,而e22靠近ssdna,但不靠近dsdna。(c)emsa证明了,在mg2+或未标记的ps30存在或不存在的情况下,mif结合到其单链5’生物素标记的dna结合基序(ps30)。mif与其dna底物的结合被mif抗体破坏,而mif突变体e22a、e22q、p16a、d17a、d17q仍然与其dna底物结合。使用从三次独立制备中纯化的mif蛋白重复实验四次。
图19.用于结合和切割测定的不同生物素标记的dna底物的二级结构。
图20.mif具有结构特异性核酸酶活性地切割茎环ssdna。(a)使用dsps100作为底物的mif核酸酶测定。(b)使用ssps100及其互补链ssps100r作为底物的mif核酸酶测定。(c)使用dsps30、其序列相关底物-dsrf和非相关底物-dsl3,mif(1-4μm)对双链dna没有明显的核酸酶活性。(d)mif(0.5-4μm)未能以浓度依赖的方式切割dsps30、dsrf、dsl3。(e)使用ssps30作为底物的mif核酸酶活性需要mg2+。(f-h)mif(2μm)以浓度和时间依赖的方式切割ssps30。
图21.mif与aif相互作用并共转位到细胞核。(a)结合测定中使用的gst-aif截短蛋白的示意图。(b)通过使用抗mif抗体的蛋白质印迹显示的gst拉下测定(上方图块)。gst融合aif截短蛋白的考马斯蓝染色用于拉下实验(下方图块)。(c)通过使用抗mif抗体的蛋白质印迹显示的aif突变体的拉下测定。(d)谷胱甘肽珠上的gst-mif及其变体拉下了aif蛋白。这些实验在三个独立的试验中重复进行。(e-g)在存在或不存在parp抑制剂,dpq(30μm)的情况下,mnng处理后hela细胞中aif和mif的核转位,其通过(e)免疫染色和(f-g)亚细胞分离(subcellularfractionation)来确定。比例尺,20μm。实验在三个独立的试验中重复。***p<0.001,单向anova。(h-j)在有或无parp抑制剂dpq或nnos抑制剂硝基精氨酸(n-arg,100μm)的情况下,nmda处理后皮层神经元中aif和mif的核转位,其通过亚细胞分离来确定。i和j中显示了mif和aif信号的强度。实验在三个独立的试验中重复。这些实验在三个独立的试验中重复。***p<0.001,单向anova。
图22.mif核酸酶活性对于在皮层神经元中nmda诱导的dna损伤和parp-1依赖性细胞死亡至关重要。(a)在表达mifwt、e22q或e22a的mifwt、ko和慢病毒转导的mifko皮层神经元中nmda诱导的细胞毒性的代表性图像。比例尺,200μm。(b-d)处理后6小时nmda引起的dna损伤的定量,通过彗星测定确定。(b)尾部阳性神经元的%,(c)尾部长度,(d)尾部中的dna的%。
图23.mif对hela细胞中mnng诱导的dna损伤至关重要。(a)通过彗星测定确定的wthela细胞、ntshrna或mifshrna慢病毒转导的hela细胞中mnng诱导的dna损伤的代表性图像。虚线表示头部和尾部的中心。比例尺,20μm。(b-d)定量(b)尾部阳性细胞的%,(c)尾部长度和(d)尾部中的dna的%。平均值±sem在b-d中示出。***p<0.001,###p<0.001,单向anova。这些实验在三个独立的试验中重复。
图24.中风中的体内依赖型细胞死亡需要mif核酸酶活性。(a)脑室内(icv)注射台盼蓝染料。(b)注射后79天aav2-mifwt在(i)皮层、(ii)纹状体和(iii和iv)海马体中的表达的代表性免疫染色图像。比例尺,50μm。(c)跨越wt(n=16)、mifko(n=12)、mifko-wt(n=11)、mifko-e22q(n=11)和mifko-e22a(n=11)小鼠中缺血区核心的侧顶叶皮层测量的激光多普勒通量。(d-e)对在45分钟mcao后1天或7天,皮层、纹状体和半球中梗塞体积的定量。(f)在45分钟mcao手术后的第1、3和7天,通过角落测试中右转弯的%来评估神经性缺陷。wtmcao(n=16),komcao(n=12),ko-wtmcao(n=16)。ko-e22qmcao(n=16),ko-e22amcao(n=16)。示出了平均值±sem已经。*与中风前对照相比,p<0.05,单向anova。(g)注射aav2-mifwt、e22q或e22a的mifwt、ko和ko小鼠在mcao手术后1天、3天或7天的通过脉冲场凝胶电泳确定的mcao后半影区中的aif(红色)和mif(绿色)的核转位和(h)dna片段化。wtmcao(n=29),komcao(n=20),ko-wtmcao(n=23)。ko-e22qmcao(n=22),ko-e22amcao(n=19)。平均值±sem在d-f中示出。*p<0.05(e),***与对照或基线相比,p<0.001(d,f),单向anova。
发明详述
本发明基于将巨噬细胞移动抑制因子(mif)鉴定为parp-1依赖性aif-相关核酸酶(paan)。
如本文所用,化合物的“治疗有效量”旨在定量用于治疗疾病或紊乱的活性成分的量。该量将达到减少或消除所述疾病或紊乱的目的。正如本领域技术人员熟知的,施用的确切剂量和频率将取决于本发明使用的特定化合物、被治疗的特定状况、被治疗状况的严重程度、特定受试者的年龄、体重和一般身体状况以及患者可能服用的其他药物。此外,根据被治疗的受试者的反应和/或根据开具本发明的化合物的处方的医生的评估,所述“治疗有效量”可以降低或增加。
如本文所用,对受试者的“治疗(treating)”或“治疗(treatment)”的提及旨在包括预防。术语“受试者”意指包括人类在内的所有哺乳动物。受试者的实例包括人、牛、狗、猫、山羊、绵羊、猪和兔子。优选地,受试者为人类。
除了本发明化合物之外,本领域技术人员将认识到,包括化疗剂、抗炎剂和治疗性抗体在内的其它治疗性化合物可以在用本发明化合物治疗之前、同时或之后使用。尽管不希望受到限制,化疗剂包括抗代谢物,如甲氨蝶呤,dna交联剂,如顺铂/卡铂;烷基化剂,例如canbusil;拓扑异构酶i抑制剂诸如达替诺米星(dactinomicin);微管抑制剂如紫杉酚(taxol)(紫杉醇(paclitaxol))等。其它化疗剂包括,例如,长春花生物碱、丝裂霉素型抗生素、博莱霉素型抗生素、抗叶酸剂、秋水仙碱、demecoline、依托泊苷、紫杉烷、蒽环类抗生素、多柔比星(doxorubicin)、柔红霉素、洋红霉素、表柔比星、伊达比星、米索蒽酮(mithoxanthrone)、4-二甲氧基柔红霉素、11-脱氧柔红霉素、13-脱氧柔红霉素、阿霉素-14-苯甲酸酯、阿霉素-14-辛酸酯、阿霉素-14-萘乙酸酯、安吖啶、卡莫司汀、环磷酰胺、阿糖胞苷、依托泊苷、洛伐他汀、美法仑、拓扑替康(topetecan)、奥沙利铂、苯丁酸氮芥、甲氨喋呤、洛莫司汀、硫鸟嘌呤、天冬酰胺酶、长春碱、长春地辛、他莫昔芬或氮芥。虽然不想受到限制,但治疗性抗体包括针对her2蛋白的抗体,如曲妥珠单抗;针对生长因子或生长因子受体的抗体,诸如靶向血管内皮生长因子的贝伐单抗,和靶向表皮生长因子的osi-774;靶向整联蛋白受体的抗体,例如vitaxin(也称为medi-522)等。适用在本发明组合物和方法中的抗癌剂类别包括但不限于:1)生物碱,包括微管抑制剂(例如长春新碱、长春碱和长春地辛等)),微管稳定剂(例如紫杉醇[紫杉酚],以及多西他赛、泰索帝(taxotere)等)和染色质功能抑制剂,包括拓扑异构酶抑制剂,例如表鬼臼毒素(例如依托泊苷[vp-16]和替尼泊苷[vm-26]等),以及靶向拓扑异构酶i的剂(例如喜树碱和isirinotecan[cpt-11]等);2)共价dna结合剂[烷化剂],包括氮芥类(例如,氮芥、苯丁酸氮芥、环磷酰胺、异环磷酰胺和白消安[马勒兰]等),亚硝脲类(例如卡莫司汀、洛莫司汀和司莫司汀(semustine)等)和其它烷化剂(例如达卡巴嗪、羟甲基三聚氰胺、噻替派和丝裂霉素等));3)非共价dna结合剂[抗肿瘤抗生素],包括核酸抑制剂(例如,更生霉素[放线菌素d]等)、蒽环类(例如柔红霉素[道诺霉素和盐酸佐柔比星]、多柔比星[阿霉素]和伊达比星(idarubicin)[依达比星(idamycin)]等),蒽二酮类(例如蒽环类似物,如[米托蒽醌]等)、博莱霉素类(bleomycins)(博莱霉素(blenoxane))等和光神霉素(plicamycin)(光辉霉素(mithramycin))等;4)抗代谢物,包括抗叶酸剂(例如甲氨喋呤、folex和mexate等)),嘌呤抗代谢物(例如6-巯基嘌呤[6-mp,嘌呤醇],6-硫鸟嘌呤[6-tg],硫唑嘌呤,阿昔洛韦,更昔洛韦,氯脱氧腺苷,2-氯脱氧腺苷[cda],和2’-脱氧助间型霉素[喷死他汀)等),嘧啶拮抗剂(例如氟嘧啶[例如,5-氟尿嘧啶(adrucil)、5-氟脱氧尿苷(fdurd)(氟尿苷(floxuridine))]等),和胞嘧啶阿拉伯糖苷(例如,赛德萨[ara-c]和氟达拉滨等);5)酶,包括l-天冬酰胺酶;6)激素,包括糖皮质激素,诸如抗雌激素(例如他莫昔芬等)),非甾族抗雄激素(例如氟他胺等))和芳香化酶抑制剂(例如阿那曲唑(anastrozole)[阿那托唑(arimidex)]等);7)铂化合物(例如顺铂和卡铂等));8)与抗癌药物、毒素和/或放射性核素等缀合的单克隆抗体;9)生物反应调节剂(例如干扰素[例如,ifn-α等)]和白细胞介素[例如,il-2等]等等);10)过继性免疫疗法;11)造血生长因子;12)诱导肿瘤细胞分化的剂(例如全反式维甲酸等));13)基因治疗技术;14)反义治疗技术;15)肿瘤疫苗;16)针对肿瘤转移的疗法(例如batimistat等));和17)血管生成抑制剂。
其它治疗剂的例子包括:环孢菌素(例如环孢菌素a)、ctla4-ig、抗体诸如icam-3、抗il-2受体(抗tac)抗体、抗cd45rb抗体、抗cd2抗体、抗cd3(okt-3)抗体、抗cd4抗体、抗cd80抗体、抗cd86抗体,阻断cd40和gp39之间相互作用的剂,诸如对cd40和/或gp39(即cd154)特异性的抗体,由cd40和gp30构建的融合蛋白(cd40ig和cd8gp30),nf-κb功能的抑制剂,例如核转位抑制剂,诸如脱氧斯格埃林(dsg)、胆固醇生物合成抑制剂如hmgcoa还原酶抑制剂(洛伐他汀和辛伐他汀)、非甾族抗炎药(nsaid)诸如布洛芬和环加氧酶抑制剂如罗非昔布、类固醇如泼尼松或地塞米松、金化合物、抗增殖剂诸如甲氨蝶呤、fk506(他克莫司,prograf)、霉酚酸酯(mycophenolatemofetil)、细胞毒性药物诸如硫唑嘌呤和环磷酰胺、tnf-a抑制剂如替尼达普、抗tnf抗体或可溶性tnf受体和雷帕霉素(西罗莫司或雷帕鸣)或其衍生物。
可以与本发明的组合物和方法联合施用的其它剂包括蛋白质治疗剂,诸如细胞因子、免疫调节剂和抗体。如本文所用,术语“细胞因子”包括趋化因子、白介素、淋巴因子、单核因子、集落刺激因子和受体相关蛋白及其功能性片段。如本文所用,术语“功能性片段”是指具有生物功能或活性的、通过确定的功能测定被鉴定的多肽或肽。
细胞因子包括内皮单核细胞活化多肽ii(emap-ii)、粒细胞-巨噬细胞-csf(gm-csf)、粒细胞-csf(g-csf)、巨噬细胞-csf(m-csf)、il-1、il-2、il-3、il-4、il-5、il-6、il-12和il-13、干扰素等,以及与细胞或细胞机制中的特定生物性、形态性或表型性改变相关的细胞因子。
针对许多器官系统中的若干毒性损伤,聚(adp-核糖)聚合酶-1(parp-1)的抑制或基因缺失具有意义深远的保护作用。奠定parp-1依赖性细胞死亡的分子机制包括线粒体凋亡诱导因子(aif)的释放和向细胞核的转位,这导致染色质溶解。aif如何诱导染色质溶解和细胞死亡尚未被知晓。本发明将巨噬细胞移动抑制因子(mif)鉴定为具有mg2+/ca2+依赖性核酸酶活性的parp-1依赖性aif相关核酸酶(paan)。aif是向细胞核招募mif所必需的,在细胞核中mif将基因组dna切割成20-50kb的片段。mif的耗尽、aif-mif相互作用的中断或在催化核酸酶结构域中e22至q22的突变在小鼠的神经元培养物和局灶性中风中阻断mif核酸酶活性、抑制谷氨酸兴奋毒性后的染色质溶解和细胞死亡。抑制mif的核酸酶活性是由parp-1过度活化引起的疾病的潜在的关键治疗靶。
mif被认为是由mnng或nmda兴奋毒性诱导的parp-1依赖性细胞死亡所必需的。
与先前的发现(13,14)一致地,endog对于parp-1依赖性的大dna片段化和mnng诱导的细胞死亡是不可缺少的(图12)。为了鉴定parp-1依赖性aif相关核酸酶(paan),用重组aif探测了16k和5k蛋白芯片(15)。最强的160个aif相互作用因子被推进到基于sirna的筛选中,以在hela细胞培养物中鉴定对由mnng诱导的依赖性细胞死亡的改变物(modifier)(这是研究依赖性细胞死亡的已充分表征的方法)(图1a和b)。这些aif相互作用因子基于它们的敲除提供等同于parp-1敲除的保护的能力以及它们是否显示出与可能的核酸酶活性一致的序列和结构同源性而被进一步分离。发现,aif相互作用因子18的敲除具有与parp-1敲除一样的保护作用(图1b)。aif相互作用因子18以前以各种同义称谓被知晓,并且它统称为巨噬细胞移动抑制因子(mif或mmif)(16,17)。针对人和小鼠mif的三种不同shrna构建体被用来证实mif的敲除阻止hela细胞中由mnng毒性诱导的依赖性细胞死亡或小鼠初级皮层神经元中的nmda兴奋性毒性诱导的依赖性细胞死亡(图13,a至f)。为了排除从shrna脱靶的效应,制备了抵抗shrna1(rshrna1)和shrna3(rshrna3)的mif构建体,并且这些mif构建体显示出其不受敲除影响(图13g)。这些抗性mif构建体在内源性mif敲除的环境中恢复nmda兴奋毒性(图13h),证实由mnng或nmda诱导的依赖性细胞死亡需要mif。
mif包含三个pd-d/e(x)k基序,这三个基序存在于许多核酸酶中(18-20)(图1c和2d),并且遍布哺乳动物物种是高度保守的(图14a)。此外,它含有cxxcxxhx(n)c锌指结构域(图1c和图14b),该结构域通常存在于在dna损伤反应蛋白中(20)。已知mif以三聚体的形式存在(21-23)。mif三聚体中的核心pd-d/e(x)k拓扑结构由紧邻2条α链的4条β链组成(图1e和图14,c至g),这与包括ecori、ecorv、exoiii和pvuii在内的已充分表征的核酸酶相似(图14,h至o)。这些序列分析和3-d建模结果表明,mif属于pd-d/e(x)k核酸酶样超家族(24,25)。
为了确定mif是否具有核酸酶活性,将pcdna质粒与重组mif一起孵育。超螺旋pcdna被mif切割成开放的圆形,并进一步切割成线性形式(图2a)。此外,mif以浓度和时间依赖的方式切割人类基因组dna(图15,a和b)。mif核酸酶活性需要添加10mmmg2+、2mmca2+或1mmmn2+(图15c),这与其它类似核酸酶体外活性所需的二价阳离子浓度一致(26)。edta阻断mif对人类基因组dna的核酸酶活性(图2b)。在不存在二价阳离子或阳离子在2-10μm时,mif没有核酸酶活性(图15c)。在mif存在下,添加200μmzn2+会沉淀基因组dna,而添加2μmzn2+则不起作用。此外,na+对mif的核酸酶活性没有影响(图15c)。重要的是,脉冲场凝胶电泳表明mif将人类基因组dna切割成与从用mnng处理的hela细胞纯化的dna相当的大片段(图2b,泳道8)。mif的shrna敲除阻止了mnng诱导的dna切割,这类似于3,4-二氢-5[4-(1-哌啶基)丁氧基]-1(2h)-异喹啉(dpq)抑制parp的作用(图2c)。由于mif被探查为具有互变异构酶活性,因此对mif互变异构酶抑制剂iso-1进行了检测(27)。iso-1未能阻止mnng诱导的dna损伤(图2c)。此外,缺乏互变异构酶活性的mifp2g互变异构酶突变体(28)对mif的核酸酶活性没有影响(图15d)。这些数据综合表明mif是一种核酸酶,并且它在parp-1依赖性dna片段化中起着重要作用。
为了鉴定对mif的核酸酶活性关键的氨基酸残基,pd-d/e(x)k结构域内关键的天冬氨酸、谷氨酸和脯氨酸残基被突变。谷氨酸22被丙氨酸(e22a)或谷氨酰胺(e22q)的取代明显抑制mif的核酸酶活性,而天冬氨酸取代(e22d)却不会(图2d、图15,e至h)。这些数据表明,mif第一个α-螺旋中的谷氨酸残基(e22)对其核酸酶活性至关重要,这与先前报道的一致,即许多核酸外切酶-核酸内切酶-磷酸酶(eep)结构域超家族核酸酶的第一个α-螺旋中的谷氨酸高度保守,并且它是核酸酶活性的活性位点(24,25)。
先前的研究表明,mif具有氧化还原酶和互变异构酶活性二者(27,29,30)。mif活性位点突变体e22q和e22a对mif的氧化还原酶或互变异构酶活性没有明显的影响(图16,a和b),表明mif核酸酶活性独立于其氧化还原酶和互变异构酶活性。此外,如通过远紫外(uv)圆二色性(cd)和近uvcd光谱测定的(分别是研究蛋白质二级和三级结构的常用方法),发现mif的蛋白质确认(proteinconfirmation)不受e22q和e22a突变的影响(图16,c至m)。mif蛋白的纯度通过考马斯蓝染色、fplc和质谱(ms)测定确认(图15g、16c、16d,材料和方法)。没有观察到外来核酸酶污染。
为了进一步研究在用dmso或mnng处理(50μm,15分钟)的hela细胞中mif是否与dna结合,进行染色质免疫沉淀(chip)测定,接下来进行深度测序(图17)。使用meme-chip,鉴定了两类mif结合基序(图3a)。第一类(序列1-3)代表高度相关家族的重叠序列(图3a和图18a)。这个家族的序列特征在序列1中得到最好的捕捉,序列1是所鉴定到的具有30个核苷酸的统计上最重要的基序,并被命名为ps30。所鉴定的第二类是多聚(a)延伸(poly(a)stretch)。
p16、d17和e22在同一pd-d/e(x)k基序中。三维计算机建模显示,mif上的p16和d17接近双链dna(dsdna),而e22接近ssdna,表明mif可能结合ssdna或dsdna或两者(图18b)。检测两类mifdna结合基序的单链和双链形式的mif结合和切割特异性。ssps30序列被合成为具有5’生物素标记,并进行电泳迁移率改变测定(emsa)(图18c)。发现mif在10mmmg2+的存在下与生物素标记的ssps30结合,形成了一种主要复合物(图18c),通过添加过量的未标记dna底物(ps30)或针对mif的多克隆抗体,该过程被完全破坏(图18c)。mife22q、e22a、p16a、p17a和p17q突变体仍然形成mif/ssps30复合物(图18c)。
由于ssps30具有在5’和3’端形成具有未配对碱基的茎环结构,因此决定确定mif是否与ssdna具有序列或结构特异性地结合。在emsa中使用了5’生物素标记的ssps30及其因去除5’端、3’端、以及5’端和3’端的未配对碱基或因去除茎环而结构不同的序列相关底物(图3b和图19)。发现完全去除3’未配对碱基(5’blf)对dna/mif复合物的形成没有明显影响(图2e和图19)。相反,除去5’未配对碱基(5’brf)减少了dna-mif的结合,尽管mif仍然以低效率与dna结合。当去除5’和3’未配对碱基(5’bsl)时,观察到相似的结果。这些数据表明mif主要与具有茎环结构的ssdna中的5’未配对碱基结合。没有茎环的多聚a序列(5’bpa30)和在茎环结构5’端的短多聚a序列(5’b3f1)也被用作底物,并且发现mif不能结合5’bpa30,但明显结合5’b3f1,表明mif-ssdna结合需要茎环(图3b和图19)。除ps30序列相关底物外,还测试了具有茎环样结构(5’bl3)的非序列相关底物,并且发现mif与5’bl3弱结合。但是5’bl3的结合效率远低于5’bps30的结合效率。这些数据表明,mif优先与具有茎环的ssdna结合,并且不太依赖于序列特异性。
与ssdna研究平行地,使用ps30、多聚a、ps30序列相关底物(5’bps30、5’bsl、5’blf、5’brf、5’bpa30和5’bpa5e)以及非相关序列(pcs和5’bl3)测试mif是否与dsdna结合(图3b和图19)。发现mif不能结合到这些双链底物中的任何一个(图3b)。
为了确定mif切割的是单链dna还是双链dna,在ps30dna结合基序的5’和3’端添加了35个随机核苷酸并将其命名为ps100,并监测对ssdna(ssps100)或dsdna(dsps100)的切割。mif充分地切割ssps100及其互补链ssps100r,但不切割dsps100(图20,a和b)。从chipseq(ps30)中鉴定的mifdna结合基序对于mif切割是足够的,因为渐增浓度的mif能够切割ssps30(图20c)。然而,渐增浓度的mif浓度(1-4μm)不能切割dsps30、其相关序列dsrf以及其非相关序列dsl3(图20c)。ssps30的mif切割需要mg2+(图20e)。mife22q和e22a突变阻断对ssps30的切割(图20e)。mif以t1/2为12分钟的时间依赖性方式切割ssp30,并且以2μm的km和41.7nm/min的vmax的浓度依赖性方式切割ssp30(图20,f至h)。这些动力学性质类似于其他pd-d/e(x)k核酸酶,诸如ecori(26,31)。mif对单链dna的偏好符合单链dna结合mif活性位点的3-d模型(图18b)和mif-dna结合测定(图3b)。
为了确定mif是否具有序列或结构特异性核酸内切酶或核酸外切酶活性,基于dna底物ssps30的二级结构合成了一系列5’和3’生物素标记的变体,并检测了mif的切割(图3c和图19)。发现mif具有3’核酸外切酶活性,并且它更偏好识别和降解ssps303’端的未配对碱基,这被3’端的生物素修饰所阻断(图3c泳道2-5和图19,表1)。mif的3’核酸外切酶活性得到通过使用5’brf底物以及5’b3e底物的切割测定的支持(图3c和图19,表1)。此外,mif的3’核酸外切酶活性允许它切割5’生物素-多聚a(5’bpa30),但不能切割3’生物素-多聚a(3’bpa30),表明mif的3’核酸外切酶活性与二级结构无关(图3c和图19)。mif还具有结构特异性核酸内切酶活性。它在靠近茎环的3’端切割ssdna的短的未配对碱基(5’bps40,3’bps40,5’b3f1,3’b3f1和5’bl3),以及靠近茎环的3’端的3’-oh/3-生物素(3’bsl和3’blf)(图3c和图19)。与其核酸外切酶活性相反,mif的核酸内切酶活性不被3’端的生物素修饰阻断(3’bsl,3’blf,3’bps40和3’b3f1)。5’bl3是ps30不相关的序列,但具有类似的被mif切割的茎环结构,不过效率较低(图3c和图19)。总之,这些结果表明mif具有3’核酸外切酶和核酸内切酶活性,并在3’端切割茎环ssdna的未配对碱基。
为了进一步研究mif在何处切割dna并避免生物素标记的可能的干扰,使用在茎环结构3’端只有1个未配对碱基的未标记ps30和3f1作为底物,并定制了基于ps30的两种不同dna梯形物(ladder)。通过将mif(2μm)与ps30孵育2小时,检测到20和22个核苷酸的两种主要产物(图3d)。此外,还观察到微弱的更高分子量条带。这些高分子量条带在生物素标记的ps30mif切割实验中更加明显,其中孵育时间为1小时(图3d)。3f1底物的mif切割仅产生29nt的条带,该条带符合切割掉茎环结构3’端的1个未配对碱基(图3,d和e)。这些数据表明,ps30最初被mif在“a23↓t24↓t25”之后同时使用3’核酸外切酶和核酸内切酶活性切割(图3e左侧图块)。然后得到的产物形成更稳定的结构(图3e右侧图块),且mif在“g20↓g21↓g22”后切割茎环结构3’端新的未配对碱基。总之,mif利用3’核酸外切酶和核酸内切酶活性,在靠近茎环的3’端在+1至+3位置处切割未配对碱基。
为了证实mif是aif的相互作用蛋白,进行了gst拉下实验。野生型gst-aif将内源性mif拉下,且野生型gst-mif将内源性aif拉下(图4a和图21,a至d)。然后mif-aif结合结构域被映射。发现mif在aa567-592处与aif结合(图21,a至c)。相反,mife22a突变体在gst拉下中与aif的结合显著减少,而e22d和e22q仍然与aif结合(图4,a和b以及图21d)。此外,另一个pd-d/e(x)k和c57a;c60a突变仍然与aif结合(图21d)。这些数据表明mife22对aif结合至关重要。根据gst拉下数据,在用500μmnmda处理的皮层神经元中,aif免疫共沉淀mif,但在未处理的培养物中几乎检测不到(图4,c和d)。
mif主要定位于hela细胞(图21e)和皮层神经元(图4e)的胞液。mif和aif都转位到细胞核,并且在hela细胞被mnng刺激和皮层神经元被nmda刺激后共同定位在细胞核内。aif的敲除导致mif丧失向细胞核的转位,但是mif的敲除并不能阻止aif在nmda暴露后向细胞核的转位(图4e)。亚细胞分离为核部分和核后部分,证实了mif和aif在皮层神经元培养物的nmda暴露后向细胞核的转位,并且aif是mif转位所必需的(图4,f和g)。dpq阻止在皮层神经元中的nmda施用以及hela细胞中的mnng处理后mif和aif在细胞核中的累积(图21,e至j)。与nmda兴奋毒性涉及一氧化氮产生的观点一致地,一氧化氮合成酶抑制剂硝基精氨酸(n-arg)阻止mif和aif在细胞核中的累积(图21h-j)。
mif广泛分布在整个大脑中,并且mif敲除小鼠之前已经被描述过(图4h)(32)。用携带mif-wt-flag、mif-e22q-flag和mif-e22a-flag的慢病毒转导来自mif敲除小鼠的原代皮质培养物以确认aif/mif结合对于nmda施用后mif的细胞核累积的需求。与gst拉下实验一致(图4a),野生型mif和e22q与aif相互作用,但是mife22a不与aif结合(图4i)。在未转导的mif敲除培养物中以及在用mif-wt-flag、mif-e22q-flag和mif-e22a-flag转导的mif敲除培养物中,aif在施用nmda后转位至细胞核(图4j)。mif野生型和mife22q也都转位到细胞核;然而,aif结合缺陷突变体mife22a未能如此(图4j)。亚细胞分离为核部分和核后部分,证实了通过免疫荧光得到的观察结果(图4,k和l)。综上所述,这些结果表明mif与aif的相互作用是mif核转位所必需的。
为了确定依赖性细胞死亡是否需要mif的核酸酶活性和aif介导的招募,用核酸酶缺陷型mife22q突变体和aif结合缺陷型mife22a突变体转导mif敲除培养物。与shrna敲除实验一致,mif敲除的皮层培养物抵抗nmda兴奋毒性(图5a和图22a)。用野生型mif转导完全恢复nmda兴奋毒性,相反,mife22q和mife22a都没有恢复nmda兴奋毒性(图5a和图22a)。通过彗星测定,发现在野生型皮层神经元中nmda的施用导致大量具有彗星尾部的神经元,尾部长度和尾部中的dna增加,而mif敲除的神经元没有明显的彗星尾部阳性神经元(图5b和图22,b至d)。用野生型mif而不是用mife22q或mife22a转导的敲除型神经元在施用nmda后恢复了彗星尾部,增加了尾部长度和尾部中的dna(图5b和图22,b至d)。施用mnng后,与非靶向shrna相比,用两种不同shrna进行对hela细胞中mif的shrna敲除导致具有彗星尾部的细胞数量减少,尾部长度和尾部中的dna减少(图23,a至d)。基因组dna的脉冲场凝胶电泳测定证实,施用nmda在野生型皮层神经元中造成大的dna片段,但在mif敲除的皮层神经元中并不这样(图5c)。在用mife22q或mife22a转导的mif敲除的神经元中没有观察到明显的大dna片段(图5c)。用野生型mif转导敲除型神经元恢复nmda诱导的大dna片段(图5c)。这些结果综合起来表明,mif是参与由mnng或nmda诱导的依赖性细胞死亡引起的大规模dna片段化的主要核酸酶。
为了在评估由于依赖性细胞死亡引起的体内细胞死亡中,对mif核酸酶活性和mif结合aif的需要,用核酸酶缺陷型mife22q突变体和aif结合缺陷型mife22a突变体通过注射新生小鼠的侧脑室区转导mif敲除小鼠。然后,对两个月大的雄性小鼠进行45分钟的大脑中动脉短暂阻塞(mcao)。在成年小鼠中的皮层、纹状体和海马体中,mif-flag的免疫染色证实了转导的有效性(图24,a和b)。尽管缺血损伤的强度相似(图24c),mif敲除小鼠的皮层、纹状体和半球的梗塞体积与野生型对应物相比,减少了约30%(图5,d和e和图24,d和e)。此外,mif敲除小鼠的神经保护作用保持了至少7天(图5e和图24e)。在mif敲除小鼠中,野生型mif而不是mife22q或mife22a的表达将梗塞体积恢复到野生型水平(图5,d和e和图24,d和e)。mcao后1天、3天和7天,通过开放场地任务(openfieldtask)中的自发型活动评估神经行为。与梗塞数据一致,与野生型相比,mif敲除小鼠具有提高的神经行为评分。表达野生型mif的mif敲除小鼠具有等同于野生型小鼠的神经行为评分,而mife22q或mife22a的表达与mif敲除小鼠没有显著差异(图5f和g)。在3天和7天内,mif敲除小鼠的神经行为评分相对于野生型小鼠而言仍然受到保护(图5f和g)。角落测试(conrnertest)数据显示,所有小鼠在mcao手术前都没有表现出侧位偏爱(sidepreference)。然而,野生型小鼠和表达野生型mif的mif敲除小鼠在mcao后的第1、3和7天具有转向未受损侧的显著增加(图24f),表明这些小鼠具有更严重的感觉和运动缺陷。在mif敲除小鼠和表达mife22q或mife22a的mif敲除小鼠中没有观察到偏好(图24f)。在中风的半影区中aif和mif的定位通过共聚焦显微镜检测(图24g)。与在皮层神经元中的观察结果一致,在mcao后1、3和7天,在mif野生型、敲除型以及注射mif野生型、e22q和e22a的mif敲除小鼠中,aif转位到细胞核(图24g)。mif野生型和mife22q也都在mcao后1、3和7天转移到细胞核;然而,aif结合缺陷突变型mife22a没有未能如此(图24g)。通过脉冲场凝胶电泳评估的dna损伤在mcao后第1、3和7天观察到,在第3天显示出在野生型小鼠或表达野生型mif的mifko小鼠中dna损伤最严重(图24h)。在mifko小鼠和表达e22q或e22amif的mifko小鼠中dna损伤减少(图24h)。这些数据表明,aif介导的神经毒性和dna切割需要mif,并且体内mif转位需要aif。
本发明的一个主要发现是将mif鉴定为paan。使用分子建模,显示mif三聚体包含与pd-d/e(x)k核酸酶超家族相同的拓扑结构,中心的四个链状的混合的β-片层紧邻两个α-螺旋(24,25)。mif具有3’核酸外切酶和核酸内切酶活性。它与具有茎环结构的ssdna的5’未配对的碱基结合,并切割其3’未配对碱基。aif与mif相互作用,并将mif招募到细胞核中,在细胞核中mif结合基因组dna并将其切割成大片段,这些大片段的尺寸与通过激活依赖性细胞死亡的应激物(stressor)诱导的尺寸相似。mif的敲除显著减少了激活parp-1依赖性细胞死亡的刺激引起的dna片段化。突变pd-d/e(x)k基序中的关键氨基酸残基消除了mif的核酸酶活性,并在体外和体内保护细胞免于依赖性细胞死亡。aif和mif蛋白-蛋白相互作用的中断阻止mif从胞液向细胞核的转位,这也在体外和体内阻止了parp-1依赖性细胞死亡。mif的硫醇-蛋白质氧化还原酶活性或互变异构酶活性都不参与其作为核酸酶的作用。mif的敲除,mif核酸酶缺陷突变体和mif的aif结合缺陷突变体都减少了梗塞体积,并且在小鼠中风的局部缺血模型中具有持久的行为性拯救作用。因此,mif是长期寻求的paan,其在由parp-1的活化和aif的释放导致的细胞死亡中是重要的(2)。
像parp一样,抑制mif核酸酶活性对于急性神经系统紊乱是一个有吸引力的靶。然而,它在慢性神经退行性疾病中可能优于parp抑制,其中长期的抑制parp会损害dna损伤反应和修复。抑制mif的核酸酶活性可以绕过这一可能的担忧,并可以为各种紊乱提供重要的治疗机会。
发现mif具有3’核酸外切酶和核酸内切酶活性以及其核酸酶活性偏好的dna序列。该序列被固定在dna-bind板上,并在有或没有来自大环化合物文库的池的情况下与重组mif一起孵育,并与生物素化的互补dna杂交。该序列通过分光计测量的色度变化来检测。如果池含有mif抑制剂,黄色底物颜色将被保持。如果mif是有活性的,dna将被切割,且颜色将丢失(图6)。
大环天然产物fk506和雷帕霉素是具有重要生物活性的被批准的免疫抑制药物。在结构上,fk506和雷帕霉素享有相似的fkbp结合结构域,但是它们的效应结构域不同。交换fk506和雷帕霉素的效应结构域可以提供靶从钙调磷酸酶到mtor的变化。因此,在功能上取代效应结构域以靶向人类蛋白质组中的蛋白质是可能的。设计并生成了一个新的大环文库以靶向新的蛋白质,称为rapafucins的该文库含有合成的fkbp-结合结构域和四肽基效应结构域(图7)。在筛选文库后,已经鉴定到几种有效抑制mif核酸酶活性的击中物。
混合大环文库由在15个不同化合物的池中的45000种化合物。筛选了38块板(约3000个池),并用切割测定完成了对这些汇集的文库的筛选(图8)。阳性池中的化合物已在切割测定中被单独地测试,并进一步评估了在用mnng作为依赖性细胞死亡的诱导剂处理的hela细胞中的体外神经保护作用(图9)。最初选择了12个阳性候选物并在剂量反应dna切割测定和mnng诱导的细胞死亡测定中对它们进行了测试,然后最后选择了4个候选物(c7;12b3-11,c8;12b3-11,c11;17a5-1,c12;17a5-2)。
阳性候选物被推进到tbe凝胶中的剂量反应dna切割测定(图10a)和在用mnng处理的hela细胞中的神经保护作用(图10b)。此外,阳性候选物以α-synucelin预先形成的原纤维(α-synpff)神经毒性被测试。对重组的错误折叠的α-synpff的处理提供了能够研究α突触核蛋白在体内和体外的传播和毒性的帕金森病的模型系统。原代皮质培养物被暴露于pff±mif抑制剂14天。通过hoechst/碘化丙啶阳性细胞的计算机辅助细胞计数来测定细胞活力。这里,c8和c12显示出pff诱导的毒性方面的最大保护作用,并且这两个击中物在pff毒性的剂量反应中被证实(图11)。
材料和方法
人蛋白芯片的高通量筛选
将通过将16000或5000种高度纯化的蛋白质点样到专门的硝酸纤维素包被的载玻片(15)上制备出的16k和5k人类蛋白质芯片在含有50mmtris-hcl、ph8.0、100mmnacl、1mmdtt、0.3%吐温20的复性缓冲液中于4℃孵育1小时。在室温下用5%无脂肪奶粉封闭1小时后,将蛋白质芯片与纯化的小鼠aif蛋白质(50nm,np_036149)在1%牛奶中孵育1小时。然后,蛋白质相互作用通过依次与兔抗aif抗体(9,11)和alexa
parp-1依赖性细胞活力的基于sirna反向转染形式的筛选(reversetransfectionformatsirna-basedscreen)。
在来自dharmacon的96孔板中定制靶向从人蛋白芯片高通量筛选产生的aif相互作用蛋白的on-targetplustm
核酸酶测定
在含有10mm氯化镁和1mmdtt的10mmtris-hcl缓冲液(ph7.0)或如所示的特殊缓冲液中,将人类基因组dna(200ng/反应,promega)、pcdna(200ng/反应)或ps30及其相关和非相关底物(1μm)与所指示的终浓度为0.25-8μm的野生型mif或其变体在37℃下孵育1小时(用pcdna和小dna底物)或4小时(用人类基因组dna)。用含有10mmedta的上样缓冲液和冰上孵育来终止反应。立即在0.5xtbe缓冲液中的1.2%脉冲场认证琼脂糖(pulsefieldcertifiedagarose)上分离人基因组dna样品,在6v/cm持续12小时,初始切换时间(switchtime)为1.5秒,且最终切换时间为3.5秒。pcdna样品用1%琼脂糖凝胶测定。在15%或25%tbe-尿素聚丙烯酰胺(page)凝胶或20%tbepage凝胶上分离小dna底物。然后凝胶用0.5μg/ml溴化乙锭(etbr)染色,接下来电泳转移到尼龙膜上。然后,使用化学发光核酸检测模块(chemiluminescentnucleicaciddetectionmodule)(thermoscientific)通过化学发光进一步检测生物素标记的dna。
电泳迁移率变化测定(emsa)
emsa测定按照制造商的说明,使用lightshiftchemiluminescentemsa试剂盒(thermoscientific)进行。简言之,将纯化的mif蛋白(2μm)与生物素标记的dna底物(10nm)在含有10mmmgcl2的结合缓冲液中于冰上孵育30分钟。然后在6%缓滞聚丙烯酰胺(retardationpolyacrylamide)上分离样品,接下来电泳转移到尼龙膜上。然后,使用化学发光核酸检测模块(thermoscientific)通过化学发光进一步检测生物素标记的dna。
彗星测定
彗星测定按照trevigen(gaitherburg,md)提供的方案进行。简而言之,在处理后6小时,用冰冷的pbs洗涤经或未经mnng处理的hela细胞和经或未经nmda处理的皮层神经元,在720g离心10分钟后收获,并以1×105细胞/ml重新悬浮在冰冷的pbs(不含ca2+和mg2+)中。然后将细胞与pbs(42℃)中1%的低熔点琼脂糖以1∶10(v/v)的比例混合,并将50μl细胞-琼脂糖混合物立即用移液器转移到cometslide上,并平坦地放置在4℃下于黑暗中持续30分钟以增强附着。在裂解缓冲液中裂解后,在室温下用碱性解旋溶液(200mmnaoh,ph>13,1mmedta)浸泡载玻片1小时。将彗星载玻片转移并在水平电泳设备中用1l碱性解旋溶液在21伏下电泳30分钟。排出多余的电泳缓冲液后,用dh2o冲洗载玻片两次,然后用70%乙醇固定5分钟,并用sybrgreen在4℃下染色5分钟。使用zeiss落射荧光显微镜(axiovert200m)捕获细胞图像,并用casp软件(版本1.2.2)进行图像分析。测量了每个样品的“彗星尾部”长度,其被定义为从核的边缘到彗星尾部末端的长度。
蛋白质表达与纯化
人endog(nm_004435)、亲环素a(nm_021130)、小鼠aif(nm_012019)、人mif(nm_002415)cdna及其变体通过ecori和xhoi限制性位点被亚克隆到谷胱甘肽s转移酶(gst)标记的pgex-6p-1载体(gehealthcare)中,并通过测序验证。该蛋白从从大肠杆菌(escherichiacoli)中表达并通过谷胱甘肽琼脂糖纯化。gst标签随后被蛋白水解去除用于核酸酶测定。mif点突变体通过聚合酶链式反应(pcr)构建,并通过测序验证。用于核酸酶测定的mif蛋白的纯度通过质谱进一步确定。通过fplc纯化的mif蛋白也用于核酸酶测定中并且在fplcmif和非fplcmif蛋白之间没有观察到明显差异。gst蛋白被用作核酸酶检测中的阴性对照。
大脑中动脉闭塞(mcao)
如前所述,脑缺血通过45分钟可逆mcao诱导(33)。成年雄性mifko小鼠(2至4个月大,20-28g)用异氟醚麻醉,并通过反馈控制加热系统将体温保持在36.5±0.5℃。制备腹侧颈部正中切口,通过将一根7.0尼龙单丝经颈外动脉残端从颈内动脉/翼腭动脉分叉处插入右侧颈内动脉6-8mm进行单侧mcao。对假手术动物进行了同样的外科手术,但是缝线没有进入颈内动脉。再灌注1天、3天或7天后,mifwt和ko小鼠灌注pbs并用氯化三苯基四唑(ttc)染色。用4%pfa进一步固定大脑,并切片用于免疫组织化学染色(9,11,15,34)。
chip-seq
如前所述(35,36)地进行chip-seq。简言之,hela细胞首先用dmso或mnng处理(50μm,15分钟)。在mnng处理后5小时,在37℃下用1%甲醛交联细胞20分钟,并在0.125m甘氨酸中终止。超声波处理前进行染色质提取。使用抗mif抗体(ab36146,abcam),从超声处理的细胞裂解物中免疫沉淀dna。这些文库是根据伴随dna样品试剂盒提供的illumina的说明来制备的,并使用illuminahiseq2000测序,产生50bp的单端读段(single-endread)。
详细程序如下。hela细胞用dmso或mnng(50μm)处理15分钟,并在新鲜培养基中再培养5小时。然后在37℃下用1%甲醛交联细胞10分钟,并在室温下在0.125m甘氨酸中持续20分钟猝灭反应。使用来自cellsignalingtechnology的
chip-seq数据分析
来自hiseq2000的原始数据使用casavav1.8转换成fastq,并被解复用(demultiplex)。使用默认参数,使用bowltie2(2.0.5版)将读段映射至人类基因组(hg19)。转换后的sam文件被传递给macs(1.4.1版),以便使用默认参数的峰值调用。来自dmso和mnng处理的文库的峰以bed格式报告,并提供在geo中。在dmso和mnng处理组中差异化地鉴定到的峰由自定义r脚本解析。在仅经mnng处理但未经dmso处理的文库中鉴定到的峰对应的序列被输入到sesimcmc_4_36、chipmunkv4.3+和memechipv4.9.0中,以便使用默认参数发现基序。
数据传输:casavav1.8软件用于将原始文件转换为fastq文件,并对通道解复用。
dmso_mif:jhutd01001/jhutd01001_001_dpan1/raw
dmso_input:jhutd01001/jhutd01001_002_dinput1/raw
mnng_mif:jhutd01001/jhutd01001_003_mpan1/raw
mnng_input:jhutd01001/jhutd01001_004_minput1/raw
分析:以下是分析步骤的列表以及该步骤使用的参数。所有基序查找软件都是使用默认设置运行的。
1.比对管道(alignmentpipeline)
a.bowdtie2-2.0.5,使用默认参数对hg19基因组进行片段比对,生成单独的sam文件
jhutd01001/jhutd01001_001_dpan1/dpan1_hg19_alignment.sam
jhutd01001/jhutd01001_002_dinput1/dinput1_hg19_alignment.sam
jhutd01001/jhutd01001_003_mpan1/mpan1_hg19_alignment.sam
jhutd01001/jhutd01001_004_minput1/minput1_hg19_alignment.sam
b.使用samtools-0.1.18/对sam文件进行排序并将其转换为bam文件
jhutd01001/jhutd01001_001_dpan1/dpan1_hg19_alignment.bam
jhutd01001/jhutd01001_002_dinput1/dinput1_hg19_alignment.bam
jhutd01001/jhutd01001_003_mpan1/mpan1_hg19_alignment.bam
jhutd01001/jhutd01001_004_minput1/minput1_hg19_alignment.bam
2.使用macs-1.4.1使用默认参数的峰值调用
a.峰值调用
jhutd01001/jhutd01001_000_analysis/macs/dpan1_vs_dinput_peaks.bed
jhutd01001/jhutd01001_000_analysis/macs/mpan1_vs_minput_peaks.bed
b.带注释的(annotated)峰值调用
jhutd01001/jhutd01001_000_analysis/macs/dpan1_vs_dinput_annotation.txt
jhutd01001/jhutd01001_000_analysis/macs/mpan1_vs_minput_annotation.txt
c.根据基因自定义脚本执行差异化峰值调用(differentialpeakcalls)
jhutd01001/jhutd01001_000_analysis/macs/intersections.bothsamples.dpan1.mpan1.txt
jhutd01001/jhutd01001_000_analysis/macs/intersectionsdpan1_not_mpan1.txt
jhutd01001/jhutd01001_000_analysis/macs/intersectionsmpan1_not_dpan1.txt
d.带注释的差异化峰值调用
jhutd01001/jhutd01001_000_analysis/macs/only_dpan1_annotation.txt
jhutd01001/jhutd01001_000_analysis/macs/only_mpan1_annotation.txt
3.覆盖轨道以查看通过igvtools创建的比对
jhutd01001/jhutd01001_000_analysis/coverage_analysis/dpan1.tdf
jhutd01001/jhutd01001_000_analysis/coverage_analysis/dinput1.tdf
jhutd01001/jhutd01001_000_analysis/coverage_analysis/mpan1.tdf
jhutd01001/jhutd01001_000_analysis/coverage_analysis/minput1.tdf
4.使用三种不同的软件找到了基序
a.sesimcmc_4_36
jhutd01001/jhutd01001_000_analysis/motif/sesimcmc_motif_dpan1.txt
jhutd01001/jhutd01001_000_analysis/motif/sesimcmc_dpan1_logo.png
jhutd01001/jhutd01001_000_analysis/motif/sesimcmc_mpan1_motif.txt
jhutd01001/jhutd01001_000_analysis/motif/sesimcmc_mpan1_logo.pdf
b.chipmunkv4.3+
jhutd01001/jhutd01001_000_analysis/motif/dpan1_chipmunk_motif.txt
jhutd01001/jhutd01001_000_analysis/motif/mpan1_chipmunk_motif.txt
c.memechipv4.9.0
jhutd01001/jhutd01001_000_analysis/motif/memechip_dpan1.webarchive
jhutd01001/jhutd01001_000_analysis/motif/memechip_mpan1.webarchive
jhutd01001/jhutd01001_000_analysis/motif/only_mpan1_meme_chip.webarchive
5.ceas软件生成用于区域注释、基因中心化注释和基因组特征附近的平均信号分布的图
jhutd01001/jhutd01001_000_analysis/ceas/dpan1.pdf
jhutd01001/jhutd01001_000_analysis/ceas/mpan1.pdf
jhutd01001/jhutd01001_000_analysis/ceas/mpan1_only.pdf
mif-dna对接方法
使用hex-8.0蛋白质-dna对接程序(39,40)将dna双链结构(37)(pdb登录号1bna)和单链dna结构(pdb登录号2rpd(38))对接到mif表面(pdb登录号1fim(23))。hex程序使用表面互补算法来鉴定蛋白质和dna之间的接触。mif表面是使用pymol生成的。所有图像都用pdb浏览器,pymol查看和标记。如从hex程序获得的一样,示出了mif-dna对接模型。
慢病毒、腺相关病毒(aav)的构建和病毒产生
小鼠mif-wt-flag(nm_010798)、mif-e22q-flag和mif-e22a-flag通过agei和ecori限制性位点亚克隆到慢病毒cfugw载体中,且其表达由人泛素c(hubc)启动子驱动。人类mif和小鼠mifshrna是使用网站<http://katahdin.cshl.org/sirna/rnai.cgi?type=shrna>设计的。该程序给出了97nt寡核苷酸序列,用于产生shrnamir。使用pacisme2正向引物5’cagaaggttaattaaaaggtatattgctgttgacagtgagcg3’和nheisme2反向引物5’ctaaagtagccccttgctagccgaggcagtaggca3’。进行pcr以产生第二条链,并添加paci和nhei限制性位点以将产物克隆到psme2中,psme2是一种将空shrna表达盒到psme2载体中、修改的限制性位点插入到cfugw骨架中的构建体。该载体表达gfp。慢病毒是通过将重组cfugw载体与三种包装载体:plp1、plp2和pvsv-g(1.3∶1.5∶1∶1.5)一起瞬时转染到293ft细胞中产生的。转染后48小时和72小时收集病毒上清液,并通过在50,000g超速离心2小时浓缩。
mif-wt-flag、mif-e22q-flag和mif-e22a-flag通过bamhi和ecori限制性位点亚克隆到aav-wpre-bgh(044am/cba-pi-wpre-bgh)载体中,且其表达由鸡β-肌动蛋白(cba)启动子驱动。所有aav2病毒都是由vectorbiolabs产生。
mif底物、模板和引物的序列
用于shrna构建体和点突变构建体的mif底物、模板和引物的序列如下。
ps100
ps100r
hmifshrna1-
hmifshrna2-
hmifshrna3-
mmifshrna1-
mmifshrna-
mmifshrna3-
aifshrna1-
rsh1-mmif-fw-
rsh1-mmif-re-
rsh3-mmif-fw-
rsh3-mmif-re-
括号中的三个字母指示pam序列,且其他序列(20nt)是靶位点。
细胞培养、转染、慢病毒转导和细胞毒性
hela细胞在补充有10%胎牛血清(hyclone)的dulbecco的改良的eagle’s培养基(invitrogen)中培养。v5标记的mif用lipofectamineplus(invitrogen)转染。如前所述(9)制备来自皮层的原代神经元培养物。简而言之,在0.027%胰蛋白酶/盐水溶液(gibco-brl)中消化10分钟后,解剖皮层并通过在改良的eagle’s培养基(mem)、20%马血清、30mm葡萄糖和2mml-谷氨酰胺中研磨来分离细胞。神经元被铺在包被着聚鸟氨酸的15-mm多孔板或包被着聚鸟氨酸的盖玻片上。将神经元保持在mem、10%马血清、30mm葡萄糖和2mml-谷氨酰胺中,于7%co2加湿的37℃培养箱中。生长培养基每周更换两次。在成熟培养物中,神经元占细胞总数的70%到90%。在体外天数(div)的第7-9天,神经元被携带有mif-wt-flag、mif-e22q-flag或mif-e22a-flag[1x109单位(tu)/ml)的慢病毒感染72小时。依赖性细胞死亡通过mnng(sigma)在hela细胞中诱导或通过nmda(sigma)在神经元中诱导。hela细胞被暴露于mnng(50μm)15分钟,且神经元(div10至14)用对照盐溶液[css,含有120mmnacl、5.4mmkcl、1.8mmcacl2、25mmtris-cl和20mm葡萄糖(ph7.4)]洗涤,暴露于css中的500μmnmda和10μm甘氨酸5分钟,然后暴露于含有10%马血清、30mm葡萄糖和2mml-谷氨酰胺的mem多次,然后固定、免疫细胞化学染色和激光共聚焦扫描显微镜前。第二天,在用7μmhoechst33342(invitrogen)染色所有细胞核和用2μm碘化丙啶(invitrogen)染色死细胞核后,通过无偏见的客观计算机辅助细胞计数来确定细胞活力。用axiovision4.6软件(carlzeiss)计算总细胞数和死亡细胞数。使用至少六个单独的孔进行至少三次独立的实验,每个数据点计数至少15000到20000个神经元或细胞。对于神经元毒性评估,胶质细胞核发出的荧光强度不同于神经元细胞核并被门控排除(gateout)。细胞死亡的百分比被确定为活细胞与死细胞的比率,与对照孔中的细胞死亡的百分比进行比较以解释由于培养物的机械刺激引起的细胞死亡。
拉下、免疫共沉淀和免疫印迹
对于拉下测定,gst标记的mif或aif蛋白质固定的谷胱甘肽琼脂糖珠与500μghela细胞裂解物一起孵育,在裂解缓冲液中洗涤,并在蛋白质上样缓冲液中洗脱。对于免疫共沉淀,在蛋白a/g琼脂糖(santacruzbiotechnology)存在下,将1mg全细胞裂解物与aif抗体(1μg/ml)孵育过夜,接下来用小鼠抗flag抗体(clonem1,sigma)、小鼠抗v5(v8012,sigma)或山羊抗mif(ab36146,abcam)进行免疫印迹测定。蛋白质在变性sds-page上分离,并转移到硝酸纤维素膜上。将膜封闭并用初级抗体(50ng/ml;小鼠抗flag;兔抗aif;或山羊抗mif)在4℃下孵育,接下来用辣根过氧化物酶(hrp)缀合的驴抗小鼠、驴抗兔或驴抗山羊在室温下孵育1小时。洗涤后,通过supersignalwestpico化学发光底物(pierce)检测免疫复合物。
亚细胞分离
核提取物(n)和核后细胞提取物(pn)(其是从去除核蛋白质后的全细胞裂解物中制备的部分)在低渗缓冲液中分离(9,11)。通过监测组蛋白h3和mnsod或tom20免疫反应性(9,11)确定核和核后亚细胞部分的完整性。
免疫细胞化学、免疫组织化学和共聚焦显微术
对于免疫细胞化学,将细胞在mnng或nmda处理4小时后用4%多聚甲醛固定,用0.05%tritonx-100渗透,并用在pbs中的3%bsa封闭。aif通过驴抗兔cy3或驴抗兔647观察。mif通过驴抗小鼠cy2(2μg/ml)、驴抗山羊cy2或驴抗山羊647观察。用针对flag的抗体进行免疫组织化学。免疫荧光分析如所述地用lsm710共聚焦激光扫描显微镜(carlzeiss)进行。
fplc
利用已知分子量的天然标记蛋白的洗脱体积和分子量(kda)之间的标准校准曲线,在aktabasicfplc(amersham-pharmacialimited)中使用superdex20010/300gl柱(gehealthcare,lifesciences)确定纯化的重组mif的天然状态和纯度。在标准pbs缓冲液中以0.5ml/min的流速运行凝胶过滤柱。使用以下分子量标准:分别为铁蛋白(440kda)、醛缩酶(158kda)、伴白蛋白(75kda)、卵清蛋白(43kda)、碳酸酐酶(29kda)和核糖核酸酶(13.7kda)(gehealthcare,lifesciences)。含有mif的洗脱级分在12%sds-page上分离,并用考马斯蓝染色以检查蛋白质的纯度。
mif蛋白纯度的质谱分析
用于核酸酶测定的mif蛋白也通过质谱检测,以排除任何来自其他已知核酸酶的可能污染。使用不同标准在95%和更低的置信水平下进行分析,以捕捉核酸酶的任何极小可能性。使用所有物种对ncbi数据库的分析和搜索揭示,在核酸酶测定中使用的mif蛋白中没有检测到已知的能够消化单链或双链dna的核酸酶。
圆二色(cd)光谱
cd光谱在aviv420cd光谱仪(biomedicalinc.,lakewood,nj,usa)上进行。于室温下,使用0.5cm路径长度的石英比色皿,记录浓度为2mg/ml的蛋白质样品在240-320nm之间的近紫外cd光谱。于室温下,使用0.1cm路径长度的夹心石英比色皿(quartzsandwichcuvette),记录浓度为0.2mg/ml的蛋白质样品在196-260nm之间的远紫外cd光谱(41)。将蛋白悬浮在有或没有氯化镁(5.0mm)和/或氯化锌(0.2mm)的pbs缓冲液中。cd光谱是从0.5nm的数据节距(datapitch)中获取,1nm/3秒的扫描速度和0.5秒的响应时间被选择用于记录。
氧化还原酶活性测定
如前所述(30),使用胰岛素作为底物测量mif的硫醇-蛋白质氧化还原酶活性。简言之,胰岛素测定基于胰岛素的还原和随后胰岛素α-链的不溶解。浊度的时间依赖性的增加随后在650nm处用分光光度法测量。反应通过加入5μm溶解在20mm磷酸钠缓冲液(ph7.2)中的mifwt、e22a、e22q、c57a;c60a或和p2g突变体并将200mm还原型谷胱甘肽(gsh)加入含有1mg/ml胰岛素、100mm磷酸钠缓冲液(ph7.2)和2mmedta的冰冷反应混合物开始的。在同一实验中,mif胰岛素还原通过对比对照溶液(含有gsh)来测量。
互变异构酶活性测定
如前所述(42),使用d-多巴色素互变异构酶作为底物测量互变异构酶活性。简言之,通过在室温下将2mml-3,4二羟基苯丙氨酸甲酯与4mm过氧化钠混合5min制备d-多巴色素甲酯的新鲜溶液,然后在使用前直接置于冰上。,通过向200μl制备在互变异构酶测定缓冲液(50mm磷酸钾,1mmedta,ph6.0)中的mifwt、e22a、e22q、c57a;c60a(最终浓度5μm)和/或p2g突变体中加入20μl多巴色素甲酯底物在25℃启动酶促反应。利用分光光度计,通过od475nm的半连续减少来确定活性。
脑室内(icv)注射
将3μlaav2-mifwt、e22q和e22a(1x1013gc/ml,vectorbiolabs)注射到新生mifko小鼠(34)脑室两侧内。通过免疫组织化学验证mcao手术后mif及其变体8-16周龄期间的表达。
神经行为活动性
通过将动物置于小鼠笼中5分钟来评估mcao后1天、3天和7天的自发运动活动性。在笼子顶部安装了摄像机以记录笼子里小鼠的活动性。神经性缺陷由对动物的治疗和基因型不知情的观察者以0-5(0,无神经性缺陷;5,严重神经性缺陷)的量表进行评估。以下标准被用于对缺陷打分:0=小鼠看起来正常,探索笼子环境,并在笼子里自由活动;1=小鼠在笼子里犹豫地移动,但是偶尔会碰到笼子的壁,2=小鼠显示出姿势和运动异常,并且不接近笼子的所有侧面,3=小鼠显示出姿势和运动异常,并且在笼子的中心进行中等尺寸的圆形活动,4=小鼠有姿势异常,并且就地做非常小的圆形活动,5=小鼠不能在笼子里移动并且停留在中心。记录值由对动物的治疗和基因型不知情的观察者评估。
mcao后1天、3天和7天进行角落测试,以评估皮层和纹状体损伤后的感觉和运动缺陷。在笼子的顶部安装摄像机以持续5分钟记录小鼠在笼子里的活动性。将小鼠放置在两块纸板之间,每块纸板的尺寸为30cm×20cm×0.5mm,从边缘彼此30°角相互连接。一旦进入角落,小鼠通常后退,然后向左或向右转弯。中风前,小鼠不会表现出侧位偏爱。中风后具有感觉和运动缺陷的小鼠会转向没有受损的一侧(右侧)。计算并比较右转%=右转/总转弯x100。记录值由对动物的治疗和基因型不知情的观察者评估。
动物
约翰·霍普金斯医疗机构(johnshopkinsmedicalinstitutions)完全由美国实验动物护理认证协会(aaalac)认证。本研究中执行的所有研究程序都由约翰·霍普金斯医疗机构的动物护理机构和使用委员会(iacuc)按照《动物福利法》条例和公共卫生服务(phs)政策批准。所有动物研究都是以不知情方式进行的。小鼠基因型由k.n.确定。中风手术是由r.a.进行。中风手术、小鼠行为测试和数据分析后,小鼠基因型被解码。根据它们的基因型,小鼠被分为wt、ko、ko-wt、ko-e22q和ko-e22a的组。在每组内,小鼠被随机分配到包括假中风、1天后中风、3天后中风或7天后中风的亚组。
统计分析
除非另有说明,统计评估是通过两组之间的学生t-检验和单向方差分析(anova)进行的,接下来使用graphpadprism软件利用bonferroni检验在多组内进行事后比较。数据显示为平均值±sem。p<0.05被认为是显著的。定量实验以不知情方式进行。为了确保检测效果的足够能力,对所有分子生物化学研究进行至少3次独立测试,并使用来自3个不同窝的至少5只小鼠用于动物研究。
对endog、mif蛋白结构、mif突变体、mif蛋白纯度、chip测序数据和mif-aif相互作用的额外分析。
endog对parp-1依赖型细胞死亡是不可缺少的
为了证实如前所述(13,14)的,内切酶对于依赖性细胞死亡是不可缺少的,crispr/cas9系统用于从人sh-sy5y细胞中敲除endog(图12a)。发现endog的敲除未能阻断mnng诱导的依赖性细胞死亡(图12b)和大的dna片段化(图12c),证实了依赖性细胞死亡不需要endog(13,14)。
mif蛋白结构分析
核酸酶的核心pd-d/e(x)k拓扑结构由靠近双螺旋的4个β链组成(18)。两个β链彼此平行,而另外两个是反向平行的(图14c,自(18)修改)。mif先前的3-d晶体结构表明它以三聚体的形式存在(21-23)。mif的三聚体结构使得每个单体的β链与其它单体相互作用,从而形成pd-d/e(x)k结构,该结构由与2条α链相邻的4条β链组成(图1e和图14d至g)。两个β-链(β-4和β-5)平行,而另两条链(β-6和β-7)(来自相邻单体)反向平行(图14,d至g)。pd-d/e(x)k基序的拓扑结构在mif三聚体中具有的β链相对于α螺旋的朝向与已被充分表征的核酸内切酶,ecorv非常相似(图14,h至k)。重要的是,基于mif三聚体结构的pd-d/e(x)k基序在结构上类似于ii型不依赖atp的限制性内切酶,诸如ecori和ecorv,以及exoiii族嘌呤/无嘧啶(ap)核酸内切酶,诸如exoiii(图1e和图14,l至n)。此外,mif也具有与pvuii核酸内切酶相似的拓扑结构,并且在其pd-d/e(x)k基序的相同位置,其β-7链的尺寸与pvuii核酸内切酶在其pd-d/e(x)k基序中的相同位置处的β-链相似(图14o)。这些3-d建模结果综合起来表明,mif属于pd-d/e(x)k核酸酶样超家族(24,25)。
对mif核酸酶活性关键的残基的鉴定
为了鉴定对mif的核酸酶活性至关重要的氨基酸残基,pd-d/e(x)k结构域内的关键性天冬氨酸和谷氨酸残基被突变为丙氨酸。丙氨酸对谷氨酸22的取代(e22a)明显但不完全地降低mif的核酸酶活性,而在其它天冬氨酸盐和谷氨酸包括d17a、d45a、e55a、e86a、d93a和d101a处的丙氨酸取代,没有实质性影响(图15e)。mif的cxxcxxhx(n)c锌指结构域突变为c57a;c60a没有明显的效果(图15e)。由于mife22a降低了核酸酶活性,在e22周围进行了另外的保守性突变(图15,f和g)。发现mife22q没有核酸酶活性(图2d和图15,d和h),而e22d具有与野生型等同的核酸酶活性(图2d)。这些数据表明,mif第一个α-螺旋中的谷氨酸残基(e22)对其核酸酶活性至关重要,这符合先前的报道,即许多核酸外切酶-内切核酸酶-磷酸酶(eep)结构域超家族核酸酶的第一个α-螺旋中的谷氨酸高度保守,并且它是核酸酶活性的活性位点(24,25)。基于三维结构建模(图1e),可能的mifdna结合位点被突变,包括:p16a、p44a、r87q、r89q、p92a、d45q、d17q、e55q和d93q(图15h)。发现p16a或d17q阻止mif核酸酶活性(图15h)。基于mif的序列比对和三维结构建模,数据揭示p16、d17和e22位于相同的pd-d/e(x)k基序中,且每个残基的突变足以阻断mif的核酸酶活性。考虑到第一个α-螺旋中的e22跨物种高度保守的事实,并且之前已经报道过它是pd-d/e(x)k核酸酶样超家族核酸酶活性的活性位点(24,25),e22突变体在随后的研究中被聚焦。
endog和亲环素a不直接参与parp-1依赖性大dna片段化
endog和亲环素a以前被认为是aif相关的核酸酶(43-45)。脉冲场凝胶电泳表明,endog将dna切割成小片段,这与在依赖性细胞死亡中观察到的更大dna片段化模式不一致(图15d)。相反,mif以与mnng诱导的dna片段相似的模式将dna切割成大片段(图2b和图15d)(13,14)。亲环素a和aif没有明显的核酸酶活性,谷胱甘肽s-转移酶(gst)作为阴性对照(图15d)。
mif核酸酶活性独立于其氧化还原酶和互变异构酶活性
先前的研究表明mif具有氧化还原酶和互变异构酶活性(27,29,30)。使用胰岛素作为底物测量野生型和mif突变体的氧化还原酶活性,其中还原的胰岛素在野生型mif存在下显示650nm的光密度值(图16a)。e22q、e22a、c57a;c60amif突变体和互变异构酶p2gmif突变体对mif的氧化还原酶活性没有明显的影响(图16a)。mif的互变异构酶活性也被测量。e22q、e22a、c57a;c60amif突变体对mif的互变异构酶活性没有明显影响,而p2gmif突变体显著降低了mif的互变异构酶活性(图16b)。这些结果综合起来表明mif活性位点突变体e22q和e22a对mif的氧化还原酶或互变异构酶活性没有明显的影响。
纯化的mif蛋白没有外来核酸酶污染
为了证实重组mif制剂不含外来核酸酶,进行了fplc。fplc显示在约37kd分子量处的仅一个峰,与mif以三聚体形式存在一致。与三聚体结构一致地,mife22q和e22a也在37kd洗脱,表明这些突变不会明显影响mif的确认(图16c)。在fplc纯化后的蛋白质(图16d)以及未经fplc纯化的蛋白质(图15g)中,考马斯蓝染色显示只有一条条带。经过fplc纯化和没有经过fplc纯化的两种类型的蛋白都在核酸酶测定中使用,且都没有观察到明显的差异。在核酸酶测定中使用的所有这些重组mif蛋白的纯度也通过两个独立的质谱(ms)测定得到证实。通过ms鉴定的肽的大多数是mif。没有鉴定到能切割单链或双链dna的来自所有物种的已知核酸酶。因此,mif制备品是高纯度的,且不存在外来核酸酶。
mif蛋白的确认不受e22q、e22a和c57a;c60a突变的影响
研究蛋白质二级结构的常用方法,远紫外(uv)圆二色(cd)光谱显示,野生型mif由α-螺旋和β-片层的混合物组成,与之前报道的mif晶体结构一致(22)。mif突变体e22q、e22a和c57a;c60a显示出与野生型mif相似的cd光谱,表明这些突变未显著影响mif的构象(图16e)。向野生型mif或mif突变体添加mg2+时没有观察到显著变化(图16f-i)。然而,zn2+的加入促进了光谱的巨大变化,表明野生型mif蛋白在zn2+结合时结构的显著变化(图16f)。mife22q和e22a在zn2+存在下显示出与野生型mif相似的cd光谱(图16,g和h),然而zn2+加入c57a;c60a突变体没有引起cd光谱的变化,表明mif在mif的cxxcxxhx(n)c锌指结构域结合zn2+(图16i)。
近紫外cd光谱被用于进一步分析mif和mif突变体的三级结构。纯化的蛋白质具有正确折叠的三级结构,因为在近uvcd光谱中有明显的苯丙氨酸和酪氨酸的峰(图16j)。mif突变体e22q、e22a和c57a;c60a,显示出与野生型相似的近uvcd光谱,表明这些突变不会显著影响mif的三级结构(图16,j至m)。向野生型mif或mif突变体c57a;c60a添加mg2+引起三级结构的显著变化,表明mg2+的结合(图16,j和m)。e22a在mg2+存在下显示出微小变化,而e22q在近紫外cd光谱中没有显示出显著变化,表明mg2+在e22处或附近结合(图16,k和l),这符合我们的发现,即mif的核酸酶活性需要mg2+,e22q和e22a突变体可以完全或部分阻断其核酸酶活性。向野生型mif或mif突变体e22a和e22q添加zn2+导致三级结构的显著变化,表明zn2+结合,而mifc57a;c60a突变体未显示显著变化,符合zn2+与cxxcxxhx(n)c锌指结构域结合(图16,j至m)。
mif-dna结合特性的chip测序分析
因为数据显示mif具有核酸酶活性,接下来研究了用在dmso或mnng(50μm,15分钟)处理的hela细胞中mif是否与dna结合。处理后5小时,细胞被交联,并准备进行chip测定,接下来是深度测序。测试并确认剪切的基因组dna的质量和使用mif抗体的chip的特异性(图17,a和b)。排除dmso处理的样品中的重叠峰后,在mnng处理后,总映射读段的0.1%显示出mif峰(图17c)。mnng处理后,mif优先结合启动子和5’utr区域(图17d)。mif在基因组上的富集的代表性igv可视化图以两种不同的窗口尺寸示出(250kb(图17e)和50kb(图17f))。mif峰之间的平均距离间隔约为15kb至60kb,这与通过脉冲凝胶电泳观察到的依赖性细胞死亡期间的dna片段的尺寸一致。chip-qpcr进一步证实在mnng处理后,mif结合55101、66005、65892、36229、46426和62750处的峰区域,但是它不结合非峰区域(图17g)。
映射aif-mif相互作用
为了证实mif是aif相互作用蛋白,进行了gst拉下实验。野生型gst-aif拉下了内源性mif,而野生型gst-mif拉下了内源性aif(图4a和图21,a至d)。结合mif的结构域用各种gst标记的aif结构域通过gst拉下进一步明确(图21a)。mif结合gst-c2baif(aa551-590)和gstc2eaif(aa571-612)(图21a和b)。mif不与gst-c2aif、gst-c2caif、gst-c2daif或gst结合,表明mif在所用的实验条件下不与gst非特异性结合(图21,a和b)。将aa567-592突变成聚丙氨酸(aifm567-592)或从全长中删除aa567-592(aifδ567-592)完全废除了mif和aif的结合(图21c),表明mif在aa567-592处与aif结合。
先前对mif的结晶研究通过3-d建模证明,mif在结构上类似于pd-d/e(x)k核酸酶。含有pd-d/e(x)k结构域的蛋白质属于核酸酶样超家族((对于综述,参见(24,25)),进一步提供证据证明mif是核酸酶。这个核酸酶超家族包含来自所有生命领域的核酸酶。这些蛋白质中的大多数属于原核生物体,但这个结构域被多种脊椎动物核酸酶(24,25)包含。mif中的pd-d/e(x)k结构域在脊椎动物中高度保守。mif第一个α-螺旋中的谷氨酸残基(e22)对其核酸酶活性至关重要,这符合先前的报道,即许多核酸外切酶-核酸内切酶-磷酸酶(eep)结构域超家族核酸酶的第一个α-螺旋中的该谷氨酸高度保守,且它是核酸酶活性的活性位点(24,25)。
核心pd-d/e(x)k结构由紧挨着两个α-螺旋的4个β-链组成。两条β-链相互平行,而另外两条则反向平行(18,24)。有趣的是,具有伪2-重对称性的mif单体不包含核心pd-d/e(x)k结构,因为该mif单体具有紧挨着2个α-螺旋的4个β-链,并且分离的单体中的β-链的朝向不符合pd-d/e(x)k拓扑结构的要求(22)。然而,基于具有3-重对称性的mif三聚体的结构-活性分析表明,每种单体的β-链与其它单体的相互作用形成由紧挨着2条α链的4条β链组成的mifpd-d/e(x)k结构(22)。两条β-链是平行的(β-4和β-5),而另外两条β-链(β-6和β-7)(来自相邻单体)是反向平行的。这种拓扑结构精巧地支持mif的核酸酶活性需要三聚体的观点,因为单体不支持所需的拓扑结构,并且与mif作为三聚体存在一致。mif三聚体的这种拓扑结构将含有活性残基谷氨酸22的α-1螺旋置于β-链旁,但这并不是前所未有的(18,24)。例如,ecorv,一种已被充分表征的核酸内切酶具有pd-d/e(x)k基序,其β链相对于α螺旋的朝向不同于通常的pd-d/e(x)k基序,而类似于mif的朝向。mif与ecorv拓扑结构的相似性表明mif与该充分表征的限制性内切酶极为相似。事实上,来自第一个α-螺旋的核心α-螺旋(通常地)谷氨酸的保守型酸性残基至少在pd-d/e(x)k家族的亚组中经常有助于形成活性位点,这类似于对于mif所报道的(24)。基于mif三聚体结构的pd-d/e(x)k基序也具有与核酸酶exoiii、ecori和ecorv非常相似的结构。此外,mif具有类似于pvuii核酸内切酶的拓扑结构,并且mif的β-7链的尺寸与pvuii核酸内切酶在其pd-d/e(x)k基序中的相同位置处的β-链相似(46)。根据结构分析,mif应该被归类为核酸酶。
mif具有各种多效性性作用。它作为非经典的分泌的细胞因子发挥功能,其中它在癌症生物学、免疫反应和炎症中发挥重要作用(16,17)。mif在细胞应激和凋亡中也有重要作用(47,48)。mif的敲除也被证明在局部缺血中具有神经保护作用(49)。结果证实,mif的敲除针对局部缺血进行保护,并显示mif通过其与aif的结合以及与其作为paan的功能相一致的核酸酶活性促进了局部缺血中的神经元损伤。mif还具有硫醇-蛋白质氧化还原酶活性和互变异构酶活性。emsa和chip都表明mif与dna结合。尽管mif结合高度相关家族的重叠序列,但是结构-活性实验表明基于其结构mif优先结合ssdna,并且它不太依赖于序列特异性。mif结合具有茎环结构的ssdna的5’未配对碱基,并且具有3’核酸外切酶和核酸内切酶活性二者,并切割茎环ssdna3’末端的未配对碱基。三维计算建模显示,催化性的e22接近ssdna的模拟结合结构域。如此处表明的,mif的核酸酶活性与它的氧化还原酶和互变异构酶活性是能够明显分开的。
先前的鉴定aif相关核酸酶的尝试最初集中在endog,一种线粒体基质蛋白(43)。在秀丽隐杆线虫中,cps-6,哺乳动物endog的同源物,是wah-1(aif同源物)的细胞死亡诱导特性所必需的。然而,在哺乳动物中,endog在包括parp-1依赖性缺血性细胞死亡的许多细胞死亡模型中是不可缺少的(13,14,50)。重要的是,,在大脑中动脉闭塞后,在endog敲除小鼠中存在与野生型对照相比等同量的dna片段化(13)。与这些观察结果一致,证实了endog的敲除不能阻断mnng诱导的依赖性细胞死亡和大dna片段化,证实了endog对于依赖性细胞死亡不是必要的(13,14)。相反的,mif的敲除型、mif核酸酶缺陷突变体和mifaif结合缺陷突变体阻止了parp-1活化后的体外和体内细胞死亡和大dna片段化。因此,endog不是哺乳动物中的paan,而mif符合这一角色的所有标准。最近,教导了aif与亲环素a产生活性dna降解复合物(45),但该复合物中的核酸酶尚未被鉴定。
表1.用于核酸酶测定的mif底物的概述。(y:是;n:否)
尽管已经参考上述实施例描述了本发明,但是应当理解,修改和变化包含在本发明的精神和范围内。因此,本发明仅由随后的权利要求书限制。
参考文献
以下参考文献各自被依赖并以其整体并入本文。
1.p.bai,biologyofpoly(adp-ribose)polymerases:thefactotumsofcellmaintenance.molcell58,947(jun18,2015).
2.a.a.fatokun,v.l.dawson,t.m.dawson,parthanatos:mitochondrial-linkedmechanismsandtherapeuticopportunities.brjpharmacol171,2000(apr,2014).
3.y.wang,v.l.dawson,t.m.dawson,poly(adp-ribose)signalstomitochondrialaif:akeyeventinparthanatos.expneurol218,193(aug,2009).
4.p.pacher,c.szabo,roleoftheperoxynitrite-poly(adp-ribose)polymerasepathwayinhumandisease.amjpathol173,2(jul,2008).
5.c.szabo,v.l.dawson,roleofpoly(adp-ribose)synthetaseininflammationandischaemia-reperfusion.trendspharmacolsci19,287(jul,1998).
6.s.martire,l.mosca,m.d′erme,parp-linvolvementinneurodegeneration:afocusonaizheimer′sandparkinson′sdiseases.mechageingdev146-148c,53(mar,2015).
7.l.virag,a.robaszkiewicz,j.m.rodriguez-vargas,f.j.oliver,poly(adp-ribose)signalingincelldeath.molaspectsmed34,1153(dec,2013).
8.h.wangetal.,apoptosis-inducingfactorsubstitutesforcaspaseexecutionersinnmda-triggeredexcitotoxicneuronaldeath.jneurosci24,10963(dec1,2004).
9.y.wangetal.,poly(adp-ribose)(par)bindingtoapoptosis-inducingfactoriscriticalforparpolymerase-1-dependentcelldeath(parthanatos).scisignal4,ra20(2011).
10.s.w.yuetal.,apoptosis-inducingfactormediatespoly(adp-ribose)(par)polymer-inducedcelldeath.procnatlacadsciusa103,18314(nov28,2006).
11.s.w.yuetal.,mediationofpoly(adp-ribose)polymerase-1-dependentcelldeathbyapoptosis-inducingfactor.science297,259(jul12,2002).
12.x.wang,c.yang,j.chai,y.shi,d.xue,mechanismsofaif-mediatedapoptoticdnadegradationincaenorhabditiselegans.science298,1587(nov22,2002).
13.z.xuetal.,endonucleasegdoesnotplayanobligatoryroleinpoly(adp-ribose)polymerase-dependentcelldeathaftertransientfocalcerebralischemia.amjphysiolregulintegrcompphysiol299,r215(jul,2010).
14.k.k.david,m.sasaki,s.w.yu,t.m.dawson,v.l.dawson,endogisdispensableinembryogenesisandapoptosis.celldeathdiffer13,1147(jul,2006).
15.s.huetal.,profilingthehumanprotein-dnainteractomerevealserk2asatranscriptionalrepressorofinterferonsignaling.cell139,610(oct30,2009).
16.t.calandra,t.roger,macrophagemigrationinhibitoryfactor:aregulatorofinnateimmunity.natrevimmunol3,791(oct,2003).
17.m.merk,r.a.mitchell,s.endres,r.bucala,d-dopachrometautomerase(d-dtormif-2):doublingthemifcytokinefamily.cytokine59,10(jul,2012).
18.j.kosinski,m.feder,j.m.bujnicki,thepd-(d/e)xksuperfamilyrevisited:identificationofnewmembersamongproteinsinvolvedindnametabolismandfunctionalpredictionsfordomainsof(hitherto)unknownfunction.bmcbioinformatics6,172(2005).
19.c.mackayetal.,identificationofkiaa1018/fan1,adnarepairnucleaserecruitedtodnadamagebymonoubiquitinatedfancd2.cell142,65(jul9,2010).
20.k.kratzetal.,deficiencyoffancd2-associatednucleasekiaa1018/fan1sensitizescellstointerstrandcrosslinkingagents.cell142,77(ju19,2010).
21.h.sugimoto,m.suzuki,a.nakagawa,i.tanaka,j.nishihira,crystalstructureofmacrophagemigrationinhibitoryfactorfromhumanlymphocyteat2.1aresolution.febslett389,145(jul1,1996).
22.h.w.sun,j.bernhagen,r.bucala,e.lolis,crystalstructureat2.6-aresolutionofhumanmacrophagemigrationinhibitoryfactor.procnatlacadsciusa93,5191(may28,1996).
23.m.suzukietal.,crystalstructureofthemacrophagemigrationinhibitoryfactorfromratliver.natstructbiol3,259(mar,1996).
24.k.steczkiewicz,a.muszewska,l.knizewski,l.rychlewski,k.ginalski,sequence,structureandfunctionaldiversityofpd-(d/e)xkphosphodiesterasesuperfamily.nucleicacidsres40,7016(aug,2012).
25.m.laganeckas,m.margelevicius,c.venclovas,identificationofnewhomologsofpd-(d/e)xknucleasesbysupportvectormachinestrainedondataderivedfromprofile-profilealignments.nucleicacidsres39,1187(mar,2011).
26.v.pingoudetal.,onthedivalentmetaliondependenceofdnacleavagebyrestrictionendonucleasesoftheecorifamily.jmolbiol393,140(oct16,2009).
27.j.b.lubetskyetal.,thetautomeraseactivesiteofmacrophagemigrationinhibitoryfactorisapotentialtargetfordiscoveryofnovelanti-inflammatoryagents.jbiolchem277,24976(jul12,2002).
28.g.fingerle-rowsonetal.,atautomerase-nullmacrophagemigration-inhibitoryfactor(mif)geneknock-inmousemodelrevealsthatproteininteractionsandnotenzymaticactivitymediatemif-dependentgrowthregulation.molcellbiol29,1922(apr,2009).
29.e.rosengrenetal.,themacrophagemigrationinhibitoryfactormifisaphenylpyruvatetautomerase.febslett417,85(nov3,1997).
30.a.kudrinetal.,humanmacrophagemigrationinhibitoryfactor:aprovenimmunomodulatorycytokine?jbiolchem281,29641(oct6,2006).
31.g.zhao,b.zhao,z.tong,r.mu,y.guan,effectsof2′-o-methylnucleotidesubstitutiononecoriendonucleasecleavageactivities.plosone8,e77111(2013).
32.m.bozzaetal.,targeteddisruptionofmigrationinhibitoryfactorgenerevealsitscriticalroleinsepsis.jexpmed189,341(jan18,1999).
33.s.a.andrabietal.,idunaprotectsthebrainfromglutamateexcitotoxicityandstrokebyinterferingwithpoly(adp-ribose)polymer-inducedcelldeath.natmed17,692(jun,2011).
34.j.j.glascocketal.,deliveryoftherapeuticagentsthroughintracerebroventricular(icv)andintravenous(iv)injectioninmice.jvisexp,(2011).
35.y.chenetal.,systematicevaluationoffactorsinfluencingchip-seqfidelity.natmethods9,609(jun,2012).
36.j.feng,t.liu,b.qin,y.zhang,x.s.liu,identifyingchip-seqenrichmentusingmacs.natprotoc7,1728(sep,2012).
37.h.r.drewetal.,structureofab-dnadodecamer:conformationanddynamics.procnatlacadsciusa78,2179(apr,1981).
38.t.masuda,y.ito,t.terada,t.shibata,t.mikawa,anon-canonicaldnastructureenableshomologousrecombinationinvariousgeneticsystems.jbiolchem284,30230(oct30,2009).
39.a.w.ghoorah,m.d.devignes,m.smail-tabbone,d.w.ritchie,proteindockingusingcase-basedreasoning.proteins,(oct7,2013).
40.g.macindoe,l.mavridis,v.venkatraman,m.d.devignes,d.w.ritchie,hexserver:anfft-basedproteindockingserverpoweredbygraphicsprocessors.nucleicacidsres38,w445(jul,2010).
41.p.p.reddyetal.,moleculardynamicsoftheneuronalef-handca2+-sensorcaldendrin.plosone9,e103186(2014).
42.k.bendratetal.,biochemicalandmutationalinvestigationsoftheenzymaticactivityofmacrophagemigrationinhibitoryfactor.biochemistry36,15356(dec9,1997).
43.x.wang,c.yang,j.chai,y.shi,d.xue,mechanismsofaif-mediatedapoptoticdnadegradationincaenorhabditiselegans.science298,1587(nov22,2002).
44.c.candeetal.,aifandcyclophilinacooperateinapoptosis-associatedchromatinolysis.oncogene23,1514(feb26,2004).
45.c.artusetal.,aifpromoteschromatinolysisandcaspase-independentprogrammednecrosisbyinteractingwithhistoneh2ax.emboj29,1585(may5,2010).
46.a.athanasiadisetal.,crystalstructureofpvuiiendonucleaserevealsextensivestructuralhomologiestoecorv.natstructbiol1,469(jul,1994).
47.j.d.hudsonetal.,aproinflammatorycytokineinhibitsp53tumorsuppressoractivity.jexpmed190,1375(nov15,1999).
48.r.kleemannetal.,intracellularactionofthecytokinemiftomodulateap-1activityandthecellcyclethroughjab1.nature408,211(nov9,2000).
49.a.r.inacio,k.ruscher,l.leng,r.bucala,t.deierborg,macrophagemigrationinhibitoryfactorpromotescelldeathandaggravatesneurologicdeficitsafterexperimentalstroke.jcerebbloodflowmetab31,1093(apr,2011).
50.r.a.irvineetal.,generationandcharacterizationofendonucleasegnullmice.molcellbiol25,294(jan,2005).
51.chipseqdata(gse65110)
http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?token=atktuskchzmbvgz&acc=gse65110.
52.chipseq.bamfiles:
https://drive.google.com/folderview?id=0b5rxnsjjvi-9fjjis1fxbu9vmofurizbsc0yykswz3njyzzmvvbiyuoysg5yovrzqtzkw8&usp=sharing.
- 还没有人留言评论。精彩留言会获得点赞!
- SPNP‑I在制备Nav1.7通道工具试剂中的应用的制造方法与工艺
- SPNP‑I在制备Nav1.2通道工具试剂中的应用的制造方法与工艺
- SPNP‑IX在制备Nav1.3通道抑制剂中的应用的制造方法与工艺
- SPNP‑IX在制备Kv4.2通道工具试剂中的应用的制造方法与工艺
- SPNP‑IX在制备Kv4.3通道工具试剂中的应用的制造方法与工艺
- SPNP‑27在制备Nav1.5通道抑制剂中的应用的制造方法与工艺
- SPNP‑27在制备Nav1.3通道抑制剂中的应用的制造方法与工艺
- 一种多肽在制备Nav1.3钠通道工具试剂中的应用的制造方法与工艺
- SPKP‑XI在制备Kv4.2通道抑制剂中的应用的制造方法与工艺
- SGLT‑2糖尿病抑制剂及其中间体的制备方法与流程