一种基于深度神经网络的蛋白质接触图预测方法与流程
本发明涉及蛋白质图谱预测的技术领域,尤其涉及一种基于深度神经网络的蛋白质接触图预测方法。
背景技术:
蛋白质作为一种大分子化合物,是生命体的重要组成部分,并且参与了多种多样的生命活动。对蛋白质的研究兼具巨大的理论及现实意义。蛋白质的种类数量巨大,具有的功能各不相同,而决定蛋白质功能的蛋白质结构不仅有氨基酸脱水缩合后组成肽链,一条或多条肽链折叠后成为各种蛋白质独特的空间结构,还包括蛋白质序列,即20种常见氨基酸的排列组合。序列与结构之间的数量缺口使得学界开始尝试使用计算方法预测蛋白质结构。目前通过序列预测蛋白质结构的方法大致分为三类:同源建模法、折叠识别法和基于第一性原理的方法,然而蛋白质结构的复杂导致对其的预测十分困难,以上这些方法单独使用或存在各种各样的缺陷,难以取得令人满意的结果。
蛋白质接触图预测算法可以分为基于协同进化的统计学方法、基于传统机器学习的方法、基于深度学习的方法。目前,基于深度学习的方法得到的预测精度更好。但蛋白质残基接触预测的主要目的是辅助三级结构预测。接触预测结果对结构预测帮助的大小不仅仅取决于预测精度,还受到预测残基接触位置的影响。实际上,一个长距离的接触相对一个短距离能够给一个初步预测蛋白质结构带来更多的约束,也就对结构预测精度影响更大。此外,离散的接触预测结果比集中的预测结果更有效也是同理。所以对于预测结果好坏的衡量指标应该是多方面的,即包括精度与离散度。作为一个分类问题,蛋白质接触预测任务存在一定特殊性:一、定义蛋白质接触阈值不具备生物学意义上的特殊性。蛋白质结构测定的残原子位置不完全准确。二、蛋白质接触预测的主要作用是用来辅助接触图预测,在其他分类问题上使用的平均准确率、精确率、auc等并不适合用来衡量预测结果的优劣。三、正负样本不平衡。这些特殊性使得常用的机器学习模型在该问题上可能存在一些改进空间。然而学界其他预测器并没有针对其做出改进。
技术实现要素:
本部分的目的在于概述本发明的实施例的一些方面以及简要介绍一些较佳实施例。在本部分以及本申请的说明书摘要和发明名称中可能会做些简化或省略以避免使本部分、说明书摘要和发明名称的目的模糊,而这种简化或省略不能用于限制本发明的范围。
鉴于上述现有存在的问题,提出了本发明。
因此,本发明解决的技术问题是:提出一种基于深度神经网络的蛋白质接触图预测方法,能够在蛋白质接触图预测方面得到更高的预测精度。
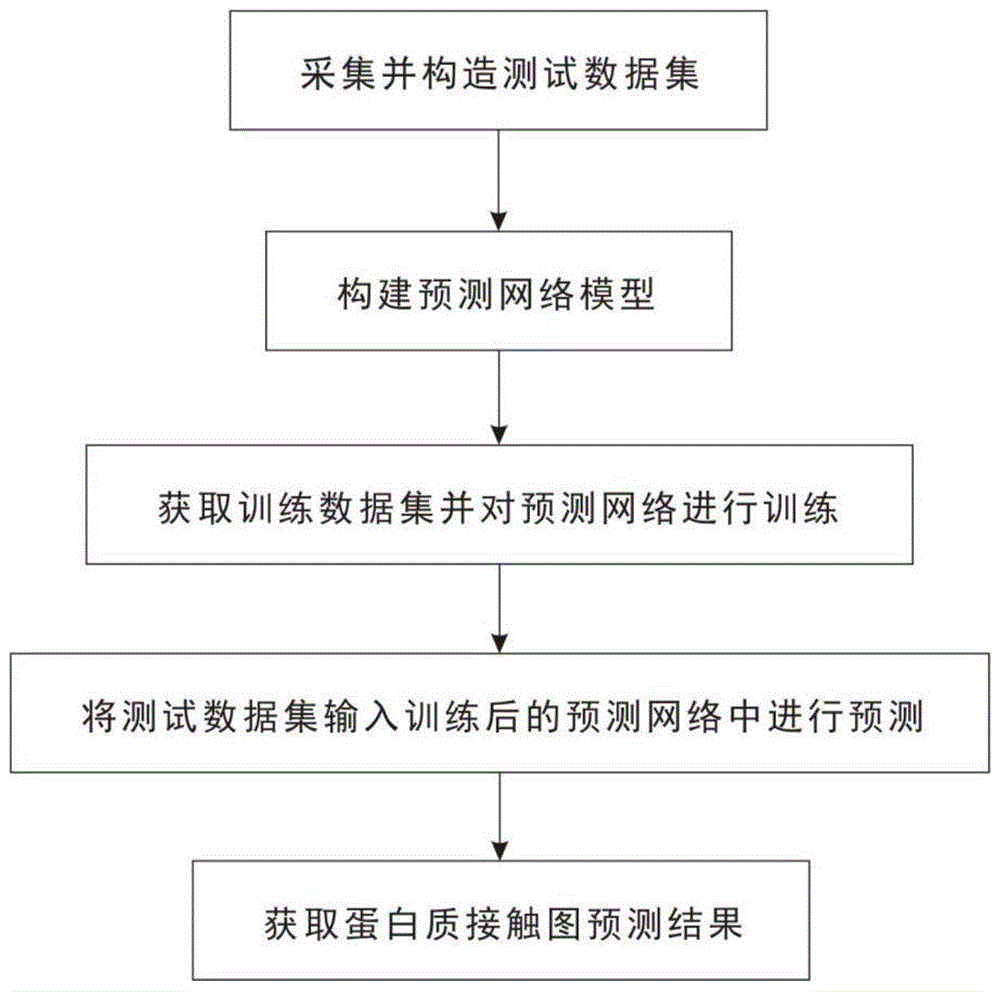
为解决上述技术问题,本发明提供如下技术方案:一种基于深度神经网络的蛋白质接触图预测方法,包括,采集并构造测试数据集;构建预测网络模型;获取训练数据集并对预测网络进行训练;将测试数据集输入训练后的预测网络中进行预测;获取蛋白质接触图预测结果。
作为本发明所述的基于深度神经网络的蛋白质接触图预测方法的一种优选方案,其中:所述采集数据还包括对蛋白质进行测序,得到蛋白质序列。
所作为本发明所述的基于深度神经网络的蛋白质接触图预测方法的一种优选方案,其中:所述预测网络模型包括卷积层、残差块和激活函数,且预测网络模型中的卷积层包括3×3的卷积核。
所作为本发明所述的基于深度神经网络的蛋白质接触图预测方法的一种优选方案,其中:所述残差块还包括卷积层、归一化层和激活函数,且卷积层为96个3×3的卷积核。
所作为本发明所述的基于深度神经网络的蛋白质接触图预测方法的一种优选方案,其中:所述预测网络模型的激活函数包括sigmoid函数和elu函数,sigmoid函数计算公式为,
所作为本发明所述的基于深度神经网络的蛋白质接触图预测方法的一种优选方案,其中:所述残差块的激活函数包括elu函数,其计算公式为,
所作为本发明所述的基于深度神经网络的蛋白质接触图预测方法的一种优选方案,其中:所述训练数据集包括蛋白家族数据库pfam中的蛋白质、casp11测试蛋白质、cameo困难目标和跨膜蛋白。
所作为本发明所述的基于深度神经网络的蛋白质接触图预测方法的一种优选方案,其中:所述训练预测网络还包括,初始化预测网络模型和训练数据集;将训练数据集分批输入至预测网络模型中;计算损失函数的值并优化网络;重复训练直至达到预测精度不再增加,输出训练后的预测网络模型。
所作为本发明所述的基于深度神经网络的蛋白质接触图预测方法的一种优选方案,其中:所述训练中的损失函数为焦点损失函数,其计算公式为,
其中,jfocalloss的值越小,代表训练中过程中预测网络的预测结果精度越高。
本发明的有益效果:本发明提出的基于深度神经网络的蛋白质接触图预测方法,能够弥补传统深度学习方法中交叉熵损失函数与蛋白质接触图预测之间的不和谐,得到的预测网络模型能够对蛋白质接触图进行更加准确的预测,具有很好的实用性。
附图说明
为了更清楚地说明本发明实施例的技术方案,下面将对实施例描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动性的前提下,还可以根据这些附图获得其它的附图。其中:
图1为本发明所述基于深度神经网络的蛋白质接触图预测方法的整体流程示意图;
图2为本发明所述基于深度神经网络的蛋白质接触图预测方法中预测网络的结构示意图;
图3为本发明中作为测试数据输入预测网络中的蛋白质序列的结构示意图;
图4为本发明中输入一种pdb目标进行预测后预测网络输出的蛋白质接触图。
具体实施方式
为使本发明的上述目的、特征和优点能够更加明显易懂,下面结合说明书附图对本发明的具体实施方式做详细的说明,显然所描述的实施例是本发明的一部分实施例,而不是全部实施例。基于本发明中的实施例,本领域普通人员在没有做出创造性劳动前提下所获得的所有其他实施例,都应当属于本发明的保护的范围。
在下面的描述中阐述了很多具体细节以便于充分理解本发明,但是本发明还可以采用其他不同于在此描述的其它方式来实施,本领域技术人员可以在不违背本发明内涵的情况下做类似推广,因此本发明不受下面公开的具体实施例的限制。
其次,此处所称的“一个实施例”或“实施例”是指可包含于本发明至少一个实现方式中的特定特征、结构或特性。在本说明书中不同地方出现的“在一个实施例中”并非均指同一个实施例,也不是单独的或选择性的与其他实施例互相排斥的实施例。
本发明结合示意图进行详细描述,在详述本发明实施例时,为便于说明,表示器件结构的剖面图会不依一般比例作局部放大,而且所述示意图只是示例,其在此不应限制本发明保护的范围。此外,在实际制作中应包含长度、宽度及深度的三维空间尺寸。
同时在本发明的描述中,需要说明的是,术语中的“上、下、内和外”等指示的方位或位置关系为基于附图所示的方位或位置关系,仅是为了便于描述本发明和简化描述,而不是指示或暗示所指的装置或元件必须具有特定的方位、以特定的方位构造和操作,因此不能理解为对本发明的限制。此外,术语“第一、第二或第三”仅用于描述目的,而不能理解为指示或暗示相对重要性。
本发明中除非另有明确的规定和限定,术语“安装、相连、连接”应做广义理解,例如:可以是固定连接、可拆卸连接或一体式连接;同样可以是机械连接、电连接或直接连接,也可以通过中间媒介间接相连,也可以是两个元件内部的连通。对于本领域的普通技术人员而言,可以具体情况理解上述术语在本发明中的具体含义。
实施例1
参照图1的示意,示意为本实施例提出的一种基于深度神经网络的蛋白质接触图预测方法的整体流程示意图,具体包括以下步骤,
s1:采集并构造测试数据集;所述采集数据还包括对蛋白质进行测序,得到蛋白质序列。蛋白质序列可以通过蛋白质测序仪进行获取,获取到的蛋白质序列用于后续输入预测网络中进行蛋白质接触图的预测。
s2:构建预测网络模型;
具体的,参照图2的示意,所述预测网络模型包括卷积层、残差块和激活函数,且预测网络模型中的卷积层包括3×3的卷积核,分别是96个3×3的卷积核构成的卷积层和1个3×3的卷积核构成的卷积层。
预测网络的精度随残差块的增加而逐渐缓慢地提高,且随着模型宽度的增大,即每层卷积核的个数增大,网络的精度也呈上升趋势。但是残差块或模型宽度增加的同时,网络模型的计算效率会变低,以及需要更多的计算资源,因此最终构建的预测网络模型中残差块的选择是基于计算速度和精度的折中,本实施例中的预测网络包括10个残差块,
其中,所述预测网络模型的激活函数包括sigmoid函数和elu函数,sigmoid函数计算公式为,
进一步的,参照图2,所述残差块还包括卷积层、归一化层和激活函数,且所述残差块的激活函数包括elu函数,其计算公式为,
s3:获取训练数据集并对预测网络进行训练;
其中,训练数据集包括蛋白家族数据库pfam中的蛋白质、casp11测试蛋白质、cameo困难目标和跨膜蛋白。训练数据可以通过公开数据集pdb25进行获取,要求其任意两条蛋白质序列的相似度小于25%,此外还需要将满足以下条件的蛋白质也剔除出训练数据集,包括长度小于28或大于700的蛋白质;分辨率小于
具体的,所述训练预测网络还包括以下步骤,
初始化预测网络模型和训练数据集;
将训练数据集分批输入至预测网络模型中;
计算损失函数的值并优化网络;所述训练中的损失函数为焦点损失函数,其计算公式为,
其中,jfocalloss的值越小,代表训练中过程中预测网络的预测结果精度越高。
重复训练直至达到预测精度不再增加,输出训练后的预测网络模型。若训练后的预测结果精度相比于之前仍有增加,则说明还应继续进行训练以提高预测精度;若预测精度不再增加,则说明已经经过充分训练,再训练不会增加预测网络的预测精度,反而可能会降低其预测精度,因此可以输出预测网络。此外训练过程中还添加了l2正则项以抑制过拟合。
s4:将测试数据集输入训练后的预测网络中进行预测;
输入特征为l×l×487,随后该特征后输入给一个深度全卷积神经网络。首先经过一个包含96个卷积核的3×3卷积层和elu激活函数。因为高维的输入特征需要大量的运算,该层可以起到降维的作用。随后是30个残差块,输入经过卷积运算后被批标准化、激活函数之后是第二个卷积和批标准化,和进入该残基块时的输入相加后经过激活函数得到输出结果。其中卷积层包括96个3×3的卷积核,三十个残差块过后,经过sigmoid函数给出预测的蛋白质接触图。
s5:获取蛋白质接触图预测结果。
参照图3~4的示意,以一个蛋白质作为测试数据并将其输入至预测网络中进行预测,pdb是存放蛋白质数据的国际公开数据库,其中的每个蛋白质都有一个pdbid,本实施例中输入的蛋白质pdbid为3hw9a,图3为该蛋白质的实际结构示意图。经过预测网络的处理,输出预测结果的蛋白质接触图可参照图4的示意,所标注出来的为topl/2的预测。
具体的,由于图3表示了输入蛋白质的真实结构,因此必定可以得到其真实的接触图,而图4中的点表示准确预测的接触,叉表示错误预测的接触,可以看出,预测网络准确预测了大部分接触,在该目标上的topl/2精度和topl/5精度分别为92.3%95.5%。此外,由于出现在多个不同位置的接触能对结构预测提供有效的信息,反之如果预测结果都被局限在小范围内,帮助则不大。而图4表示的预测结果呈现高离散度,也就代表着预测网络能够为结构预测提供有效的信息,具有较大的实用价值。
因此,对于给定的蛋白质输入序列,预测网络能够输出对应的蛋白质接触图,预测出的蛋白质接触图可以后续辅助进行蛋白质三维结构的预测。
场景一:
为了验证本实施例所述基于深度神经网络的蛋白质接触图预测方法在实际应用中相比于传统方法的优势,进行了如下的对比实验。
首先,在传统的二分类预测模型以及其他接触预测模型中,通常使用交叉熵损失函数,而本实施例中训练使用的损失函数为focalloss,为了对比二者带来的差异,使用的训练数据集所述的6767条蛋白质数据,在500条蛋白质构成的数据集上进行比较,获取方式参照训练数据集的获取。训练数据集来自pdb25公开数据集,并且为减小随机性带来的差异,例如随机初始化网络参数以及gpu运算过程中存在的随机性,结果皆为重新训练模型三次后取得的平均数,实验结果如下表1所示,
表1:不同损失函数训练得到的网络模型预测精度对比表
可以看出,实验结果说明了使用focalloss作为训练损失函数,能够改进对于蛋白质接触预测的准确性。
进一步的,为了验证本实施例所述基于深度神经网络的蛋白质接触图预测方法中构建的预测网络,相比于传统预测方法在预测效果上的优势,进行如下的实验,实验中除本实施例所述方法的构建的预测网络模型,还包括了evfold、psicov、ccmpred、plmdca、gremlin和metapsicov预测器,其中,其中evfold、psicov、ccmpred、plmdca和gremlin使用的是如互信息、协方差矩阵等统计学算法从多序列联配出发来进行预测,metapsicov综合使用了机器学习综合多种特征,使用的是浅层神经网络。
实验中,使用的数据集包括150个pfam家族的蛋白质、105个casp11的测试蛋白质、76个cameo困难目标和398个跨膜蛋白。其中pfam数据集包含来自不同家族的蛋白质,所以在该数据集上可以测试预测器对来自不同家族蛋白质的预测精度。casp11数据集是包含在国际蛋白质预测竞赛中的公开自由建模目标,这些目标缺乏同源蛋白质来进行同源建模或折叠识别。所以在该数据集上可以评估预测器对这类自由建模目标的预测精度。在cameo数据集同样包括一些困难目标。跨膜蛋白是蛋白质中较为重要的一类,所以本实验中也分出一个数据集进行测试。经过实验,得到的测试结果如下表2~5所示,
表2:在150个pfam目标上的预测结果对比表
表3:在105个casp11目标上的预测结果对比表
表4:在76个cameo目标上的预测结果对比表
表5:在398个跨膜蛋白上的预测结果对比表
根据表2~5的对比结果可以看出,本实施例所述基于深度神经网络的蛋白质接触图预测方法相比于传统的预测方法,预测精度有了明显的提升,基于序列的蛋白质接触图预测这一任务输入与输出之间存在的高阶复杂关系在拟合能力较强的机器学习模型的帮助下能够取得较好的结果。此外可以看出,由各个数据集之间所获得的精度存在差别,特别在150个pfam家族的蛋白质上取得较高精度,这是由于这些蛋白质本身在数据集中存在较多同源蛋白;而在76个cameo困难目标和398个跨膜蛋白上预测精度较低,是由于这些蛋白本身同源性低。本实施例中提出的方法对于跨膜蛋白的结构预测来说,如果在仅仅包含该类蛋白的数据集上训练将取得较好的预测结果。
应当认识到,本发明的实施例可以由计算机硬件、硬件和软件的组合、或者通过存储在非暂时性计算机可读存储器中的计算机指令来实现或实施。所述方法可以使用标准编程技术-包括配置有计算机程序的非暂时性计算机可读存储介质在计算机程序中实现,其中如此配置的存储介质使得计算机以特定和预定义的方式操作——根据在具体实施例中描述的方法和附图。每个程序可以以高级过程或面向对象的编程语言来实现以与计算机系统通信。然而,若需要,该程序可以以汇编或机器语言实现。在任何情况下,该语言可以是编译或解释的语言。此外,为此目的该程序能够在编程的专用集成电路上运行。
此外,可按任何合适的顺序来执行本文描述的过程的操作,除非本文另外指示或以其他方式明显地与上下文矛盾。本文描述的过程(或变型和/或其组合)可在配置有可执行指令的一个或多个计算机系统的控制下执行,并且可作为共同地在一个或多个处理器上执行的代码(例如,可执行指令、一个或多个计算机程序或一个或多个应用)、由硬件或其组合来实现。所述计算机程序包括可由一个或多个处理器执行的多个指令。
进一步,所述方法可以在可操作地连接至合适的任何类型的计算平台中实现,包括但不限于个人电脑、迷你计算机、主框架、工作站、网络或分布式计算环境、单独的或集成的计算机平台、或者与带电粒子工具或其它成像装置通信等等。本发明的各方面可以以存储在非暂时性存储介质或设备上的机器可读代码来实现,无论是可移动的还是集成至计算平台,如硬盘、光学读取和/或写入存储介质、ram、rom等,使得其可由可编程计算机读取,当存储介质或设备由计算机读取时可用于配置和操作计算机以执行在此所描述的过程。此外,机器可读代码,或其部分可以通过有线或无线网络传输。当此类媒体包括结合微处理器或其他数据处理器实现上文所述步骤的指令或程序时,本文所述的发明包括这些和其他不同类型的非暂时性计算机可读存储介质。当根据本发明所述的方法和技术编程时,本发明还包括计算机本身。计算机程序能够应用于输入数据以执行本文所述的功能,从而转换输入数据以生成存储至非易失性存储器的输出数据。输出信息还可以应用于一个或多个输出设备如显示器。在本发明优选的实施例中,转换的数据表示物理和有形的对象,包括显示器上产生的物理和有形对象的特定视觉描绘。
如在本申请所使用的,术语“组件”、“模块”、“系统”等等旨在指代计算机相关实体,该计算机相关实体可以是硬件、固件、硬件和软件的结合、软件或者运行中的软件。例如,组件可以是,但不限于是:在处理器上运行的处理、处理器、对象、可执行文件、执行中的线程、程序和/或计算机。作为示例,在计算设备上运行的应用和该计算设备都可以是组件。一个或多个组件可以存在于执行中的过程和/或线程中,并且组件可以位于一个计算机中以及/或者分布在两个或更多个计算机之间。此外,这些组件能够从在其上具有各种数据结构的各种计算机可读介质中执行。这些组件可以通过诸如根据具有一个或多个数据分组(例如,来自一个组件的数据,该组件与本地系统、分布式系统中的另一个组件进行交互和/或以信号的方式通过诸如互联网之类的网络与其它系统进行交互)的信号,以本地和/或远程过程的方式进行通信。
应说明的是,以上实施例仅用以说明本发明的技术方案而非限制,尽管参照较佳实施例对本发明进行了详细说明,本领域的普通技术人员应当理解,可以对本发明的技术方案进行修改或者等同替换,而不脱离本发明技术方案的精神和范围,其均应涵盖在本发明的权利要求范围当中。
- 还没有人留言评论。精彩留言会获得点赞!