模块标记物挖掘方法、装置、计算机设备及存储介质与流程
本发明涉及一种模块标记物挖掘方法、装置、计算机设备及存储介质,属于模块标记物的挖掘领域。
背景技术:
复杂疾病是受多种遗传因子控制的,基于生物学和医学相关理论的推测,认为该类疾病的发病是受多个基因影响的,但每个基因的作用都很微弱,无主效应基因,称该现象为“微效性”,为找到这些微效基因,目前对于该类疾病的模块标物挖掘算法主要集中在基因层面,即mrna层面。
然而,此类分析没有考虑其他因素对于基因表达变化的影响,只是单一层面的分析,不够全面。而该层面的表达变化在一定程度上是受microrna(通常缩写为mirna)控制的,mirna是一类小的、非编码rna,通过转录后调控其靶基因的表达发挥作用,与其靶基因的表达呈现负相关,故基因的微效性也受其影响。
技术实现要素:
有鉴于此,本发明提供了一种模块标记物挖掘方法、装置、计算机设备及存储介质,其结合了mirna与mrna两个层面的数据,并充分利用二者之间的生物学关系,挖掘复杂疾病的mirna模块标记物,具有更加全面、系统和可移植性的特点。
本发明的第一个目的在于提供一种模块标记物挖掘方法。
本发明的第二个目的在于提供一种模块标记物挖掘装置。
本发明的第三个目的在于提供一种计算机设备。
本发明的第四个目的在于提供一种存储介质。
本发明的第一个目的可以通过采取如下技术方案达到:
一种模块标记物挖掘方法,所述模块标记物为复杂疾病的mirna模块标记物,所述方法包括:
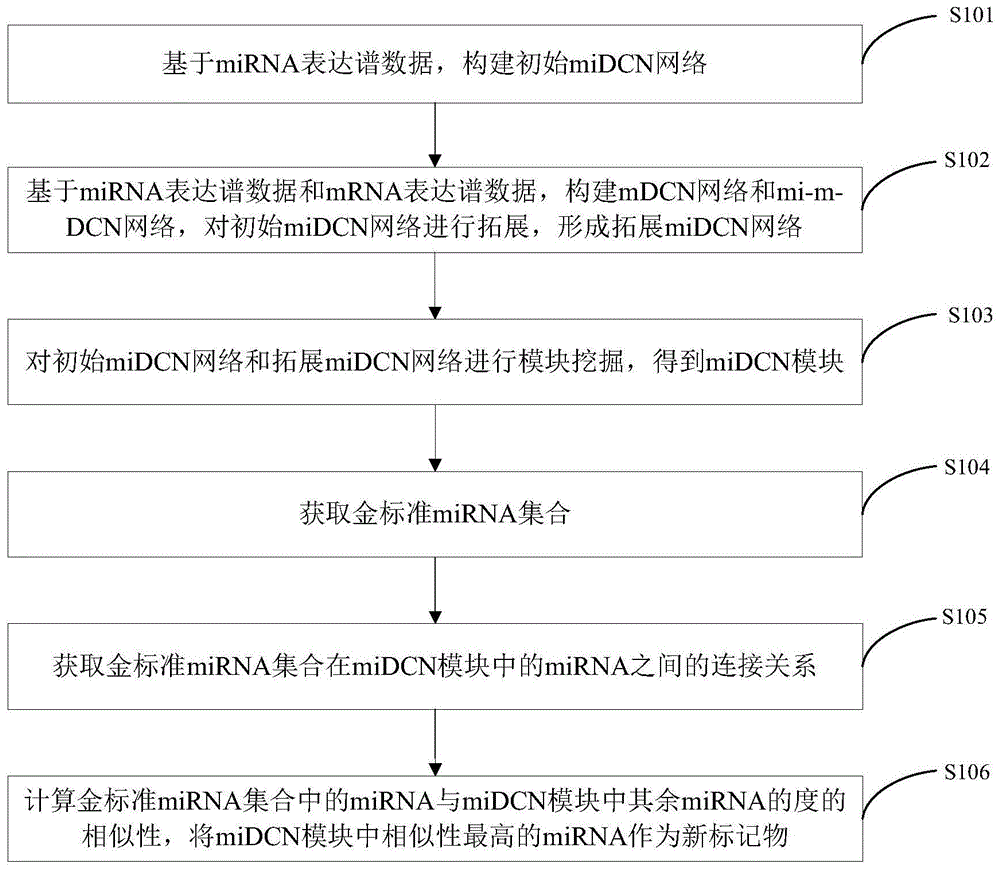
基于mirna表达谱数据,构建初始midcn网络;
基于mirna表达谱数据和mrna表达谱数据,构建mdcn网络和mi-m-dcn网络,对初始midcn网络进行拓展,形成拓展midcn网络;
对初始midcn网络和拓展midcn网络进行模块挖掘,得到midcn模块;
获取金标准mirna集合;其中,所述金标准mirna集合包括公开数据库中与所研究疾病有关的mirna;
获取金标准mirna集合在midcn模块中的mirna之间的连接关系;
计算金标准mirna集合中的mirna与midcn模块中其余mirna的度的相似性,将midcn模块中相似性最高的mirna作为新标记物。
进一步的,所述基于mirna表达谱数据,构建初始midcn网络,具体包括:
根据mirna表达谱数据,生成x行z列的矩阵;其中,每行表示一个mirna,每列表示一个样本,设e表示mirna表达谱数据,s表示所研究疾病的m个样本组成的疾病组表达谱数据,c表示对照组的z-m+1个样本组成的对照组表达谱数据;
用i-j表示任意一对mirna,计算i-j在疾病组表达谱数据s、对照组表达谱数据c中的相关系数r与代表统计学意义的p值,其中在疾病组表达谱数据s中计算所得的相关系数r和p值分别记为rs,i-j及ps,i-j,在对照组表达谱数据c中计算所得的相关系数r和p值分别记为rc,i-j及pc,i-j;
若i-j在疾病组表达谱数据s中的p值和i-j在对照组表达谱数据c中的p值至少有一个小于或等于设定阈值,则进行dci-j分数的计算;
将所有具有dc值的mirna对构建为初始midcn网络;其中,网络节点是mirna,边的权重是dc值。
进一步的,所述若i-j在疾病组表达谱数据s中的p值和i-j在对照组表达谱数据c中的p值至少有一个小于或等于设定阈值,则进行dci-j分数的计算,具体包括:
若i-j在疾病组表达谱数据s中的p值和i-j在对照组表达谱数据c中的p值均小于或等于设定阈值,则按照下式计算dci-j分数:
dci-j=|rs,i-j*(1-ps,i-j)-rc,i-j*(1-pc,i-j)|
若i-j在疾病组表达谱数据s中的p值小于或等于设定阈值,且i-j在对照组表达谱数据c中的p值大于设定阈值,则按照下式计算dci-j分数:
dci-j=|rs,i-j*(1-ps,i-j)-rc,i-j*pc,i-j|
若i-j在对照组表达谱数据c中的p值小于或等于设定阈值,且i-j在疾病组表达谱数据s中的p值大于设定阈值,则按照下式计算dci-j分数:
dci-j=|rs,i-j*ps,i-j-rc,i-j*(1-pc,i-j)|。
进一步的,所述将所有具有dc值的mirna对构建为初始midcn网络,具体包括:
构建疾病tbd-midcn网络;其中,所述疾病tbd-midcn网络包括所有ps,i-j及pc,i-j均具有统计学意义的mirna关系对;
构建疾病生成-midcn网络:其中,所述疾病生成-midcn网络包括所有ps,i-j具有统计学意义的mirna关系对;
构建疾病缺失-midcn网络:其中,所述疾病缺失-midcn网络包括所有pc,i-j具有统计学意义的mirna关系对。
进一步的,所述基于mirna表达谱数据和mrna表达谱数据,构建mdcn网络和mi-m-dcn网络,对初始midcn网络进行拓展,形成拓展midcn网络,具体包括:
根据mrna表达谱数据,生成x行z列的矩阵;其中,每行表示一个mirna,每列表示一个样本,设e1表示mrna表达谱数据,s1表示所研究疾病的m个样本组成的疾病组表达谱数据,c1表示对照组的z-m+1个样本组成的对照组表达谱数据;
用i1-j1表示任意一对mrna,计算i1-j1在疾病组表达谱数据s1、对照组表达谱数据c1中的相关系数r与代表统计学意义的p值,其中在疾病组表达谱数据s1中计算所得的相关系数r和p值分别记为rs1,i1-j1及ps1,i1-j1,在对照组表达谱数据c1中计算所得的相关系数r和p值分别记为rc1,i1-j1及pc1,i1-j1;
若i1-j1在疾病组表达谱数据s1中的p值和i1-j1在对照组表达谱数据c1中的p值至少有一个小于或等于设定阈值,则进行dci1-j1分数的计算;
将所有具有dc值的mrna对构建为mdcn网络;其中,网络节点是mrna,边的权重是dc值;
根据mirna表达谱数据和mrna表达谱数据,生成x行z列的矩阵;其中,每行表示一个一个mirna-mrna对,每列表示一个样本,设e2表示mirna-mrna表达谱数据,s2表示所研究疾病的m个样本组成的疾病组表达谱数据,c2表示对照组的z-m+1个样本组成的对照组表达谱数据;
用i2-j2表示任意一个mirna-mrna对,计算i2-j2在疾病组表达谱数据s2、对照组表达谱数据c2中的相关系数r与代表统计学意义的p值,其中在疾病组表达谱数据s2中计算所得的相关系数r和p值分别记为rs2,i2-j2及ps2,i2-j2,在对照组表达谱数据c2中计算所得的相关系数r和p值分别记为rc2,i2-j2及pc2,i2-j2;
若i2-j2在疾病组表达谱数据s2中的p值和i2-j2在对照组表达谱数据c2中的p值至少有一个小于或等于设定阈值,且相关系数r<0,则进行dci2-j2分数的计算;
将所有具有dc值的mirna-mrna对构建为mi-m-dcn-e网络,并选取若干个mirna靶基因预测算法的结果,为每个mirna-mrna对计算一个tnet分数,即靶基因预测算法将该mirna-mrna对预测为靶向关系的次数,构建一个mi-m-dcn-t网络;其中,网络节点是mirna-mrna对,边的权重是tnet分数;
利用初始midcn网络、mdcn网络、mi-m-dcn-e网络/mi-m-dcn-t网络作为输入,利用以节点的相似性关系为计算依据的一类计算机算法,预测新的mirna-mirna关系,加入midcn网络,形成拓展midcn网络。
进一步的,所述将所有具有dc值的mrna对构建为mdcn网络,具体包括:
构建疾病tbd-mdcn网络;其中,所述疾病tbd-mdcn网络包括所有ps1,i1-j1及pc1,i1-j1均具有统计学意义的mrna关系对;
构建疾病生成-mdcn网络:其中,所述疾病生成-mdcn网络包括所有ps1,i1-j1具有统计学意义的mrna关系对;
构建疾病缺失-mdcn网络:其中,所述疾病缺失-mdcn网络包括所有pc1,i1-j1具有统计学意义的mrna关系对。
所述将所有具有dc值的mirna-mrna对构建为mi-m-dcn-e网络,具体包括:
构建疾病tbd-mi-m-dcn-e网络;其中,所述疾病tbd-mi-m-dcn-e网络包括所有ps2,i2-j2及pc2,i2-j2均具有统计学意义的mirna-mrna对;
构建疾病生成-mi-m-dcn-e网络:其中,所述疾病生成-mi-m-dcn-e网络包括所有ps2,i2-j2具有统计学意义的mirna-mrna对;
构建疾病缺失-mi-m-dcn-e网络:其中,所述疾病缺失-mi-m-dcn-e网络包括所有pc2,i2-j2具有统计学意义的mirna-mrna对。
进一步的,所述对初始midcn网络和拓展midcn网络进行模块挖掘,得到midcn模块,具体包括:
针对初始midcn网络和拓展midcn网络,利用模块挖掘算法进行模块挖掘;
舍弃所有包含大于设定百分比的初始midcn网络节点的模块或仅有一条边的模块,对拓展midcn网络的midcn模块进行筛选;
通过网络文献数据库,将筛选得到的midcn模块中包含的mirna与所研究疾病作为关键词进行检索,按照功能将midcn模块中的mirna进行分类。
本发明的第二个目的可以通过采取如下技术方案达到:
一种模块标记物挖掘装置,所述模块标记物为复杂疾病的mirna模块标记物,所述装置包括:
构建单元,用于基于mirna表达谱数据,构建初始midcn网络;
拓展单元,用于基于mirna表达谱数据和mrna表达谱数据,构建mdcn网络和mi-m-dcn网络,对初始midcn网络进行拓展,形成拓展midcn网络;
挖掘单元,用于对初始midcn网络和拓展midcn网络进行模块挖掘,得到midcn模块;
第一获取单元,用于获取金标准mirna集合;其中,所述金标准mirna集合包括公开数据库中与所研究疾病有关的mirna;
第二获取单元,用于获取金标准mirna集合在midcn模块中的mirna之间的连接关系;
计算单元,用于计算金标准mirna集合中的mirna与midcn模块中其余mirna的度的相似性,将midcn模块中相似性最高的mirna作为新标记物。
本发明的第三个目的可以通过采取如下技术方案达到:
一种计算机设备,包括处理器以及用于存储处理器可执行程序的存储器,所述处理器执行存储器存储的程序时,实现上述的模块标记物挖掘方法。
本发明的第四个目的可以通过采取如下技术方案达到:
一种存储介质,存储有程序,所述程序被处理器执行时,实现上述的模块标记物挖掘方法。
本发明相对于现有技术具有如下的有益效果:
本发明利用mirna表达谱数据和mrna表达谱数据,构建初始midcn网络,并构建mdcn网络和mi-m-dcn网络,对初始midcn网络进行拓展,形成拓展midcn网络,对初始midcn网络和拓展midcn网络进行模块挖掘,得到midcn模块,结合两个生物学层面的数据、利用多种经典方法进行分析,所得到的结果更加准确,由于充分考虑了数据间的内在生物学联系,因此所得结果更加具有实际意义,可以用于多基因遗传病,如精神分裂等复杂疾病相关的mirna模块标记物的挖掘。
附图说明
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单的介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图示出的结构获得其他的附图。
图1为本发明实施例1的模块标记物挖掘方法的流程图。
图2为本发明实施例1的模块标记物挖掘方法的原理图。
图3为本发明实施例1的构建初始midcn网络的流程图。
图4为本发明实施例1的对初始midcn网络进行拓展的流程图。
图5为本发明实施例2的模块标记物挖掘装置的结构框图。
图6为本发明实施例3的计算机设备的结构框图。
具体实施方式
为使本发明实施例的目的、技术方案和优点更加清楚,下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整的描述,显然,所描述的实施例是本发明的一部分实施例,而不是全部的实施例,基于本发明中的实施例,本领域普通技术人员在没有作出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
实施例1:
如图1~图2所示,本实施例提供了一种模块标记物挖掘方法,模块标记物为精神分裂症的mirna模块标记物,该方法包括以下步骤:
s101、基于mirna表达谱数据,构建初始midcn网络。
基于mirna的表达谱数据可来源于深度测序数据或芯片数据,本实施例从公开数据库中获取mirna表达谱数据,dcn网络是指datacommunicationnetwork,即数据通信网络,midcn网络即为基于mirna的数据通信网络,可以理解的,后面的mdcn网络为基于mrna的数据通信网络,mi-m-dcn网络为基于mirna与mrna的数据通信网络。
如图3所示,该步骤s101具体包括:
(1)数据拆分与组合计算:根据mirna表达谱数据,生成x行z列的矩阵,每行表示一个mirna,每列表示一个样本,设e表示mirna表达谱数据,s表示所研究疾病的m个样本组成的疾病组表达谱数据,c表示对照组的z-m+1个样本组成的对照组表达谱数据,x个mirna的所有可能的组合则为
本实施例在对表达谱数据e进行生物信息学常规预处理后,x=230,z=30,m=15,即包含230行(230个mirna),30列(30个样本),其中,s组包含精神分裂症患者的15个样本组成的疾病组表达谱数据,c组包含健康人的15个样本组成的对照组表达谱数据,230个mirna的所有可能的组合则为
(2)相关性计算:用i-j表示任意一对mirna,利用斯皮尔曼秩相关或皮尔斯相关系数等方法计算i-j在疾病组表达谱数据s、对照组表达谱数据c中的相关系数r与代表统计学意义的p值。
本实施例利用斯皮尔曼秩相关计算i-j在疾病组表达谱数据s、对照组表达谱数据c中的相关系数r与代表统计学意义的p值,在疾病组表达谱数据s中计算所得的相关系数r和p值分别记为rs,i-j及ps,i-j,在对照组表达谱数据c中计算所得的相关系数r和p值分别记为rc,i-j及pc,i-j。
(3)dc值计算:若i-j在疾病组表达谱数据s中的p值和i-j在对照组表达谱数据c中的p值至少有一个小于或等于设定阈值,则进行dci-j分数的计算。
针对步骤(2)所得的p值,设定阈值为0.05,选择至少有一个p值小于或等于设定阈值的mirna对,进行dci-j分数的计算,具体为:
若i-j在疾病组表达谱数据s中的p值和i-j在对照组表达谱数据c中的p值均小于或等于设定阈值,则按照下式计算dci-j分数:
dci-j=|rs,i-j*(1-ps,i-j)-rc,i-j*(1-pc,i-j)|
若i-j在疾病组表达谱数据s中的p值小于或等于设定阈值,且i-j在对照组表达谱数据c中的p值大于设定阈值,则按照下式计算dci-j分数:
dci-j=|rs,i-j*(1-ps,i-j)-rc,i-j*pc,i-j|
若i-j在对照组表达谱数据c中的p值小于或等于设定阈值,且i-j在疾病组表达谱数据s中的p值大于设定阈值,则按照下式计算dci-j分数:
dci-j=|rs,i-j*ps,i-j-rc,i-j*(1-pc,i-j)|
从以上三式可知,如果在s和c中都显著,则分别将r值与1-p相乘,再将二者相减,得到一个差值的绝对值;如果仅在s或c中p值显著,则不显著的p值与r值直接相乘,再计算差值的绝对值,总结为下式:
(4)构建初始midcn网络:将所有具有dc值的mirna对构建为初始midcn网络;其中,网络节点是mirna,边的权重是dc值。
根据步骤(3)中dc值的计算方式,构建三个midcn网络,如下:
a、构建疾病tbd-midcn网络(tobedetermined,待确定组),包括3375个所有ps,i-j及pc,i-j均具有统计学意义的mirna关系对,记为raw-tbd-midcn。
b、构建疾病生成-midcn网络,包括5274个ps,i-j具有统计学意义的mirna关系对。
c、构建疾病缺失-midcn网络,包括3293个所有ps,i-j具有统计学意义的mirna关系对。
s102、基于mirna表达谱数据和mrna表达谱数据,构建mdcn网络和mi-m-dcn网络,对初始midcn网络进行拓展,形成拓展midcn网络。
如图4所示,该步骤s102具体包括:
(1)构建mdcn网络,利用步骤s101的方法,如下:
a、数据拆分与组合计算:根据mrna表达谱数据,生成x行z列的矩阵;其中,每行表示一个mirna,每列表示一个样本,设e1表示mrna表达谱数据,s1表示所研究疾病的m个样本组成的疾病组表达谱数据,c1表示对照组的z-m+1个样本组成的对照组表达谱数据。
b、相关性计算:用i1-j1表示任意一对mrna,计算i1-j1在疾病组表达谱数据s1、对照组表达谱数据c1中的相关系数r与代表统计学意义的p值,其中在疾病组表达谱数据s1中计算所得的相关系数r和p值分别记为rs1,i1-j1及ps1,i1-j1,在对照组表达谱数据c1中计算所得的相关系数r和p值分别记为rc1,i1-j1及pc1,i1-j1。
c、dc值计算:若i1-j1在疾病组表达谱数据s1中的p值和i1-j1在对照组表达谱数据c1中的p值至少有一个小于或等于设定阈值,则进行dci1-j1分数的计算。
d、构建mdcn网络:将所有具有dc值的mrna对构建为mdcn网络;其中,网络节点是mrna,边的权重是dc值。
在本实施例中,从公开数据库中获取mrna表达谱数据e1,进行生物信息学常规预处理后,x=1311,z=30,m=18,即包含1311行(1311个mrna),30列(30个样本),其中,s组包含精神分裂症患者的18个样本组成的疾病组表达谱数据,c包含健康人的12个样本组成的对照组表达谱数据,1311个mirna的所有可能的组合则为
(2)构建mi-m-dcn网络,利用步骤s101的方法,如下:
a、数据拆分与组合计算:根据mirna表达谱数据和mrna表达谱数据,生成x行z列的矩阵;其中,每行表示一个一个mirna-mrna对,每列表示一个样本,设e2表示mirna-mrna表达谱数据,s2表示所研究疾病的m个样本组成的疾病组表达谱数据,c2表示对照组的z-m+1个样本组成的对照组表达谱数据。
b、相关性计算:用i2-j2表示任意一个mirna-mrna对,计算i2-j2在疾病组表达谱数据s2、对照组表达谱数据c2中的相关系数r与代表统计学意义的p值,其中在疾病组表达谱数据s2中计算所得的相关系数r和p值分别记为rs2,i2-j2及ps2,i2-j2,在对照组表达谱数据c2中计算所得的相关系数r和p值分别记为rc2,i2-j2及pc2,i2-j2。
c、dc值计算:若i2-j2在疾病组表达谱数据s2中的p值和i2-j2在对照组表达谱数据c2中的p值至少有一个小于或等于设定阈值,且相关系数r<0,则进行dci2-j2分数的计算。
d、构建mi-m-dcn网络:将所有具有dc值的mirna-mrna对构建为mi-m-dcn-e网络,并选取若干个mirna靶基因预测算法的结果,为每个mirna-mrna对计算一个tnet分数,即靶基因预测算法将该mirna-mrna对预测为靶向关系的次数,构建一个mi-m-dcn-t网络;其中,网络节点是mirna-mrna对,边的权重是tnet分数。
在本实施例中,基于步骤s101和步骤s102的(1)中的mirna与mrna的表达谱数据,1311个mrna与230个mirna之间所有的可能组合为1311*230=301530,选择所有r<0且p≤0.05的mirna-mrna对,构建两个mi-m-dcn-e网络:“疾病缺失-mi-m-dcn-e”含32369条边(记为raw-lost-mi-m-dcn-e)、“疾病生成-mi-m-dcn-e”网络含13210条边(记为raw-gain-mi-m-dcn-e),由于没有mirna-mrna对满足“疾病tbd-mi-m-dcn-e”的构建条件,故此并没有构建此网络;此外,选取10个mirna靶基因预测算法(diana-microt,mirsvr,pictar5,rna22,rnahybrid,targetscan,pita,mirtarget2,targetminer,miranda)的结果,为每一对mirna-mrna组合计算一个tnet分数,共有82071对mirna-mrna组合的tnet≥1,用于构建mi-m-dcn-t网络(记为raw-mi-m-dcn-e)。
(3)拓展midcn网络:利用初始midcn网络、mdcn网络、mi-m-dcn-e网络/mi-m-dcn-t网络作为输入,利用以节点的相似性关系为计算依据的一类计算机算法,预测新的mirna-mirna关系,加入midcn网络,形成拓展midcn网络。
其中,计算机算法采用基于随机游走的发表于2017年methods学术期刊的算法(文章详见pengw,lanw,zhongj,etal.anovelmethodofpredictingmicrorna-diseaseassociationsbasedonmicrorna,disease,geneandenvironmentfactornetworks[j].methods,2017,124:69-77.),预测两种新的mirna-mirna关系,分别加入所构建的两个midcn网络,形成两种共计四个新的拓展midcn网络:extended-lost-midcn-e,extended-gain-midcn-e,extended-lost-midcn-t,extended-gain-midcn-t。
s103、对初始midcn网络和拓展midcn网络进行模块挖掘,得到midcn模块。
(1)模块挖掘:针对初始midcn网络和拓展midcn网络,利用模块挖掘算法进行模块挖掘。
本实施例中,针对上述七个midcn网络,如下:
raw-tbd-midcn,raw-lost-midcn,extended-lost-midcn-e,extended-lost-midcn-t,raw-gain-midcn,extended-gain-midcn-e,extended-gain-midcn-t
利用计算机的各种模块挖掘算法进行模块挖掘,如利用r语言的igraph包里的挖掘网络模块的方法‘cluster_label_prop’进行模块挖掘。
(2)模块筛选:舍弃所有包含大于设定百分比的初始midcn网络节点的模块或仅有一条边的模块,对拓展midcn网络的midcn模块进行筛选。
本实施例中,设定百分比为90%,舍弃所有包含≥90%的初始midcn网络节点的模块或仅有一条边的模块,最终在五个midcn网络中(raw-tbd-midcn,raw-lost-midcn,extended-lost-midcn-e,raw-gain-midcn,extended-gain-midcn-e)筛选出十二个midcn模块。
(3)模块功能注释:通过网络文献数据库,将筛选得到的midcn模块中包含的mirna与所研究疾病作为关键词进行检索,按照功能将midcn模块中的mirna进行分类。
本实施例中,通过网络文献数据库,以筛选得到的midcn模块中包含的mirna与“schizophrenia”作为关键词进行检索,将midcn模块中的mirna根据功能分为3类:与精神分裂症相关、与其他精神类疾病相关、无证据证实相关。
接下来的步骤s104~s106为新标记物的验证步骤,根据步骤s103所得的midcn模块中的mirna分类,为midcn模块的功能进行概括,并为midcn模块中无功能注释的mirna进行功能预测,具体地,根据midcn模块中的mirna自身的生物学的意义,比如与精神分裂的发生直接相关、与除了精神分裂外的其他精神类疾病相关、无证据证明与精神类疾病有关,如果一个midcn模块内包含的80%的mirna都是与精神分裂的发生直接相关的,那么就可以概括这个midcn模块的功能是与精神分裂的发生直接相关,预测midcn模块中其他的20%可能也与精神分裂的发生相关,但可能不是直接相关,而是一种间接相关。
s104、获取金标准mirna集合。
具体地,选取公开数据库中证实的与所研究的疾病有关的mirna作为金标准mirna集合。
s105、获取金标准mirna集合在midcn模块中的mirna之间的连接关系。
s106、计算金标准mirna集合中的mirna与midcn模块中其余mirna的度的相似性,将midcn模块中相似性最高的mirna作为新标记物。
具体地,利用皮尔斯相关系数、斯皮尔曼秩相关等方法计算金标准集合中的mirna与模块中其余mirna的度的相似性,选取相关系数最大且p值显著的作为新标记物。
本实施例中,获取金标准mirna集合在步骤s103中所得的midcn模块中的mirna之间的连接关系,利用斯皮尔曼秩相关计算金标准集合中的mirna与模块中其余mirna的度的相似性,在一个midcn模块中发现金标准mirna集合中的has-mir-346与has-mir-184的度相似性最高,为r=1,p=0,经文献检索,发现mir-184与mdd(majordepressivedisorder)[pmid:27468165]有关。故选择mir-184作为新标记物。
在执行上述步骤s101~s106之后,对新标记物进行验证,本实施例利用mk-801对sd大鼠进行精神分裂症的建模。经水迷宫等行为学验证造模成功后,取大鼠脑组织,利用pcr实验证实了mir-184在精神分裂症的大鼠脑组织中表达上调,验证了与精神分裂症的相关性,说明了本实施例技术方案的准确性。
其中,pcr的结果如下表:
计算方法如下:
(1)步骤1:内参均一化样本差异,方法为:ct内参-ct目标=△ct
(2)步骤2:其他样本和对照样本比较,方法为:△ctcase组-△ctcontrol组=△△ct
(3)步骤3:使用公式计算,方法为:倍数变化=2-△△ct
根据上述步骤,计算得到的三次重复的结果分别为:1.972465,2.789487,2.928171,证实了mir-184在精神分裂症的脑组织中表达上调。
本领域技术人员可以理解,实现上述实施例方法中的全部或部分步骤可以通过程序来指令相关的硬件来完成,相应的程序可以存储于计算机可读取存储介质中。
应当注意,尽管在附图中以特定顺序描述了上述实施例的方法操作,但是这并非要求或者暗示必须按照该特定顺序来执行这些操作,或是必须执行全部所示的操作才能实现期望的结果。相反,描绘的步骤可以改变执行顺序。附加地或备选地,可以省略某些步骤,将多个步骤合并为一个步骤执行,和/或将一个步骤分解为多个步骤执行。
实施例2:
如图5所示,本实施例提供了一种模块标记物挖掘装置,模块标记物为复杂疾病的mirna模块标记物,该装置包括构建单元501、拓展单元502、挖掘单元503、第一获取单元504、第二获取单元505和计算单元506,各个单元的具体功能如下:
构建单元501,用于基于mirna表达谱数据,构建初始midcn网络。
拓展单元502,用于基于mirna表达谱数据和mrna表达谱数据,构建mdcn网络和mi-m-dcn网络,对初始midcn网络进行拓展,形成拓展midcn网络。
挖掘单元503,用于对初始midcn网络和拓展midcn网络进行模块挖掘,得到midcn模块。
第一获取单元504,用于获取金标准mirna集合;其中,所述金标准mirna集合包括公开数据库中与所研究疾病有关的mirna。
第二获取单元505,用于获取金标准mirna集合在midcn模块中的mirna之间的连接关系。
计算单元506,用于计算金标准mirna集合中的mirna与midcn模块中其余mirna的度的相似性,将midcn模块中相似性最高的mirna作为新标记物。
本实施例中各个单元的具体实现可以参见上述实施例1,在此不再一一赘述;需要说明的是,本实施例提供的装置仅以上述各功能单元的划分进行举例说明,在实际应用中,可以根据需要而将上述功能分配给不同的功能单元完成,即将内部结构划分成不同的功能单元,以完成以上描述的全部或者部分功能。
实施例3:
如图6所示,本实施例提供了一种计算机设备,该计算机设备可以是计算机,包括通过装置总线601连接的处理器602、存储器、输入装置603、显示器604和网络接口605;其中,处理器602用于提供计算和控制能力,存储器包括非易失性存储介质606和内存储器607,该非易失性存储介质606存储有操作装置、计算机程序和数据库,该内存储器1207为非易失性存储介质606中的操作装置和计算机程序的运行提供环境,计算机程序被处理器602执行时,实现上述实施例1的模块标记物挖掘方法,如下:
基于mirna表达谱数据,构建初始midcn网络;
基于mirna表达谱数据和mrna表达谱数据,构建mdcn网络和mi-m-dcn网络,对初始midcn网络进行拓展,形成拓展midcn网络;
对初始midcn网络和拓展midcn网络进行模块挖掘,得到midcn模块;
获取金标准mirna集合;其中,所述金标准mirna集合包括公开数据库中与所研究疾病有关的mirna;
获取金标准mirna集合在midcn模块中的mirna之间的连接关系;
计算金标准mirna集合中的mirna与midcn模块中其余mirna的度的相似性,将midcn模块中相似性最高的mirna作为新标记物。
实施例4:
本实施例提供一种存储介质,该存储介质为计算机可读存储介质,其存储有计算机程序,所述计算机程序被处理器执行时,实现上述实施例1的模块标记物挖掘方法,如下:
基于mirna表达谱数据,构建初始midcn网络;
基于mirna表达谱数据和mrna表达谱数据,构建mdcn网络和mi-m-dcn网络,对初始midcn网络进行拓展,形成拓展midcn网络;
对初始midcn网络和拓展midcn网络进行模块挖掘,得到midcn模块;
获取金标准mirna集合;其中,所述金标准mirna集合包括公开数据库中与所研究疾病有关的mirna;
获取金标准mirna集合在midcn模块中的mirna之间的连接关系;
计算金标准mirna集合中的mirna与midcn模块中其余mirna的度的相似性,将midcn模块中相似性最高的mirna作为新标记物。
需要说明的是,本实施例的计算机可读存储介质可以是计算机可读信号介质或者计算机可读存储介质或者是上述两者的任意组合。计算机可读存储介质例如可以是——但不限于——电、磁、光、电磁、红外线、或半导体的装置、装置或器件,或者任意以上的组合。计算机可读存储介质的更具体的例子可以包括但不限于:具有一个或多个导线的电连接、便携式计算机磁盘、硬盘、随机访问存储器(ram)、只读存储器(rom)、可擦式可编程只读存储器(eprom或闪存)、光纤、便携式紧凑磁盘只读存储器(cd-rom)、光存储器件、磁存储器件、或者上述的任意合适的组合。在本公开中,计算机可读存储介质可以是任何包含或存储程序的有形介质,该程序可以被指令执行装置、装置或者器件使用或者与其结合使用。而在本本实施例中,计算机可读信号介质可以包括在基带中或者作为载波一部分传播的数据信号,其中承载了计算机可读的程序。这种传播的数据信号可以采用多种形式,包括但不限于电磁信号、光信号或上述的任意合适的组合。计算机可读信号介质还可以是计算机可读存储介质以外的任何计算机可读存储介质,该计算机可读信号介质可以发送、传播或者传输用于由指令执行装置、装置或者器件使用或者与其结合使用的程序。计算机可读存储介质上包含的计算机程序可以用任何适当的介质传输,包括但不限于:电线、光缆、rf(射频)等等,或者上述的任意合适的组合。
综上所述,本发明利用mirna表达谱数据和mrna表达谱数据,构建初始midcn网络,并构建mdcn网络和mi-m-dcn网络,对初始midcn网络进行拓展,形成拓展midcn网络,对初始midcn网络和拓展midcn网络进行模块挖掘,得到midcn模块,结合两个生物学层面的数据、利用多种经典方法进行分析,所得到的结果更加准确,由于充分考虑了数据间的内在生物学联系,因此所得结果更加具有实际意义,可以用于多基因遗传病,如精神分裂等复杂疾病相关的mirna模块标记物的挖掘。
以上所述,仅为本发明专利较佳的实施例,但本发明专利的保护范围并不局限于此,任何熟悉本技术领域的技术人员在本发明专利所公开的范围内,根据本发明专利的技术方案及其发明构思加以等同替换或改变,都属于本发明专利的保护范围。
- 还没有人留言评论。精彩留言会获得点赞!