基于肿瘤样本的多组学联合分析方法和系统与流程
1.本技术涉及肿瘤致病机制分析领域,特别涉及一种基于肿瘤样本的多组学联合分析方法和系统。
背景技术:
2.肿瘤的治疗一直是医学不断探索的领域,而对于肿瘤致病机制的调控关系,以及致病因子的分析是肿瘤治疗及预后的关键所在。
3.基于肿瘤样本基因测序数据和样本的表达量数据的联合分析,挖掘肿瘤致病驱动的候选调控因子。这类分析的本质是基于样本的基因组学数据和转录组数据的联合分析,找出基因组学不同类型的调控因子(顺式作用元件)对转录组学层面基因表达量(反式作用因子)的调控关系。基于多组学数据联合分析进行肿瘤致病驱动的候选调控因子的挖掘可以确定不同的基因组变异事件对基因转录表达层面的影响,可以为肿瘤的发病机制以及致病因子的提供新思路。
技术实现要素:
4.(一)申请目的基于此,本技术通过基因组数据和转录组数据的联合分析,挖掘了肿瘤致病驱动的候选调控因子,对由转录调控影响变化而造成的疾病进展,疾病发病机制的调控关系,揭示了新的肿瘤致病基因。本技术公开了以下技术方案。
5.(二)技术方案本技术公开了一种基于肿瘤样本的多组学联合分析方法,其特征在于,包括:根据肿瘤样本的转录组学数据构建共表达基因模块;基于肿瘤样本的基因组数据获取scnv基因列表;对所述共表达基因模块中的基因和所述scnv基因列表中的基因进行相关性分析,生成scnv基因列表中基因的评分结果;基于所述评分结果,利用go注释和快速功能注释从所述scnv基因列表中获取肿瘤的致病新发基因;基于肿瘤核心通路变化因子构建肿瘤致病新发基因调控网络;基于所述肿瘤致病新发基因调控网络验证所述肿瘤的致病新发基因。
6.在一种可能的实施方式中,所述根据肿瘤样本的转录组学数据构建共表达基因模块,包括:根据肿瘤样本的转录组学数据生成差异表达基因列表;将所述差异表达基因列表中的差异表达基因进行聚类,得到若干基因簇;基于所述基因簇的软阈值构建基因簇共表达网络;重构所述基因簇共表达网络中的基因,生成共表达基因模块。
7.在一种可能的实施方式中,所述肿瘤样本的转录组学数据为rna-seq的表达量数
据。
8.在一种可能的实施方式中,所述rna-seq的表达量数据为rpkm数。
9.在一种可能的实施方式中,所述将所述差异表达基因列表中的差异表达基因进行聚类采用的聚类方法为双向聚类法。
10.在一种可能的实施方式中,所述重构所述基因簇共表达网络中的基因,生成共表达基因模块,包括:利用相异度算法对所述基因簇共表达网络中的基因进行层次聚类;基于所述层次聚类结果,建立分层聚类树;将所述分层聚类树剪切成若干模块并进行模块合并,生成若干个共表达基因模块。
11.在一种可能的实施方式中,所述将所述分层聚类树剪切成若干模块利用动态剪切法。
12.在一种可能的实施方式中,所述进行模块合并具体为,将相关性系数大于0.75的模块进行合并。
13.在一种可能的实施方式中,所述对所述共表达基因模块中的基因和所述scnv基因列表中的基因进行相关性分析,生成scnv基因列表中基因的评分结果,包括:利用决策树分析法对所述共表达基因模块中的基因和所述scnv基因列表中的基因进行相关性分析,生成scnv基因列表中基因的初步评分结果;根据cnv的拷贝类型对scnv扩增基因和scnv缺失基因按照评分进行排序,生成scnv基因列表中基因的评分结果;其中,所述评分结果包括scnv扩增基因评分结果和scnv缺失基因评分结果。
14.在一种可能的实施方式中,所述scnv基因列表中基因的评分越高,与所述共表达模块中的基因的相关性越高。
15.在一种可能的实施方式中,所述基于所述评分结果,利用go注释和快速功能注释从所述scnv基因列表中获取肿瘤的致病新发基因,包括:利用go注释工具选取第一致病新发基因;将进行标注的所述第一致病新发基因输入icmdb数据库进行快速功能注释,获取肿瘤的致病新发基因。
16.在一种可能的实施方式中,还包括:结合临床样本的预后及生存数据,对所述肿瘤的致病新发基因进行验证。
17.作为本技术的第二方面,本技术还公开了一种基于肿瘤样本的多组学联合分析系统,包括:基因构建模块,用于根据肿瘤样本的转录组学数据构建共表达基因模块;列表获取模块,用于基于肿瘤样本的基因组数据获取scnv基因列表;评分模块,用于对所述共表达基因模块中的基因和所述scnv基因列表中的基因进行相关性分析,生成scnv基因列表中基因的评分结果;新发基因获取模块,用于基于所述评分结果,利用go注释和快速功能注释从所述scnv基因列表中获取肿瘤的致病新发基因;网络构建模块,用于基于肿瘤核心通路变化因子构建肿瘤致病新发基因调控网
络;验证模块,用于基于所述肿瘤致病新发基因调控网络验证所述肿瘤的致病新发基因。
18.在一种可能的实施方式中,所述基因构建模块,包括:差异列表生成子模块,用于根据肿瘤样本的转录组学数据生成差异表达基因列表;聚类子模块,用于将所述差异表达基因列表中的差异表达基因进行聚类,得到若干基因簇;网络构建子模块,用于基于所述基因簇的软阈值构建基因簇共表达网络;基因构建子模块,用于重构所述基因簇共表达网络中的基因,生成共表达基因模块。
19.在一种可能的实施方式中,所述肿瘤样本的转录组学数据为 rna-seq的表达量数据。
20.在一种可能的实施方式中,所述rna-seq的表达量数据为rpkm数。
21.在一种可能的实施方式中,所述将所述差异表达基因列表中的差异表达基因进行聚类采用的聚类方法为双向聚类法。
22.在一种可能的实施方式中,所述基因构建子模块,包括:聚类单元,用于利用相异度算法对所述基因簇共表达网络中的基因进行层次聚类;聚类树建立单元,用于基于所述层次聚类结果,建立分层聚类树;基因构建单元,用于将所述分层聚类树剪切成若干模块并进行模块合并,生成若干个共表达基因模块。
23.在一种可能的实施方式中,所述将所述分层聚类树剪切成若干模块利用动态剪切法。
24.在一种可能的实施方式中,所述进行模块合并具体为,将相关性系数大于0.75的模块进行合并。
25.在一种可能的实施方式中,所述评分模块,包括:初步评分子模块,用于利用决策树分析法对所述共表达基因模块中的基因和所述scnv基因列表中的基因进行相关性分析,生成scnv基因列表中基因的初步评分结果;评分子模块,用于根据cnv的拷贝类型对scnv扩增基因和scnv缺失基因按照评分进行排序,生成scnv基因列表中基因的评分结果;其中,所述评分结果包括scnv扩增基因评分结果和scnv缺失基因评分结果。
26.在一种可能的实施方式中,所述scnv基因列表中基因的评分越高,与所述共表达模块中的基因的相关性越高。
27.在一种可能的实施方式中,所述新发基因获取模块,包括:第一新发基因获取子模块,用于利用go注释工具选取第一致病新发基因;新发基因获取子模块,用于将进行标注的所述第一致病新发基因输入icmdb数据库进行快速功能注释,获取肿瘤的致病新发基因。
28.在一种可能的实施方式中,还包括:临床验证模块,用于结合临床样本的预后及生
存数据,对所述肿瘤的致病新发基因进行验证。
29.(三)有益效果本技术通过基因组数据和转录组数据的联合分析,挖掘了肿瘤致病驱动的候选调控因子,对由转录调控影响变化而造成的疾病进展,疾病发病机制的调控关系,揭示了新的肿瘤致病基因。
附图说明
30.以下参考附图描述的实施例是示例性的,旨在用于解释和说明本技术,而不能理解为对本技术的保护范围的限制。
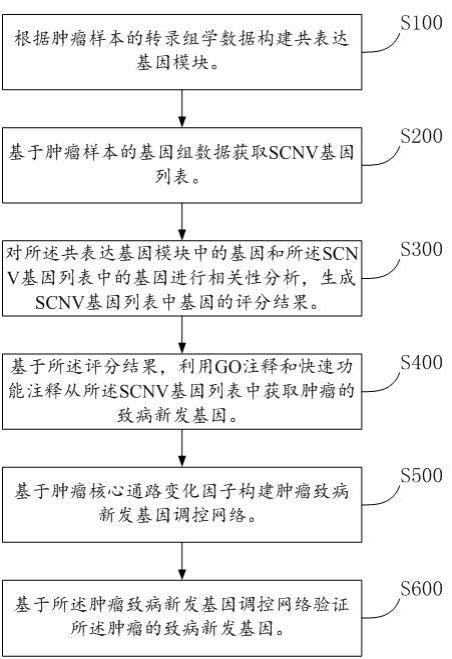
31.图1是本技术公开的基于肿瘤样本的多组学联合分析方法的流程示意图。
32.图2是本技术公开的基于肿瘤样本的多组学联合分析系统的结构框图。
具体实施方式
33.为使本技术实施的目的、技术方案和优点更加清楚,下面将结合本技术实施例中的附图,对本技术实施例中的技术方案进行更加详细的描述。
34.下面参考图1详细描述本技术公开的基于肿瘤样本的多组学联合分析方法实施例。如图1所示,本实施例公开的方法主要包括有以下步骤s100至s600。
35.s100,根据肿瘤样本的转录组学数据构建共表达基因模块。
36.其中s100包括s110-s140。
37.s110,根据肿瘤样本的转录组学数据生成差异表达基因列表。
38.在一个实施例中,转录组学数据为rna-seq的表达量数据,进一步,所述rna-seq的表达量数据为rpkm数,对肿瘤样本的bokm数值矩阵进行t-n的差异表达分析,得到差异表达的基因列表。
39.s120,将所述差异表达基因列表中的差异表达基因进行聚类,得到若干基因簇。
40.具体的,将s110中差异表达的基因列表中的基因表达矩阵作为输入文件,通过双向聚类法(two-way clusting)进行聚类,将基因分为若干个基因簇。
41.s130,基于所述基因簇的软阈值构建基因簇共表达网络。
42.s140,重构所述基因簇共表达网络中的基因,生成共表达基因模块。
43.其中,s140还包括s141-s143。
44.s141,利用相异度算法对所述基因簇共表达网络中的基因进行层次聚类。
45.s142,基于所述层次聚类结果,建立分层聚类树。
46.s143,将所述分层聚类树剪切成若干模块并进行模块合并,生成若干个共表达基因模块。
47.具体的,利用动态剪切法将所述分层聚类树剪切成若干模块,将相关性系数大于0.75即相异性系数小于0.25的模块合并掉。所述模块的最小基因数为30。
48.s200,基于肿瘤样本的基因组数据获取scnv基因列表。
49.具体的,基于肿瘤样本基因组的cnv片段数据得到发生拷贝数变化的scnv基因列表。其中,cnv为copy-number variations(拷贝数变化),scnv为somatic copy-number variations(肿瘤中体细胞突变相关的拷贝数变化)。
50.s300,对所述共表达基因模块中的基因和所述scnv基因列表中的基因进行相关性分析,生成scnv基因列表中基因的评分结果。
51.具体的,s300还包括s310-s320。
52.s310,利用决策树分析法对所述共表达基因模块中的基因和所述scnv基因列表中的基因进行相关性分析,生成scnv基因列表中基因的初步评分结果。
53.具体的,利用决策树对所述scnv基因列表中的基因和所述共表达基因模块里的基因进行相关性分析,建立cnv调控因子的评分体系,评分考虑到每个调控因子在共表达基因模块中的被分配到的子决策树的数量,生成scnv基因列表中基因的初步评分结果。
54.s320,根据cnv的拷贝类型对scnv扩增基因和scnv缺失基因按照评分进行排序,生成scnv基因列表中基因的评分结果。
55.具体的,scnv基因列表中基因的评分越高,则所述scnv基因列表中基因与所述共表达模块中的基因的相关性越高。
56.其中,所述评分结果包括scnv扩增基因评分结果和scnv缺失基因评分结果。
57.s400,基于所述评分结果,利用go注释和快速功能注释从所述scnv基因列表中获取肿瘤的致病新发基因。
58.具体的,s400还包括s410-s420。
59.s410,利用go注释工具选取第一致病新发基因。
60.具体的,利用go注释工具进行go分析,将和肿瘤进展相关的go-term所在共表达基因模块中的基因簇标注出,同时标注对应的基因。
61.s420,将进行标注的所述第一致病新发基因输入icmdb数据库进行快速功能注释,获取肿瘤的致病新发基因。
62.具体的,结合icmdb数据库,对模块网络中的scna列表中的基因(顺式作用元件)及go-term中的基因(反式调控因子)进行搜索注释,快速得到和肿瘤发生、进展等过程相关的新的基因组变异事件及其调控的基因。
63.s500,基于肿瘤核心通路变化因子构建肿瘤致病新发基因调控网络。
64.具体的,肿瘤核心通路变化因子即为现有的已知的与肿瘤治病的相关因子,从肿瘤核心通路变化因子库中选取在scnv列表中排名靠前的肿瘤核心通路变化因子作为目标肿瘤核心通路变化因子,基于所述目标肿瘤核心通路变化因子构建新发基因调控网络。
65.s600,基于所述肿瘤致病新发基因调控网络验证所述肿瘤的致病新发基因。
66.基于构建的新发基因调控网络为激活或抑制关系网络,以及它们在样品中的基因拷贝数变化水平。可以阐明scnv中的致病新发基因可能存在的调控模式。
67.在一个实施例中,本技术的验证方式还包括:用于结合临床样本的预后及生存数据,对所述肿瘤的致病新发基因进行验证。
68.具体的,结合临床样本的预后及生存资料数据,进行scnv致病新发基因的验证。通过生存分析,构建高评分候选基因拷贝数分组,分布将scnv增加和正常的分为两组进行比较,将scnv较少和正常的进行分组。在群里样本中进行生产分析。算出每个高评分候选基因在群体中的p值。挑选出p≤0.05的候选基因进行分析。对致病新发基因事件进行验证。
69.下面参考图2详细描述本技术公开的基于肿瘤样本的多组学联合分析方法系统实施例。如图2所示,本实施例公开的系统包括:
基因构建模块1,用于根据肿瘤样本的转录组学数据构建共表达基因模块;列表获取模块2,用于基于肿瘤样本的基因组数据获取scnv基因列表;评分模块3,用于对所述共表达基因模块中的基因和所述scnv基因列表中的基因进行相关性分析,生成scnv基因列表中基因的评分结果;新发基因获取模块4,用于基于所述评分结果,利用go注释和快速功能注释从所述scnv基因列表中获取肿瘤的致病新发基因;网络构建模块5,用于基于肿瘤核心通路变化因子构建肿瘤致病新发基因调控网络;验证模块6,用于基于所述肿瘤致病新发基因调控网络验证所述肿瘤的致病新发基因。
70.在一种实施方式中,所述基因构建模块1,包括:差异列表生成子模块11,用于根据肿瘤样本的转录组学数据生成差异表达基因列表;聚类子模块12,用于将所述差异表达基因列表中的差异表达基因进行聚类,得到若干基因簇;网络构建子模块13,用于基于所述基因簇的软阈值构建基因簇共表达网络;基因构建子模块14,用于重构所述基因簇共表达网络中的基因,生成共表达基因模块。
71.在一种实施方式中,所述肿瘤样本的转录组学数据为rna-seq的表达量数据。
72.在一种实施方式中,所述rna-seq的表达量数据为rpkm数。
73.在一种实施方式中,所述将所述差异表达基因列表中的差异表达基因进行聚类采用的聚类方法为双向聚类法。
74.在一种实施方式中,所述基因构建子模块14,包括:聚类单元141,用于利用相异度算法对所述基因簇共表达网络中的基因进行层次聚类;聚类树建立单元142,用于基于所述层次聚类结果,建立分层聚类树;基因构建单元143,用于将所述分层聚类树剪切成若干模块并进行模块合并,生成若干个共表达基因模块。
75.在一种实施方式中,所述将所述分层聚类树剪切成若干模块利用动态剪切法。
76.在一种实施方式中,所述进行模块合并具体为,将相关性系数大于0.75的模块进行合并。
77.在一种实施方式中,所述评分模块3,包括:初步评分子模块31,用于利用决策树分析法对所述共表达基因模块中的基因和所述scnv基因列表中的基因进行相关性分析,生成scnv基因列表中基因的初步评分结果;评分子模块32,用于根据cnv的拷贝类型对scnv扩增基因和scnv缺失基因按照评分进行排序,生成scnv基因列表中基因的评分结果;其中,所述评分结果包括scnv扩增基因评分结果和scnv缺失基因评分结果。
78.在一种实施方式中,所述scnv基因列表中基因的评分越高,与所述共表达模块中的基因的相关性越高。
79.在一种实施方式中,所述新发基因获取模块4,包括:第一新发基因获取子模块41,用于利用go注释工具选取第一致病新发基因;新发基因获取子模块42,用于将进行标注的所述第一致病新发基因输入icmdb数据库进行快速功能注释,获取肿瘤的致病新发基因。
80.在一种实施方式中,还包括:临床验证模块7,用于结合临床样本的预后及生存数据,对所述肿瘤的致病新发基因进行验证。
81.以上所述,仅为本技术的具体实施方式,但本技术的保护范围并不局限于此,任何熟悉本技术领域的技术人员在本技术揭露的技术范围内,可轻易想到的变化或替换,都应涵盖在本技术的保护范围之内。因此,本技术的保护范围应以所述权利要求的保护范围为准。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1