使用核酸卡尺的高通量结构测定的制作方法
本申请要求于2014年4月23日提交的美国临时申请No.61/982,914的权益,其全部内容通过引用并入本文。
发明领域
本发明涉及并提供用于在单分子基础上测量分子间和分子内距离的组合物、装置和方法。
发明背景
X射线晶体学和溶液NMR是为大分子结构测定提供强大的工具的成熟领域。尽管如此,结构表征对于许多目标仍然存在巨大的挑战。例如,由多蛋白复合物的非同步群体探索的多样的构象转变可混淆大量分析方法。CryoEM具有单分子成像的优势;然而需要计算平均值来恢复高分辨率结构。对于表现出构象异质性的样品,在低剂量成像下的差的信噪比导致在粒子的类分配过程中的误差,从而损害重建的有效分辨率。因此,对于可补充标准结构生物学方法的新技术存在巨大的需求。这种附加数据的有价值的来源是远程距离约束,因为这些的集合显著简化了用于结构测定的计算方法的构象搜索空间。从短期来看,远程距离约束可用于将从先前测定的x射线或NMR研究得到的良好定义的亚基的对接的模型精细化至更大的复合物。从长期来看,这些数据可用作指导从头计算折叠预测的实验约束的主要来源。单分子FRET是产生远程距离约束的有前景的方法,然而其目前需要大量的半胱氨酸工程化以及复杂的仪器和分析来获得即使适中数量的这些距离。因此,目前不存在用于在单分子水平上低成本、高通量收集远程距离约束的方法。
发明概述
本发明特别地提供了用于目标靶(包括大分子,例如蛋白质、核酸或多个蛋白质和/或核酸的复合物)的高通量结构测定的方法。所述方法包括测量(例如通过力谱法)目标靶上随机选择的点之间的远程距离,然后通过第二测量来鉴定附接点。与用于测量远程距离约束的现有技术方法相比,这些方法提供了大得多的通量并易于实现。这些方法还能够表征通过张力稳定的结构中间体(其原本会是短暂的,因而实际上是不可观察的)。这些方法可用于测定一级序列已知的靶的结构,或者它们可用于解析新设计或分离的靶的结构。
因此,在一个方面,本公开内容提供了一种系统,其包括:
(1)具有靶结构域的单链核酸卡尺,其中所述靶结构域包含与靶夹板互补的非连续核苷酸序列TS1和TS2以及各自与侧接靶的不同单链核酸柄TH1和TH2互补的非连续核苷酸序列TT1和TT2,
(2)靶夹板,其是具有与所述单链核酸卡尺的部分序列互补性(其中参照和靶夹板结合所述单链核酸卡尺的分开的不同序列)和当所述靶夹板结合至所述单链核酸卡尺时保持单链的TS支点序列(toehold sequence)的单链寡核苷酸。
在另一方面,本公开内容提供了一种系统,其包括:
(1)具有参照结构域和靶结构域的单链核酸卡尺,
其中(a)参照结构域包含与参照夹板互补的非连续核苷酸序列RS1和RS2以及各自与侧接参照分子的不同单链核酸柄RH1和RH2互补的非连续核苷酸序列RR1和RR2,和(b)靶结构域包含与靶夹板互补的非连续核苷酸序列TS1和TS2以及各自与侧接靶的不同单链核酸柄TH1和TH2互补的非连续核苷酸序列TT1和TT2,
(2)参照夹板,其是具有与单链核酸卡尺的部分序列互补性和当参照夹板与单链核酸卡尺结合时保持单链的RS支点序列的单链寡核苷酸,
(3)靶夹板,其是具有与所述单链核酸卡尺的部分序列互补性(其中参照和靶夹板结合所述单链核酸卡尺的分开的不同序列)和当所述靶夹板结合至所述单链核酸卡尺时保持单链的TS支点序列的单链寡核苷酸,和
(4)侧接有两个单链核酸柄RH1和RH2的参照分子。
在一些实施方案中,前述系统中的任一个还包含侧接有两个单链核酸柄TH1和TH2的靶。
在一些实施方案中,单链核酸卡尺在第一末端与珠粒缀合。在一些实施方案中,单链核酸卡尺在第一末端与珠粒缀合,并在第二末端与表面缀合。在一些实施方案中,珠粒是微珠。在一些实施方案中,珠粒是磁珠。在一些实施方案中,单链核酸将卡尺附接至固定表面。
在一些实施方案中,所述系统还包含与RS支点序列的序列互补的RS置换链。
在一些实施方案中,系统还包括与TS支点序列的序列互补的TS置换链。
在一些实施方案中,靶是蛋白质。在一些实施方案中,靶是具有已知一级氨基酸序列的蛋白质。在一些实施方案中,靶是具有未知一级氨基酸序列的蛋白质。在一些实施方案中,靶是结合于结合伴侣的蛋白质。
在一些实施方案中,单链柄TS1和TS2在未修饰的表面赖氨酸处附接至靶。在一些实施方案中,单链柄TS1和TS2在突变的表面半胱氨酸处附接至靶。在一些实施方案中,单链柄TS1和TS2在未修饰的表面色氨酸处附接至靶。
在一些实施方案中,靶是核酸纳米结构。
在一些实施方案中,单链柄TS1和TS2各自包含发夹条形码序列和环序列,其中发夹条形码序列在TS1与TS2之间相同,并且环序列的长度在TS1与TS2之间不同。
在一些实施方案中,单链柄TS1和TS2各自包含条形码序列。在一些实施方案中,条形码序列可通过链置换存取。在一些实施方案中,条形码序列存在于嵌套环(nested loop)中。条形码可是线性条形码或嵌套条形码。
在另一方面,本公开内容提供多个任何前述系统。在一些实施方案中,参照分子、参照夹板、RS1、RS2、RH1、RH2、RR1、RR2、TS1、TS2、TT1、TT2、TH1、TH2、TS支点和RS支点在多个系统之间是相同的。
在一些实施方案中,单链核酸卡尺在第一末端附接至表面,并在第二末端附接至珠粒。
在一些实施方案中,多个系统存在于离心力显微镜中。在一些实施方案中,离心力显微镜是反射干涉对比离心力显微镜(RIC-CFM)。
在一些实施方案中,单链核酸卡尺各自包含形成独特长度的环状结构的独特序列。
在另一方面,本公开内容提供了方法,其包括:
(a)在张力下测量当与侧接有单链核酸柄的靶和靶夹板结合时,在第一末端附接至表面并在第二末端附接至珠粒的任何前述核酸卡尺的珠粒至表面距离(BSD-背景),
(b)从所述核酸卡尺移除所述靶夹板,
(c)在张力下测量当与侧接有单链核酸柄结合但不与靶夹板结合时,所述核酸卡尺的珠粒至表面距离(BSD-靶),以及
(d)将BSD-靶与BSD-背景之间的差异确定为当靶处于其天然(非变性)构象时与靶结合的单链核酸柄的附接点之间的距离的量度。
在另一方面,本发明提供了一种方法,其包括:
(a)在张力下测量当与参照分子、侧接有单链核酸柄的靶和靶夹板结合但不与参照夹板结合时,在第一末端附接至表面并在第二末端附接至珠粒的任何前述核酸卡尺的珠粒至表面距离(BSD-ref),
(b)从所述核酸卡尺移除所述靶夹板,并将参照夹板与所述核酸卡尺杂交,
(c)在张力下测量当与参照分子、侧接有单侧核酸柄的靶、参照夹板结合但不与靶夹板结合时,所述核酸卡尺的珠粒至表面距离(BSD-靶),以及
(d)将BSD-靶与BSD-ref之间的差异确定为当靶处于其天然(非变性)构象时与靶结合的单链核酸柄的附接点之间的距离的量度。
在一些实施方案中,所述方法还包括在张力和变性条件下测量当与靶、任选地与参照和参照夹板(如果卡尺含有参照结构域的话)结合时,所述核酸卡尺的珠粒至表面距离,以获得当靶处于其变性构象时与靶结合的单链核酸柄的附接点之间的距离。
在一些实施方案中,靶是蛋白质。在一些实施方案中,靶是具有已知一级氨基酸序列的蛋白质。在一些实施方案中,靶是具有未知一级氨基酸序列的蛋白质。在一些实施方案中,靶是核酸纳米结构。
在一些实施方案中,所述方法还包括在张力下并且在第一置换核酸的存在下,测量当与靶、任选地与参照和参照夹板(如果核酸卡尺包含参照结构域的话)和第一置换核酸结合时,所述核酸卡尺的珠粒至表面距离,以鉴定单链核酸柄与靶的第一附接点。
在一些实施方案中,所述方法还包括在张力下并且在第二置换核酸的存在下,测量当与靶、任选地参照和参照夹板(如果所述卡尺包含参照结构域的话)和第二置换核酸结合时,所述核酸卡尺的珠粒至表面距离,以鉴定单链核酸柄与靶的第二附接点。
在一些实施方案中,张力包括离心力。
在一些实施方案中,使用结合反射干涉对比的离心力显微镜术(RIC-CFM)来测量珠粒至表面距离。
在一些实施方案中,在张力下包括在磁力下。在一些实施方案中,在张力下包括在重力下。
在一些实施方案中,所述柄共价附接至靶。在一些实施方案中,在张力下意指约300-1000pN的力。在一些实施方案中,在张力下意指小于约10pN的力。
在另一方面,本公开内容提供了一种方法,其包括:
(a)在张力下测量当与多单元靶的第一单元的Xaa残基和第二单元的Xaa残基结合时,在第一末端附接至表面并在第二末端附接至珠粒的核酸卡尺的珠粒至表面距离,其中第一和第二单元的Xaa残基附接至相同序列的单链核酸柄,
(b)将第一单元的第二Yaa残基附接至所述核酸卡尺,并在张力和变性条件下测量当与第一单元的Xaa和Yaa残基结合时,所述核酸卡尺的珠粒至表面距离,以鉴定所述第一单元上的Xaa和Yaa残基,和
(c)从所述核酸卡尺解离所述第一单元,将第二单元的第二Yaa残基附接至所述核酸卡尺,并在张力和变性条件下测量当与第二单元的Xaa和Yaa残基结合,所述核酸卡尺的珠粒至表面距离,以鉴定所述第二单元上的Xaa和Yaa残基。
在一些实施方案中,核酸卡尺包含参照结构域,并且当核酸卡尺结合参照和参照夹板时,进行(a)、(b)和(c)的测量。
在一些实施方案中,多单元靶是多单元蛋白质。在一些实施方案中,第一和第二单元是具有已知一级氨基酸序列的蛋白质。在一些实施方案中,第一和第二单元是具有未知一级氨基酸序列的蛋白质。
在一些实施方案中,Xaa是赖氨酸,Yaa是突变型半胱氨酸。
在一些实施方案中,张力包括离心力。在一些实施方案中,使用结合反射干涉对比的离心力显微镜术(RIC-CFM)来测量珠粒至表面距离。
在一些实施方案中,张力包括磁力。在一些实施方案中,张力包括重力。在一些实施方案中,使用链置换使第一单元与核酸卡尺解离。
在一些实施方案中,变性条件是SDS的存在。
在一些实施方案中,所述柄共价连接至靶。
在另一方面,本公开内容提供了一种系统,其包括:
(1)具有成环结构域的核酸卡尺,其中成环结构域侧接有与参照夹板序列RSS1和RSS2互补的序列,
(2)参照夹板,其包含具有第一参照夹板序列RSS1和第二参照夹板序列RSS2的单链寡核苷酸,以及靶分子,其在第一位置处附接至RSS1与RSS2序列之间的参照夹板并在第二位置处附接至第三参照夹板序列RSS3,其中第二和第三参照夹板序列RSS1和RSS3在序列上相同。当与第一和第二参照夹板序列(RSS1和RSS2)杂交时以及当与第一和第三参照夹板序列(RSS1和RSS3)杂交时,核酸卡尺形成两种不同的环状态。
在一些实施方案中,核酸卡尺是部分双链的。
在一些实施方案中,核酸卡尺在第一末端与珠粒缀合。在一些实施方案中,核酸卡尺在第一末端与珠粒缀合并在第二末端与表面缀合。在一些实施方案中,珠粒是微珠。在一些实施方案中,珠粒是磁珠。在一些实施方案中,核酸卡尺附接至固定表面。
在一些实施方案中,靶是蛋白质。在一些实施方案中,靶是具有已知一级氨基酸序列的蛋白质。在一些实施方案中,靶是具有未知一级氨基酸序列的蛋白质。
在一些实施方案中,环状态是双链环状态。在一些实施方案中,在力被减小或移除后,环状态可再生。
在另一方面,本公开内容提供了一种方法,其包括:
(a)在张力下测量当与参照夹板的第一参照夹板序列RSS1和第二参照夹板序列RSS2杂交时,在第一末端附接至表面并在第二末端附接至珠粒的前述核酸卡尺的珠粒至表面距离(BSD-RS),
(b)在张力下测量当与参照夹板的第一参照夹板序列RSS1和第三参照夹板序列RSS3杂交时,所述核酸卡尺的珠粒至表面距离(BSD-靶),
(c)在张力下测量当与参照夹板的第一参照夹板序列RSS1杂交而非与第二参照夹板序列RSS2或第三参照夹板序列RSS3杂交时,所述核酸卡尺的珠粒至表面距离,以及
(d)将BSD-靶和BSD-RS之间的差异确定为靶的长度的度量。
在一些实施方案中,靶是蛋白质。在一些实施方案中,靶是具有已知一级氨基酸序列的蛋白质。在一些实施方案中,靶是具有未知一级氨基酸序列的蛋白质。在一些实施方案中,靶是核酸纳米结构。
在一些实施方案中,在张力下意指在离心力下。
在一些实施方案中,使用结合反射干涉对比的离心力显微镜术(RIC-CFM)测量珠粒至表面距离。
在一些实施方案中,在张力下意指在磁力下。在一些实施方案中,在张力下意指在重力下。
在另一方面,本公开内容提供了一种方法,其包括:
(a)在张力下测量当结合至靶的Xaa残基和第一Yaa残基时,在第一末端附接至表面并在第二末端附接至株粒的核酸卡尺的珠粒至表面距离,其中所述Xaa和Yaa残基附接至单链核酸柄,
(b)使核酸卡尺与第一Yaa残基解离并将核酸卡尺附接至第二Yaa残基,
(c)在张力下测量当与靶的Xaa和第二Yaa残基结合时,核酸卡尺的珠粒至表面距离,以及
(d)重复步骤(a)-(c)。
在一些实施方案中,在张力下意指约300-1000pN的力。
在一些实施方案中,靶是蛋白质。
在一些实施方案中,该方法在变性条件下进行。在一些实施方案中,变性条件包括SDS的存在。
在一些实施方案中,所述柄包含条形码序列。在一些实施方案中,条形码序列可通过链置换存取。在一些实施方案中,条形码序列被嵌套在核酸环中。条形码可以是线性条形码或嵌套条形码。
在一些实施方案中,靶是具有未知一级氨基酸序列的蛋白质。
在一些实施方案中,Xaa是赖氨酸,Yaa是突变型半胱氨酸。
在一些实施方案中,在张力下意指在离心力下。在一些实施方案中,使用结合反射干涉对比的离心力显微镜术(RIC-CFM)测量珠粒至表面距离。
在一些实施方案中,在张力下意指在磁力下。在一些实施方案中,在张力下意指在重力下。
在一些实施方案中,使用链置换使第一Yaa与核酸卡尺解离。
在一些实施方案中,在小于约10pN的力下进行步骤(a)至(c)多次,然后在约300-1000pN的力下进行步骤(a)至(c)多次。
在一些实施方案中,所述柄共价附接至靶。
在一些实施方案中,靶是大分子复合物。在一些实施方案中,大分子复合物是蛋白质组。在一些实施方案中,大分子复合物是转录组。
在一些实施方案中,靶是多糖并且残基是糖。
在另一方面,本公开内容提供了一种方法,包括:
(a)提供多个靶核酸,每个靶核酸包含模板链,
(b)在脱氧尿苷存在下对多个靶核酸进行有限的聚合酶反应,从而产生具有模板链和包含脱氧尿苷的互补链的双链靶核酸,
(c)将多个双链靶核酸暴露于尿嘧啶DNA糖基化酶以产生1个核苷酸的间隙,
(d)将所述多个双链靶核酸暴露于外切核酸酶以加宽所述1个核苷酸的间隙,
(e)将带有条形码的可交联的末端保护的寡核苷酸连接至加宽的间隙中,
(f)将所述寡核苷酸与模板链交联,
(g)在第一和第二带条形码的位置处将单个所得带条形码的靶核酸与核酸卡尺接触,
(h)测量所述第一和第二带条形码的位置之间的距离,并鉴定所述位置处的条形码,
(i)将所述卡尺从所述第二带条形码的位置释放并且使所述卡尺与第三带条形码的位置重新缔合,以及
(j)重复步骤(g)至(i),每次维持所述卡尺在一个位置上与靶核酸的附接,并将所述卡尺在新位置处附接至靶核酸。
在另一方面,本公开内容提供了一种方法,包括:
将包含位置C1和C2的核酸卡尺在位置X1和Y1处附接至靶,由此C1附接至X1并且C2附接至Y1,
在张力和非变性条件下测量X1与Y1之间的距离,
从C2释放Y1,和任选地在卡尺的另一位置C3处附接或维持Y1,
将卡尺上的位置C4附接至靶上的另外的位置X2,并在张力下和任选地在变性条件下测量X1与X2之间的距离,从X2释放C4,并重复直至获得足够的X1的一级序列信息,以及
从C1释放X1,
将卡尺上的位置C5附接至靶上的另外的位置Y2,并在张力和任选地在变性条件下测量Y1与Y2之间的距离,从Y2释放C5,并重复直至获得足够的Y1的一级序列信息,其中靶上的每个位置具有独特的条形码。
在一些实施方案中,C2和C4是相同的位置。在一些实施方案中,C1和C5是相同的位置。
在一些实施方案中,其中条形码是线性或嵌套条形码。
在一些实施方案中,在张力和非变性条件下包括小于10pN的力。在一些实施方案中,在张力和变性条件下包括在300-1000pN范围内的力。
在一些实施方案中,卡尺在第一末端附接至表面并在第二末端附接至珠粒。
在一些实施方案中,卡尺内的连接是共价的。
在一些实施方案中,靶上的位置用单链核酸柄标记。在一些实施方案中,单链核酸柄包含独特的条形码。
在一些实施方案中,靶是蛋白质。在一些实施方案中,靶是多组分靶。
卡尺可以是本文所述的任何卡尺。
在各种前述系统和卡尺的实施方案中,各种序列和结构域之间的连接可以是共价的。
在本文中更详细地描述了本文提供的这些和其它方面及实施方案。
附图概述
图1.卡尺测量远程距离约束的黑盒示意图。
图2A.基于DNA的纳米卡尺CLP-I。夹板链的移除通过微流体引入与该夹板链完全互补的链来实现。如果需要,可将互补寡核苷酸(未示出)对ssDNA主链的暴露区域退火,以使不想要的二级结构减少至最少。
图2B.核酸纳米结构的结构测定。链的微流体引入将组装体从一个状态推动至下一个状态。在状态1中,参照(绿色)(例如,长度已知的dsDNA)被接合并决定“参照”珠粒高度。在状态2中,靶(红色)(例如,DNA纳米结构)被接合并决定珠粒高度。在状态3中,条形码环在面向表面的柄上被打开,从而允许珠粒升高。在状态4中,条形码环在面向珠粒的柄上被打开。珠粒高度的精确上升量揭示了条形码的身份。右方的图是条形码环的结构。链置换导致开环。可使用相同的链打开所有面向表面的柄。可使用相同的链打开所有面向珠粒的柄。用于打开面向表面的柄的链将不同于用于打开面向珠粒的柄的链。
图3.使用基于DNA的纳米卡尺CLP-II测量靶复合物距离约束和鉴定多蛋白复合物的附着位点。掩蔽链的移除通过微流体引入与该掩蔽链完全互补的链来实现。为了简洁和清楚起见,此处省略了整个参照(绿色)结构域以及用于使靶-复合物结构域闲置的外环(如图2A中所示的),但可将其包括在实际装置中。
图4.在离心力显微镜(CFM)上进行的大规模平行单分子测量。CFM示意图。旋转显微成像臂使得能够在观察期间向样品施加离心力。通过拉动经由单一生物分子系连至盖玻片的官能化的珠粒而将力施加于单分子复合物。
图5A-D.3D反射干涉对比显微镜术的概述。(A)具有单色源、偏振器(p1和p2)、分束器(bs)、物镜(o)和四分之一波片(qw)的成像光路的示意图。在底部显示了在三个不同高度处的7.5μm珠粒的RICM图案(10μm的比例尺)。(B)显示操作原理的简单模型。(C)来自微球的径向强度分布和与本文提供的模型的拟合的实例。(D)在不存在力的情况下(下图)和在具有施加的来自两个激光电源处的光学陷阱的张力的情况下(上图)所系连的珠粒的高分辨率3D位置图(Heinrich等,2008)。
图6.力-带有条形码的DNA纳米开关的假定设计,其由在力作用下断裂打开的多个嵌套环组成。这使得能够独特鉴定多达100万个不同的纳米卡尺复合物。
图7A-C.DNA卡尺系统的实验设计。(A)接合参照夹板。(B)接合靶。(C)环被打开。
图8A-D.在双光学陷阱设置中进行的初步实验的实验设计,其中分开地接合夹板的两个不同部分以形成可通过力转变为开环(C)的环(A和B)。(D)来自单个系链的重复测量的环长度测量的直方图。与RICM中的基于干扰的测量相反,此设置由于其基于图像的位置跟踪而具有较低的空间分辨率。
图10A-B.(A)设置中的珠粒底部位置的计算分布,其中用200nm dsDNA系链将珠粒系连至表面。(B)相同设置的模拟数据。
图11.图2B中概述的实验设置针对50nm的参照长度和40nm的靶长度的模拟时序数据。蓝色(外围)点是模拟数据,橙色(中间)点是20帧运行平均值。平稳段代表不同的状态。从左到右(和从最低至最高),它们为如下:状态1代表接合的参照;状态2代表接合的靶;状态3代表解锁和展开的面向盖玻片的条形码;状态4代表解锁和展开的两种条形码。
发明详述
本文提供的方法用于测定目标靶的结构特征(包括完整3D结构)。此类靶包括但不限于核酸纳米结构、蛋白质、多蛋白复合物、蛋白质-核酸复合物等。可通过定向(即,非随机方式)附接至核酸来表面修饰的任何靶可根据本文提供的方法进行分析。
本文提供的某些方法利用纳米级核酸卡尺拉伸个体靶诸如蛋白质和多蛋白复合体,并且在拉伸后使用高通量、高分辨率装置例如但不限于离心力显微镜术测量此类卡尺的距离。重要的是,所述方法可在单分子水平上进行,因此不受某些现有方法的大群体分析的阻碍。
所述方法可用于测量在不同张力水平下拉伸或以另一方式操作(包括例如与结合伴侣的缔合或解离)的靶上的距离。这些方法还可用于对来自先前解析的亚单元组分的大的多组分复合物的动力学进行建模,或在一些情况下可用于预测从头结构。
下面更详细地描述的这些方法可仅用少量的样品(或靶)进行,其中最小限度地制备或修饰此类样品或靶。例如,在蛋白质结构测定的背景中,最小限度的半胱氨酸工程化或无半胱氨酸工程化是必需的。可以以毫秒时间分辨率对每小时数千个加载靶的卡尺或对每小时数百万个加载靶的卡尺收集空间分辨率上的测量(每一个在100ms的时期内进行整合)。
本文提供的各种方法可使用结合反射干涉对比的离心力显微镜术(CFM)(RIC-CFM)来进行。RIC-CFM可用于实现各自附接在珠粒与表面之间的加载靶的个体纳米卡尺的平行高分辨率分析,从而提供高通量和易于实施。RIC-CFM能够实现埃-空间和毫秒-时间分辨率。
一般性的远程距离测量
本文提供的远程距离测量方法的基本逻辑示于图1中。还可参照图2和7,其更具体地概述了该方法。
靶(诸如靶蛋白、核酸或复合物)在其两个表面残基处被紧固至核酸(在本文中有时称为卡尺或纳米卡尺)。核酸本身在一端附接至表面,并在另一端附接至珠粒如微珠。可使用离心力(或者在磁珠的情况下,使用磁力)将珠粒从表面移开。可使用重力从表面移开珠粒。在进行方法期间在不同的时间测量珠粒至表面距离,并且这样的距离的变化用于测定靶上的两个附接点之间的距离,并因此测定靶的结构。
在某些实施方案中,相对于参照状态测量珠粒至表面距离(BSD),如现在所描述的。参照状态是相对其中安装的靶测定BSD的状态的其中安装的校准参照测定BSD的状态。该差异测量可用于推断靶上的两个附接点之间的距离。
本公开内容考虑了其中两个附接点都是已知的情况。例如,附接点可是两个特定的残基,其在蛋白质的一级结构中的位置是已知的。
本公开内容还考虑了其中在已知类型但未知位置的残基处进行附接的情况。例如,附接点可以是两个赖氨酸(因所使用的附接化学),但是这些赖氨酸在靶蛋白的一级氨基酸序列中的位置是未知的。换句话说,在一些情况下,附接点将是赖氨酸,但为靶蛋白中的哪个具体赖氨酸将是未知的。
在这些后面的情况下,将测量在变性条件下延伸至其伸直长度的靶的附加BSD。该距离将取决于一级序列中的间插残基的数目,因此可用于推断附接对的身份(即,在靶蛋白的情况下,一级氨基酸序列内的位置)。重要的是,该变性驱动的附接点的鉴定显著降低了对靶蛋白进行位点特异性标记的需要。
如本文将讨论的,第二种情况也可应用于测定单链(ss)核酸“柄”在核酸纳米结构上的位置和距离。
如本文还将更详细讨论的,该方法还可被扩展至多蛋白复合物,其中复合物中的个体亚单元可表达为单半胱氨酸突变体,每个标记有两个不同的单链核酸“柄”,然后重构成完整的多组分组装体。这允许测定复合物的不同亚单元上的残基之间的距离。
单一单元的靶的高通量结构分析
通过单链核酸柄(为了简洁起见和作为实例,其在本文中可称为ssDNA“柄”)的附接,在特定位点修饰待分析的靶。所述位点可以是靶表面上的位点的子集(例如,所有表面赖氨酸)。
所述方法用于测量附接至靶蛋白上的两个位点的这些短ssDNA“柄”之间的距离。可使用多种化学物质(每种具有氨基酸特异性)将所述柄附接至靶蛋白。作为实例,靶蛋白表面上的两个随机选择的赖氨酸与NHS-官能化的寡核苷酸反应,以形成具有附接至表面可及的随机赖氨酸的两个ssDNA柄的靶蛋白。类似地,其它化学物质可用于附接至其它表面残基。例如,硫醇特异性试剂可用于附接至半胱氨酸,胺特异性试剂可用于附接至蛋白质的氨基末端或附接至赖氨酸,羧基特异性试剂可用于附接至蛋白质的羧基末端或附接至天冬氨酸或谷氨酸,胍特异性试剂可用于附接至精氨酸,咪唑特异性试剂可用于附接至组氨酸,酚特异性试剂可用于附接至酪氨酸,吲哚特异性试剂可用于附接至色氨酸,氨基末端特异性试剂可用于附接至蛋白质的氨基末端,以及羧基末端特异性试剂可用于附接至羧基末端。Basle等,Chemistry&Biology,17:213描述了这样的各种化学物质,并且通过引用并入本文。
第一测量是两个柄之间的距离,其代表两个表面残基之间的距离。第二测量是在变性条件下两个柄之间的距离。该后一测量鉴定那些残基在靶蛋白的一级氨基酸序列中的位置。
称为CLP-I的纳米卡尺的实例示于图2A中。该纳米卡尺由安装在表面与珠粒之间的通过外力拉伸拉紧(例如,以5-30pN牵拉的光学陷阱或离心力)的长ssDNA组成。该链被组织成两个独立的结构域(“靶”(红色)结构域和“参照”(绿色)结构域),每个结构域由嵌套环组成。每个内环的颈部由客体分子桥接。靶内环与靶结合。参照内环与已知长度的校准标准物(诸如但不限于dsDNA)结合。ssDNA柄和纳米卡尺被设计来具有互补序列,从而使得能够在纳米卡尺的特定区域结合靶,从而形成环。靶夹板也被设计来具有与纳米卡尺互补的序列。靶夹板可起到促进靶与纳米卡尺的结合和/或稳定加载靶的纳米卡尺的作用。靶夹板可以例如通过链置换的过程移除,以便测量靶中的距离。
在一些情况下,纳米卡尺包含参照区域,如图2A中所示的。在其它情况下,纳米卡尺不包含参照区域。
当使用具有参照区域的纳米卡尺时,参照分子类似地将具有ssDNA柄。这些柄和纳米卡尺也将被设计来具有互补序列,从而使得能够在纳米卡尺的特定区域结合参照分子,从而形成环。还可使用被设计来具有与纳米卡尺互补的序列的参照夹板。参照夹板可起到促进参照分子与纳米卡尺的结合和/或稳定加载参照的纳米卡尺的作用。参照夹板可例如通过链置换的过程移除,以便测量参照中的距离,从而校准纳米卡尺。
因此,对于每个靶,将存在包括卡尺、待附接至靶(并且具有与卡尺的互补序列)的单链寡核苷酸和用作靶夹板的单链寡核苷酸的系统。另外,系统还可包含待附接至参照(或其部分)(并且具有与卡尺的互补序列)的单链寡核苷酸和用作参照夹板的单链寡核苷酸。另外,系统还可包含参照分子本身。该系统还可包含用于置换靶夹板和参照夹板的单链寡核苷酸。靶和参照夹板的核苷酸序列将是不同的,因此用于置换靶和参照夹板的寡核苷酸的核苷酸序列也将是不同的。类似地,靶ssDNA柄的核苷酸序列将不同于参照ssDNA柄。如从图2A将容易明白的,每个夹板结合卡尺上的非连续序列,从而形成环。类似地,每个靶和参照结合卡尺上的非连续序列,从而形成环。
应当理解,为了简洁起见,术语ssDNA在本公开内容中以非限制性方式使用,并且旨在一般性地表示单链核酸,包括但不限于单链DNA。类似地,术语纳米卡尺在本公开内容中以非限制性方式使用,并且旨在表示具有足以如本文所述起作用的长度的单链核酸。术语卡尺和纳米卡尺可互换使用。
当夹板链存在时,结构域处于“闲置”。这在图2A中示为“状态0”。BSD表示在无来自客体分子的干扰的情况下的卡尺本身的长度(即,所附接的客体的长度不影响结构域或整个卡尺的长度)。
当移除(例如,通过链置换)参照夹板时,BSD的变化表示参照分子上的ssDNA柄之间的距离。这在图2A中被示为“状态1”。通常,参照分子将是已知的,其附接的ssDNA之间的距离也将是已知的。参照分子可以是蛋白质,并且在一些情况下其可被拉伸至其伸直长度。在不存在参照夹板的情况下测量BSD后,则可将参照夹板重新引入并重新与卡尺杂交,从而重新形成参照环。
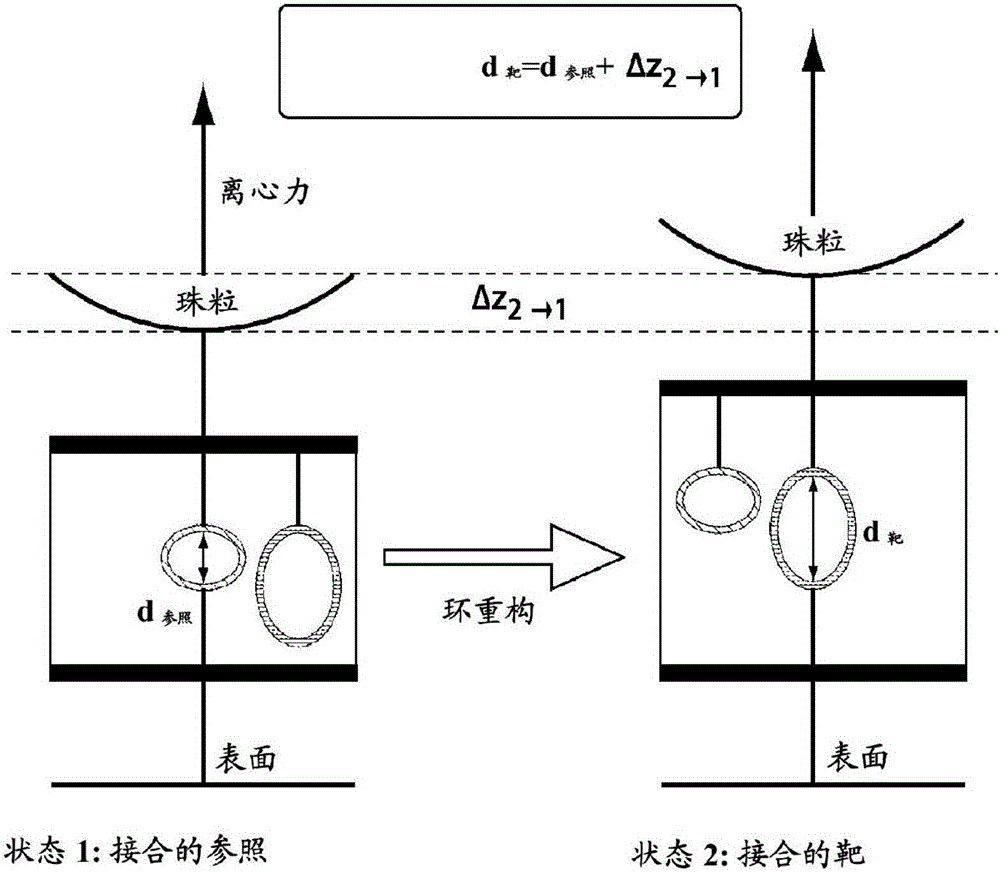
然后移除(例如,通过链置换)靶夹板,并再次测量BSD。这在图2A中被示为“状态2”。可测量状态2(其中仅释放靶(红色)结构域的外环)与状态1(其中仅释放参照(绿色)结构域的外环)之间的BSD中的差异。该差在本文中称为Δz2→1。然后,可如图2A所示,按照测量的与已知距离d参照的偏移恢复表示以其非变性形式存在的靶上的柄之间的未知距离的距离d靶。
因此,在与卡尺混合后,将从每个靶中随机选择两个柄官能化的赖氨酸用于在卡尺上对接。然后,在记录Δz2→1之后,可使靶经历变性条件以形成状态3,如图2A所示。变性条件将取决于靶的性质。蛋白质变性可在例如SDS存在的情况下进行。然后可测量状态3与状态2之间的BSD的差异(称为Δz3→2)。这表示靶在变性后至其伸直长度的延伸。因此,在靶蛋白的情况下,一级氨基酸序列中分开的残基的数目n可推断为:
n=([d参照+Δz2→1+Δz3→2]-2d赖氨酸侧链)/dcα-cα
其中dcα-cα是在施加的外力下延伸的多肽链中的相邻α-碳之间的距离。该间插残基数目n将独特地鉴定赖氨酸对,或者至少将大大限制可能的配对。
如基于本公开内容将理解的,对于单体靶诸如单体蛋白质,不需要靶的半胱氨酸工程化。然而,可使用中间体柄标记方法,其包括产生具有单半胱氨酸突变体的靶,然后将一个马来酰亚胺-ssDNA柄(特异于半胱氨酸突变体)和一个NHS-ssDNA柄(特异于赖氨酸)附接于那些靶中的每一个靶。将不必除去天然半胱氨酸,因为n的测定可用于推断哪个半胱氨酸-赖氨酸对已用ssDNA柄标记。
靶上的任何其它化学不稳定位置(例如,氨基末端或酪氨酸,例如在靶蛋白的情况下)可用作ssDNA柄的附接点。关于可用于靶向靶蛋白中的各种残基的反应性部分或基团,可参见Baslé等,Protein chemical modification on endogenous amino acids.Chem Biol.17,213–227,2010。
核酸纳米结构的高通量结构分析
基于核酸纳米结构的装置显示出许多应用的前景(Pinheiro等,Nat.Nanotechnol.6:763-772,2011)。本公开内容的方法可用于测定核酸纳米结构的原子分辨率结构。类似于前述方面,本公开内容的此方面提供了用于核酸纳米结构诸如使用折纸合成法形成的DNA纳米结构的表面上展示的核酸(例如ssDNA)柄的配对之间的距离的埃分辨率测量的高通量方法。在此方法中,两个柄结合并形成一段长ssDNA的环,其进而悬挂在通过离心力(或磁力或重力)拉开的表面和珠粒(诸如微珠)之间。每个珠粒的静止高度反映了柄之间的距离,并且可每小时恢复数百万个珠粒的位置。这些单珠(和从而单纳米结构)测量提供关于DNA纳米结构的静态和动态异质性的细节。
该方法用于测量在靶核酸纳米结构(NNS)(图2B中的上方的或红色的矩形)表面上展示的两个ssDNA柄之间的距离。为了实现此,将ssDNA柄与单链核酸(本文称为卡尺)杂交,以使得卡尺的一段环出,如图2B所示。然后将卡尺的一个末端附接至表面,并将另一个末端附接至珠粒诸如微珠。在施加力以使珠粒拉伸远离表面之后,可测定珠粒的位置并将其用于推断靶上的两个柄之间的距离。可在校准或参照存在或不存在的情况下进行该方法。图2B显示了包含靶和参照结构域的卡尺(类似于图2A)。参照分子上的柄之间的距离是已知的。此类参照的一个实例是确定长度的dsDNA(图2B中的下方的或绿色的矩形)。
本公开内容考虑了仅单次折叠靶NNS,其中同时展示多个面向表面的柄和多个面向珠粒的柄。然后个体靶与个体卡尺的结合将随机选择单个面向表面的柄和单个面向珠粒的柄。在测定这两个柄之间的距离之后,可通过读出嵌入柄的条形码来识别两个柄的身份(即,各自附接至哪个订书钉链(staple strand))。此处每个柄具有环出的“条形码”结构域,其可例如通过链置换来释放。柄可被设计来使得所有面向表面的柄将用相同的链打开,并且类似地,所有面向珠粒的柄将用链打开。因此,面向表面的柄将具有相同的发夹结构域,但它们将在环出序列的长度上彼此不同。类似地,面向珠粒的柄将具有相同的发夹结构域,但它们在环出序列的长度上彼此不同。正是环出序列的长度“鉴定了”所述柄。在图2B中,首先打开面向表面的柄(状态3),然后打开面向珠粒的柄(状态4)。在触发环的打开后,珠粒可上升至新的位置以占据释放的松弛部分。
为了估计图2B中描述的系统的性能,进行这样的实验,该实验模拟表面结合的珠粒-系链系统(利用系链的现实聚合物模型并且包括珠粒的平移和旋转自由度)的热噪声分布。图9A是对于具有200nm的伸直长度的dsDNA系链、半径为1.5μm的珠粒以及8pN的力,由在室温下的热效应导致的珠粒底部的典型位置分布。可从RICM图案测量的高度的标准偏差被计算为1.8nm。图9B显示了从该分布采样的模拟数据。使用100Hz的帧速率和1秒的观察时间,可能实现小于2埃的平均值标准误差。根据先前的研究,仪器分辨率预期为1-2埃(Wong等,MRS Proceedings第790卷P5 1(Cambridge Univ Press 2003))。另外,还考虑使用先前开发的利用图像模糊分析的成像技术以进一步改善定位(Wong和Halvorsen,Optics Express,14:12517-12531,2006)。
通过使用这些计算,模拟了如图2B所述的完整实验。在图10B中,显示了考虑了分离的dsDNA、ssDNA、参照和靶区域的一组模拟时序数据(数据:蓝色(分散的外围点),20帧移动平均值:橙色(中心点))。这些更详细的计算还确认了图2B中的设计的埃解析能力。
多组分靶的远程距离测量
对于多蛋白复合物,如果两个不同的亚单元附接至卡尺,则变性(例如,利用SDS)将破坏这两个亚单元之间的连接。然而,如果可从重组亚单元重构复合物,则仍然可使用用于鉴定附接残基的变性方法,如图3概述的。在这种情况下,复合物由3个以红色、灰色和橙色表示的亚单元组成。红色和橙色亚单元单独地表达为各自具有独特半胱氨酸(或任何其它独特反应性,例如氨基末端)的突变体。然后将“细红色”马来酰亚胺柄附接至红色亚单元的独特半胱氨酸,并将“粗红色”NHS柄附接至红色亚单元的随机赖氨酸。类似地,将“细橙色”马来酰亚胺柄和“粗橙色”NHS柄分别附接至橙色蛋白质的独特半胱氨酸和随机赖氨酸。所有四个柄具有不同的序列。接下来,将官能化的红色、灰色亚单元和官能化的橙色亚单元重建成完全复合物,然后通过“粗红色”(赖氨酸附接的红色亚单元)和“粗橙色”(赖氨酸附接的橙色亚单元)柄在被称为CLP-II的纳米卡尺上进行对接,以实现状态2,如图3所示的。
可将具有如前述段落中所描述的接合的靶复合物(状态2)相对具有接合的校准参照(状态1,为了简明和清楚起见从图3省略)的珠粒z-位置的比较用于恢复“粗红色”(赖氨酸附接的红色亚单元)柄与“粗橙色”(赖氨酸附接的橙色亚单元)柄之间的距离d靶复合物。
d靶复合物=d参照+Δz2→1
下一步是鉴定附接至“粗红色”(赖氨酸附接的红色亚单元)柄的赖氨酸残基。首先,使复合物变性(例如通过SDS),并且通过链置换将“细红色”(半胱氨酸附接的红色亚单元)柄去遮蔽,随后将其在“粗橙色”(赖氨酸附接的橙色亚单元)柄所结合的位点下方与纳米卡尺对接,以实现状态3。现在,可将“粗红色”(赖氨酸附接的红色亚单元)和“细红色”(半胱氨酸附接的红色亚单元)柄之间的残基数恢复为:
n红色=([d靶复合物+Δz3→2+Δz+]–d赖氨酸铡链–d半胱氨酸侧链)/dcα-cα
其中Δz+是由与状态3或状态4相比在状态2中存在的额外长度引起的校正因子。
最后一步是鉴定附接至“粗橙色”(赖氨酸附接的橙色亚单元)柄的赖氨酸残基。通过链置换将“细橙色”(半胱氨酸附接的橙色亚单元)柄去遮蔽并将其对接至“粗红色”(赖氨酸附接的红色亚单元)柄上方的位置,然后通过链置换完全移除红色靶以实现状态4。现在,“细橙色”(半胱氨酸附接的橙色亚单元)和“粗橙色”(赖氨酸附接的橙色亚单元)柄之间的残基数目可恢复为
n橙色=([d靶复合物+Δz4→2+Δz+]–d赖氨酸侧链–d半胱氨酸侧链)/dcα-cα
力触发的DNA卡尺
本公开内容还考虑和提供了另一种DNA卡尺设计,其可用于使用力触发的可重构DNA系链来进行大分子距离测量。该实施方案不需要链置换来起作用,因此其允许用我们现有的单分子仪器进行DNA卡尺测量,而不实施集成的流体交换系统。该方法的一个示例示于图7A-C中。简言之,将珠粒连接至表面的DNA系链可形成两种不同的可能的环状态:其中靶分子(或靶)在力线内的环状态,和其中靶分子不存在的环状态。通过施加力打开这两种不同的环状态,并且比较这两种状态之间的长度变化,可测定靶分子上的两个附接点之间的距离。
如图7A-C所示,向珠粒施加静止向上的力(~8pN)以拉伸DNA系链。存在附接于系链的结构,其由两个不同序列(以蓝绿色显示)中的ssDNA夹板组成,所述夹板通过互补杂交闭锁系链上的两个区域以形成环。(该结构在图7A中显示为与环的上游和下游的序列杂交的两个序列)。靶分子(红色矩形)附接至两个序列之间的夹板的内部点。靶分子上的另一点具有ssDNA柄(以红色显示,并且在本文中称为靶柄),其在序列上与夹板的一侧(以绿色显示)相同。与靶分子或其部分结合的ssDNA柄在图7A中显示为从矩形延伸,因此不与系链杂交,并且在图7B中与已置换链(现显示为延伸和未杂交的)的系链杂交。通过执行以下步骤进行测量:
步骤1:当系链处于环状态时测量珠粒至表面距离(z1或z2)。
步骤2:通过施加力,环被破坏,并且对于非环状态再次测量珠粒至表面距离(z3)。
步骤3:将这两个测量之间的差作为此循环的环长度测量(ΔL,参见图7A-C中的公式)。
步骤4:松弛系链并重新形成环以重复该循环。将随机地附接靶柄或夹板的一侧。
分析:解析环长度测量的两个群体(ΔL1和ΔL2),并且平均值之间的距离给出靶的长度(即,矩形的长度)。珠粒在每个循环达到高度z3,这为终端用户提供了检查测量之间偏移的能力。
作为前述设计的变型的初步实验使用比图7A-C中所示的空间分辨率设置低的空间分辨率设置(即,使用比使用RICM可实现的分辨率低的分辨率)来进行。实验的设计提供在图8A-C中,并且数据提供于图8D中。简言之,已在由两个光学陷阱保持的两个珠粒之间捕获了M13DNA形式的系链。所述系链在一个末端通过生物素-链霉抗生物素蛋白相互作用并在另一个末端通过地高辛-抗地高辛抗体相互作用附接至珠粒。系链上的成环ssDNA夹板由三部分组成:与系链上的某一区域互补的40个碱基长的锚定部分(以青色显示,最左侧序列)和与系链上的不同区域互补的两个相同的20个碱基长的部分(以红色和绿色显示,中间和最右边的序列)。将系链的其余部分用互补的DNA片段平铺以除去任何二级结构(成环夹板的5'和3'序列)。
如下进行实验:
步骤1:当系链处于环状态时,测量珠粒之间的距离(D1或D2)。
步骤2:通过施加力(~50pN),夹板的20个碱基对的一侧被脱离以测量系链的全长(D3)。
步骤3:将这两个测量之间的差作为该循环的环长度测量(ΔL,参见图8A-C中的公式)。
步骤4:将珠粒移得更近,以松开系链,这使得环通过杂交20个碱基长的片段之一来快速(~30秒)重新形成。
该循环可每系链重复多次(例如,10-20次或更多次),或直至在结构中(通常在其它地方)观察到结构失效。通过使用共价附接可避免此类结构失效。图8D是来自这些初步实验之一的长度测量的直方图。如可看出的,即使利用如此少量的测量,也可分辨相隔约15nm的两个峰,这是在该特定双珠粒光学陷阱设置的~2nm的预期分辨率内的20个碱基对ssDNA分子的预期长度。该分辨率可通过使用RICM来增加,该系统例如在公开的专利申请US 20130288349中进行了描述。该实验的结果还揭示了在所使用的特定结构内的空间效应,其导致一个部分相对于另一个部分更可能的结合。
条形码
本文所述的核酸系统可称为“DNA-操纵的卡尺”或DPC。DPC可用于各种应用。在描述这样的应用之前,提供DPC的以下简要功能描述:
DPC的操作涉及循环两个原始动作:
(1)重构(经由链置换或其它扰动)以将靶(或分析物)的新取向接合至受力路径中,并将其它靶(或分析物)脱离至每个设备的非受力路径或分支中,和
(2)对于每个装置的受力路径的力-延伸曲线的高通量、高分辨率采样。
DPC可用于测定关于生物聚合物及其复合物的三类信息:
(1)1D序列指纹,其是侧链类型确定的残基在生物聚合物链的一级序列内的一组成对距离(例如赖氨酸至赖氨酸的距离,半胱氨酸至赖氨酸的距离);
(2)3D表面指纹,其是天然靶表面上的残基之间的一组成对距离,以及这些残基中的每一个的一级序列身份的鉴定;和
(3)条形码读取,其是嵌入附接至靶分析物的ssDNA条形码内的信息的解码。
如下所述,3D表面指纹分析通过条形码读取以及通过1D序列指纹分析来辅助。
指纹分析通过在随机取样的残基上利用用作卡尺的潜在附接点的ssDNA柄标记靶来介导。ssDNA柄可额外地包括对信息诸如其所附接于的残基类型(例如,半胱氨酸、赖氨酸等)进行数字编码的DPC可解码的条形码。另一种重要的信息类型是随机选择的独特条形码,其可用于鉴定先前由卡尺采样的柄(类似于在穿过迷宫时在交叉点处掉落的独特着色的旗)。其它的信息类型可包括靶的历史,诸如在重组产生其的情况下其具有什么突变(即,其相对基因型),它经历了什么样的环境条件(例如,在过去经受了应激),其在何时何地被标记等。
1D序列指纹可通过双腿分子爬行器(即,卡尺)来测定,所述爬行器随机地抓取单个链上的两个柄,用相对高的力(例如,300-1000pN)拉动以将该间插片段拉伸至近似其伸直长度,然后报告该长度以使得能够推断一级序列中的距离。残基类型(例如半胱氨酸、赖氨酸等)可通过解码嵌入柄中的条形码来读出。此时还可读出独特的标识符条形码。然后爬行器释放一个柄,并随机抓取同一生物聚合物链上的另一个柄,并且重复该循环。这样,可获得大量的等同于链的部分序列的一级序列相关性。
3D表面指纹可通过随机抓取靶上的两个柄的双腿分子爬行器(即,卡尺)来测定,然而这次它使用相对低的力(即,小于10pN,包括例如大约8pN),以使得其不会使靶变性。此时也可读出独特的标识符条形码。然后,爬行器释放一个柄,随机抓取同一靶上的另一个柄,并重复该循环。这样,可对在单个靶的表面上采样的点进行大量的成对距离测量。在随后的阶段中,在相同的靶上以较高的力重复分析以测定复合物中每个成分链的1D序列指纹。在1D指纹分析阶段期间读出的标识符条形码的索引允许在3D表面指纹分析期间抓取的每个柄的序列鉴定的分配。应注意的是,卡尺将仅能够获得在使组分靶链变性的条件下操作时其不释放的链上的1D序列指纹。因此,卡尺应当具有多个臂以保持多个链,以使得可通过1D序列指纹分析依次分析给定靶的许多链。
因此,作为实例,本文所述的卡尺可用于使用两步法来对靶(诸如多组分靶)作图。首先,允许卡尺本身随机地附接至靶上的第一位置X1和第二位置Y1。卡尺在C1和C2(即,C1和C2是卡尺上的位置或部位)处结合至X1和Y1。通常在非变性条件下测量X1与Y1之间的距离。位置X1和Y1中的每一个可使用条形码诸如线性或嵌套条形码来鉴定。卡尺与Y1脱离,同时保持其与X1的附接。卡尺可完全或部分地从Y1脱离。部分脱离意味着卡尺从C2卡尺位置释放Y1,但是卡尺不完全释放Y1,而是替代地在另一卡尺位置C3处接合Y1。然后使用卡尺来以顺序方式从X1或Y1开始接合另外的位点。例如,卡尺保持其与X1(通过C1)的附接,然后结合另外的位置X2、X3、X4、X5等,并且测量X1与这些位置中的每一个之间的距离。这提供了关于X1位置周围的一级序列的信息。然后,卡尺结合相对于Y1位置的另外的位置(即,另外的位置Y2、Y3、Y4、Y5等),并且测量Y1与这些另外的位置之间的距离。这提供了关于Y1位置周围的一级序列的信息。X1与Y1之间的初始测量可在维持复合物的天然状态的条件下进行。可在变性条件下(例如,通过使变性剂流过反应室)进行X1与X2、X3、X4、X5等之间以及Y1与Y2、Y3、Y4、Y5等之间的随后测量。
逐步伸长条形码:此处描述了条形码结构,其可作为ssDNA链的一系列预编程的长度增加读出,每个长度增加通过置换链的流体引入来驱动。这示于图11中。一种结构是长支架链的一系列区段,每个区段通过订书钉链环出。我们将在每个区段上通过流体引入分别环出该区段的一半长度或整个长度的订书钉链编码“0”或“1”。例如,假设区段长度为100nt。则定义桥接两个20nt的子区段并具有60nt的端环的“1”订书钉链,从而将末端至末端的区段距离减小到0nt加上桥的跨度。然后定义桥接另外两个20nt的子区段并具有10nt的端环的“0”订书钉链,所述10nt的端环嵌套在先前定义的60nt的端环内;该“0”订书钉链(“1”订书钉链不存在)将末端至末端的区段距离减小至50nt加上桥的跨度。每个订书钉链具有独特的序列,因此每个环可通过流体引入与订书钉链的一半互补的置换链来独立地打开,以及通过经由流体引入与置换链互补的恢复链移除置换链来重新闭合。
例如,假设存在n个串联独特的区段。则对于每个区段,存在独特的“0”订书钉链和独特的“1”订书钉链。为创建特定的条形码,对于每个区段,选择“0”或“1”订书钉链以包括在折叠中。则存在2n个潜在条形码。通过连续引入置换链同时观察长度的变化来读出条形码。任何时候,订书钉链介导的桥接只有当该订书钉链已在其初始构建期间被包含在条形码上时才可能被破坏,然后可观察到长度增加。
简化的替代设计通过订书钉链的存在来表示“1”,以及通过其不存在来表示“0”。
一种替代读出方法是使用力而非链置换来触发条形码内的长度变化。每个比特(环+订书钉链)可设计成使得订书钉链在特定的力水平(利用例如从最低有效比特至最高有效比特所需的递增水平的力)下断裂。通过使每条订书钉链在一侧上比在另一侧上更强(使得能够在力减小时重新退火),可使读出过程可逆。该设计的一个优势是不需要流动来读出条形码,从而使得该条形码能够与标准单分子力探针仪器一起使用。
第二种考虑的结构包含嵌套环。这也示于图11(右图)中。与第一设计一样,条形码可由于链置换而读出为一系列长度增加。这些嵌套环可采用大环的形式,其中多个订书钉链以不同的尺寸闭合环。每个订书钉链的存在或不存在可编码“1”或“0”。与第一结构一样,读出可使用链置换(以依次探测每个比特)或者使用力(将环从最低有效比特拉开至最高有效比特)来实现。
与常规的基于DNA的条形码不同,信息不直接存储在条形码的序列中,而是存储在其几何形状中。因此,不需要DNA合成和测序来写入和读取条形码。相反,杂交是写入条形码所全部需要的,并且观察几何形状和长度的变化是读取条形码所全部需要的。
创建逐步伸长条形码的组合文库:
可使用拆分和组合合成方法来创建条形码的文库。首先,将支架链附接至珠粒。然后对于每个区段,将珠粒库分成两份,并且将“0”订书钉链添加至一个子库,并将“1”订书钉链添加至另一个子库。然后从每个子库洗去多余的订书钉链,并将子库组合在一起。对于每个区段重复拆分和组合循环。
或者,对于其中“1”表示订书钉链的存在并且“0”表示其不存在的设计,可通过将条形码与订书钉链的集合混合(以使得每个条形码仅结合至订书钉链的子集)来随机生成条形码的集合。如果制备足够的独特组合,这将足以独特地鉴定给定的大分子上的每个柄。其可类似于井号标签(hashtag),具有小但非零概率的两个相同的带有条形码的柄终止于同一分子上(被称为“碰撞”)。
成像和测量技术
BSD可使用多种技术(包括离心力显微镜术(CFM)、磁力镊、前向散射照明、光镊、声镊等)测定。在公开的专利申请US20130288349及其母专利中更详细地描述了CFM,这两篇专利文件通过引用并入本文。在图4中示意性地显示了CFM。CFM可用于并行执行数千个单分子力实验。
CFM的一种变型(称为RIC-CFM)将反射干涉对比显微镜术(RICM)整合至CFM中。用于以亚纳米(埃)精度跟踪3D中的单个微球的成像光学器件、光学模型和算法示于图5中。通过将必要的光学器件整合至CFM中,该方法提供了许多珠粒的同时亚纳米级分辨率跟踪,包括在100Hz下以2埃分辨率跟踪多达400个珠粒(例如,与现代的400万像素、100Hz sCMOS相机一起),或者使用高速、低分辨率照相机在10,000Hz或更高下以2埃分辨率跟踪1个珠粒。
另外,可将所使用的CFM或任何成像技术与流体控制系统(诸如微流体控制系统)耦合,以促进引入和去除本文所述方法中使用的核酸和变性剂。此外,可向CFM添加高速平移台以实现样品的快速扫描。
本公开内容还考虑了扩展高分辨率CFM测定法以产生用于表征纳米卡尺构建体的状态的大规模多重平台。虽然CFM测定固有地是高度并行的,但为了在单一测定中表征许多不同的纳米卡尺构建体(例如,不同的独特的半胱氨酸突变体),而不是相同构建体的多个拷贝,该方法提供了鉴定每个独特相互作用的手段。这通过使用利用预编程的DNA纳米开关的力-延伸行为的条形码技术来完成。
构建了DNA纳米开关构建体的大家族,每个构建体可通过其力-延伸行为来独特鉴定。在其最简单的形式中,可通过添加与纳米卡尺构建体串联的不同大小的环来产生该构建体家族,所述环被设计来在施加规定的机械力下打开。可使用例如CFM来区分不同的环大小。或者,可用被设计来在增大的力下打开的嵌套环的集合代替单个DNA环(图6)。该方法将使得能够在CFM中实时观察数百万个独特的条形码(例如,如果可区分多于100个不同的环大小,则三个嵌套环将实现大约106个不同组合)。该嵌套环结构可用作本文所述的CLP-I卡尺的替代设计。可将校准环与靶蛋白并联而非串联放置,并且卡尺结构的重构可通过力介导的链融合而非链置换来触发。
靶
可使用本公开内容的方法分析多种靶。对靶的唯一限制是其必须适于与核酸直接或间接结合。靶可以是但不限于蛋白质、多肽、肽、核酸、病毒样颗粒、类固醇、蛋白聚糖、脂质、碳水化合物及其类似物、衍生物、混合物、融合物、组合或缀合物。
一类靶是基于肽的靶,诸如(单链或多链)蛋白质和肽。基于肽的靶的实例包括但不限于抗体、单链抗体、抗体片段、酶、辅因子、受体、配体、转录因子和其它调节因子、一些抗原(如下所论述的)、细胞因子、趋化因子、激素等。
在一些实施方案中,可分析无机或合成试剂。此类无机或合成试剂包括无机非颗粒和合成聚合物。
靶的修饰
靶(例如多蛋白复合物)的表面可用ssDNA柄共价装饰以产生与卡尺的附接点。碳二亚胺活化随后与胺反应可用于天冬氨酸和谷氨酸残基的特异性修饰,尽管优选地预先消耗或保护赖氨酸以防止不期望的交叉反应。Mendoza和Vachet,Probing Protein Structure by Amin Acid-Specific Covalent Labeling and Mass Spectrometry;Mass Spectrom Rev,28(5):785-815,2009,报道了如下8种残基的氨基酸特异性修饰的方法:
A.精氨酸(例如与苯甲酰甲醛反应)
B.羧化物(天冬氨酸和谷氨酸)(例如通过碳二亚胺活化,然后与氨基-氧基反应)
C.半胱氨酸(例如与马来酰亚胺反应)
D.组氨酸(例如与焦碳酸二乙酯反应)
E.赖氨酸(例如与NHS酯反应)
F.色氨酸(例如与2-羟基-5-硝基苄基溴反应)
G.酪氨酸(例如与四硝基甲烷、碘或N-乙酰基咪唑反应)
如果仅需要分析变性状态下的靶,则可在变性条件下,例如在6M GuCl、8M尿素或1%SDS的存在下进行利用ssDNA柄的装饰。因此,包埋在天然结构内部的位置可用于标记。
应用和用途
本文提供的方法可用于测定(或作图)一级氨基酸序列已知的蛋白质的表面结构。在这些情况下,与靶蛋白的附接点是已知的(由于用于附接的试剂的已知反应性和特异性)。例如,将知道附接点是赖氨酸,因为将使用NHS反应基团来附接。最初,当靶蛋白处于其天然构象时,测定赖氨酸之间的距离。该距离用于对靶蛋白的表面结构作图。将不知道牵涉哪个特定的赖氨酸,因为与靶蛋白的附接是随机的并且可以是可用于反应的任何表面赖氨酸。然而,靶蛋白随后在变性条件下(诸如但不限于在SDS的存在下)被拉伸,从而允许测定当靶蛋白变性时两个赖氨酸之间的距离。然后将该后一种距离用于通过与已知的一级氨基酸序列比较来鉴定牵涉哪个赖氨酸。
所述方法还可用于测定(或作图)一级氨基酸序列未知的蛋白质的表面结构。可使用与上述类似的方法,除了过程的更多重复可能是必要的。在该过程中,还将部分测定一级氨基酸序列。
鉴定靶的能力允许在样品中或作为事件的结果来确定其存在。因此,所述方法可用作确定靶的存在(或不存在)的检测或诊断方法。这可能具有广泛的用途,包括临床用途。
所述方法还可用于测定作为结合到已知或未知结合伴侣的结果的靶的结构变化,或测定响应于所施加的力的结构变化。通常,当处于未结合状态时,靶结构是已知的或确定的。结合伴侣的实例包括推定的药物候选物,例如变构抑制剂或激活剂(例如,酶诸如激酶的激活剂)。以这种方式,所述方法可用于大规模平行药物筛选测定。
所述方法还可用于测定作为结合至已知或未知结合伴侣的结果的结构对靶的变化,或测定响应于所施加的力的结构变化。通常,当处于未结合状态时的靶结构是已知的或确定的。结合伴侣的实例包括假定的药物候选物,诸如变构抑制剂或激活剂(例如酶诸如激酶的激活剂)。这样,所述方法可以用于大规模并行药物筛选测定。
所述方法还可用于单颗粒检测或蛋白质组学,例如鉴定病毒。它们可以进一步用于合成生物学装置(诸如人工蛋白质机器)的快速结构表征。更具体的应用描述如下。
单细胞蛋白质组学
DPC可用于解决细胞生物学的关键技术挑战:单细胞蛋白质组的随机取样的计数、时空跟踪和结构测定。目前,这可在单个细胞中对于个体核酸在一定程度上进行(由于它们的基于探针杂交或测序的鉴定)。另一方面,对于蛋白质,质谱或其它方法不能在蛋白质组规模上提供任何接近单分子的灵敏度。相反地,DPC可通过在每个多肽(或核酸)上收集1D序列指纹来扩展蛋白质组规模上的蛋白质的单分子鉴定和计数。这包括在单分子水平上测量个体蛋白质上存在的任何翻译后修饰(或个体RNA上的转录后修饰),所述蛋白质可通过ssDNA柄特异性标记;例如,丝氨酸/苏氨酸磷酸化可通过在碱性pH下的β-消除,随后通过硫醇标记的ssDNA柄的迈克尔加成来靶向;去乙酰化的赖氨酸可通过NHS标记的ssDNA柄来监测;抗体或适体也可用于指导ssDNA柄与靶位点的连接。此外,DPC可通过收集每个靶的3D表面指纹,不仅返回有关蛋白质组(和转录组)中的个体大分子复合物的身份的信息,而且还返回关于其天然结构的信息,从而还可监测与化学组成无关的构象异质性。因此,DPC可实现单靶、单细胞蛋白质组学(包括关于大分子构象的信息)。
翻译后修饰的单分子指纹分析本身就是前所未有的和有价值的应用本身。批量方法提供了翻译后修饰而非单生物聚合物链上的相关修饰的统计图。
空间解析的单细胞蛋白质组学
大量随机选择的靶(每个用独特的ssDNA标签随机地加条形码)可使用DPC在单细胞内通过空间和时间跟踪。时间分辨率也可通过使用时间编码条形码的脉冲标记来实现。可使用随机选择的条形码作为独特标识符来标记单细胞内的蛋白质,并且使用超分辨率显微镜术跟踪这些条形码在空间和时间中的位置。然后可从该细胞中提取所有蛋白质,然后使用DPC来鉴定每个条形码及由该条形码标记的靶的3D表面轮廓,以及存在于该靶中的多肽的1D序列指纹。因此,时空解析的单细胞蛋白质组学可通过DPC来实现。
单分子核酸测序,包括重复区域的测序
本文提供的卡尺和方法可用于核酸(例如DNA)测序。以下是可用于对核酸测序的方法。测序方法可用于检测和鉴定基因组的“黑暗区域”。基因组的“黑暗区域”是对DNA测序仍然不可适用的区域,通常是因为它们具有高水平的重复序列。DPC可用于单分子DNA测序中的两个关键操作,特别是用于读取重复区域。这些关键操作是(1)读取附接至靶DNA的带条形码的序列标签之间的精确距离;和(2)读出带条形码的序列标签的序列身份。这可如下实现:
1.提供一群(或多个)靶DNA
2.进行一轮(或循环)的聚合酶复制,其将脱氧尿苷掺入一条链,但不掺入另一条链
3.通过用尿嘧啶DNA糖基化酶处理核酸引入1nt的间隙
4.用外切核酸酶加宽一个或多个间隙
5.将带条形码的、可交联的、末端保护的5聚体连接入一个或多个间隙
6.将所述5聚体与模板链交联以产生条形码镶嵌的靶DNA
7.将条形码镶嵌的靶DNA捕获在卡尺上(即,每个卡尺随机抓取两个条形码)
8.读出条形码之间的距离,然后读出每个条形码的身份
9.释放一个条形码,抓取另一个条形码,然后重复;这样,获得单个条形码镶嵌的靶DNA上的许多条形码的成对距离和身份
10.如果需要,通过对5聚体的末端去保护,然后连接另一组,来进行带条形码的5聚体的另一轮连接
DPC可用于将条形码定位至每1kb的1bp的精确度。这可例如通过施加大的力(例如,高达1nN或更高,特别是在干燥条件下,诸如在空气或有机溶剂中,以防止力偶合水解)来实现。通过这样做,有可能实现5聚体条形码之间的距离的0.1%精确度(例如,1千碱基靶上的1个碱基内)。
多糖的单分子指纹分析
在此应用中,多糖内的个体糖(或糖类)单体用其中嵌入有编码关于糖类型、随机选择的标识符等的信息的条形码的柄标记。然后,测量柄之间的成对距离。
使用指纹分析的靶鉴定
DPC可用于通过产生样品内靶的距离指纹来测定目标靶(包括小的目标靶)诸如蛋白质、DNA构建体、病毒、其它大分子等的身份和/或量。每个指纹将由在单个靶上进行的多个距离测量组成。这些测量可以以与先前对于结构测定应用所描述的类似的方式进行。例如,可进行以下步骤:
i)将多个核酸(例如,DNA)柄附接至样品内的目标靶,其中每个柄潜在地包括条形码;
ii)将柄-标记的靶附接至DPC构建体;
iii)将靶偶联的DPC构建体作为系链附接在珠粒与表面之间以实现单分子距离测量;
iv)通过柄-附接、距离测量和柄脱离的重复循环在每个卡尺上进行多个距离测量。
这些步骤不需要按上面所示的顺序进行。例如,可将卡尺在附接至待鉴定的靶之前附接至珠粒。
与上文针对结构测定所描述的相比,此应用是简化的方法,部分因为其不需要从每个样本获得同样多的信息,因为目标不是测定从头结构,而是简单地鉴定靶(即,结构或“指纹”可能是已知的)。此外,计算要求远低于结构测定的要求,因为该方法需要将每个样品靶的距离指纹与指纹数据库进行比较来鉴定靶。鉴定数据库可通过实际上使用本文提供的方法对广泛多样的已知靶进行测量或通过基于已知结构的计算方法来产生。
可对折叠的天然结构以及对变性结构进行蛋白质指纹分析和鉴定。当对变性蛋白质进行指纹分析时,以多种方式放松实验要求:(1)由于蛋白质已被变性,因此不用关注使用在执行本文所述的方法中施加的力使其变性。这允许使用甚至更大的力,这又可减少热噪声。(2)分辨率要求较低,因为单氨基酸分辨率(~3-4埃)是所全部需要的,并且可以以甚至更低的分辨率进行概率鉴定。(3)可使用更宽范围的缓冲剂和环境条件(例如盐、pH、温度等),其中可能包括一些变性剂诸如SDS或尿素以保持肽变性。(4)即使蛋白质被片段化,我们仍然可潜在地使用与用于质谱蛋白质组学的类似算法来鉴定蛋白质。
由于本文提供的方法在单个分子水平上起作用,因此可在非常小的体积上进行靶鉴定和性质分析,所述非常小的体积包括i)来自单细胞的裂解物,和ii)小体积样品,诸如但不限于小体积的体液诸如血液、尿液和唾液。因此,DPC可应用于单细胞蛋白质组学和超低体积检测。
还应当注意,本文所述的所有链置换致动可通过将支点结构域掺入要移除的链中来介导。
高通量单分子大分子结构测定
通过DPC测量的大分子复合物的3D表面指纹可用于精细化这些复合物的骨架结构模型,或在一些情况下可为从头骨架结构测定提供足够的实验约束,而无需任何额外的实验数据。此外,可测量在成对的表面点处施加的力的刚度和力依赖性构象转变,并且以这种方式可获得与大分子复合物相关的额外信息。
因为DPC是高通量的,所以其可用于测量靶的复杂混合物的3D表面指纹。例如,可产生蛋白质的重组形式的文库,每个具有不同组的突变,每个具有附接的编码关于基因型的信息的条形码。此外,可在变化的环境条件(诸如pH、温度、盐浓度、去垢剂的存在、变性剂的存在、外部场、不同配体的存在、大分子结合伴侣的存在)下重复混合物的3D表面指纹分析。这仅对于具有足够高的通量的方法(如DPC)是可能的。
如部分地在本文中所述的,结构测定考虑了将卡尺附接至靶上的各种柄。可在附接至相同蛋白质的柄之间进行几个这样的测量。柄可包括条形码,以使得每个柄可被独特地鉴定和定位。当蛋白质的结构是已知的时,可将结合位点映射至该已知结构。此外,由于可使用DPC获得的高分辨率结构细节,有可能分析和鉴定大分子(或复合物)群体中的结构异质性。
分析的高通量性质也有助于获得关于文库的结构信息,例如,每个单或双重突变,特别是因为可能对文库的每个成员的身份加条形码。还存在可被改变以便进一步分析的变体。这些包括盐、温度、压力、配体、化学修饰、结合伴侣、降解,力等的变化。
本文提供的方法还可用于拟合结构模型。此类模型可使用结构测定过程例如但不限于本文提供的那些方法来生成。然后可针对使用本文提供的方法获得的实验数据对此类模型评分。该过程可产生额外数据以针对静态结构中不存在的进行评分,例如其可提供与靶诸如蛋白质或大分子结构的折叠和解折叠相关的信息。使用本文提供的方法产生的数据还产生关于靶在力或其它环境条件下的响应和顺应性的信息。这些是不能使用静态结构分析来研究的性质。基于与可使用本文提供的方法测量的材料性质拟合,还可能更好地区分正确的静态模型与诱饵静态模型(即,相似的但具有微小缺陷的模型)。
足迹分析
如本文所使用的,足迹分析是允许鉴定靶上的不可用于被探针(诸如DPC中使用的柄)修饰的区域的方法。通过观察哪些残基不能被柄标记,可推断什么残基不可用,可能是因为它们位于靶的内部,或可能由于化学阻断(例如赖氨酸的乙酰化)。DPC使得能够进行单分子足迹分析,从而鉴定单个靶中或其上的隐藏残基。
映射变构界面和转换
本文提供的方法还可用于研究和/或鉴别由施加力或任何其它扰动(例如配体、盐、温度等)诱导的构象变化。这允许鉴定不同的构象状态及其诱导物和使其相关联。所述方法还可用于研究和/或鉴定靶的一个或多个点突变的效应包括结构效应。这进而可用于鉴定治疗靶上的用于变构药物或药剂的位点。
单分子拉下:测定配体的结合配伴侣,以及形成的复合物的结构
药物发现的重要挑战如下:(1)确定药物的治疗靶(包括脱靶)的身份,(2)测定药物:靶相互作用的动力学和热力学,(3)测定药物:靶复合物的结构,(4)测定靶的药物诱导的构象变化。
为了确定靶的身份,典型的策略是将药物偶联至固体支持物,结合靶,洗脱靶,然后使用质谱或相关方法鉴定靶。然而,该方法不能在单分子检测限上鉴定靶。其也不提供有关其余挑战的信息。另一方面,DPC能够实现所有四个步骤。在DPC中,卡尺被系连至药物和靶上的随机位点,测定这两个附接之间的距离,然后鉴定靶和靶上的随机位点。如本文的通过指纹分析的靶鉴定部分中所述,可使用以小的力或大的力附接和拉伸的多个回合来分别测定3D天然指纹或1D序列指纹。为了实现此,可用核酸(例如DNA)柄标记药物。或者,如果对药物-靶结合的干扰是个问题,则药物可用较小的标签(例如,叠氮化物点击标签)以及用于将药物与靶交联的可交联部分(例如,胺反应性系链)来标记。在通过系链将药物与靶交联后,柄可被附接至叠氮化物点击标签。
超分辨率显微镜术
DPC还可用作超分辨率成像的标准方法的替代方案。与使用荧光团的定位来鉴定目标特征的位置不同,通过核酸(例如DNA)柄来跟踪附接至目标特征的小珠粒的位置。与标准荧光测定相比,这提供了许多优势。可以以3D中的高分辨率测量珠粒的位置。我们已证实了每100fps视频帧x和y中的~1nm以及z中的~0.2nm的珠粒跟踪分辨率,这超过了现有的超分辨率技术的分辨率,这是由于珠粒跟踪相对于荧光成像的更高的信噪比、无漂白等导致的。每个珠粒可通过从一个DNA标记位点脱离和附接至另一个DNA标记位点的多个循环来报告许多不同的DNA标记位点的位置。
此外,可在施加力(或改变力)的情况下测量珠粒位置,这可用于减小珠粒的热噪声,从而增加分辨率,以及用于测量所观察的物体的顺从性和力依赖性变形。还可将不同的条形码整合入柄中以使得能够鉴定和定位不同类型的位点。此外,由于可创建和鉴定大量的不同条形码(例如,超过100万个),因此将可能区分比当前利用荧光成像可实现的多得多的特征。
一般性的核酸纳米结构方法学
本文所述的结构测定方法可应用于任何数量和类型的核酸纳米结构。核酸纳米结构可使用任何种类的核酸折叠方法合成,包括但不限于DNA折纸和DNA单链瓦片(SST)。一种这样的方法是DNA折纸(Rothemund,2006,Nature,440:297-302,通过引用整体并入本文)。在DNA折纸方法中,通过较长的“支架”核酸链经由与多个较短的“订书钉”寡核苷酸杂交的折叠来产生结构,每个“订书钉”寡核苷酸与支架链内的两个或更多个非连续区域杂交。在一些实施方案中,支架链的长度为至少100个核苷酸。在一些实施方案中,支架链长度为至少500个、至少1000个、至少2000个、至少3000个、至少4000个、至少5000个、至少6000个、至少7000个或至少8000个核苷酸。支架链可以是天然或非天然存在的。通常使用的支架是M13mp18病毒基因组DNA,其约为7kb。可使用其它单链支架,包括例如λ基因组DNA。订书钉链长度通常小于100个核苷酸;然而,取决于应用和取决于支架链的长度,它们可更长或更短。在一些实施方案中,订书钉链长度可为约15至约100个核苷酸。在一些实施方案中,订书钉链长度为约25至约50个核苷酸。
这些技术是本领域已知的,并且在美国专利No.7,745,594和7,842,793;美国专利公开No.2010/00696621;和Goodman等,Nature Nanotechnology中进行了更详细的描述。
在一些实施方案中,可在不存在支架链的情况下(例如,无支架结构)装配核酸结构。例如,可组装多个寡核苷酸(例如,长度<200个核苷酸或小于100个核苷酸)以形成核酸纳米结构。该方法描述于WO 2013/022694和WO 2014/018675(其各自通过引用整体并入本文)中。
用于组装核酸结构的其它方法是本领域已知的,本文可使用其中的任何一种。(参见例如Kuzuya和Komiyama,2010,Nanoscale,2:310-322)。还应当理解,这些方法的组合或杂合也可用于产生本文公开的核酸结构。
核酸
核酸结构可包含天然存在的和/或非天然存在的核酸。如果是天然存在的,核酸可从天然来源分离,或者它们可在其天然存在的来源之外合成。非天然存在的核酸是人造的。
术语“核酸”、“寡核苷酸”和“链”可互换使用,意指以连续方式彼此连接的多个核苷酸。核苷酸是包含连接至磷酸基团和可交换有机碱基的糖(例如脱氧核糖)的分子,所述可交换有机碱基是嘧啶(例如,胞嘧啶(C)、胸腺嘧啶(T)或尿嘧啶(U))或嘌呤(例如,腺嘌呤(A)或鸟嘌呤(G))。在一些实施方案中,核酸可以是L-DNA。在一些实施方案中,核酸不是RNA或寡核糖核苷酸。在这些实施方案中,核酸结构可称为DNA结构。然而,DNA结构可仍然包含碱基、糖和主链修饰。
修饰
核酸结构可由DNA、经修饰的DNA及其组合构成。用于产生核酸结构或存在于核酸结构中的寡脱氧核糖核苷酸(本文中也称为寡核苷酸,并且其可以是订书钉链、连接器链等)可具有同质或异质(即,嵌合)主链。主链可以是天然存在的主链诸如磷酸二酯主链或其可包含主链修饰。在一些情况下,由于核酸酶介导的降解减少,因此主链修饰导致寡核苷酸的更长的半衰期。这进而导致更长的半衰期。合适的主链修饰的实例包括但不限于硫代磷酸酯修饰、二硫代磷酸酯修饰、对乙氧基修饰、甲基膦酸酯修饰、硫代磷酸甲酯修饰、烷基-和芳基-磷酸酯(其中带电荷的膦酸酯氧被烷基或芳基替代)、烷基磷酸三酯(其中带电荷的氧部分被烷基化)、肽核酸(PNA)主链修饰、锁核酸(LNA)主链修饰等。这些修饰可彼此组合使用和/或与磷酸二酯主链键联组合使用。
或者或另外地,寡核苷酸可包含其它修饰,包括在碱基或糖部分上的修饰。实例包括具有共价连接至低分子量有机基团而非3'位的羟基且非5'位的磷酸基团的糖(例如2'-O-烷基化核糖)的核酸,具有诸如阿拉伯糖而非核糖的糖的核酸。核酸还包括取代的嘌呤和嘧啶,诸如C-5丙炔修饰的碱基(Wagner等,Nature Biotechnology 14:840-844,1996)。其它嘌呤和嘧啶包括但不限于5-甲基胞嘧啶、2-氨基嘌呤、2-氨基-6-氯嘌呤、2,6-二氨基嘌呤、次黄嘌呤。其它此类修饰是本领域技术人员公知的。
可使用利用氨基磷酸酯或H-膦酸酯化学物质的自动化技术来合成修饰的主链,诸如硫代磷酸酯。可以例如如美国专利No.4,469,863中所述产生芳基-和烷基-膦酸酯,以及可通过使用商购可得的试剂的自动化固相合成来制备烷基磷酸三酯(其中如美国专利No.5,023,243和欧洲专利No.092574中所述烷基化带电荷的氧部分)。已经描述了用于产生其它DNA主链修饰和取代的方法(Uhlmann,E.和Peyman,A.,Chem.Rev.90:544,1990;Goodchild,J.,Bioconjugate Chem.1:165,1990)。
可使用本领域已知的许多方法中的任何一种来从头合成核酸,这些方法包括例如b-氰基乙基亚磷酰胺法(Beaucage和Caruthers 22:1859,1981)和核苷H-膦酸酯法(Garegg等,Tet.Let.27:4051-4054,1986;Froehler等,Nucl.Acid.Res.14:5399-5407,1986;Garegg等,Tet.Let.27:4055-4058,1986,Gaffney等,Tet.Let.29:2619-2622,1988)。这些化学法可通过市场上可获得的各种自动化核酸合成仪来执行。这些核酸被称为合成核酸。修饰和未修饰的核酸也可从商业来源诸如IDT和Bioneer购买。
如本文所用的分离是指所需实体(例如,核酸结构等)从其通常或天然存在的环境或产生其的环境中物理分离。分离可以是部分的或完全的。分离的核酸通常是指从在自然界中通常与其缔合的组分分离的核酸。作为实例,分离的核酸可以是从细胞、细胞核、线粒体或染色质分离的核酸。
可分离和/或纯化核酸纳米结构。可如下进行核酸纳米结构的分离:在凝胶上电泳杂交反应混合物,和分离以特定分子量迁移从而区别于杂交反应的核酸底物和假产物的核酸结构。作为另一个实例,可使用浮力密度梯度、沉降梯度离心或通过过滤装置进行核酸结构的分离。
参考文献
1.Pinheiro AV,Han D,Shih WM,Yan H.Challenges and opportunities for structural DNA nanotechnology.Nat Nanotechnol.6,763-72,2011.
2.Halvorsen K,Wong WP.Massively parallel single-molecule manipulation using centrifugal force.Biophys J.98,L53–55,2010.
3.Otto O,Czerwinski F,Gornall JL,Stober G,Oddershede LB,Seidel R,Keyser UF.Real-time particle tracking at 10,000 fps using optical fiber illumination.Opt.Express,18,22722–22733,2010.
4.Lansdorp,B.M.,Tabrizi,S.J.,Dittmore,A.,&Saleh,O.A.A high-speed magnetic tweezer beyond 10,000 frames per second.Review of Scientific Instruments,84,044301–044301,2013.
5.Heinrich V,Wong WP,Halvorsen K,Evans E.Imaging biomolecular interactions by fast three-dimensional tracking of laser-confined carrier particles.Langmuir 24,1194–1203,2008.
6.Kim K,Saleh OA.A high-resolution magnetic tweezer for single-molecule measurements.Nucleic Acids Research,37,e136–e136,2009.
- 还没有人留言评论。精彩留言会获得点赞!