木葡聚糖内糖基转移酶变体以及编码其的多核苷酸的制作方法
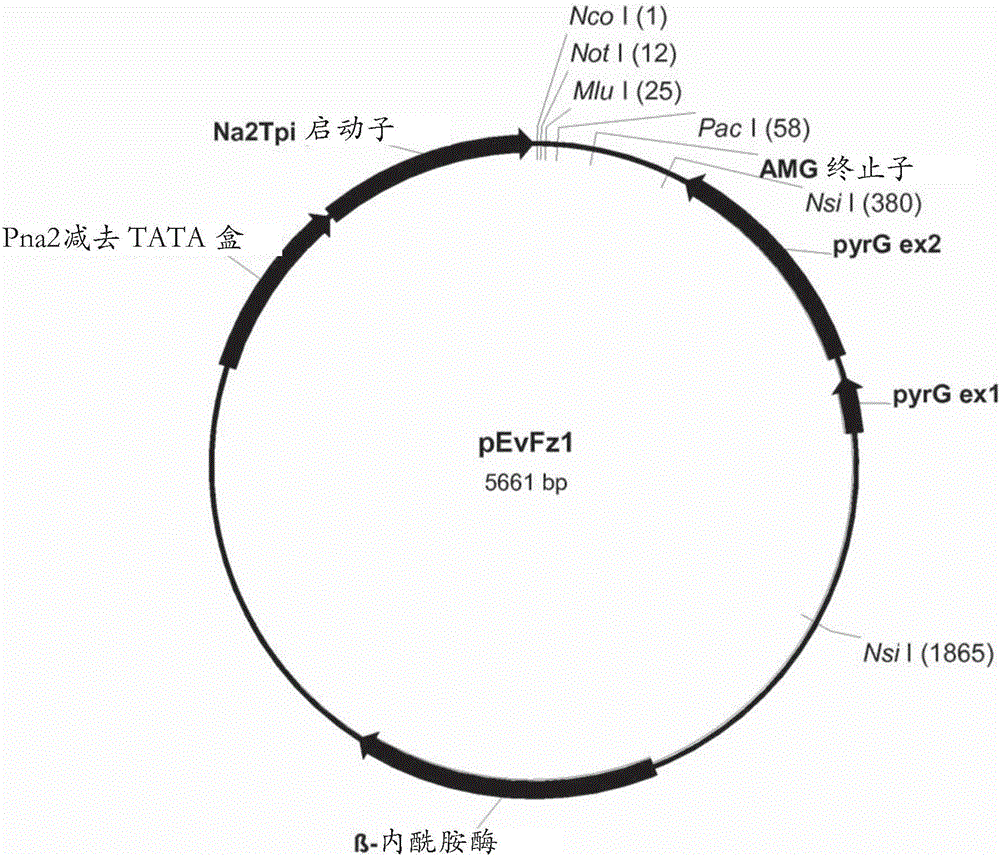
本申请包含计算机可读形式的序列表,将其通过引用结合在此。发明背景发明领域本发明涉及木葡聚糖内糖基转移酶变体、编码这些变体的多核苷酸、产生这些变体的方法以及使用这些变体的方法。相关领域描述木葡聚糖内糖基转移酶(XET)是一种催化木葡聚糖(植物细胞壁的结构多糖)的内切-转糖基作用的酶。该酶存在于大多数植物中,并且具体地是陆生植物。已经从双子叶植物和单子叶植物中提取出XET。还没有在工业微生物中实现商业相关水平的木葡聚糖内糖基转移酶的异源表达。在本领域中,对改进木葡聚糖内糖基转移酶在工业重要微生物中的表达存在需要。本发明提供了与其亲本相比具有增加的表达产量的木葡聚糖内糖基转移酶变体。发明概述本发明涉及分离的木葡聚糖内糖基转移酶变体,这些变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280相对应的一个或多个(例如,若干个)位置处的取代,其中这些变体具有木葡聚糖内糖基转移酶活性。本发明还涉及编码这些变体的分离的多核苷酸,包含这些多核苷酸的核酸构建体、载体和宿主细胞,以及产生这些变体的方法。本发明还涉及用于获得这些变体的方法和增加木葡聚糖内糖基转移酶的表达产量的方法。本发明进一步涉及包括这些变体的组合物。附图简要说明图1示出了pMMar27的限制性图谱。图2示出了pEvFz1的限制性图谱。图3示出了pDLHD0006的限制性图谱。图4示出了pDLHD0044的限制性图谱。图5示出了pDau571的限制性图谱。图6示出了pDLHD0075的限制性图谱。图7示出了pDLHD0095的限制性图谱。定义等位基因变体:术语“等位基因变体”意指占用同一染色体位点的一种基因的两个或更多个替代形式中的任一者。等位基因变异由突变天然产生,并且可以导致群体内多态性。基因突变可以是沉默的(在所编码的多肽中没有改变)或可编码具有改变的氨基酸序列的多肽。多肽的等位基因变体是由基因的等位基因变体编码的多肽。cDNA:术语“cDNA”意指可以通过从获得自真核或原核细胞的成熟的、剪接的mRNA分子进行反转录而制备的DNA分子。cDNA缺乏可以存在于对应基因组DNA中的内含子序列。早先的初始RNA转录本是mRNA的前体,其在呈现为成熟的剪接的mRNA之前要经一系列的步骤进行加工,包括剪接。编码序列:术语“编码序列”意指直接指明变体的氨基酸序列的多核苷酸。编码序列的边界一般由一个开放阅读框架决定,该开放阅读框架从一个起始密码子(如ATG、GTG或TTG)开始并且以一个终止密码子(如TAA、TAG或TGA)结束。编码序列可以是基因组DNA、cDNA、合成DNA或其组合。控制序列:术语“控制序列”意指对于表达编码本发明的变体的多核苷酸所必需的核酸序列。每个控制序列对于编码该变体的多核苷酸来说可以是原生的(即,来自相同基因)或外源的(即,来自不同基因),或相对于彼此是原生的或外源的。这些调控序列包括但不局限于前导子、聚腺苷酸化序列、前肽序列、启动子、信号肽序列和转录终止子。至少,控制序列包括启动子,以及转录和翻译终止信号。出于引入有利于将这些控制序列与编码变体的多核苷酸的编码区连接的特异性限制酶切位点的目的,这些控制序列可以提供有多个接头。表达:术语“表达”包括涉及变体产生的任何步骤,包括但不限于,转录、转录后修饰、翻译、翻译后修饰以及分泌。表达载体:术语“表达载体”意指线性或环状DNA分子,该分子包括编码变体的多核苷酸并且该多核苷酸可操作地与提供用于其表达的控制序列相连接。片段:术语“片段”意指具有从成熟多肽的氨基和/或羧基末端缺失一个或多个(例如,数个)的氨基酸的多肽;其中该片段具有木葡聚糖内糖基转移酶活性。在一个方面中,片段包含成熟多肽的至少85%、至少90%、或至少95%的氨基酸残基。宿主细胞:术语“宿主细胞”意指易于用包含本发明的多核苷酸的核酸构建体或表达载体转化、转染、转导等的任何细胞类型。术语“宿主细胞”涵盖由于复制期间发生的突变而与亲本细胞不同的亲本细胞的任何后代。改进的特性:术语“改进的特性”意指与变体相关的与亲本相比得到改进的特征。在本发明中,改进的特性是相对于亲本,变体的增加的表达产量。增加的表达产量:术语“增加的表达产量”意指相对于在相同的培养条件下,培养每升表达亲本基因的相同的宿主细胞产生的分泌的活性酶的量(g),来自培养每升表达变体基因的宿主细胞的培养基的分泌的活性酶的更高量(g)。在一个方面中,与亲本酶相比,该变体具有至少1.05、至少1.10、至少1.20、至少1.30、至少1.40、至少1.50、至少1.60、至少1.70、至少1.80、至少1.90、至少2、至少2.25、至少2.50、至少2.75、至少3.00、至少3.25、至少3.50、至少3.75、至少4、至少4.25、至少4.50、至少4.75、至少5、至少6、至少7、至少8、至少9、或至少10倍的增加的表达产量。分离的:术语“分离的”意指处于自然界中不存在的形式或环境中的物质。分离的物质的非限制性实例包括(1)任何非天然存在的物质,(2)包括但不限于任何酶、变体、核酸、蛋白、肽或辅因子的任何物质,该物质至少部分地从与其本质相关的一种或多种或所有天然存在的成分中去除;(3)相对于天然发现的物质通过人工修饰的任何物质;或(4)通过增加该物质相对于与其天然相关的其他组分的量而修饰的任何物质(例如,宿主细胞中的重组体产生;编码该物质的基因的多个拷贝;以及使用比编码该物质的基因天然相关的启动子强的启动子)。成熟多肽:术语“成熟多肽”意指在翻译和任何翻译后修饰如N-末端加工、C-末端截短、糖基化作用、磷酸化作用等之后处于其最终形式的多肽。在一个方面中,基于预测SEQIDNO:2的氨基酸1至20是信号肽的SignalP3.0程序(本特森(Bendtsen)等人,2004,分子生物学杂志(J.Mol.Biol.)340:783-795),该成熟多肽是SEQIDNO:2的氨基酸21至292。在另一个方面中,基于预测SEQIDNO:4的氨基酸1至27是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:4的氨基酸28至287。在另一个方面中,基于预测SEQIDNO:6的氨基酸1至22是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:6的氨基酸23至294。在另一个方面中,基于预测SEQIDNO:8的氨基酸1至24是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:8的氨基酸25至297。在另一个方面中,基于预测SEQIDNO:10的氨基酸1至22是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:10的氨基酸23至294。在另一个方面中,基于预测SEQIDNO:12的氨基酸1至26是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:12的氨基酸27至285。在另一个方面中,基于预测SEQIDNO:14的氨基酸1至22是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:14的氨基酸23至323。在另一个方面中,基于预测SEQIDNO:16的氨基酸1至28是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:16的氨基酸29至299。在另一个方面中,基于预测SEQIDNO:18的氨基酸1至24是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:18的氨基酸25至275。在另一个方面中,基于预测SEQIDNO:20的氨基酸1至18是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:20的氨基酸19至330。在另一个方面中,基于预测SEQIDNO:22的氨基酸1至24是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:22的氨基酸25至297。在另一个方面中,基于预测SEQIDNO:24的氨基酸1至24是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:24的氨基酸25至288。在另一个方面中,基于预测SEQIDNO:26的氨基酸1至24是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:26的氨基酸25至311。在另一个方面中,基于预测SEQIDNO:28的氨基酸1至28是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:28的氨基酸29至291。在另一个方面中,基于预测SEQIDNO:30的氨基酸1至25是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:30的氨基酸26至280。在另一个方面中,基于预测SEQIDNO:32的氨基酸1至21是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:32的氨基酸22至290。在另一个方面中,基于预测SEQIDNO:34的氨基酸1至30是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:34的氨基酸31至302。在另一个方面中,基于预测SEQIDNO:36的氨基酸1至27是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:36的氨基酸28至291。在另一个方面中,基于预测SEQIDNO:38的氨基酸1至29是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:38的氨基酸30至299。在另一个方面中,基于预测SEQIDNO:40的氨基酸1至26是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:40的氨基酸27至290。在另一个方面中,基于预测SEQIDNO:42的氨基酸1至28是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:42的氨基酸29至304。在另一个方面中,基于预测SEQIDNO:44的氨基酸1至21是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:44的氨基酸22至292。在另一个方面中,基于预测SEQIDNO:46的氨基酸1至30是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:46的氨基酸31至283。在另一个方面中,基于预测SEQIDNO:48的氨基酸1至22是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:48的氨基酸23至283。在另一个方面中,基于预测SEQIDNO:50的氨基酸1至28是信号肽的SignalP3.0程序,该成熟多肽是SEQIDNO:50的氨基酸29至290。本领域已知,宿主细胞可以产生由同一多核苷酸表达的两种或更多种不同成熟多肽(即,具有不同C-末端和/或N-末端氨基酸)的混合物。本领域还已知,不同的宿主细胞不同地加工多肽,并且因此一个表达多核苷酸的宿主细胞当与另一个表达相同多核苷酸的宿主细胞相比时可以产生不同的成熟多肽(例如,具有不同的C-末端和/或N-末端氨基酸)。成熟多肽编码序列:术语“成熟多肽编码序列”意指编码具有木葡聚糖内糖基转移酶活性的成熟多肽的多核苷酸。在一个方面中,基于预测SEQIDNO:1的核苷酸1至60编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:1的核苷酸61至876。在另一个方面中,基于预测SEQIDNO:3的核苷酸1至81编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:3的核苷酸82至861。在另一个方面中,基于预测SEQIDNO:5的核苷酸1至66编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:5的核苷酸67至882。在另一个方面中,基于预测SEQIDNO:7的核苷酸1至72编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:7的核苷酸73至891。在另一个方面中,基于预测SEQIDNO:9的核苷酸1至66编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:9的核苷酸67至882。在另一个方面中,基于预测SEQIDNO:11的核苷酸1至78编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:11的核苷酸79至855。在另一个方面中,基于预测SEQIDNO:13的核苷酸1至66编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:13的核苷酸67至969。在另一个方面中,基于预测SEQIDNO:15的核苷酸1至84编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:15的核苷酸85至897。在另一个方面中,基于预测SEQIDNO:17的核苷酸1至72编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:17的核苷酸73至825。在另一个方面中,基于预测SEQIDNO:19的核苷酸1至54编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:19的核苷酸55至990。在另一个方面中,基于预测SEQIDNO:21的核苷酸1至72编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:21的核苷酸73至891。在另一个方面中,基于预测SEQIDNO:23的核苷酸1至72编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:23的核苷酸73至864。在另一个方面中,基于预测SEQIDNO:25的核苷酸1至72编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:25的核苷酸73至933。在另一个方面中,基于预测SEQIDNO:27的核苷酸1至84编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:27的核苷酸85至873。在另一个方面中,基于预测SEQIDNO:29的核苷酸1至75编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:29的核苷酸76至840。在另一个方面中,基于预测SEQIDNO:31的核苷酸1至63编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:31的核苷酸64至870。在另一个方面中,基于预测SEQIDNO:33的核苷酸1至90编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:33的核苷酸91至906。在另一个方面中,基于预测SEQIDNO:35的核苷酸1至81编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:35的核苷酸82至873。在另一个方面中,基于预测SEQIDNO:37的核苷酸1至87编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:37的核苷酸88至897。在另一个方面中,基于预测SEQIDNO:39的核苷酸1至78编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:39的核苷酸79至870。在另一个方面中,基于预测SEQIDNO:41的核苷酸1至84编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:41的核苷酸85至912。在另一个方面中,基于预测SEQIDNO:43的核苷酸1至63编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:43的核苷酸64至876。在另一个方面中,基于预测SEQIDNO:45的核苷酸1至90编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:45的核苷酸91至849。在另一个方面中,基于预测SEQIDNO:47的核苷酸1至66编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:47的核苷酸67至849。在另一个方面中,基于预测SEQIDNO:49的核苷酸1至84编码信号肽的SignalP3.0程序,该成熟多肽编码序列是SEQIDNO:49的核苷酸85至870。在以上各方面中,术语“成熟多肽编码序列”应理解成包括基因组DNA序列的cDNA序列或cDNA序列的基因组DNA序列。突变体:术语“突变体”意指编码变体的多核苷酸。核酸构建体:术语“核酸构建体”意指单-链或双链的核酸分子,该核酸分子是从天然存在的基因中分离的,或以本来不存在于自然界中的方式被修饰成包含核酸的区段,或是合成的,该核酸分子包括一个或多个控制序列。可操作地连接:术语“可操作地连接”意指如下的构造,其中,控制序列相对于多核苷酸的编码序列安置在适当位置,从而使得该控制序列指导该编码序列的表达。亲本或亲本木葡聚糖内糖基转移酶:术语“亲本”或“亲本木葡聚糖内糖基转移酶”意指在一个或多个(例如,若干个)位置对木葡聚糖内糖基转移酶进行改变,即取代、插入和/或缺失,以产生本发明的木葡聚糖内糖基转移酶变体。该亲本可以是天然存在的(野生型)多肽或其变体或片段。序列一致性:用参数“序列一致性”来描述两个氨基酸序列之间或两个核苷酸序列之间的相关性。出于本发明的目的,使用如在EMBOSS包(EMBOSS:欧洲分子生物学开放软件套件(TheEuropeanMolecularBiologyOpenSoftwareSuite),赖斯(Rice)等人,2000,遗传学趋势(TrendsGenet.)16:276-277)(优选5.0.0版或更新版本)的尼德尔(Needle)程序中所实施的尼德尔曼-翁施(Needleman-Wunsch)算法(尼德尔曼(Needleman)和翁施(Wunsch),1970,分子生物学杂志(J.Mol.Biol.)48:443-453)来确定两个氨基酸序列之间的序列一致性。使用的参数是空位开放罚分10、空位扩展罚分0.5以及EBLOSUM62(BLOSUM62的EMBOSS版本)取代矩阵。使用Needle标记为“最高同一性(longestidentity)”的输出结果(使用-nobrief选项获得)作为同一性百分比,并计算如下:(相同残基x100)/(比对的长度-在比对中的空位总数)。出于本发明的目的,使用如在EMBOSS包(EMBOSS:欧洲分子生物学开放软件套件,赖斯等人,2000,同上)(优选5.0.0版或更新版本)的尼德尔程序中所实施的尼德尔曼-翁施算法(尼德尔曼和翁施,1970,同上)来确定两个脱氧核糖核苷酸序列之间的序列一致性。使用的参数是空位开放罚分10、空位扩展罚分0.5,以及EDNAFULL(NCBINUC4.4的EMBOSS版本)取代矩阵。使用Needle标记为“最高同一性(longestidentity)”的输出结果(使用-nobrief选项获得)作为同一性百分比,并计算如下:(一致的脱氧核糖核苷酸x100)/(比对长度-比对中的空位总数)严格条件:术语“非常低严格条件”是指对于长度为至少100个核苷酸的探针而言,遵循标准DNA印迹程序,在42℃下在5XSSPE、0.3%SDS、200微克/ml剪切并变性的鲑鱼精子DNA和25%甲酰胺中预杂交和杂交12至24小时。载体材料最终使用0.2XSSC、0.2%SDS,在45℃下洗涤三次,每次15分钟。术语“低严格条件”意指对于长度为至少100个核苷酸的探针而言,遵循标准DNA印迹程序,在42℃下在5XSSPE、0.3%SDS、200微克/ml剪切并变性的鲑鱼精子DNA和25%甲酰胺中预杂交和杂交12至24小时。载体材料最终使用0.2XSSC、0.2%SDS,在50℃下洗涤三次,每次15分钟。对于至少100个核苷酸长度的探针,术语“中严格性条件”意指按照标准DNA印迹程序在42℃在5XSSPE、0.3%SDS、200微克/ml剪切和变性的鲑精DNA和35%甲酰胺中预杂交和杂交12至24小时。载体材料最终使用0.2XSSC、0.2%SDS,在55℃下洗涤三次,每次15分钟。术语“中-高严格条件”意指对于长度为至少100个核苷酸的探针而言,遵循标准DNA印迹程序,在42℃下在5XSSPE、0.3%SDS、200微克/毫升剪切并变性的鲑鱼精子DNA以及35%甲酰胺中预杂交和杂交12至24小时。载体材料最终使用0.2XSSC、0.2%SDS,在60℃下洗涤三次,每次15分钟。术语“高严格条件”意指对于长度为至少100个核苷酸的探针而言,遵循标准DNA印迹程序,在42℃下在5XSSPE、0.3%SDS、200微克/ml剪切并变性的鲑精DNA和50%甲酰胺中预杂交和杂交12至24小时。载体材料最终使用0.2XSSC、0.2%SDS,在65℃下洗涤三次,每次15分钟。术语“非常高严格条件”意指对于长度为至少100个核苷酸的探针而言,遵循标准DNA印迹程序,在42℃下在5XSSPE、0.3%SDS、200微克/ml剪切并变性的鲑鱼精子DNA和50%甲酰胺中预杂交和杂交12至24小时。载体材料最终使用0.2XSSC、0.2%SDS,在70℃下洗涤三次,每次15分钟。子序列:术语“子序列”意指缺失成熟多肽编码序列的5'和/或3'端的一个或多个(例如,若干个)核苷酸的多核苷酸;其中该子序列编码具有木葡聚糖内糖基转移酶活性的片段。在一个方面中,子序列包含成熟多肽编码序列的至少85%、至少90%、或至少95%的核苷酸。变体:术语“变体”意指在一个或多个(例如,若干个)位置包括改变(即,取代、插入和/或缺失)的具有木葡聚糖内糖基转移酶活性的多肽。取代意指占据一个位置的氨基酸由不同的氨基酸代替;缺失意指除去占据一个位置的氨基酸;以及插入意指在占据一个位置的氨基酸的毗邻处和紧邻处添加一个氨基酸。本发明的变体具有亲本的木葡聚糖内糖基转移酶活性的至少20%,例如至少40%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%、或至少100%。野生型木葡聚糖内糖基转移酶:术语“野生型”木葡聚糖内糖基转移酶意指由天然存在的有机体(如在自然界中发现的植物、细菌、酵母或丝状真菌)表达的木葡聚糖内糖基转移酶。木葡聚糖内糖基转移酶:术语“木葡聚糖内糖基转移酶”意指木葡聚糖:木葡聚糖内糖基转移酶(EC2.4.1.207),该酶催化木葡聚糖骨架中β-(1→4)键的裂解,并且转移该木葡聚糖基区段到受体非还原末端葡萄糖残基的O-4上,该受体可以是木葡聚糖或木葡聚糖的寡糖。木葡聚糖内糖基转移酶又称木葡聚糖内糖基转移酶/水解酶或内切木葡聚糖转移酶。一些木聚糖内糖基转移酶可以具有不同活性,这些活性包括木葡聚糖和甘露聚糖内糖基转移酶活性。例如,来自成熟的木瓜水果的木聚糖内糖基转移酶可以使用杂木聚糖,如小麦阿拉伯糖基木聚糖、桦木葡糖醛酸木聚糖、及其他作为供体分子。这些木聚糖可能与木葡聚糖发挥类似的作用,同时成本便宜很多,因为它们可以,例如,从纸浆厂废液和/或未来生物质生物炼制中提取。通过本领域中的那些技术人员,使用任何以下方法,可以评估木葡聚糖内糖基转移酶活性。在木葡聚糖内糖基转移酶的存在下,通过用摩尔过量的木葡聚糖低聚物孵育木葡聚糖聚合物时,木葡聚糖聚合物的平均分子量的减少可以通过液相层析(苏鲁瓦(Sulova)等人,2003,植物生理生化(PlantPhysiol.Biochem.)41:431-437)或通过乙醇沉淀(山中(Yaanaka)等人,2000,食品胶体(FoodHydrocolloids)14:125-128),随后通过重量或纤维素结合分析(弗里(Fry)等人,1992,生物化学杂志(Biochem.J.)282:821-828)来确定,或可以在碱性条件下通过与碘结合在比色上来进行评估(苏鲁瓦(Sulova)等人,1995,分析生物化学(AnalyticalBiochemistry)229:80-85)。变体命名规则出于本发明的目的,披露于SEQIDNO:2中的全长木葡聚糖内糖基转移酶被用来确定在另一个木葡聚糖内糖基转移酶内的相应氨基酸残基。将另一种木葡聚糖内糖基转移酶的氨基酸序列与在SEQIDNO:2中披露的全长多肽比对,并且基于比对,使用尼德曼-翁施算法(尼德尔曼和翁施,1970,分子生物学杂志48:443-453)如在EMBOSS软件包(EMBOSS:欧洲分子生物学开放软件包,赖斯等人,2000,遗传学趋势16:276-277)的尼德尔程序,优选地5.0.0版或更新版本中所执行的,确定与SEQIDNO:2的全长多肽中的任何氨基酸残基相应的氨基酸位置编号。使用的参数是空位开放罚分10、空位扩展罚分0.5以及EBLOSUM62(BLOSUM62的EMBOSS版本)取代矩阵。氨基酸位置的编号是基于SEQIDNO:2的全长多肽(例如,包括信号肽),其中位置1是信号肽的第一个氨基酸(即,Met)并且位置21(即,Ala)是SEQIDNO:2的成熟多肽的第一个位置。另一种木葡聚糖内糖基转移酶中对应的氨基酸残基的鉴定可以通过使用若干计算机程序使用其对应的缺省参数比对多个多肽序列来确定,这些计算机程序包括但不限于MUSCLE(通过对数期望值的多序列比较;3.5版或更新版本;埃德加(Edgar),2004,核酸研究(NucleicAcidsResearch)32:1792-2797);MAFTT(6.857版或更新版本;加藤(Katoh)和库玛(Kuma),2002,核酸研究30:3059-3066;加藤等人,2005,核酸研究33:511-518;加藤和朝都(Toh),2007,生物信息学(Bioinformatics)23:372-374;加藤等人,2009,分子生物学中的方法(MethodsinMolecularBiology)537:39-64;加藤和朝都,2010,生物信息学(Bioinformatics)26:1899-1900);以及采用ClustalW的EMBOSSEMMA(1.83版或更新版本;汤普森(Thompson)等人,1994,核酸研究(NucleicAcidsResearch)22:4673-4680)。当另一种木葡聚糖内糖基转移酶与SEQIDNO:2的全长多肽相背离,这样使得传统的基于序列的比较无法检测它们的相互关系时(林达尔(Lindahl)和埃洛氟松(Elofsson),2000,分子生物学杂志295:613-615),可以使用其他成对序列比较算法。在基于序列的搜索中的更大灵敏度可以使用搜索程序来获得,这些搜索程序利用多肽家族的概率表示(特征曲线)来搜索数据库。例如,PSI-BLAST程序通过迭代数据库搜索过程来产生多个谱,并且能够检测远距离同源物(阿特休尔(Atschul)等人,1997,《核酸研究》25:3389-3402)。如果多肽的家族或超家族在蛋白结构数据库中具有一个或多个代表,则可以实现甚至更大的灵敏度。程序如GenTHREADER(琼斯(Jones),1999,分子生物学杂志(J.Mol.Biol.)287:797-815;麦古芬(McGuffin)和琼斯,2003,生物信息学(Bioinformatics)19:874-881)利用来自不同来源(PSI-BLAST、二级结构预测、结构比对谱以及溶剂化势)的信息作为预测查询序列的结构折叠的神经网络的输入。类似地,高夫(Gough)等人,2000,分子生物学杂志(J.Mol.Biol.)313:903-919的方法可以用于比对未知结构的序列与存在于SCOP数据库中的超家族模型。这些比对进而可以用于产生多肽的同源性模型,并且使用出于该目的而开发的多种工具可以评定此类模型的准确度。对于已知结构的蛋白,若干工具和资源可用于检索并产生结构比对。例如,蛋白的SCOP超家族已经在结构上进行比对,并且那些比对是可访问的并且可下载的。可以使用多种算法如距离比对矩阵(奥尔姆(Holm)和桑德(Sander),1998,蛋白质(Proteins)33:88-96)或组合延伸(辛迪亚洛夫(Shindyalov)和伯恩(Bourne),1998,蛋白质工程(ProteinEngineering)11:739-747)比对两种或更多种蛋白质结构,并且这些算法的实施可以另外用于查询具有感兴趣结构的结构数据库,以便发现可能的结构同源物(例如,奥尔姆和帕克(Park),2000,生物信息学(Bioinformatics)16:566-567)。在描述本发明的变体中,以下所述的命名法适于方便参考。采用了已接受的IUPAC单个字母和三字母的氨基酸缩写。取代。对于氨基酸取代,使用以下命名法:初始氨基酸、位置、取代氨基酸。因此,在位置226处的苏氨酸被丙氨酸取代表示为“Thr226Ala”或者“T226A”。多个突变由加号(“+”)分开,例如“Gly205Arg+Ser411Phe”或“G205R+S411F”代表分别在位置205和位置411处甘氨酸(G)被精氨酸(R)取代,并且丝氨酸(S)被苯丙氨酸(F)取代。缺失。对于氨基酸缺失,使用以下命名法:初始氨基酸、位置、*。因此,在位置195处的甘氨酸缺失表示为“Gly195*”或“G195*”。多个缺失由加号(“+”)分开,例如“Gly195*+Ser411*”或“G195*+S411*”。插入。对于氨基酸插入,使用以下命名法:初始氨基酸、位置、初始氨基酸、插入氨基酸。因此,在位置195处的甘氨酸之后插入赖氨酸被表示为“Gly195GlyLys”或“G195GK”。多个氨基酸的插入被表示为[初始氨基酸,位置,初始氨基酸,插入氨基酸#1、插入氨基酸#2;等]。例如,在位置195处的甘氨酸之后插入赖氨酸和丙氨酸被表示为“Gly195GlyLysAla”或“G195GKA”。在此类情况下,通过将小写字母添加至在所插入的一个或多个氨基酸残基之前的氨基酸残基的位置编号中来对所插入的一个或多个氨基酸残基进行编号。在以上实例中,该序列因此将是:亲本:变体:195195195a195bGG-K-A多种改变。包括多种改变的变体由加号(“+”)分开,例如“Arg170Tyr+Gly195Glu”或者“R170Y+G195E”代表在位置170和位置195处的精氨酸和甘氨酸分别被酪氨酸和谷氨酸取代。不同改变。可以在一个位置上引入不同的改变时,这些不同的改变由逗号分开,例如“Arg170Tyr,Glu”代表在位置170上的精氨酸被酪氨酸或谷氨酸取代。因此,“Tyr167Gly,Ala+Arg170Gly,Ala”表示以下变体:“Tyr167Gly+Arg170Gly”、“Tyr167Gly+Arg170Ala”、“Tyr167Ala+Arg170Gly”、和“Tyr167Ala+Arg170Ala”。发明详述本发明涉及分离的木葡聚糖内糖基转移酶变体,这些变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280相对应的一个或多个(例如,若干个)位置处的取代,其中这些变体具有木葡聚糖内糖基转移酶活性。变体在一个实施例中,这些变体与亲本木葡聚糖内糖基转移酶的氨基酸序列具有至少60%,例如至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%、但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、或50的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:2的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:4的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:6的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:8的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:10的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:12的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:14的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:16的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:18的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:20的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:22的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:24的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:26的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:28的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:30的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:32的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:34的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:36的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:38的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:40的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:42的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:44的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:46的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:48的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在另一个实施例中,这些变体与SEQIDNO:50的成熟多肽具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。在一个方面中,在本发明的变体中的取代数目是1至17个,例如1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、或17个取代。在另一个方面中,变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280相对应的一个或多个(例如,若干个)位置处的取代。在另一个方面中,变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280中的任一个相对应的两个位置处的取代。在另一个方面中,变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280中的任一个相对应的三个位置处的取代。在另一个方面中,变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280中的任一个相对应的四个位置处的取代。在另一个方面中,变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280中的任一个相对应的五个位置处的取代。在另一个方面中,变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280中的任一个相对应的六个位置处的取代。在另一个方面中,变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280中的任一个相对应的七个位置处的取代。在另一个方面中,变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280中的任一个相对应的八个位置处的取代。在另一个方面中,变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280中的任一个相对应的九个位置处的取代。在另一个方面中,变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280中的任一个相对应的十个位置处的取代。在另一个方面中,变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280中的任一个相对应的十一个位置处的取代。在另一个方面中,变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280中的任一个相对应的十二个位置处的取代。在另一个方面中,变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280中的任一个相对应的十三个位置处的取代。在另一个方面中,变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280中的任一个相对应的十四个位置处的取代。在另一个方面中,变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280中的任一个相对应的十五个位置处的取代。在另一个方面中,变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280中的任一个相对应的十六个位置处的取代。在另一个方面中,变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280相对应的各个位置处的取代。在另一个方面中,该变体包括与位置10相对应的位置处的取代或由其组成。在另一个方面中,与位置10相对应的位置处的氨基酸被Ala、Arg、Asn、Asp、Cys、Gln、Glu、Gly、His、Ile、Leu、Lys、Met、Phe、Pro、Ser、Thr、Trp、Tyr或Val取代,优选被Ala取代。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代I10A或由其组成。在另一个方面中,该变体包括与位置30相对应的位置处的取代或由其组成。在另一个方面中,与位置30相对应的位置处的氨基酸被Ala、Arg、Asn、Asp、Cys、Gln、Glu、Gly、His、Ile、Leu、Lys、Met、Phe、Pro、Ser、Thr、Trp、Tyr、或Val取代,优选被Glu取代。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代P30E或由其组成。在另一个方面中,该变体包括与位置40相对应的位置处的取代或由其组成。在另一个方面中,与位置40相对应的位置处的氨基酸被Ala、Arg、Asn、Asp、Cys、Gln、Glu、Gly、His、Ile、Leu、Lys、Met、Phe、Pro、Ser、Thr、Trp、Tyr、或Val取代,优选被Gly取代。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代A40G或由其组成。在另一个方面中,该变体包括与位置51相对应的位置处的取代或由其组成。在另一个方面中,与位置51相对应的位置处的氨基酸被Ala、Arg、Asn、Asp、Cys、Gln、Glu、Gly、His、Ile、Leu、Lys、Met、Phe、Pro、Ser、Thr、Trp、Tyr、或Val取代,优选被Thr取代。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代S51T或由其组成。在另一个方面中,该变体包括与位置53相对应的位置处的取代或由其组成。在另一个方面中,与位置53相对应的位置处的氨基酸被Ala、Arg、Asn、Asp、Cys、Gln、Glu、Gly、His、Ile、Leu、Lys、Met、Phe、Pro、Ser、Thr、Trp、Tyr、或Val取代,优选被Ala或Val取代。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代I53A,V或由其组成。在另一个方面中,该变体包括与位置60相对应的位置处的取代或由其组成。在另一个方面中,与位置60相对应的位置处的氨基酸被Ala、Arg、Asn、Asp、Cys、Gln、Glu、Gly、His、Ile、Leu、Lys、Met、Phe、Pro、Ser、Thr、Trp、Tyr、或Val取代,优选被Ser取代。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代Y60S或由其组成。在另一个方面中,该变体包括与位置99相对应的位置处的取代或由其组成。在另一个方面中,与位置99相对应的位置处的氨基酸被Ala、Arg、Asn、Asp、Cys、Gln、Glu、Gly、His、Ile、Leu、Lys、Met、Phe、Pro、Ser、Thr、Trp、Tyr、或Val取代,优选被Glu或Asn取代。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代T99E,N或由其组成。在另一个方面中,该变体包括与位置102相对应的位置处的取代或由其组成。在另一个方面中,与位置102相对应的位置处的氨基酸被Ala、Arg、Asn、Asp、Cys、Gln、Glu、Gly、His、Ile、Leu、Lys、Met、Phe、Pro、Ser、Thr、Trp、Tyr、或Val取代,优选被Gly取代。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代E102G或由其组成。在另一个方面中,该变体包括与位置117相对应的位置处的取代或由其组成。在另一个方面中,与位置117相对应的位置处的氨基酸被Ala、Arg、Asn、Asp、Cys、Gln、Glu、Gly、His、Ile、Leu、Lys、Met、Phe、Pro、Ser、Thr、Trp、Tyr、或Val取代,优选被Glu取代。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代Q117E或由其组成。在另一个方面中,该变体包括与位置130相对应的位置处的取代或由其组成。在另一个方面中,与位置130相对应的位置处的氨基酸被Ala、Arg、Asn、Asp、Cys、Gln、Glu、Gly、His、Ile、Leu、Lys、Met、Phe、Pro、Ser、Thr、Trp、Tyr、或Val取代,优选被Arg取代。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代K130R或由其组成。在另一个方面中,该变体包括与位置136相对应的位置处的取代或由其组成。在另一个方面中,与位置136相对应的位置处的氨基酸被Ala、Arg、Asn、Asp、Cys、Gln、Glu、Gly、His、Ile、Leu、Lys、Met、Phe、Pro、Ser、Thr、Trp、Tyr、或Val取代,优选被Trp取代。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代R136W或由其组成。在另一个方面中,该变体包括与位置157相对应的位置处的取代或由其组成。在另一个方面中,与位置157相对应的位置处的氨基酸被Ala、Arg、Asn、Asp、Cys、Gln、Glu、Gly、His、Ile、Leu、Lys、Met、Phe、Pro、Ser、Thr、Trp、Tyr、或Val取代,优选被His取代。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代Y157H或由其组成。在另一个方面中,该变体包括与位置162相对应的位置处的取代或由其组成。在另一个方面中,与位置162相对应的位置处的氨基酸被Ala、Arg、Asn、Asp、Cys、Gln、Glu、Gly、His、Ile、Leu、Lys、Met、Phe、Pro、Ser、Thr、Trp、Tyr、或Val取代,优选被Cys取代。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代Y162C或由其组成。在另一个方面中,该变体包括与位置175相对应的位置处的取代或由其组成。在另一个方面中,与位置175相对应的位置处的氨基酸被Ala、Arg、Asn、Asp、Cys、Gln、Glu、Gly、His、Ile、Leu、Lys、Met、Phe、Pro、Ser、Thr、Trp、Tyr或Val取代,优选被Ser、Gly、或Gln取代。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代N175S,G,Q或由其组成。在另一个方面中,该变体包括与位置183相对应的位置处的取代或由其组成。在另一个方面中,与位置183相对应的位置处的氨基酸被Ala、Arg、Asn、Asp、Cys、Gln、Glu、Gly、His、Ile、Leu、Lys、Met、Phe、Pro、Ser、Thr、Trp、Tyr、或Val取代,优选被Ile取代。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代F183I或由其组成。在另一个方面中,该变体包括与位置254相对应的位置处的取代或由其组成。在另一个方面中,与位置254相对应的位置处的氨基酸被Ala、Arg、Asn、Asp、Cys、Gln、Glu、Gly、His、Ile、Leu、Lys、Met、Phe、Pro、Ser、Thr、Trp、Tyr、或Val取代,优选被Glu取代。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代A254E或由其组成。在另一个方面中,该变体包括与位置280相对应的位置处的取代或由其组成。在另一个方面中,与位置280相对应的位置处的氨基酸被Ala、Arg、Asn、Asp、Cys、Gln、Glu、Gly、His、Ile、Leu、Lys、Met、Phe、Pro、Ser、Thr、Trp、Tyr或Val取代,优选被Gly或Glu取代。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代S280G,E或由其组成。在以上各方面中,该变体在包括与作为亲本的其他木葡聚糖内糖基转移酶中的SEQIDNO:2的全长多肽相对应的位置处的上述一个或多个取代或由其组成。在以下各方面中,该变体包括与其他木葡聚糖内糖基转移酶中的SEQIDNO:2的全长多肽相对应的位置处的或SEQIDNO:2的全长多肽的位置处的下述一个或多个取代或由其组成。在另一个方面中,该变体包括选自下组的一个或多个(例如,若干个)取代或由其组成,该组由以下各项组成:I10A;P30E;A40G;S51T;I53A,V;Y60S;T99E,N;E102G;Q117E;K103R;R136W;Y157H;Y162C;N175S,G,Q;F183I;A254E;以及S280G,E。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代A40G+N175S或由其组成。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代A40G+F183I或由其组成。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代N175G+S280G或由其组成。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代A40G+I53A+N175S或由其组成。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代A40G+N175S+F183I或由其组成。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代I10A+I53A+E102G或由其组成。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代A40G+E102G+Q117E或由其组成。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代N175Q+A254E+S280E或由其组成。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代P30E+S51T+Y60S+T99N或由其组成。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代A40G+T99E+E102G+K130R或由其组成。在另一个方面中,该变体包括SEQIDNO:2的全长多肽的取代I53V+R136W+Y157H+Y162C+N175S或由其组成。这些变体可以进一步在一个或多个(例如,若干个)其他位置处包括一个或多个另外的改变,例如,取代、插入或缺失。氨基酸改变可以具有次要性质,即不显著影响蛋白质的折叠和/或活性的保守氨基酸取代或插入;典型地1-30个氨基酸的小缺失;小的氨基-或羧基末端延伸,如氨基末端蛋氨酸残基;至多20-25个残基的小接头肽;或通过改变净电荷或另一功能来促进纯化的小延伸,如多组氨酸序列、抗原性表位或结合结构域。保守取代的实例在下组之内:碱性氨基酸(精氨酸、赖氨酸和组氨酸)、酸性氨基酸(谷氨酸和天冬氨酸)、极性氨基酸(谷氨酰胺和天冬酰胺)、疏水氨基酸(亮氨酸、异亮氨酸和缬氨酸)、芳族氨基酸(苯丙氨酸、色氨酸和酪氨酸)、以及小氨基酸(甘氨酸、丙氨酸、丝氨酸、苏氨酸以及甲硫氨酸)。一般不会改变比活性的氨基酸取代是本领域已知的并且例如由H.诺伊拉特(Neurath)和R.L.希尔(Hill),1979,在蛋白质(TheProteins),学术出版社(AcademicPress),纽约中描述。常见取代是Ala/Ser、Val/Ile、Asp/Glu、Thr/Ser、Ala/Gly、Ala/Thr、Ser/Asn、Ala/Val、Ser/Gly、Tyr/Phe、Ala/Pro、Lys/Arg、Asp/Asn、Leu/Ile、Leu/Val、Ala/Glu、以及Asp/Gly。可替代地,氨基酸变化是这样一种性质,使得多肽的理化性质被改变。例如,氨基酸改变可以改进多肽的热稳定性、改变底物特异性、改变最适pH等。可以根据本领域已知的方法,如定点诱变或丙氨酸扫描诱变鉴定多肽中的必需氨基酸(康宁汉(Cunningham)和韦尔斯(Wells),1989,科学(Science)244:1081-1085)。在后一项技术中,在该分子中的每个残基处引入单个丙氨酸突变,并且测试所得突变体分子的木葡聚糖内糖基转移酶活性以鉴定对于该分子的活性至关重要的氨基酸残基。还参见希尔顿(Hilton)等人,1996,生物化学杂志(J.Biol.Chem.)271:4699-4708。酶或其他生物学相互作用的活性部位还可通过对结构的物理分析来确定,如由下述技术确定:核磁共振、晶体学(crystallography)、电子衍射、或光亲和标记,连同对推定的接触位点(contractsite)氨基酸进行突变。参见,例如德沃斯(deVos)等人,1992,科学(Science)255:306-312;史密斯(Smith)等人,1992,分子生物学杂志(J.Mol.Biol.)224:899-904;乌乐达维尔(Wlodaver)等人,1992,欧洲生物化学学会联盟通讯(FEBSLett.)309:59-64。也可以从与相关多肽的比对推断必需氨基酸的身份。在SEQIDNO:2的氨基酸21至292的序列中的必需活性位点氨基酸位于位置105、107、和109。本发明还涉及增加木葡聚糖内糖基转移酶的表达产量的方法,该方法包括在与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、和280相对应的一个或多个位置处将取代引入亲本木葡聚糖内糖基转移酶中以产生变体,其中该变体的表达产量相对于亲本是增加的;并任选地回收该变体。在一个实施例中,与亲本酶相比,该变体具有至少1.05、至少1.10、至少1.20、至少1.30、至少1.40、至少1.50、至少1.60、至少1.70、至少1.80、至少1.90、至少2、至少2.25、至少2.50、至少2.75、至少3.00、至少3.25、至少3.50、至少3.75、至少4、至少4.25、至少4.50、至少4.75、至少5、至少6、至少7、至少8、至少9、或至少10倍的增加的表达产量。亲本木葡聚糖内糖基转移酶该亲本木葡聚糖内糖基转移酶可以是(a)与SEQIDNO:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、或50的成熟多肽具有至少60%序列一致性的多肽;(b)由以下多核苷酸编码的多肽,该多核苷酸在低严格条件下与(i)SEQIDNO:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、或49的成熟多肽编码序列,或(ii)(i)的全长互补体杂交;或(c)由以下多核苷酸编码的多肽,该多核苷酸与SEQIDNO:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、或49的成熟多肽编码序列具有至少60%序列一致性。在一个实施例中,该亲本与SEQIDNO:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、或50的成熟多肽具有至少60%,例如至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%、或100%序列一致性,这些成熟多肽具有木葡聚糖内糖基转移酶活性。在另一个实施例中,该亲本的氨基酸序列与SEQIDNO:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、或50的成熟多肽相差多达10个氨基酸,例如1、2、3、4、6、6、7、8、9或10个。在另一个实施例中,该亲本包括SEQIDNO:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、或50的氨基酸序列或由其组成。在另一个实施例中,该亲本包括SEQIDNO:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、或50的成熟多肽或由其组成。在另一个实施例中,该亲本是SEQIDNO:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、或50的成熟多肽的片段,该片段包含该亲本的至少85%,例如至少90%和至少95%的氨基酸残基。在另一个实施例中,该亲本是SEQIDNO:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、或50的成熟多肽的等位基因变体。在另一个实施例中,该亲本由如下的多核苷酸编码,该多核苷酸在非常低严格条件、低严格条件、中严格条件、中-高严格条件、高严格条件或非常高严格条件下与(i)SEQIDNO:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、或49的成熟多肽编码序列,或(ii)(i)的全长互补体杂交(萨拉布鲁克(Sambrook)等人,1989,分子克隆实验指南(MolecularCloning:ALaboratoryManual),第二版,冷泉港(ColdSpringHarbor),纽约)。SEQIDNO:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、或49的多核苷酸或其子序列,连同SEQIDNO:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48或50的多肽或其片段可被用于设计核酸探针以根据本领域熟知的方法来鉴定并克隆编码来自不同属或种的亲本的DNA。具体而言,可以根据标准DNA印迹程序,使用这类探针与感兴趣的细胞的基因组DNA或cDNA杂交,以便鉴定和分离其中的对应基因。此类探针可以明显短于完整序列,但是长度应为至少15,例如至少25、至少35、或至少70个核苷酸。优选地,核酸探针具有至少100个核苷酸长度,例如至少200个核苷酸长度、至少300个核苷酸长度、至少400个核苷酸长度、至少500个核苷酸长度、至少600个核苷酸长度、至少700个核苷酸长度、至少800个核苷酸长度、或至少900个核苷酸长度。DNA和RNA探针二者均可使用。典型地将探针进行标记(例如,用32P、3H、35S、生物素、或抗生物素蛋白),以检测相应的基因。本发明涵盖此类探针。可以针对与上文所述的探针杂交并编码亲本的DNA来筛选由这类其他属或种制备的基因组DNA或cDNA文库。可以通过琼脂糖或聚丙烯酰胺凝胶电泳,或通过其他分离技术分离基因组或其他DNA。可以将来自文库的DNA或分离的DNA转移至硝化纤维素或其他适合的载体材料并且固定于其上。为了鉴定与SEQIDNO:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、或49或其子序列杂交的克隆或DNA,将载体材料用于DNA印迹中。出于本发明的目的,杂交指示该多核苷酸在非常低到非常高严格度条件下和与以下各项相对应的标记的核酸探针杂交:(i)1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、或49;(ii)其成熟多肽编码序列;(iii)其全长互补体;或(iv)其子序列。可以使用例如X-射线胶片或本领域已知的任何其他检测手段来检测在这些条件下核酸探针杂交的分子。在一个方面中,该核酸探针是SEQIDNO:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、或49的成熟多肽编码序列。在另一个方面中,该核酸探针是编码SEQIDNO:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、或50的多肽;其成熟多肽;或其片段的多核苷酸。在另一个方面中,该核酸探针是SEQIDNO:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、或49。在另一个实施例中,该亲本由以下多核苷酸编码,该多核苷酸与SEQIDNO:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、或49的成熟多肽编码序列具有至少60%,例如至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%、或100%序列一致性。该亲本可以是一种杂合多肽(嵌合体),其中该亲本的一个区域被另一个多肽的区域替换。该亲本可以是一种融合多肽或可切割的融合多肽,其中另一个多肽融合在该亲本的N-末端或C-末端处。通过将编码另一种多肽的多核苷酸与本发明多核苷酸融合而产生融合多肽。用于产生融合多肽的技术是本领域已知的,并且包括连接编码多肽的编码序列,使得它们在框内,并且融合多肽的表达处于相同的启动子和终止子的控制之下。也可以使用内含肽技术构建融合多肽,其中以翻译后方式产生融合多肽(库珀(Cooper)等人,1993,欧洲分子生物学学会杂志(EMBOJ.)12:2575-2583;道森(Dawson)等人,1994,科学(Science)266:776-779)。融合多肽可以进一步包括两种多肽之间的切割位点。在融合蛋白分泌之时,该位点被切割,从而释放出这两种多肽。切割位点的实例包括但不限于在如下文献中披露的位点:马丁(Martin)等人,2003,工业微生物学生物技术杂志(J.Ind.Microbiol.Biotechnol.)3:568-576;Svetina等人,2000,生物技术杂志(J.Biotechnol.)76:245-251;拉斯马森-威尔逊(Rasmussen-Wilson)等人,1997,应用与环境微生物学(Appl.Environ.Microbiol.)63:3488-3493;沃德(Ward)等人,1995,生物技术(Biotechnology)13:498-503;以及孔特雷拉斯(Contreras)等人,1991,生物技术9:378-381;伊顿(Eaton)等人,1986,生物化学(Biochemistry)25:505-512;Collins-Racie等人,1995,生物技术13:982-987;卡特(Carter)等人,1989,蛋白质:结构、功能和遗传学(Proteins:Structure,Function,andGenetics)6:240-248;和史蒂文斯(Stevens),2003,世界药物发现(DrugDiscoveryWorld)4:35-48。该亲本可以从任何属的植物获得。出于本发明的目的,如在此结合给定的来源使用的术语“从……获得(或获得自)”应意指由多核苷酸编码的亲本是由该来源产生的。在一个方面中,该亲本是胞外分泌的。该亲本可以是任何植物木葡聚糖内糖基转移酶。在一个实施例中,该亲本是从豆科(同义词:豆科(Leguminosae和Papilionaceae))的子叶中获得,优选地菜豆属,具体地,绿豆。优选的单子叶植物是非禾本科的单子叶植物和百合的单子叶植物。木葡聚糖内糖基转移酶还可以从苔藓和苔类中来提取,如在弗里(Fry)等人,1992,生物化学杂志(Biochem.J.)282:821-828中所描述的。例如,该木葡聚糖内糖基转移酶可以从子叶中获得,即,双子叶植物或单子叶植物,具体地是选自下组的双子叶植物,该组由以下各项组成:花椰菜、大豆、番茄、马铃薯、油菜、向日葵、棉花、以及烟草,或选自下组的单子叶植物,该组由以下各项组成:小麦、水稻、玉米和甘蔗。参见,例如,WO2003/033813和WO97/023683。在另一个实施例中,该亲本从以下种获得:拟南芥属(Arabidopsis)、番木瓜属(Carica)、香瓜属(Cucumis)、胡萝卜属(Daucus)、羊茅属(Festuca)、大豆属(Glycine)、大麦属(Hordeum)、番茄属(Lycopersicon)、苜蓿属(Medicago)、稻属(Oryza)、杨属(Populus)、慈菇属(Sagittaria)、高粱属(Sorghum)、豇豆属(Vigna)、或玉蜀黍属(Zea)。在另一个实施例中,该亲本从拟南芥、番木瓜、黄瓜、野胡萝卜、草地羊茅、大豆、大麦、番茄、蒺藜状苜蓿、水稻、欧洲山杨、矮慈姑(Sagittariapygmaea)、双色高粱、红豆(Vignaangularis)、或玉米(Zeamays)获得。在另一个实施例中,该亲本是拟南芥(GENESEQP:AOE11231、GENESEQP:AOE93420、GENESEQP:BAL03414、GENESEQP:BAL03622、或GENESEQP:AWK95154);番木瓜(GENESEQP:AZR75725);黄瓜(GENESEQP:AZV66490);野胡萝卜(GENESEQP:AZV66139);草地羊茅(GENESEQP:AZR80321);大豆(GENESEQP:AWK95154或GENESEQP:AYF92062);大麦(GENESEQP:AZR85056、GENESEQP:AQY12558、GENESEQP:AQY12559、或GENESEQP:AWK95180);番茄(GENESEQP:ATZ45232);蒺藜状苜蓿(GENESEQP:ATZ48025);水稻(GENESEQP:ATZ42485、GENESEQP:ATZ57524、或GENESEQP:AZR76430);欧洲山杨(GENESEQP:AWK95036);矮慈菇(GENESEQP:AZV66468);双色高粱(GENESEQP:BAO79623或GENESEQP:BAO79007);红豆(GENESEQP:ATZ61320);或玉蜀黍(GENESEQP:AWK94916)木葡聚糖内糖基转移酶,其中将这些登录号以其全部内容结合在此。可以通过筛选基因组DNA或cDNA文库或混合的DNA样品来获得编码亲本的多核苷酸。一旦已经用一个或多个探针检测到编码亲本的多核苷酸,则可以通过利用对本领域的普通技术人员来说已知的技术来分离或克隆该多核苷酸(参见,例如,萨拉布鲁克(Sambrook)等人,1989,见上文)。变体的制备本发明还涉及用于获得木葡聚糖内糖基转移酶变体的方法,该方法包括:(a)将与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280相对应的一个或多个(例如,若干个)位置处的取代引入亲本木葡聚糖内糖基转移酶,其中该变体具有木葡聚糖内糖基转移酶活性,并且任选地(b)回收该变体。可以使用本领域已知的任何诱变程序来制备这些变体,如定点诱变、位点饱和诱变、合成基因构建、半合成基因构建、随机诱变、改组等。定点诱变是在编码该亲本的多核苷酸中的一个或多个限定位点处引入一个或多个(例如,若干个)突变的技术。通过使用涉及包含所希望的突变的寡核苷酸引物的PCR可以体外实现定点诱变。也可以通过盒式诱变进行体外定点诱变,所述盒式诱变涉及由限制酶在包括编码亲本的多核苷酸的质粒中的位点处切割并且随后将包含突变的寡核苷酸连接在多核苷酸中。通常,消化该质粒与该寡核苷酸的限制酶是相同的,以允许该质粒的粘性末端以及插入片段彼此连接。参见,例如谢勒(Scherer)和戴维斯(Davis),1979,美国国家科学院院刊(Proc.Natl.Acad.Sci.USA)76:4949-4955;和巴顿(Barton)等人,1990,核酸研究(NucleicAcidsRes.)18:7349-4966。还可以通过本领域已知的方法体内实现定点诱变。参见,例如,美国专利申请公开号2004/0171154;斯托西(Storici)等人,2001,自然生物技术(NatureBiotechnol.)19:773-776;凯伦(Kren)等人,1998,自然医学(Nat.Med.)4:285-290;以及卡里萨诺(Calissano)和曼奇诺(Macino),1996,真菌遗传学通讯(FungalGenet.Newslett.)43:15-16。在本发明中可以使用任何定点诱变程序。存在可用于制备变体的很多可商购的试剂盒。位点饱和诱变在一个或多个(例如,若干个)特定位置处将多肽编码序列系统性地替代为编码全部19个氨基酸的序列(帕里克(Parikh)和松村(Matsumura),2005,分子生物学杂志(J.Mol.Biol.)352:621-628)。合成基因构建需要体外合成设计的多核苷酸分子以编码感兴趣的多肽。基因合成可以利用多种技术来进行,如由田(Tian)等人(2004,自然(Nature)432:1050-1054)所述的基于多路微芯片的技术、以及其中在光可编程的微流芯片上合成并组装寡核苷酸的类似技术。通过组合合成基因构建、和/或定点诱变、和/或随机诱变、和/或改组的多个方面来实现半合成基因构建。半合成构建典型地是利用合成的多核苷酸片段的过程结合PCR技术。因此,基因的限定的区域可以从头合成,而其他区域可以使用位点特异性诱变引物来扩增,而还有其他区域可以经受易错PCR或非易错PCR扩增。然后可以对多核苷酸子序列进行改组。可以做出单个或多个氨基酸取代、缺失和/或插入并且使用已知的诱变、重组和/或改组方法进行测试,随后进行有关筛选程序,如由里德哈尔-奥尔森(Reidhaar-Olson)和萨奥尔(Sauer),1988,科学(Science)241:53-57;博维(Bowie)和萨奥尔,1989,美国科学院院刊(Proc.Natl.Acad.Sci.USA)86:2152-2156;WO95/17413;或WO95/22625所披露的那些。可以使用的其他方法包括易错PCR、噬菌体展示(例如,罗曼(Lowman)等人,1991,生物化学(Biochemistry)30:10832-10837;美国专利号5,223,409;WO92/06204)和区域定向诱变(德比舍尔(Derbyshire)等人,1986,基因(Gene)46:145;Ner等人,1988,DNA7:127)。诱变/改组方法可以与高通量自动化筛选方法组合以检测由宿主细胞表达的克隆的诱变多肽的活性(奈斯(Ness)等人,1999,自然生物技术(NatureBiotechnology)17:893-896)。编码活性多肽的诱变的DNA分子可以回收自宿主细胞,并且使用本领域的标准方法对其进行迅速测序。这些方法允许迅速确定多肽中单个氨基酸残基的重要性。多核苷酸本发明还涉及编码本发明的变体的分离的多核苷酸。核酸构建体本发明还涉及包括编码本发明的变体的、可操作地连接至一个或多个控制序列上的多核苷酸的核酸构建体,该一个或多个控制序列在与控制序列相容的条件下指导编码序列在适合的宿主细胞中的表达。可以按多种方式来操纵该多核苷酸以提供变体的表达。取决于表达载体,在其插入载体以前操纵多核苷酸可以是希望的或必需的。用于利用重组DNA方法修饰多核苷酸的技术是本领域熟知的。该控制序列可以是一个启动子,即,被宿主细胞识别以对编码本发明的变体的多核苷酸进行表达的一种多核苷酸。启动子包含介导该变体的表达的转录控制序列。启动子可以是在宿主细胞中显示出转录活性的任何多核苷酸,包括突变型、截短型及杂合型启动子,并且可以是由编码与该宿主细胞同源或异源的细胞外或细胞内多肽的基因获得。在丝状真菌宿主细胞中,用于指导本发明的核酸构建体的转录的适合启动子的实例是从以下各项的基因获得的启动子:构巢曲霉乙酰胺酶、黑曲霉中性α-淀粉酶、黑曲霉酸稳定性α-淀粉酶、黑曲霉或泡盛曲霉葡萄糖淀粉酶(glaA)、米曲霉TAKA淀粉酶、米曲霉碱性蛋白酶、米曲霉丙糖磷酸异构酶、尖孢镰刀菌胰蛋白酶-样蛋白酶(WO96/00787)、镶片镰孢菌淀粉葡糖苷酶(WO00/56900)、镶片镰孢菌Daria(达莉亚)(WO00/56900)、镶片镰孢菌Quinn(奎恩)(WO00/56900)、米黑根毛霉脂肪酶、米黑根毛霉天冬氨酸蛋白酶、里氏木霉β-葡糖苷酶、里氏木霉纤维二糖水解酶I、里氏木霉纤维二糖水解酶II、里氏木霉内切葡聚糖酶I、里氏木霉内切葡聚糖酶II、里氏木霉内切葡聚糖酶III、里氏木霉内切葡聚糖酶V、里氏木霉木聚糖酶I、里氏木霉木聚糖酶II、里氏木霉木聚糖酶III、里氏木霉β-木糖苷酶,以及里氏木霉翻译延伸因子,连同NA2-tpi启动子(来自编码中性α-淀粉酶的曲霉属基因的修饰的启动子,其中已经用来自编码丙糖磷酸异构酶的曲霉属基因的未翻译的前导子替换未翻译的前导子;非限制性实例包括来自编码中性α-淀粉酶的黑曲霉基因的修饰的启动子,其中已经用来自编码丙糖磷酸异构酶的构巢曲霉或米曲霉基因的未翻译的前导子替换未翻译的前导子);及其突变型、截短型及杂合型启动子。其他启动子在美国专利号6,011,147中描述。在酵母宿主中,从针对以下各项的基因获得有用的启动子:酿酒酵母烯醇酶(ENO-1)、酿酒酵母半乳糖激酶(GAL1)、酿酒酵母乙醇脱氢酶/甘油醛-3-磷酸脱氢酶(ADH1,ADH2/GAP)、酿酒酵母磷酸丙糖异构酶(TPI)、酿酒酵母金属硫蛋白(CUP1)、和酿酒酵母3-磷酸甘油酸激酶。在罗马诺斯(Romanos)等人,1992,酵母(Yeast)8:423-488中描述了酵母宿主细胞的其他有用启动子。控制序列也可以是由宿主细胞识别以终止转录的转录终止子。该终止子可操作地连接至编码该变体的多核苷酸的3'-末端。在宿主细胞中有功能的任何终止子可以用于本发明中。用于丝状真菌宿主细胞的优选终止子是从以下各项的基因获得:构巢曲霉乙酰胺酶、构巢曲霉邻氨基苯甲酸合酶、黑曲霉葡糖淀粉酶、黑曲霉α-葡糖苷酶、米曲霉TAKA淀粉酶、尖镰孢胰蛋白酶样蛋白酶、里氏木霉β-葡糖苷酶、里氏木霉纤维二糖水解酶I、里氏木霉纤维二糖水解酶II、里氏木霉内切葡聚糖酶I、里氏木霉内切葡聚糖酶II、里氏木霉内切葡聚糖酶III、里氏木霉内切葡聚糖酶V、里氏木霉木聚糖酶I、里氏木霉木聚糖酶II、里氏木霉木聚糖酶III、里氏木霉β-木糖苷酶以及里氏木霉翻译延长因子。酵母宿主细胞的优选终止子从针对以下各项的基因获得:酿酒酵母烯醇化酶、酿酒酵母细胞色素C(CYC1)和酿酒酵母甘油醛-3-磷酸脱氢酶。在罗马诺斯(Romanos)等人,1992,上文中描述了酵母宿主细胞的其他有用终止子。该控制序列也可以是前导子,该前导子是对宿主细胞翻译重要的mRNA的非翻译区。该前导子可操作地连接至编码该变体的多核苷酸的5'-末端。可以使用在宿主细胞中有功能的任何前导子。从米曲霉TAKA淀粉酶和构巢曲霉磷酸丙糖异构酶的基因获得用于丝状真菌宿主细胞的优选前导子用于丝状真菌宿主细胞的优选前导子。从酿酒酵母烯醇化酶(ENO-1)、酿酒酵母3-磷酸甘油酸激酶、酿酒酵母醹-因子和酿酒酵母醇脱氢酶/甘油醛-3-磷酸脱氢酶(ADH2/GAP)的基因获得用于酵母宿主细胞的合适的前导子。控制序列也可以是多聚腺苷化序列,一种与多核苷酸3’-末端可操作地连接并在转录时由宿主细胞识别为向转录的mRNA添加聚腺苷酸残基的信号序列。可以使用在宿主细胞中起作用的任何聚腺苷酸化序列。用于丝状真菌宿主细胞的优选聚腺苷酸化序列是从以下各项的基因获得:构巢曲霉邻氨基苯甲酸合酶、黑曲霉葡糖淀粉酶、黑曲霉α-葡糖苷酶、米曲霉TAKA淀粉酶以及尖镰孢胰蛋白酶样蛋白酶。对于酵母宿主细胞有用的聚腺苷酸化序列在郭(Guo)和谢尔曼(Sherman),1995,分子细胞生物学(Mol.CellularBiol.)15:5983-5990中得以描述。该控制序列还可以是信号肽编码区,编码与变体的N-端连接的信号肽,并且引导该变体进入细胞的分泌通路。该多核苷酸的编码序列的5’-端可以固有地包含信号肽编码序列,该信号肽编码序列在翻译阅读框中与编码该变体的编码序列的区段天然地连接在一起。可替代地,编码序列5’-端可以包含对于该编码序列是外源的信号肽编码序列。在编码序列不天然地包含信号肽编码序列的情况下,可能需要外源信号肽编码序列。可替代地,外源信号肽编码序列可以简单地置换天然信号肽编码序列,以便增加变体的分泌。然而,可以使用指导表达的变体进入宿主细胞的分泌通路的任何信号肽编码序列。用于丝状真菌宿主细胞的有效信号肽编码序列是从黑曲霉中性淀粉酶、黑曲霉葡糖淀粉酶、米曲霉TAKA淀粉酶、特异腐质霉纤维素酶、特异腐质霉内切葡聚糖酶V、柔毛腐质霉脂肪酶和米黑根毛霉天冬氨酸蛋白酶的基因获得的信号肽编码序列。从酿酒酵母α-因子和酿酒酵母转化酶的基因获得用于酵母宿主细胞的有用信号肽。其他的有用的信号肽编码序列由罗曼诺斯(Romanos)等人(1992,上文)描述。该控制序列还可以是编码位于变体的N-末端处的前肽的前肽编码序列。生成的多肽被称为前体酶(proenzyme)或多肽原(或在一些情况下被称为酶原(zymogen))。多肽原通常是无活性的并且可以通过催化切割或自身催化切割来自多肽原的前肽而转化为活性多肽。前肽编码序列可以从以下各项的基因获得:嗜热毁丝霉漆酶(WO95/33836)、米黑根毛霉天冬氨酸蛋白酶和酿酒酵母α-因子。在信号肽序列和前肽序列二者都存在的情况下,该前肽序列定位成紧邻该变体的N-末端并且该信号肽序列定位成紧邻该前肽序列的N-末端。还令人希望的可以是添加相对于宿主细胞的生长来调节该变体的表达的调节序列。调节序列的实例是使得基因的表达响应于化学或物理刺激(包括调控化合物的存在)而开启或关闭的那些序列。在酵母中,可以使用ADH2系统或GAL1系统。在丝状真菌中,可以使用黑曲霉葡糖淀粉酶启动子、米曲霉TAKAα-淀粉酶启动子和米曲霉葡糖淀粉酶启动子、里氏木霉纤维二糖水解酶I启动子以及里氏木霉纤维二糖水解酶II启动子。调节序列的其它实例是那些允许基因扩增的序列。在真核系统中,这些调控序列包括在甲氨蝶呤存在下被扩增的二氢叶酸还原酶基因以及用重金属扩增的金属硫蛋白基因。在这些情况下,编码该变体的多核苷酸将可操作地连接至该调节序列。表达载体本发明还涉及包括编码本发明的变体的多核苷酸、启动子、以及转录和翻译终止信号的重组表达载体。不同的核苷酸和控制序列可以连接在一起以产生重组表达载体,这一重组表达载体可以包括一个或多个便利的限制酶切位点以允许在这些位点处插入或取代编码该变体的多核苷酸。可替代地,可以通过将多核苷酸或包含该多核苷酸的核酸构建体插入用于表达的适当载体中而表达该多核苷酸。在产生该表达载体时,该编码序列位于该载体中,这样使得该编码序列与该供表达的适当控制序列可操作地连接。重组表达载体可以是任何载体(例如,质粒或病毒),其能够方便地进行重组DNA程序,并且能够引起多核苷酸的表达。典型地,载体的选择将取决于载体与向其中待引入载体的宿主细胞的相容性。该载体可以是线性的或闭合的环状质粒。该载体可以是自主复制载体,即,作为染色体外实体存在的载体,其复制独立于染色体复制,例如,质粒、染色体外元件、微染色体或人工染色体。该载体可以包含任何用以保证自我复制的要素。可替代地,该载体可以是这样载体,当它被引入该宿主细胞中时,被整合到基因组中并且与其中已整合了它的一个或多个染色体一起复制。此外,可以使用单一载体或质粒或两个或更多个载体或质粒(这些载体或质粒共同包含待引入到宿主细胞的基因组中的总DNA)或转座子。该载体优选包含一个或多个允许方便地选择转化细胞、转染细胞、转导细胞等细胞的选择性标记。选择性标记是这样一种基因,该基因的产物提供了杀生物剂抗性或病毒抗性、重金属抗性、营养缺陷型的原养型等。用于酵母宿主细胞的适合标记包括,但不限于,ADE2、HIS3、LEU2、LYS2、MET3、TRP1、和URA3。用于在丝状真菌宿主细胞中使用的选择性标记包括但不限于,adeA(磷酸核糖酰氨基咪唑-琥珀羧胺合酶)、adeB(磷酸核糖酰-氨基咪唑合酶)、amdS(乙酰胺酶)、argB(鸟氨酸氨甲酰基转移酶)、bar(草丁膦乙酰转移酶)、hph(潮霉素磷酸转移酶)、niaD(硝酸还原酶)、pyrG(乳清酸核苷-5'-磷酸脱羧酶)、sC(硫酸腺苷基转移酶)、以及trpC(邻氨基苯甲酸合酶)、连同其等效物。优选地用于曲霉细胞中的是构巢曲霉或米曲霉amdS和pyrG基因以及吸水链霉菌bar基因。在木霉属细胞中优选使用的是adeA、adeB、amdS、hph以及pyrG基因。选择性标记可以是如在WO2010/039889中描述的双选择性标记系统。在一个方面中,双选择性标记是hph-tk双选择性标记系统。载体优选包含允许载体整合到宿主细胞的基因组中或载体在细胞中独立于基因组自主复制的一个或多个元件。对于整合到该宿主细胞基因组中,该载体可以依靠编码该变体的多核苷酸序列或者用于通过同源或非同源重组整合到该基因组中的该载体的任何其他元件。可替代地,该载体可以包含用于指导通过同源重组而整合到宿主细胞基因组中的一个或多个染色体中的一个或多个精确位置的另外的多核苷酸。为了增加在精确位置整合的可能性,这些整合元件应包含足够数量的核酸,例如100至10,000个碱基对、400至10,000个碱基对、以及800至10,000个碱基对,这些碱基对与对应的靶序列具有高度的序列一致性以提高同源重组的可能性。这些整合元件可以是与宿主细胞的基因组内的靶序列同源的任何序列。此外,这些整合元件可以是非编码多核苷酸或编码多核苷酸。在另一个方面,该载体可以通过非同源重组整合到宿主细胞的基因组中。对于自主复制,该载体可以进一步包括使该载体能够在所讨论的宿主细胞中自主复制的复制起点。复制起点可以是在细胞中起作用的介导自主复制的任何质粒复制子。术语“复制起点”或“质粒复制子”意指使质粒或载体能够在体内复制的多核苷酸。用于在酵母宿主细胞中使用的复制起点的实例是2微米复制起点、ARS1、ARS4、ARS1与CEN3的组合、以及ARS4与CEN6的组合。在丝状真菌细胞中有用的复制起点的实例是AMA1和ANS1(格姆斯(Gems)等人,1991,基因(Gene)98:61-67;卡伦(Cullen)等人,1987,核酸研究(NucleicAcidsRes.)15:9163-9175;WO00/24883)。AMA1基因的分离和包括该基因的质粒或载体的构建可以根据披露于WO00/24883中的方法完成。可以将本发明的多核苷酸的多于一个的拷贝插入到一个宿主细胞中以增加变体的产生。通过将序列的至少一个另外的拷贝整合到宿主细胞基因组中或通过包括一个与该多核苷酸一起的可扩增的选择性标记基因可以获得多核苷酸的增加的拷贝数目,其中通过在适当的选择性试剂的存在下培养细胞可以选择包含选择性标记基因的经扩增的拷贝的细胞、以及由此该多核苷酸的另外的拷贝。用于连接以上所描述的元件以构建重组表达载体的程序是本领域的普通技术人员熟知的(参见,例如,萨姆布鲁克(Sambrook)等人,1989,见上文)。宿主细胞本发明还涉及重组宿主细胞,这些重组宿主细胞包括编码本发明的变体的、可操作地连接至一个或多个控制序列的多核苷酸,该一个或多个控制序列指导本发明的变体的产生。将包括多核苷酸的构建体或载体引入宿主细胞中,这样使得该构建体或载体被维持作为染色体整合体或作为自主复制的染色体外载体,如早前所述。术语“宿主细胞”涵盖由于复制过程中发生的突变与亲本细胞不同的亲本细胞的任何后代。宿主细胞的选择在很大程度上将取决于编码该变体的基因及其来源。宿主细胞可以是真核生物,如哺乳动物、昆虫、植物或真菌细胞。该宿主细胞可以是真菌细胞。如在此所用的“真菌”包括子囊菌门(Ascomycota)、担子菌门(Basidiomycota)、壶菌门(Chytridiomycota)和接合菌门(Zygomycota)以及卵菌门(Oomycota)和所有有丝分裂孢子真菌(如霍克斯沃思(Hawksworth)等人定义,引自:安斯沃斯(Ainsworth)和比斯比(Bisby)的真菌大词典(DictionaryofTheFungi),第8版,1995,国际CAB,大学出版社(UniversityPress),剑桥(Cambridge),英国)。真菌宿主细胞可以是酵母细胞。如在此使用的“酵母”包括产子嚢酵母(内孢霉目)、产担子酵母和属于半知菌类(芽孢纲)的酵母。由于酵母的分类可能在将来变化,为了本发明的目的,酵母应当如酵母的生物学与活性(BiologyandActivitiesofYeast)(斯金纳(Skinner),帕斯莫尔(Passmore)和达文波特(Davenport)编著,应用细菌学学会专题论文集系列9(Soc.App.Bacteriol.SymposiumSeriesNo.9),1980)所描述那样定义。酵母宿主细胞可以是假丝酵母属细胞、汉逊酵母属细胞、克鲁维酵母属细胞、毕赤酵母属细胞、酵母菌属细胞、裂殖酵母或耶罗维亚酵母属细胞、如乳酸克鲁维酵母细胞、卡氏酵母细胞、酿酒酵母细胞、糖化酵母细胞、道格拉斯酵母(Saccharomycesdouglasii)细胞、克鲁弗酵母细胞、诺地酵母细胞、卵形酵母细胞或解脂耶罗维亚酵母细胞。真菌宿主细胞可以是丝状真菌细胞。“丝状真菌”包括真菌门(Eumycota)和卵菌门(Oomycota)亚类的所有丝状形式(如霍克斯沃思等人,1995,上文定义)。丝状真菌通常的特征在于由几丁质、纤维素、葡聚糖、壳聚糖、甘露聚糖、以及其他复杂多糖构成的菌丝体壁。营养生长是通过菌丝延伸,而碳分解代谢是专性需氧的。相反,酵母(如酿酒酵母)的营养生长是通过单细胞菌体的出芽(budding),而碳分解代谢可以是发酵的。丝状真菌宿主细胞可以是枝顶孢霉属、曲霉属、短梗霉属、烟管霉属(Bjerkandera)、拟腊菌属、金孢子菌属、鬼伞属、革盖菌属(Coriolus)、隐球菌属、线黑粉菌科(Filibasidium)、镰孢属、腐质霉属、梨孢菌属(Magnaporthe)、毛霉属、毁丝霉属、新美鞭菌属、链孢菌属、拟青霉属、青霉属、平革菌属、射脉菌属(Phlebia)、瘤胃壶菌属、侧耳属(Pleurotus)、裂褶菌属、篮状菌属、嗜热子囊菌属、梭孢壳属、弯颈霉属、栓菌属(Trametes)或木霉属细胞。例如,丝状真菌宿主细胞可以是泡盛曲霉、臭曲霉、烟曲霉、日本曲霉、构巢曲霉、黑曲霉、米曲霉、黑刺烟管菌(Bjerkanderaadusta)、干拟蜡菌(Ceriporiopsisaneirina)、卡内基拟蜡菌(Ceriporiopsiscaregiea)、浅黄拟蜡孔菌(Ceriporiopsisgilvescens)、潘诺希塔拟蜡菌(Ceriporiopsispannocinta)、环带拟蜡菌(Ceriporiopsisrivulosa)、微红拟蜡菌(Ceriporiopsissubrufa)、虫拟蜡菌(Ceriporiopsissubvermispora)、狭边金孢子菌(Chrysosporiuminops)、嗜角质金孢子菌、卢克诺文思金孢子菌(Chrysosporiumlucknowense)、粪状金孢子菌(Chrysosporiummerdarium)、租金孢子菌、女王杜香金孢子菌(Chrysosporiumqueenslandicum)、热带金孢子菌、褐薄金孢子菌(Chrysosporiumzonatum)、灰盖鬼伞(Coprinuscinereus)、毛革盖菌(Coriolushirsutus)、杆孢状镰孢、谷类镰孢、库威镰孢、大刀镰孢、禾谷镰孢、禾赤镰孢、异孢镰孢、合欢木镰孢、尖镰孢、多枝镰孢、粉红镰孢、接骨木镰孢、肤色镰孢、拟分枝孢镰孢、硫色镰孢、圆镰孢、拟丝孢镰孢、镶片镰孢、特异腐质霉、柔毛腐质霉、米黑毛霉、嗜热毁丝霉、粗糙链孢菌、产紫青霉、黄孢平革菌(Phanerochaetechrysosporium)、射脉菌(Phlebiaradiata)、刺芹侧耳(Pleurotuseryngii)、埃默森篮状菌、土生梭孢霉、长域毛栓菌(Trametesvillosa)、变色栓菌(Trametesversicolor)、哈茨木霉、康宁木霉、长枝木霉、里氏木霉或绿色木霉细胞。可以将真菌细胞通过涉及原生质体形成、原生质体转化和细胞壁重建的方法以本身公知的方式转化。用于转化曲霉属和木霉属宿主细胞的适合程序在EP238023和约尔顿(Yelton)等人,1984,美国国家科学院院刊(Proc.Natl.Acad.Sci.USA)81:1470-1474以及科里蒂森(Christensen)等人,1988,生物/技术(Bio/Technology)6:1419-1422中描述。用于转化镰刀菌属物种的适合方法在马拉迪耶(Malardier)等人,1989,基因(Gene)78:147-156和WO96/00787中描述。可以使用由如以下文献描述的程序转化酵母:贝克尔(Becker)和瓜伦特(Guarente),在阿贝尔森(Abelson),J.N.和西蒙(Simon),M.I.编,酵母遗传学与分子生物学指南,酶学方法(GuidetoYeastGeneticsandMolecularBiology,MethodsinEnzymology),第194卷,第182-187页,学术出版社有限公司(AcademicPress,Inc.),纽约;伊藤(Ito)等人,1983,细菌学杂志(J.Bacteriol.)153:163;以及哈尼恩(Hinnen)等人,1978,美国国家科学院院刊(Proc.Natl.Acad.Sci.USA)75:1920。产生方法本发明还涉及产生变体的方法,这些方法包括(a)在有助于该变体的产生的条件下培养本发明的重组宿主细胞;并且任选地(b)回收该变体。使用本领域已知的方法在适合于产生该变体的一种营养培养基中培养这些宿主细胞。例如,可以通过摇瓶培养,或者在适合的培养基中并在允许该变体表达和/或分离的条件下在实验室或工业发酵罐中进行小规模或大规模发酵(包括连续发酵、分批发酵、分批给料发酵或固态发酵)来培养该细胞。该培养是使用本领域中已知的程序,在适合的营养培养基中发生,该培养基包括碳和氮来源及无机盐。适合的培养基从商业供应商可获得或可以根据公开的组成(例如,在美国典型培养物保藏中心的目录中)制备。如果该变体被分泌到该营养培养基中,则该变体可直接从该培养基中回收。如果该变体没有分泌,则它可从细胞裂解液中回收。可以使用本领域已知的对木葡聚糖内糖基转移酶特异的方法检测这些变体。这些检测方法包括但不局限于,特异性抗体的使用、酶产物的形成或酶底物的消失。例如,酶测定法可用于确定如在此所述的变体的活性。该变体可以使用本领域已知的方法回收。例如,可以通过多种常规程序从该营养培养基中回收该变体,这些常规程序包括但不局限于收集、离心、过滤、萃取、喷雾干燥、蒸发、或沉淀。在一个方面中,回收整个发酵液。可以通过本领域中已知的多种程序来纯化变体以获得基本上纯的变体,这些程序包括但不限于色谱法(例如,离子交换色谱、亲和色谱、疏水作用色谱、色谱聚焦、以及尺寸排阻色谱)、电泳程序(例如,制备型等电点聚焦)、差别溶解度(例如,硫酸铵沉淀)、SDS-PAGE、或萃取(参见,例如,蛋白质纯化(ProteinPurification),詹森(Janson)和赖登(Ryden)编辑,VCH出版社(VCHPublishers),纽约,1989)。发酵液配制品或细胞组合物本发明还涉及包含本发明的变体的发酵液配制品或细胞组合物。发酵液产物进一步包括在发酵过程中使用的另外的成分,例如像,细胞(包括包含编码本发明的变体的基因的宿主细胞,这些宿主细胞被用于产生感兴趣的变体)、细胞碎片、生物质、发酵介质和/或发酵产物。在一些实施例中,该组合物是包含一种或多种有机酸、杀灭的细胞和/或细胞碎片以及培养基的细胞杀灭的全培养液。如在此使用的术语“发酵液”是指由细胞发酵产生、不经历或经历最低限的回收和/或纯化的制剂。例如,当微生物培养物生长至饱和,在碳限制条件下孵育以允许蛋白质合成(例如,由宿主细胞进行酶的表达)并且分泌到细胞培养基中时,产生发酵液。发酵液可以包含在发酵结束时得到的发酵材料的未分级的或分级的内容物。典型地,发酵液是未分级的并且包括用过的培养基以及例如通过离心去除微生物细胞(例如,丝状真菌细胞)之后存在的细胞碎片。在一些实施例中,发酵液包含用过的细胞培养基、胞外酶以及有活力的和/或无活力的微生物细胞。在一个实施例中,该发酵液配制品和细胞组合物包括第一有机酸组分(包括至少一种1-5碳的有机酸和/或其盐)以及第二有机酸组分(包括至少一种6碳或更多碳的有机酸和/或其盐)。在具体实施例中,该第一有机酸组分是乙酸、甲酸、丙酸、其盐,或前述两种或更多种的混合物;并且该第二有机酸组分是苯甲酸、环己烷羧酸、4-甲基戊酸、苯乙酸、其盐,或前述两种或更多种的混合物。在另一个实施例中,该组合物包含一种或多种有机酸,并且任选地进一步包含杀灭的细胞和/或细胞碎片。在一个实施例中,从细胞杀灭的全培养液中去除这些杀灭的细胞和/或细胞碎片,以提供不含这些组分的组合物。这些发酵液配制品或细胞组合物可以进一步包括防腐剂和/或抗微生物(例如,抑菌)剂,包括但不限于山梨醇、氯化钠、山梨酸钾、以及本领域中已知的其他试剂。这些发酵液配制品或细胞组合物可以进一步包括多种酶活性,如选自下组的一种或多种(例如,若干种)酶,该组由以下各项组成:水解酶、异构酶、连接酶、裂解酶、氧化还原酶以及转移酶。该细胞杀灭的全培养液或组合物可以包含在发酵结束时得到的发酵材料的未分级的内容物。典型地,该细胞杀灭的全培养液或组合物包含用过的培养基以及在微生物细胞(例如,丝状真菌细胞)生长至饱和、在碳限制条件下孵育以允许蛋白合成之后存在的细胞碎片。在一些实施例中,该细胞杀灭的全培养液或组合物包含用过的细胞培养基、胞外酶和杀灭的丝状真菌细胞。在一些实施例中,可以使用本领域已知的方法来使细胞杀灭的全培养液或组合物中存在的微生物细胞透性化和/或裂解。如在此描述的全培养液或细胞组合物典型地是液体,但是可以包含不溶性组分,例如杀灭的细胞、细胞碎片、培养基组分和/或一种或多种不溶性酶。在一些实施例中,可以去除不溶性组分以提供澄清的液体组合物。本发明的全液配制品和细胞组合物可以通过WO90/15861或WO2010/096673中所描述的方法来产生。酶组合物本发明还涉及包括本发明的一种变体的组合物。优选地,这些组合物富含这种变体。术语“富含”指示该组合物的木葡聚糖内糖基转移酶活性已经增加,例如,富集因子为至少1.1。这些组合物可以包括本发明的一种变体作为主要酶组分,例如一种单组分组合物。可替代地,这些组合物可以包括多种酶活性,如选自下组的一种或多种(例如,若干种)酶,该组由以下各项组成:水解酶、异构酶、连接酶、裂解酶、氧化还原酶、以及转移酶。这些组合物可以根据本领域已知的方法制备,并可以是液体或干燥组合物的形式。可以根据本领域中已知的方法稳定这些组合物。下文中给出本发明的组合物的优选用途的实例。组合物的剂量以及组合物使用的其他条件可以根据本领域已知的方法来确定。用途本发明的这些变体可以用于根据WO97/23683为纤维素材料(如织物或纸和纸浆产品)提供改进的强度和/或形状保持和/或抗皱特性的工艺中。本发明的这些变体还可以用于洗衣和/或织物和/或颜色护理组合物中,以根据WO2001/07556针对纤维素纤维刷新和/或恢复改进的拉伸强度,增强的抗皱、抗起球和抗缩水特性。通过以下实例进一步对本发明进行描述,但不应将其理解为对本发明范围的限制。实例培养基和溶液LB板由以下各项构成:10g的胰蛋白胨、5g的酵母提取物、5g的NaCl、15g的细菌琼脂(bacteriologicalagar)、以及加至1升的去离子水。LB培养基由以下各项构成:10g的胰蛋白胨、5g的酵母提取物、5g的NaCl、以及加至1升的去离子水。基本培养基琼脂板由以下各项构成:342.3g的蔗糖、10g的葡萄糖、4g的MgSO4·7H20、6g的NaNO3、0.52g的KCl、1.52g的KH2PO4、0.04mg的Na2B4O7·10H2O、0.4mg的CuSO4·5H2O、1.2mg的FeSO4·7H2O、0.7mg的MnSO4·2H2O、0.8mg的Na2MoO4·2H2O、10mg的ZnSO4·7H2O、500mg的柠檬酸、4mg的D-生物素、20g的纯净琼脂、以及加至1升的去离子水。合成的、缺少尿苷的确定成分培养基由以下各项构成:18mg的腺嘌呤半硫酸盐、76mg的丙氨酸、76mg的精氨酸盐酸盐、76mg的天冬酰胺一水合物、76mg的天门冬氨酸、76mg的半胱氨酸盐酸盐一水合物、76mg的谷氨酸单钠盐、76mg的谷氨酰胺、76mg的甘氨酸、76mg的组氨酸、myo-76mg的肌醇、76mg的异亮氨酸、380mg的亮氨酸、76mg的赖氨酸单盐酸盐、76mg的蛋氨酸、8mg的对-氨基苯甲酸钾盐、76mg的苯丙氨酸、76mg的脯氨酸、76mg的丝氨酸、76mg的苏氨酸、76mg的色氨酸、76mg的酪氨酸二钠盐、76mg的缬氨酸、以及加至1升的去离子水。YP+2%葡萄糖培养基由以下各项构成:10g的酵母提取物、20g的蛋白胨、20g的葡萄糖、以及加至1升的去离子水。YP+2%麦芽糊精培养基由以下各项构成:10g的酵母提取物、20g的蛋白胨、20g的麦芽糊精、以及加至1升的去离子水。2XYT+氨比西林平板由16g的胰蛋白胨,10g的酵母提取物,5g的氯化钠,15g的细菌琼脂,和加至1升的去离子水组成。在高压蒸汽处理的培养基回火到55℃后,添加1ml的100mg/ml的氨比西林。TAE缓冲液由以下各项构成:4.84g的Tris碱、1.14ml的冰醋酸、2ml的0.5MEDTA(pH8.0)、并且去离子水补足至1升。TBE缓冲液由以下各项构成:10.8g的Tris碱、5.5g硼酸、4ml的0.5MEDTA(pH8.0)、以及加至1升的去离子水。实例1:碘比色法测定来确定木葡聚糖内糖基转移酶活性木葡聚糖内糖基转移酶活性使用由布洛瓦(Sulova)等人,1995,分析生物化学(AnalyticalBiochemistry)229:80-85中描述的碘比色测定的修改版本进行测定。对于每个反应,5μl的罗望子木葡聚糖(麦格酶国际公司,布雷,英国)(5mg/ml在水中)与20μl的木葡聚糖低聚物(麦格酶国际公司,布雷,英国)(5mg/ml在水中)和10μl的400mM柠檬酸钠pH5.5合并,并且分散到96孔板中。反应通过添加5μl的液体培养液至每个孔中启动,并且在37℃孵育板10分钟。反应通过添加200μl的由14%(w/v)Na2SO4、0.2%KI、0.1MHCl和0.5%I2构成的溶液猝灭,并且在黑暗中孵育30分钟,然后在M5分光光度计(MolecularDevices公司,桑尼维尔,加利福尼亚州,美国)中在620nm测量吸光度。实例2:荧光素异硫氰酸酯标记的木葡聚糖的产生根据由周(Zhou)等人,2006,生物催化与生物转化(BiocatalysisandBiotransformation)24:107-120所描述的程序,通过木葡聚糖低聚物的还原端的还原氨化,随后在100mM碳酸氢钠(pH9.0)中在室温下将XGO的氨基基团结合到荧光素异硫氰酸酯异构体I(西格玛奥德里奇(SigmaAldrich),圣路易斯,密苏里州,美国)24小时,来产生荧光素异硫氰酸酯标记的木葡聚糖低聚物(FITC-XGO)。将结合反应产物在真空中浓缩干燥,溶解于0.5ml的去离子水,并且通过硅胶层析进行纯化,该硅胶层析用从100:0:0.04至70:30:1梯度的乙腈:水:乙酸作为流动相进行洗脱。通过蒸发该缓冲液、溶解于D2O(西格玛奥德里奇,圣路易斯,密苏里州,美国),并且使用Varian400MHzMercuryVx(安捷伦(Agilent),圣克拉拉,加利福尼亚州,美国)通过1HNMR分析来确认纯度和产物同一性。在-20℃下在黑暗中,储存干燥的FITC-XGO,并且在解冻过程中干燥。一mg的FITC-XGOs与在200μl反应中的每ml的20mM柠檬酸钠pH5.0的1mg的罗望子木葡聚糖(麦格酶公司,布雷,英国)和18mg的VaXET16一起孵育至少30分钟。合并样品混合物并且通过添加冰冷的乙醇至80%(v/v)的终浓度进行沉淀,并且在4℃下孵育过夜。沉淀的荧光素异硫氰酸标记木葡聚糖(FITC-XG)通过使用LEGENDTMRT加离心机(赛默飞世尔科技公司(ThermoScientific),沃尔瑟姆,马萨诸塞州,美国)在3000rpm离心,倾析掉乙醇进行回收,并且在室温下干燥24小时。将FITC-XG溶解在最小体积的去离子水中直到溶解并储存在-20℃。将冷冻的FITC-XG解冻并且冻干过夜。将冻干粉末溶解在5000μl的去离子水中并且通过在488nm的吸光度定量。按以下方式制备大规模批次的FITC-XG。在去离子水中制备每ml溶液7.9mg的FITC-XGO。彻底混合四十ml的10mg的罗望子木葡聚糖(麦格酶公司,布雷,英国)/ml的去离子水、452ml的7.9mg的FITC-XGO/ml的去离子水、2ml的400mM柠檬酸钠(pH5.5)、和1.2ml的1.4mg的VaXET16/ml的20mM柠檬酸钠(pH5.5),并且在室温下孵育过夜。在过夜孵育后,通过添加冰冷的乙醇至终体积110ml来沉淀FITC-XG,彻底混合,并且在4℃下整夜孵育。用水洗涤沉淀的FITC-XG,并且然后转移到圆底烧瓶(Erlenmeyerbulb)中。通过使用EZ-2Elite蒸发器(SPScientific/Genevac公司,斯通里奇,纽约,美国)蒸发4小时来去除残余的水和乙醇。将干燥的样品溶解于水中,并且用去离子水将体积调整到48ml,以产生,在所希望的100kDa的平均分子量下,5mg/ml的最终FITC-XG浓度。实例3:荧光偏振测定以确定木葡聚糖内糖基转移活性使用以下测定来确定木葡聚糖内糖基转移活性。如在实例2中所描述的制备200μl的反应,该反应包含1mg的罗望子木葡聚糖/ml、0.01mg/mlFITC-XGO,并且将10μl的适当稀释的XET在25℃下在不透明96孔微量滴定板中的20mM柠檬酸钠(pH5.5)中孵育10分钟。在这个时间段,以顶级阅读方向,用490nm的激发波长、520nm的发射波长、激发路径中495截止滤波器、高精度(100次读取)、和中光电倍增管的灵敏度,使用M5酶标仪(分子器件公司(MolecularDevices),森尼维耳市,加利福尼亚州,美国)连续监测荧光偏振。将荧光XGO以XET依赖性掺入非荧光XG中导致随着时间增加荧光偏振。使用偏振时间进程曲线的线性区域的斜率来确定该活性。实例4:质粒pMMar27的构建构建质粒pMMar27,用于表达酵母中的土生梭孢壳霉Cel6A纤维二糖水解酶II。该质粒产生自酵母表达载体的谱系:质粒pMMar27构建自质粒pBM175b;质粒pBM175b构建自质粒pBM143b(WO2008/008950)和质粒pJLin201;并且质粒pJLin201构建自pBM143b。除紧邻pBM143b中疏棉状嗜热丝孢菌脂肪酶变体基因的下游的XbaI位点突变为独特的NheI位点以外,质粒pJLin201与pBM143b相同。使用IIXL定点诱变试剂盒(Stratagenee公司,拉荷亚,加利福尼亚州,美国)将pBM143b中的XbaI序列(TCTAGA)变为NheI序列(gCTAGc)。以下示出了用来突变该位点的引物。引物999551(正义):5’-ACATGTCTTTGATAAgCTAGcGGGCCGCATCATGTA-3’(SEQIDNO:52)引物999552(反义):5’-TACATGATGCGGCCCgCTAGcTTATCAAAGACATGT-3’(SEQIDNO:53)小写代表突变的核苷酸。最终体积为50μl的PCR由以下各项构成:125ng的以上每种引物、20ng的pBM143b、1X反应缓冲液(Stratagene公司,拉荷亚,加利福尼亚州,美国)、3μl的(Stratagene公司,拉荷亚,加利福尼亚州,美国)、1μl的dNTP混合物、以及1μl的2.5单元/mlPfuUltraHFDNA聚合酶(Stratagene公司,拉荷亚,加利福尼亚州,美国)。使用热循环仪(EppendorfAG公司,汉堡,德国)仪进行该反应,程序为1个循环,在95℃下,持续1分钟;18个循环,每个循环在95℃下持续50秒,60℃持续50秒,和68℃持续6分钟6秒;以及1个循环,在68℃下,持续7分钟。在PCR反应后,将该管置于冰上2分钟。向PCR中直接添加一微升的DpnI(普洛麦格公司(Promega),菲奇堡,威斯康辛州,美国),并且在37℃下孵育1小时。根据制造商的说明,使用2μl体积的DpnI消化的反应来转化大肠杆菌XL10高效感受态细胞(Stratagene公司,拉荷亚,加利福尼亚州,美国)。在2XYT加氨比西林板上对大肠杆菌转化体进行选择。使用9600(凯杰公司,巴伦西亚,加利福尼亚州,美国)将质粒DNA从转化体中的若干种分离。通过限制性酶切和测序分析来确认具有所希望的NheI变化的一个质粒,并且指定为质粒pJLin201。为了消除由定点突变引入的可能的PCR错误,通过将包含NheI位点的片段克隆回质粒pBM143b中来构建质粒pBM175b。简言之,将质粒pJLin201用NdeI和MluI进行消化,并且将得到的片段克隆到之前用相同的酶使用快速连接试剂盒(罗氏诊断公司(RocheDiagnosticsCorporation),印第安纳波利斯,印第安纳州,美国)消化的pBM143b中。简言之,将7μl的NdeI/MluI消化的pJLin201片段和1μl的消化的pBM143b与2μl的5XDNA稀释液(罗氏诊断公司,印第安纳波利斯,印第安纳州,美国)、10μl的2XT4DNA连接缓冲液(罗氏诊断公司,印第安纳波利斯,印第安纳州,美国)、和1μl的T4DNA连接酶(罗氏诊断公司,印第安纳波利斯,印第安纳州,美国)进行混合,并且在室温下孵育15分钟。将2微升的连接转化到XL1-蓝亚克隆-级感受态细胞(Stratagene公司,拉荷亚,加利福尼亚州,美国)细胞并且散布于2XYT加氨比西林板上。使用9600从若干转化体中纯化质粒DNA,并使用3130XL遗传分析仪(应用生物系统公司(AppliedBiosystems),福斯特城,加利福尼亚州,美国)通过DNA测序进行分析,以鉴定包含所希望的构巢曲菌pyrG插入物的质粒。将一个具有预期的DNA序列的质粒指定为pBM175b。质粒pMMar27构建自pBM175b和具有设计用于插入所消化pBM175b的突出端的土生梭孢壳霉Cel6A纤维二糖水解酶II的扩增的基因。在CUPI启动子的控制下包含疏棉状嗜热丝孢菌脂肪酶变体基因的质粒pBM175b包含独特的HindIII和NheI位点,来去除脂肪酶基因。将质粒pBM175b用这些限制性内切酶进行消化,以去除脂肪酶基因。在消化后,将空载体通过使用TBE缓冲液的1.0%琼脂糖凝胶电泳进行分离,其中将大约5,215bp的片段从凝胶切离,并且使用凝胶提取试剂盒(凯杰公司,巴伦西亚,加利福尼亚州,美国)进行提取。连接反应(20μl)由1X缓冲液(BD生物科学公司(BDBiosciences),帕洛阿尔托(PaloAlto),加利福尼亚州,美国),1XBSA(BD生物科学公司,帕洛阿尔托(PaloAlto),加利福尼亚州,美国),1μl酶(1:10稀释)(BD生物科学公司,帕洛阿尔托(PaloAlto),加利福尼亚州,美国),用HindIII和NheI消化的99ngpBM175b,和36ng的纯化的土生梭孢壳霉Cel6A纤维二糖水解酶IIPCR产物。将该反应在室温下孵育30分钟。将2μl体积的反应转化到大肠杆菌高效感受态细胞(Stratagene公司,拉荷亚,加利福尼亚州,美国)中。在每ml补充有100μg的氨比西林的LB平板上选择转化体。挑选一种菌落,其包含插入pBM175b载体替代脂肪酶基因产生pMMar27的土生梭孢壳霉Cel6A(图1)。所选择的质粒在从起始密码子的位置228处包含PCR错误,TCT代替TCC,但是导致中的土生梭孢壳霉Cel6A纤维二糖水解酶II的沉默变化。实例5:pEvFz1表达载体的构建表达载体pEvFz1是通过修饰pBM120a(美国专利8,263,824)来进行构建,以包括NA2/NA2-tpi启动子、黑曲霉淀粉葡糖苷酶终止子序列(AMG终止子)、和作为选择性标志物的构巢曲霉乳清酸核苷-5’磷酸脱羧酶基因(pyrG)。通过该将构巢曲霉pyrG基因从pAlLo2(WO2004/099228)克隆到pBM120a中来产生质粒pEvFz1。将质粒pBM120a和pAlLo2用NsiI在37℃下消化过夜。将所得4176bp线性pBM120a载体片段和来自pAlLo2的1479bppyrG基因插入片段各自使用TAE缓冲液通过0.7%琼脂糖凝胶电泳纯化,从凝胶切离,并使用凝胶提取试剂盒进行提取。使用QUICKLIGATIONTM试剂盒(新英格兰生物实验室,贝弗利,马萨诸塞州,美国)将1479bppyrG基因插入片段连接至NsiI消化的pBM120a片段。连接反应由以下构成:1XQUICKLIGATIONTM反应缓冲液(新英格兰生物实验室,贝弗利,马萨诸塞州,美国)、50ng的NsiI消化的pBM120a载体、54ng的1479bpNsiI消化的pyrG基因插入物、以及1μl的T4DNA连接酶,总体积为20μl。将连接混合物在37℃下孵育15分钟,随后在50℃下孵育15分钟,并且然后放置在冰上。将1μl的连接混合物转化到ONETOP10化学感受大肠杆菌细胞(英杰公司,卡尔斯巴德,加利福尼亚州,美国)中。在2XYT加氨比西林板上对转化体进行选择。使用9600从若干转化体中纯化质粒DNA,并使用3130XL遗传分析仪通过DNA测序进行分析,以鉴定包含所希望的构巢曲菌pyrG插入物的质粒。将一个具有预期的DNA序列的质粒指定为pEvFz1(图2)。实例6:构建作为酵母/大肠杆菌/米曲霉穿梭载体的质粒pDLHD0006使用酵母重组克隆,将质粒pDLHD0006构建为基本载体,以使得米曲霉表达盒文库建立。通过使用酵母重组克隆(表1)合并三种DNA片段来产生质粒pDLHD0006:包含大肠杆菌pUC复制起点、大肠杆菌β-内酰胺酶(ampR)选择性标志物、URA3酵母选择性标志物、和来自pMMar27(实例4)的酵母2微米复制起点的片段1;包含NA2-tpi启动子(来自编码黑曲霉中性α-淀粉酶和米曲霉丙糖磷酸异构酶的基因的启动子的杂合体)、疏棉状嗜热丝孢菌脂肪酶开放阅读框(ORF)、和来自pJaL1262(WO2013/178674)的黑曲霉葡糖糖化酶终止子的片段2;和来自pEvFz1(实例3)的包含构巢曲霉pyrG选择标志物的片段5。表I.pDLHD0006PCR含量PCR模板片段1大肠杆菌ori/AmpR/URA/2微米(4.1kb)pMMar27片段2NA2-tpiPR/脂肪酶/Tamg(4.5kb)pJaL1262片段3来自pEvFz1的pyrG基因(1.7kb)pEvFz1使用以下所示的引物613017(正义)和613018(反义)扩增片段1。设计引物613017包含与片段3(小写)具有序列同源性的侧翼区,并且设计引物613018包含与片段2(小写)具有序列同源性的侧翼区以使得酵母在这三个PCR片段之间重组克隆。引物613017(正义):ttaatcgccttgcagcacaCCGCTTCCTCGCTCACTGACTC(SEQIDNO:54)引物613018(反义):acaataaccctgataaatgcGGAACAACACTCAACCCTATCTCGGTC(SEQIDNO:55)片段1在反应中通过PCR来扩增,该反应由10ng的质粒pMMar27、0.5μl的DNA聚合酶(新英格兰生物实验室公司,伊普斯威奇,马萨诸塞州,美国)、20pmol的引物613017、20pmol的引物613018、1μl的10mMdNTP、10μl的5XHF缓冲液(新英格兰生物实验室公司,伊普斯威奇,马萨诸塞州,美国)、和35.5μl的水构成。该反应在热循环仪中孵育,程序为1个循环,在98℃下持续30秒;以及30个循环,每个循环在98℃下持续10秒,在60℃下持续10秒,以及在72℃下持续1.5分钟。将得到的4.1kbPCR产物(片段1)直接使用用于与以下片段2和3的酵母重组。使用以下所示的引物613019(正义)和613020(反义)扩增片段2。设计引物613019包含与片段1(小写)具有序列同源性的侧翼区,并且设计引物613020包含与片段3(小写)具有序列同源性的侧翼区以使得酵母在这三个PCR片段之间重组克隆。613019(正义):agatagggttgagtgttgttccGCATTTATCAGGGTTATTGTCTCATGAGCGG(SEQIDNO:56)613020(反义):ttctacacgaaggaaagagGAGGAGAGAGTTGAACCTGGACG(SEQIDNO:57)片段2在反应中通过PCR来扩增,该反应由10ng的质粒pJaL1262、0.5μl的DNA聚合酶、20pmol的引物613019、20pmol的引物613020、1μl的10mMdNTP、10μl的5XHF缓冲液、和35.5μl的水构成。在热循环仪中孵育该反应,程序为1个循环,在98℃下,持续30秒;30个循环,每个循环在98℃持续10秒,60℃持续10秒,以及72℃持续2分钟;以及20℃保持。将得到的4.5kbPCR产物(片段2)直接使用用于与以上片段1和以下片段3的酵母重组。使用以下所示的引物613022(正义)和613021(反义)扩增片段3。设计引物613021包含与片段2(小写)具有序列同源性的侧翼区,并且设计引物613022包含与片段1(小写)具有序列同源性的侧翼区以使得酵母在这三个PCR片段之间重组克隆。引物613022(正义):aggttcaactctctcctcCTCTTTCCTTCGTGTAGAAGACCAGACAG(SEQIDNO:58)引物613021(反义):tcagtgagcgaggaagcggTGTGCTGCAAGGCGATTAAGTTGG(SEQIDNO:59)片段3在反应中通过PCR来扩增,该反应由10ng的质粒pEvFz1(实例5)、0.5μl的DNA聚合酶、20pmol的引物613021、20pmol的引物613022、1μl的10mMdNTP、10μl的5XHF缓冲液、和35.5μl的水构成。在热循环仪中孵育该反应,程序为1个循环,在98℃下,持续30秒;30个循环,每个循环在98℃持续10秒,60℃持续10秒,以及72℃持续2分钟;以及20℃保持。将得到的1.7kbPCR产物(片段3)直接使用用于与以上片段1和2的酵母重组。使用基于酵母同源性的重组克隆,使用以下程序来合并三个PCR片段。将三种PCR片段的每种的20μl等分试样与来自鲑鱼睾丸的100μg的单链脱氧核糖核酸(西格玛奥德里奇,圣路易斯,密苏里州,美国)、100μl的菌株YNG318的感受态酵母细胞(酿酒酵母ATCC208973)、和600μl的PLATE缓冲液(西格玛奥德里奇,圣路易斯,密苏里州,美国)合并,并且混合。将反应在30℃下以200rpm振荡孵育30分钟。然后将该反应在42℃下在没有振荡的情况下继续15分钟。将这些细胞通过在5,000xg下离心1分钟进行沉淀,并且弃去上清液。将细胞球粒悬浮于200μl的高压蒸汽处理的水中,并且分在两个包含合成的所定义的培养基缺少尿苷的琼脂平板,并且在30℃下孵育3天。将这些酵母菌落使用1ml的高压蒸汽处理的水从平板中分离。将这些细胞通过在13,000xg下离心30秒进行沉淀,并且将100μl等分试样的玻璃珠添加到该管中。将细胞和珠混合物悬浮在250μl的P1缓冲液(凯杰公司,巴伦西亚,加利福尼亚州,美国)中,并且然后涡旋1分钟来裂解这些细胞。使用旋转迷你制备型试剂盒(凯杰公司(QIAGENInc.),巴伦西亚,加利福尼亚州,美国),将质粒DNA进行纯化。根据制造商的说明书,然后将3μl等分试样的质粒DNA转化到大肠杆菌ONETOP10电转化感受态细胞(英杰公司,卡尔斯巴德,加利福尼亚州,美国)中。将50μl的转化细胞散布于每ml补充有100μg氨比西林的LB板上,并且在37℃下孵育过夜。将转化体各自挑入每ml补充有100μg的氨比西林的3ml的LB培养基中,并且在37℃下在250rpm的振荡下生长过夜。使用旋转迷你制备型试剂盒从菌落中纯化质粒DNA。将使用3130XL遗传分析仪的DNA测序用来确认指定为pDLHD0006的最终质粒中三种片段中的每种的存在(图3)。实例7:用于野生型红豆木葡聚糖内糖基转移酶16(VaXET16)在米曲霉中表达的pDLH0044的构建野生型VaXET16cDNA经密码子优化并合成用于在米曲霉中表达。构建质粒pDLHD0044以在米曲霉中多拷贝表达密码子优化的VaXET16基因。使用无缝克隆和装配试剂盒(英杰公司,卡尔斯巴德,加利福尼亚州,美国)合并这两种DNA片段来生成质粒pDLHD0044:包含载体pMA中提供的来自的VaXET16合成基因和与片段2同源的侧翼序列的片段1,以及由载体pDLHD0006的反向PCR产物组成的片段2,该载体pDLHD0006包含大肠杆菌pUC复制起点、大肠杆菌β-内酰胺酶(ampR)选择性标记、URA3酵母选择性标记、酵母2微米复制起点、NA2-tpi启动子、黑曲霉淀粉葡糖苷酶终止子序列(AMG终止子)和构巢曲霉乳清苷-5'-磷酸脱羧酶基因(pyrG)作为选择性标记。使用以下所示的引物614603(正义)和引物614605(反义)扩增片段1。这些引物被设计成包含与片段2同源的序列的侧翼区(小写),用于无连接克隆。引物614603(正义):ttcctcaatcctctatatacacaactggccATGGGCTCGTCCCTCTGGAC(SEQIDNO:60)引物614605(反义):agctcgctagagtcgacctaGATGTCCCTATCGCGTGTACACTCG(SEQIDNO:61)片段1在反应中通过PCR来扩增,该反应由10ng的pMA-VaXET16、0.5μl的DNA聚合酶、20pmol的引物614603、20pmol的引物614605、1μl的10mMdNTP、10μl的5XHF缓冲液、和35.5μl的水构成。该反应在热循环仪中孵育,程序为1个循环,在98℃下持续30秒;以及30个循环,每个循环在98℃下持续10秒,在60℃下持续10秒,以及在72℃下持续60秒。将所得0.9kbPCR产物(片段1)用1μl的DpnI进行处理,以去除质粒模板DNA。直接添加DpnI至PCR管中,充分混合,并且在37℃下孵育60分钟。使用以下所示的引物614604(正义)和引物613247(反义)扩增片段2。引物614604(正义):taggtcgactctagcgagctcgagatc(SEQIDNO:62)引物613247(反义):catggccagttgtgtatatagaggattgaggaaggaagag(SEQIDNO:63)片段2在反应中通过PCR来扩增,该反应由10ng的pDLHD0006、0.5μl的DNA聚合酶、20pmol的引物614604、20pmol的引物613247、1μl的10mMdNTP、10μl的5XHF缓冲液、和35.5μl的水构成。该反应在热循环仪中孵育,程序为1个循环,在98℃下持续30秒;以及30个循环,每个循环在98℃下持续10秒,在60℃下持续10秒,以及在72℃下持续4分钟。将所得7.3kbPCR产物(片段2)用1μl的DpnI进行处理,以去除质粒模板DNA。直接添加DpnI至PCR管中,充分混合,并且在37℃下孵育60分钟。使用无缝克隆和装配试剂盒,将以下程序用于合并两个PCR片段。将PCR片段中的每一种的5μl等分试样添加至微量离心管中,然后添加4μl的水、4μl的5X反应缓冲液(英杰公司,卡尔斯巴德,加利福尼亚州,美国)、和2μl的10X酶混合物(英杰公司,卡尔斯巴德,加利福尼亚州,美国)。将该反应在室温下孵育30分钟。根据制造商的说明书,然后将3μl等分试样的该反应混合物转化到大肠杆菌ONETOP10电转化感受态细胞中。将50μl的转化细胞散布于每ml补充有100μg氨比西林的2XYT板上,并且在37℃下孵育过夜。将转化体各自挑入每ml补充有100μg的氨比西林的3ml的LB培养基中,并且在37℃下在250rpm的振荡下生长过夜。使用旋转迷你制备型试剂盒从菌落中纯化质粒DNA。将使用3130XL遗传分析仪的DNA测序用来确认指定为质粒pDLHD0044的最终质粒中三个片段中的每个的存在(图4)。实例8:野生型红豆木葡聚糖内糖基转移酶16(VaXET16)的克隆,用于在米曲霉中表达将密码子优化的野生型VaXET16cDNA(实例7)通过酵母重组克隆克隆,克隆到酿酒酵母/米曲霉Flp/FRT穿梭载体中,生成载体pDLHD0075。表达载体pDLHD0075被构建为包含大肠杆菌pUC复制起点、大肠杆菌β-内酰胺酶(ampR)可选择标记、URA3酵母可选择标记、酵母2微米复制起点、NA2-tpi启动子、密码子优化飞VaXET16开放阅读框(ORF;DNA序列为SEQIDNO:51并且推导的氨基酸序列为SEQIDNO:2)、黑曲霉葡糖淀粉酶终止子、构巢曲霉pyrG选择标记、米曲霉TEF1启动子和米曲霉NIAD终止子之间的酿酒酵母2μm翻转酶ORF、和酿酒酵母2μm翻转酶识别目标FRT-F和FRT-F3。质粒pDLHD0075通过使用酵母重组克隆合并四个DNA片段而生成:片段1包含翻转酶表达盒、FRT-F3、和来自pDau571(图5;SEQIDNO:64)的AMG终止子、以及与片段4和2同源的侧翼序列。片段2包含大肠杆菌pUC复制起点、大肠杆菌β-内酰胺酶(ampR)选择标记、URA3酵母选择标记、来自pDLHD0044的酵母2微米复制原点、和与片段1和3同源的侧翼序列。片段3包含所述NA2-tpi启动子、来自pDLHD0044的VaXET16密码子优化基因、和与片段2和4同源的侧翼序列。片段4包含黑曲霉淀粉葡萄糖苷酶终止子序列(AMG终止子)和作为来自pDau571的可选择标记的构巢曲霉乳清苷-5’-磷酸脱羧酶基因(pyrG),以及与片段3和1同源的侧翼序列。使用以下所示的引物615726(正义)和引物615728(反义)扩增片段1。这些引物被设计成包含分别与片段4和2同源的序列的侧翼区(小写),用于PCR片段之间的无连接克隆。引物615726(正义):accgggaggaaggctggaaaGCTTACGAGAAAAGAGTTGGACTTTGAGGG(SEQIDNO:65)引物615728(反义):tgagcgaggaagcggAAGAGCGCCCAATACGCAAACCGCC(SEQIDNO:66)片段1在反应中通过PCR来扩增,该反应由10ng的pDau571、0.5μl的DNA聚合酶、20pmol的引物615726、20pmol的引物615728、1μl的10mMdNTPs、10μl的5XHF缓冲液、和35.5μl的水构成。该反应在热循环仪中孵育,程序为1个循环,在98℃下持续30秒;以及30个循环,每个循环在98℃下持续10秒,在60℃下持续10秒,以及在72℃下持续120秒。将所得3.3kbPCR产物(片段1)用1μl的DpnI进行处理,以去除质粒模板DNA。直接添加DpnI至PCR管中,充分混合,并且在37℃下孵育60分钟。使用以下所示的引物615729(正义)和引物615731(反义)扩增片段2。这些引物被设计成包含分别与片段1和3同源的序列的侧翼区(小写),用于PCR片段之间的无连接克隆。引物615729(正义):tgcgtattgggcgctcttCCGCTTCCTCGCTCACTGACTC(SEQIDNO:67)引物615731(反义):tatactttctagagaataggaactcggaataggaacttcaaGGAACAACACTCAACCCTATCTCGGTC(SEQIDNO:68)片段2在反应中通过PCR来扩增,该反应由10ng的pDLHD0044、0.5μl的DNA聚合酶、20pmol的引物615729、20pmol的引物615731、1μl的10mMdNTP、10μl的5XHF缓冲液、和35.5μl的水构成。该反应在热循环仪中孵育,程序为1个循环,在98℃下持续30秒;以及30个循环,每个循环在98℃下持续10秒,在60℃下持续10秒,以及在72℃下持续120秒。将所得4.2kbPCR产物(片段2)用1μl的DpnI进行处理,以去除质粒模板DNA。直接添加DpnI至PCR管中,充分混合,并且在37℃下孵育60分钟。使用以下所示的引物615730(正义)和引物615611(反义)扩增片段3。这些引物被设计成包含分别与片段2和4同源的序列的侧翼区(小写),用于PCR片段之间的无连接克隆。引物615730(正义):tccgagttcctattctctagaaagtataggaacttcGCATTTATCAGGGTTATTGTCTCATGAGCGG(SEQIDNO:69)引物615611(反义):tctagatctcgagtcaGATGTCCCTATCGCGTGTACACTCG(SEQIDNO:70)片段3在反应中通过PCR来扩增,该反应由10ng的pDLHD0044、0.5μl的DNA聚合酶、20pmol的引物615730、20pmol的引物615611、1μl的10mMdNTP、10μl的5XHF缓冲液、和35.5μl的水构成。该反应在热循环仪中孵育,程序为1个循环,在98℃下持续30秒;以及30个循环,每个循环在98℃下持续10秒,在60℃下持续10秒,以及在72℃下持续120秒。将所得1.7kbPCR产物(片段3)用1μl的DpnI进行处理,以去除质粒模板DNA。直接添加DpnI至PCR管中,充分混合,并且在37℃下孵育60分钟。使用以下所示的引物615610(正义)和引物615727(反义)扩增片段4。这些引物被设计成包含分别与片段3和1同源的序列的侧翼区(小写),用于PCR片段之间的无连接克隆。引物615610(正义):acacgcgatagggacatcTGACTCGAGATCTAGAGGGTGACTGAC(SEQIDNO:71)引物615727(反义):aactcttttctcgtaagcTTTCCAGCCTTCCTCCCGGTAC(SEQIDNO:72)片段4在反应中通过PCR来扩增,该反应由10ng的pDau571、0.5μl的DNA聚合酶、20pmol的引物615610、20pmol的引物615727、1μl的10mMdNTPs、10μl的5XHF缓冲液、和35.5μl的水构成。该反应在热循环仪中孵育,程序为1个循环,在98℃下持续30秒;以及30个循环,每个循环在98℃下持续10秒,在60℃下持续10秒,以及在72℃下持续120秒。将所得1.9kbPCR产物(片段4)用1μl的DpnI进行处理,以去除质粒模板DNA。直接添加DpnI至PCR管中,充分混合,并且在37℃下孵育60分钟。使用基于酵母同源性的重组克隆,使用以下程序来合并四个PCR片段。将PCR片段的每种的10μl等分试样与来自鲑鱼睾丸的100μg的单链脱氧核糖核酸(西格玛奥德里奇,圣路易斯,密苏里州,美国)、100μl的菌株YNG318的感受态酵母细胞(酿酒酵母ATCC208973)、和600μl的PLATE缓冲液(西格玛奥德里奇,圣路易斯,密苏里州,美国)合并,并且混合。将反应在30℃下以200rpm振荡孵育30分钟。然后将该反应在42℃下在没有振荡的情况下继续15分钟。将这些细胞通过在5,000xg下离心1分钟进行沉淀,并且弃去上清液。将细胞球粒悬浮于200μl的高压蒸汽处理的水中,并且分在两个包含合成的所定义的培养基缺少尿苷的琼脂平板,并且在30℃下孵育3天。将这些酵母菌落使用1ml的高压蒸汽处理的水从平板中分离。将这些细胞通过在13,000xg下离心30秒进行沉淀,并且将100μl等分试样的玻璃珠添加到该管中。将细胞和珠混合物悬浮在250μl的P1缓冲液(凯杰公司,巴伦西亚,加利福尼亚州,美国)中,并且然后涡旋1分钟来裂解这些细胞。使用旋转迷你制备型试剂盒纯化质粒DNA。根据制造商的说明,然后将3μl等分试样的质粒DNA转化到大肠杆菌ONETOP10电转化感受态细胞中。将50μl的转化细胞散布于每ml补充有100μg氨比西林的2XYT板上,并且在37℃下孵育过夜。将转化体各自挑入每ml补充有100μg的氨比西林的3ml的LB培养基中,并且在37℃下在250rpm的振荡下生长过夜。使用旋转迷你制备型试剂盒从菌落中纯化质粒DNA。将使用3130XL遗传分析仪的DNA测序用来确认指定为质粒pDLHD0075的最终质粒中三个片段中的每个的存在(图6)。实例9:在米曲霉菌株JaL1394中野生型红豆木葡聚糖内糖基转移酶16(VaXET16)以单拷贝表达的确认使用利用酿酒酵母2μm质粒翻转酶识别靶标(FRT)和重组酶(Flp)系统以产生高效率单拷贝靶向转化系统的米曲霉菌株JaL1394(WO2012/160093)用于筛选基因变体文库。酿酒酵母的Flp-FRT系统是位点特异性重组系统,其可用于将感兴趣的DNA插入感兴趣的宿主有机体的基因组中的已知位置。米曲霉菌株JaL1394先前已被工程化为在AmyB基因座中拥有FRT-F和FRT-F3翻转酶识别靶序列,其中AmyBORF已经缺失。用包含密码子优化的VaXET16基因的质粒pDLHD0075转化米曲霉JaL1394。将来自米曲霉JaL1394的大约107个孢子接种于500ml摇瓶中的补充有10mM尿苷的100ml的YP+2%葡萄糖培养基中,并且在28℃和110rpm下孵育过夜。将10ml的过夜培养物在125ml无菌真空过滤器中过滤,并且将菌丝用50ml的0.7MKCl-20mMCaCl2洗涤两次。将剩余液体通过真空过滤去除,留下垫在滤器上。将菌丝体再悬浮于10ml的0.7MKCl-20mMCaCl2中,并且转移到无菌125ml摇瓶中,该摇瓶包含20mg的200G(诺维信瑞士股份公司(NovozymesSwitzerlandAG),Neumatt,瑞士)/ml和0.2mg的几丁质酶(西格玛奥德里奇,圣路易斯,密苏里州,美国)/ml(在10ml的0.7MKCl-20mMCaCl2中)。将该混合物在37℃和100rpm下孵育30-90分钟,直到从菌丝体中产生原生质体。将该原生质体混合物通过衬有(Calbiochem公司,圣迭哥,加利福尼亚州,美国)的无菌漏斗进行过滤,到无菌50ml塑料离心管中,以除去菌丝体碎片。将在上的碎片彻底地用0.7MKCl-20mMCaCl2进行洗涤并且在2500rpm下,在20℃-23℃下,离心10分钟。去除该上清液,并且将原生质体球粒再悬浮于20ml的1M山梨醇-10mMCaCl2-10mMTris-HCl(pH6.5)中。将该步骤重复两次,并且将最终的原生质体球粒再悬浮于1M山梨醇-10mMCaCl2-10mMTris-HCl(pH6.5)中,以获得2x107/ml的最终原生质体浓度。通过添加两μg的pDLHD0075至无菌12ml塑料离心管的底部来转化原生质体。将一百μl的原生质体添加至管中,随后添加在10mMCaCl2-10mMTris-HCl(pH6.5)中的300μl的60%PEG-4000。将该管用手轻轻混合,并且在37℃下孵育30分钟。将添加五ml的1M山梨糖醇-10mMCaCl2-10mMTris-HCl(pH6.5)添加到该转化中,并且将该混合物转移到150mm基本培养基琼脂平板上。将转化板在37℃下孵育直到转化体出现。挑出单个转化体到新基本培养基琼脂平板,并且在37℃下培养4天,直到转化体形成孢子。新鲜孢子转移到48孔深孔板,这些板包含2ml的YP+2%麦芽糊精培养基,覆盖有可透气密封件,并且在没有振荡下在28℃下生长4天。在4天生长后,将每个转化体的培养基针对根据实例1的木葡聚糖内糖基转移酶活性并且针对木葡聚糖内糖基转移酶表达通过SDS-PAGE来测定。活性测定展示,这些转化体产生活性木葡聚糖内糖基转移酶。使用8%-16%无染色SDS-PAGE凝胶(伯乐实验室有限公司,赫拉克勒斯,加利福尼亚州,美国)进行SDS-PAGE,并且用无染色成像仪(伯乐实验室有限公司,赫拉克勒斯,加利福尼亚州,美国)进行凝胶成像。SDS-PAGE揭示了大约32kDa的野生型VaXET16带。实例10:红豆木葡聚糖内糖基转移酶16(VaXET16)的改进的表达变体的构建和鉴定VaXET16基因突变体文库通过位点饱和诱变构建。将VaXET16基因的突变体文库(文库的每个片段包括突变体VaXET16基因+构巢曲霉乳清苷-5’-磷酸脱羧酶pyrG选择标记与FRT-F和FRT-F3翻转酶识别靶序列)与一μg的编码米曲霉TEF1启动子和米曲霉niaD基因终止子之间的酿酒酵母2μm翻转酶ORF的载体pDLHD0095(图7)一起转化到如实例9中所述的米曲霉JaL1394的原生质体中。37℃下在基本培养基琼脂平板上原生质体恢复4天后,挑出单个菌落至包含2ml的YP+2%麦芽糖糊精培养基的48孔深孔板中,覆盖有透气密封,并且在没有振荡下在28℃下生长4天。在液体培养基生长4天后,如在实例1中所述测定木葡聚糖内糖基转移酶活性,并且更高活性变体被打分为表达命中。孢子纯化单个突变体菌株,并且如上所述再次培养以产生新鲜培养液,用于使用密码子优化基因,相对于表达野生型VaXET16的米曲霉JaL1394菌株进行再次测试。如在实例9中所述,通过SDS-PAGE分析培养液,用于增加木葡聚糖内糖基转移酶蛋白产物的生产。十一个表征的变体在来自48孔深孔板培养的4天培养液中,优于亲本基因的表达产量的相对改进在下表II中示出。相同的培养液的SDS-PAGE分析展示所有变体的优于野生型VaXET的增加强度的VaXET带,这与在活性测定中观察到的相对改进很好地相关。野生型VaXET及其变体的SDS-PAGE带为32kDa,除包含N175S突变的变体由于另外的糖基化具有大约37kDa的带。表II.实例11:米曲霉中红豆木葡聚糖内糖基转移酶16变体(VaXET16)基因的改进的表达的发酵规模确认发酵工艺被用来相对于野生型VaXET16,表达VaXET16变体、A40G+I53A+N175S和A40G+F183I。摇瓶培养基由以下构成:50g的蔗糖、10g的KH2PO4、0.5g的CaCl2、2g的MgSO4·7H2O、2g的K2SO4、2g的尿素、10g的酵母提取物、2g的柠檬酸、0.5ml的痕量金属溶液、以及去离子水补足至1升。痕量金属溶液由以下构成:13.8g的FeSO4·7H2O、14.3g的ZnSO4·7H2O、8.5g的MnSO4·H2O、2.5g的CuSO4·5H2O、3g的柠檬酸、以及去离子水补足至1升。将一百ml的摇瓶培养基添加至500ml摇瓶。用7ml的0.01%80与从固体平板培养刮下的孢子与一起接种摇瓶,并且在轨道摇床上以200rpm在34℃下孵育24小时。将50ml的烧瓶培养液用于接种3升发酵容器。每升分批发酵培养基(Fermentationbatchmedium)由以下构成:10g的酵母提取物、24g的蔗糖、5g的(NH4)2SO4、2g的KH2PO4、0.5g的CaCl2·2H2O、2g的MgSO4.7H2O、1g的柠檬酸、2g的K2SO4、0.5ml的止泡剂、和0.5ml的痕量金属溶液。每升痕量金属溶液由以下构成:13.8g的FeSO4·7H2O、14.3g的ZnSO4·7H2O、8.5g的MnSO4·H2O、2.5g的CuSO4·5H2O,和3g的柠檬酸。发酵补料培养基(fermentationfeedmedium)由麦芽糖构成。将总计1.8升的发酵分批培养基添加到三升玻璃套发酵器中。发酵补料培养基以0到8.0g/l/hr的速度给予。将发酵容器维持在34℃的温度,pH控制在6.1+/-0.1的设定点。将空气以1vvm的速率添加到容器中,并用以1100rpm旋转的Rushton叶轮搅拌培养液。在发酵运行的第2、3、4、5、6和7天取样,并且在3000xg离心以去除生物质。将上清液过滤灭菌,并且在-20℃下储存。通过荧光偏振测定(实例3)并且通过SDS-PAGE分析(实例9),相对于野生型密码子优化基因,确定VaXET16变体表达水平。相对于野生型VaXET16,对于两个变体的7天培养液,优于亲本基因,产量相对改进在下表III中示出。按比野生型VaXET163.1X更大的量产生变体A40G+I53A+N175S,而按比野生型VaXET161.2X更大的量产生变体A40G+F183I。相同的培养液的SDS-PAGE分析示出两个变体的优于野生型VaXET的增加强度的VaXET带这与在活性测定中观察到的相对改进很好地相关。在第2、3、4、5、6、和7天所取的样品的SDS-PAGE分析示出VaXET的增加的生产,并且各变体日益增强,其中在第7天最强。表III.本发明通过以下编号的段落来进一步说明:[1]一种木葡聚糖内糖基转移酶变体,该变体包括与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280相对应的一个或多个位置处的取代,其中该变体具有木葡聚糖内糖基转移酶活性,并且其中该变体与SEQIDNO:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、或50的成熟多肽序列具有至少60%,例如至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少95%、至少96%、至少97%、至少98%、或至少99%、但小于100%序列一致性。[2]如段落1所述的变体,该变体是亲本木葡聚糖内糖基转移酶的变体,其中该亲本选自下组,该组由以下各项组成:(a)一种多肽,该多肽与SEQIDNO:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、或50的成熟多肽具有至少60%序列一致性;(b)由一种多核苷酸编码的多肽,该多核苷酸在至少低严格条件下与(i)SEQIDNO:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、或49的成熟多肽编码序列,或(ii)(i)的全长互补体杂交;(c)由一种多核苷酸编码的多肽,该多核苷酸与SEQIDNO:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、或49的成熟多肽编码序列具有至少60%一致性;以及(d)SEQIDNO:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、或50的成熟多肽的片段,该片段具有木葡聚糖内糖基转移酶活性。[3]如段落2所述的变体,其中该亲本木葡聚糖内糖基转移酶与SEQIDNO:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、或50的成熟多肽具有至少60%,例如,至少65%、至少70%、至少60%、至少80%、至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%或100%序列一致性。[4]如段落2或3所述的变体,其中该亲本木葡聚糖内糖基转移酶由以下多核苷酸编码,该多核苷酸在低严格条件下、中严格条件下、中-高严格条件下、高严格条件下、或非常高严格条件下与(i)SEQIDNO:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、或49的成熟多肽编码序列或者(ii)(i)的全长互补体杂交。[5]如段落2-4中任一项所述的变体,其中该亲本木葡聚糖内糖基转移酶由以下多核苷酸编码,该多核苷酸与SEQIDNO:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、或49的成熟多肽编码序列具有至少60%,例如,至少65%、至少70%、至少60%、至少80%、至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%、或100%序列一致性。[6]如段落2-5中任一项所述的变体,其中该亲本木葡聚糖内糖基转移酶包括SEQIDNO:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、或50的成熟多肽或由其组成。[7]如段落2-6中任一项所述的变体,其中该亲本木葡聚糖内糖基转移酶是SEQIDNO:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、或50的成熟多肽的一个片段,其中该片段具有木葡聚糖内糖基转移酶活性。[8]如段落2-7中任一项所述的变体,该变体与该亲本木葡聚糖内糖基转移酶的氨基酸序列具有至少60%,例如,至少65%、至少70%、至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少95%、至少96%、至少97%、至少98%、或至少99%,但小于100%序列一致性。[9]如段落1-8中任一项所述的变体,其中该变体由亲本的至少85%、至少90%、或至少95%的氨基酸组成。[10]如段落1-9中任一项所述的变体,其中取代的数目是1-17个,例如1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16或17个取代。[11]如段落1-10中任一项所述的变体,该变体包括与位置10相对应的位置处的一个取代。[12]如段落11所述的变体,其中该取代是用Ala进行的。[13]如段落1-12中任一项所述的变体,该变体包括与位置30相对应的位置处的一个取代。[14]如段落13所述的变体,其中该取代是用Glu进行的。[15]如段落1-14中任一项所述的变体,该变体包括与位置40相对应的位置处的一个取代。[16]如段落15所述的变体,其中该取代是用Gly进行的。[17]如段落1-16中任一项所述的变体,该变体包括与位置51相对应的位置处的一个取代。[18]如段落17所述的变体,其中该取代是用Thr进行的。[19]如段落1-18中任一项所述的变体,该变体包括与位置53相对应的位置处的一个取代。[20]如段落19所述的变体,其中该取代是用Ala或Val进行的。[21]如段落1-20中任一项所述的变体,该变体包括与位置60相对应的位置处的一个取代。[22]如段落21所述的变体,其中该取代是用Ser进行的。[23]如段落1-22中任一项所述的变体,该变体包括与位置99相对应的位置处的一个取代。[24]如段落23所述的变体,其中该取代是用Glu或Asn进行的。[25]如段落1-24中任一项所述的变体,该变体包括与位置102相对应的位置处的一个取代。[26]如段落25所述的变体,其中该取代是用Gly进行的。[27]如段落1-26中任一项所述的变体,该变体包括与位置117相对应的位置处的一个取代。[28]如段落27所述的变体,其中该取代是用Glu进行的。[29]如段落1-28中任一项所述的变体,该变体包括与位置130相对应的位置处的一个取代。[30]如段落29所述的变体,其中该取代是用Arg进行的。[31]如段落1-30中任一项所述的变体,该变体包括与位置136相对应的位置处的一个取代。[32]如段落31所述的变体,其中该取代是用Trp进行的。[33]如段落1-32中任一项所述的变体,该变体包括与位置157相对应的位置处的一个取代。[34]如段落33所述的变体,其中该取代是用His进行的。[35]如段落1-34中任一项所述的变体,该变体包括与位置162相对应的位置处的一个取代。[36]如段落35所述的变体,其中该取代是用Cys进行的。[37]如段落1-36中任一项所述的变体,该变体包括与位置175相对应的位置处的一个取代。[38]如段落37所述的变体,其中该取代是用Ser、Gly或Gln进行的。[39]如段落1-38中任一项所述的变体,该变体包括与位置183相对应的位置处的一个取代。[40]如段落39所述的变体,其中该取代是用Ile进行的。[41]如段落1-40中任一项所述的变体,该变体包括与位置254相对应的位置处的一个取代。[42]如段落41所述的变体,其中该取代是用Glu进行的。[43]如段落1-42中任一项所述的变体,该变体包括与位置280相对应的位置处的一个取代。[44]如段落43所述的变体,其中该取代是用Gly或Glu进行的。[45]如段落1-44中任一项所述的变体,该变体包括与位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254和280中的任一个相对应的两个位置处的取代。[46]如段落1-44中任一项所述的变体,该变体包括与位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254和280中的任一个相对应的三个位置处的取代。[47]如段落1-44中任一项所述的变体,该变体包括与位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254和280中的任一个相对应的四个位置处的取代。[48]如段落1-44中任一项所述的变体,该变体包括与位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254和280中的任一个相对应的五个位置处的取代。[49]如段落1-44中任一项所述的变体,该变体包括与位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254和280中的任一个相对应的六个位置处的取代。[50]如段落1-44中任一项所述的变体,该变体包括与位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254和280中的任一个相对应的七个位置处的取代。[51]如段落1-44中任一项所述的变体,该变体包括与位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254和280中的任一个相对应的八个位置处的取代。[52]如段落1-44中任一项所述的变体,该变体包括与位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254和280中的任一个相对应的九个位置处的取代。[53]如段落1-44中任一项所述的变体,该变体包括与位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254和280中的任一个相对应的十个位置处的取代。[54]如段落1-44中任一项所述的变体,该变体包括与位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254和280的中的任一个相对应十一个位置处的取代。[55]如段落1-44中任一项所述的变体,该变体包括与位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254和280中的任一个相对应的十二个位置处的取代。[56]如段落1-44中任一项所述的变体,该变体包括与位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254和280中的任一个相对应的十三个位置处的取代。[57]如段落1-44中任一项所述的变体,该变体包括与位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254和280中的任一个相对应的十四个位置处的取代。[58]如段落1-44中任一项所述的变体,该变体包括与位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254和280中的任一个相对应的十五个位置处的取代。[59]如段落1-44中任一项所述的变体,该变体包括与位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254和280相对应的十六个位置处的取代。[60]如段落1-44中任一项所述的变体,该变体包括与位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254和280相对应的各个位置处的取代。[61]如段落1-60中任一项所述的变体,该变体包括选自下组的一个或多个取代,该组由以下各项组成:I10A;P30E;A40G;S51T;I53A,V;Y60S;T99E,N;E102G;Q117E;K130R;R136W;Y157H;Y162C;N175S,G,Q;F183I;A254E;以及S280G,E。[62]如段落61所述的变体,该变体包括A40G+N175S或由其组成。[63]如段落61所述的变体,该变体包括A40G+F183I或由其组成。[64]如段落61所述的变体,该变体包括N175G+S280G或由其组成。[65]如段落61所述的变体,该变体包括A40G+I53A+N175S或由其组成。[66]如段落61所述的变体,该变体包括A40G+N175S+F183I或由其组成。[67]如段落61所述的变体,该变体包括取代I10A+I53A+E102G或由其组成。[68]如段落61所述的变体,该变体包括A40G+E102G+Q117E或由其组成。[69]如段落61所述的变体,该变体包括N175Q+A254E+S280E或由其组成。[70]如段落61所述的变体,该变体包括P30E+S51T+Y60S+T99N或由其组成。[71]如段落61所述的变体,该变体包括A40G+T99E+E102G+K130R或由其组成。[72]如段落61所述的变体,该变体包括I53V+R136W+Y157H+Y162C+N175S或由其组成。[73]如段落1-72中任一项所述的变体,该变体相对于该亲本具有增加的表达产量。[74]如段落1-73中任一项所述的变体,其中相对于该亲本,该变体的表达产量是至少1.05、至少1.10、至少1.20、至少1.30、至少1.40、至少1.50、至少1.60、至少1.70、至少1.80、至少1.90、至少2、至少2.25、至少2.50、至少2.75、至少3.00、至少3.25、至少3.50、至少3.75、至少4、至少4.25、至少4.50、至少4.75、至少5、至少6、至少7、至少8、至少9、或至少10倍的增加。[75]一种编码如段落1-74中任一项所述的变体的分离的多核苷酸。[76]一种核酸构建体,其包含如段落75所述的多核苷酸。[77]一种表达载体,其包括如段落75所述的多核苷酸。[78]一种重组宿主细胞,其包含如段落75所述的多核苷酸。[79]一种产生木葡聚糖内糖基转移酶变体的方法,该方法包括:在适合于该变体表达的条件下培养如段落78所述的重组宿主细胞;以及[80]如段落79所述的方法,该方法进一步包括回收该变体。[81]一种用于获得木葡聚糖内糖基转移酶变体的方法,该方法包括将与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280相对应的一个或多个位置处的取代引入亲本木葡聚糖内糖基转移酶,其中该变体具有木葡聚糖内糖基转移酶活性。[82]如段落81所述的方法,该方法进一步包括回收该变体。[83]一种增加木葡聚糖内糖基转移酶的表达产量的方法,该方法包括将与SEQIDNO:2的全长多肽的位置10、30、40、51、53、60、99、102、117、130、136、157、162、175、183、254、以及280相对应的一个或多个位置处的取代引入亲本木葡聚糖内糖基转移酶,其中该变体的表达产量相对于亲本是增加的。[84]如段落83所述的方法,其中相对于该亲本,该变体的表达产量是至少1.05、至少1.10、至少1.20、至少1.30、至少1.40、至少1.50、至少1.60、至少1.70、至少1.80、至少1.90、至少2、至少2.25、至少2.50、至少2.75、至少3.00、至少3.25、至少3.50、至少3.75、至少4、至少4.25、至少4.50、至少4.75、至少5、至少6、至少7、至少8、至少9、或至少10倍的增加。[85]如段落84或85所述的方法,该方法进一步包括回收该变体。[86]一种酶组合物,包括如段落1-74中任一项所述的变体。[87]一种全培养液配制品或细胞培养组合物,包括如段落1-74中任一项所述的变体。[88]如段落1-74中任一项所述的变体的用途。在此描述并且要求保护的本发明不局限于在此披露的特定方面的范围,因为这些方面旨在作为本发明若干方面的说明。预期任何等效方面都处于本发明的范围内。实际上,除在此所示和描述的那些之外,本发明的不同修改对于本领域普通技术人员而言从前述描述将变得清楚。这样的修改也旨在落入所附权利要求的范围内。在有冲突的情况下,以包括定义的本披露为准。序列表<110>诺维信公司(NovozymesA/S)赫尔德(Held),丹尼尔(Daniel)<120>木葡聚糖内糖基转移酶变体以及编码其的多核苷酸<130>12915-WO-PCT<150>US62/039,744<151>2014-08-20<160>72<170>PatentIn版本3.5<210>1<211>876<212>DNA<213>红豆(Vignaangularis)<400>1atgggttcttctttgtggacttgtctgattctgttatcactggcttctgcttctttcgct60gccaacccaagaactccaattgatgtaccatttggcagaaactatgtgcctacttgggcc120tttgatcatatcaaatatctcaatggaggttctgagattcagcttcatctcgataagtac180actggtactggattccagtccaaagggtcatacttgtttggtcacttcagcatgtacata240aaattggttcctggtgattcagctggcacagtcactgctttctatttatcgtccacaaac300gcagaacatgatgaaatagacttcgagttcttgggaaacagaactgggcaaccatacatt360ttacaaacaaatgtgttcaccggaggcaaaggtgacagagagcagagaatctacctctgg420tttgaccctacgactcaataccacagatattcagtgctatggaacatgtaccagattgta480ttctatgtggatgactacccaataagggtgttcaagaacagcaatgacttgggagtgaag540ttccccttcaatcaaccaatgaaaatatacaacagtttgtggaatgcagatgactgggct600acaaggggtggtttggagaaaacagattggtccaaagcccccttcatagcctcttacaag660ggcttccacattgatgggtgtgaggcctcagtgaatgccaagttctgtgacacacaaggc720aagaggtggtgggatcaaccagagtttcgtgaccttgatgctgctcagtggcaaaaactg780gcttgggtacgcaacaaatacaccatctacaactactgcactgatcgcaaacgctactct840caagtccctccagagtgcaccagagaccgtgacatt876<210>2<211>292<212>PRT<213>红豆(Vignaangularis)<400>2MetGlySerSerLeuTrpThrCysLeuIleLeuLeuSerLeuAlaSer151015AlaSerPheAlaAlaAsnProArgThrProIleAspValProPheGly202530ArgAsnTyrValProThrTrpAlaPheAspHisIleLysTyrLeuAsn354045GlyGlySerGluIleGlnLeuHisLeuAspLysTyrThrGlyThrGly505560PheGlnSerLysGlySerTyrLeuPheGlyHisPheSerMetTyrIle65707580LysLeuValProGlyAspSerAlaGlyThrValThrAlaPheTyrLeu859095SerSerThrAsnAlaGluHisAspGluIleAspPheGluPheLeuGly100105110AsnArgThrGlyGlnProTyrIleLeuGlnThrAsnValPheThrGly115120125GlyLysGlyAspArgGluGlnArgIleTyrLeuTrpPheAspProThr130135140ThrGlnTyrHisArgTyrSerValLeuTrpAsnMetTyrGlnIleVal145150155160PheTyrValAspAspTyrProIleArgValPheLysAsnSerAsnAsp165170175LeuGlyValLysPheProPheAsnGlnProMetLysIleTyrAsnSer180185190LeuTrpAsnAlaAspAspTrpAlaThrArgGlyGlyLeuGluLysThr195200205AspTrpSerLysAlaProPheIleAlaSerTyrLysGlyPheHisIle210215220AspGlyCysGluAlaSerValAsnAlaLysPheCysAspThrGlnGly225230235240LysArgTrpTrpAspGlnProGluPheArgAspLeuAspAlaAlaGln245250255TrpGlnLysLeuAlaTrpValArgAsnLysTyrThrIleTyrAsnTyr260265270CysThrAspArgLysArgTyrSerGlnValProProGluCysThrArg275280285AspArgAspIle290<210>3<211>861<212>DNA<213>拟南芥(Arabidopsisthaliana)<400>3atggcgtgtttcgcaaccaaacagcctctgttgttgtctctcctccttgccattggcttc60tttgtggtggctgcatctgccggaaacttctatgagagctttgatatcacttggggtaat120ggtcgtgccaacatattcgagaatggacagcttctcacttgtactcttgacaaggtctcc180ggctcaggttttcaatccaagaaggagtacttgtttggtaagatcgacatgaagctcaag240cttgtcgctggaaactctgctggcaccgtcaccgcctactacctatcgtcaaaaggcacg300gcatgggatgagattgacttcgagtttttgggaaatcgcacaggacatccttacactatc360cacaccaatgtgttcaccggaggtaaaggcgaccgtgagatgcagttccgtctctggttc420gatcccactgcggatttccacacctacaccgtccactggaaccctgttaacatcatcttc480cttgtggatgggatcccaattcgggtgttcaagaacaacgagaaaaatggggtggcttac540cctaagaaccagccgatgaggatatactcaagcctttgggaagccgatgactgggctaca600gaaggcggtcgcgtgaagatcgactggagcaacgcaccattcaaggcctcttacagaaac660ttcaacgaccaaagctcatgcagcaggacatcaagctcaaaatgggtgacttgcgagcca720aacagcaactcgtggatgtggacgactctcaatcctgcccagtacggaaaaatgatgtgg780gtgcaacgagacttcatgatctacaactattgtaccgattttaagagattccctcaaggc840ctccccaaggagtgtaaactt861<210>4<211>287<212>PRT<213>拟南芥(Arabidopsisthaliana)<400>4MetAlaCysPheAlaThrLysGlnProLeuLeuLeuSerLeuLeuLeu151015AlaIleGlyPhePheValValAlaAlaSerAlaGlyAsnPheTyrGlu202530SerPheAspIleThrTrpGlyAsnGlyArgAlaAsnIlePheGluAsn354045GlyGlnLeuLeuThrCysThrLeuAspLysValSerGlySerGlyPhe505560GlnSerLysLysGluTyrLeuPheGlyLysIleAspMetLysLeuLys65707580LeuValAlaGlyAsnSerAlaGlyThrValThrAlaTyrTyrLeuSer859095SerLysGlyThrAlaTrpAspGluIleAspPheGluPheLeuGlyAsn100105110ArgThrGlyHisProTyrThrIleHisThrAsnValPheThrGlyGly115120125LysGlyAspArgGluMetGlnPheArgLeuTrpPheAspProThrAla130135140AspPheHisThrTyrThrValHisTrpAsnProValAsnIleIlePhe145150155160LeuValAspGlyIleProIleArgValPheLysAsnAsnGluLysAsn165170175GlyValAlaTyrProLysAsnGlnProMetArgIleTyrSerSerLeu180185190TrpGluAlaAspAspTrpAlaThrGluGlyGlyArgValLysIleAsp195200205TrpSerAsnAlaProPheLysAlaSerTyrArgAsnPheAsnAspGln210215220SerSerCysSerArgThrSerSerSerLysTrpValThrCysGluPro225230235240AsnSerAsnSerTrpMetTrpThrThrLeuAsnProAlaGlnTyrGly245250255LysMetMetTrpValGlnArgAspPheMetIleTyrAsnTyrCysThr260265270AspPheLysArgPheProGlnGlyLeuProLysGluCysLysLeu275280285<210>5<211>882<212>DNA<213>欧洲山杨(Populustremula)x美洲山杨(Populustremuloides)<400>5atggctgctgcttatccgtggactttgtttcttggcatgctggttatggtatctggaaca60atgggagctgccctgaggaagccagtggatgtggcgttcggtaggaactatgttcctaca120tgggcttttgaccacattaagtacttcaatggaggcaatgagattcagctgcacttggat180aaatacacaggtactggtttccaatcaaaaggttcatacttatttggccatttcagtatg240caaatgaagttggttcctggtgactcagctggaacagtcactgctttctatctatcctca300caaaactcggagcatgacgagatagactttgagttcttaggaaacaggactggccagccc360tacattttgcagacaaatgttttcacaggaggcaagggggatagagaacagaggatttac420ctctggtttgacccaaccaaggaattccactactattctgtcctctggaacatgtacatg480atagtgttcctcgtggatgacgtgccaatcagagtgttcaagaactgcaaagatttggga540gttaagtttccattcaaccagccaatgaagatctactcaagcctatggaatgccgatgat600tgggctaccaggggtggactcgagaagacagactggtccaaggcaccgttcattgcctcc660tacaggagcttccacatagatgggtgcgaggcctccgtggaagccaagttctgcgccaca720cagggtgctagatggtgggaccagaaggagttccaagatctggatgccttccagtacagg780aggctcagctgggtccgccagaaatataccatctacaattactgcactgatagatcaaga840tacccttcaatgcccccagaatgcaagagagacagagacata882<210>6<211>294<212>PRT<213>欧洲山杨(Populustremula)x美洲山杨(Populustremuloides)<400>6MetAlaAlaAlaTyrProTrpThrLeuPheLeuGlyMetLeuValMet151015ValSerGlyThrMetGlyAlaAlaLeuArgLysProValAspValAla202530PheGlyArgAsnTyrValProThrTrpAlaPheAspHisIleLysTyr354045PheAsnGlyGlyAsnGluIleGlnLeuHisLeuAspLysTyrThrGly505560ThrGlyPheGlnSerLysGlySerTyrLeuPheGlyHisPheSerMet65707580GlnMetLysLeuValProGlyAspSerAlaGlyThrValThrAlaPhe859095TyrLeuSerSerGlnAsnSerGluHisAspGluIleAspPheGluPhe100105110LeuGlyAsnArgThrGlyGlnProTyrIleLeuGlnThrAsnValPhe115120125ThrGlyGlyLysGlyAspArgGluGlnArgIleTyrLeuTrpPheAsp130135140ProThrLysGluPheHisTyrTyrSerValLeuTrpAsnMetTyrMet145150155160IleValPheLeuValAspAspValProIleArgValPheLysAsnCys165170175LysAspLeuGlyValLysPheProPheAsnGlnProMetLysIleTyr180185190SerSerLeuTrpAsnAlaAspAspTrpAlaThrArgGlyGlyLeuGlu195200205LysThrAspTrpSerLysAlaProPheIleAlaSerTyrArgSerPhe210215220HisIleAspGlyCysGluAlaSerValGluAlaLysPheCysAlaThr225230235240GlnGlyAlaArgTrpTrpAspGlnLysGluPheGlnAspLeuAspAla245250255PheGlnTyrArgArgLeuSerTrpValArgGlnLysTyrThrIleTyr260265270AsnTyrCysThrAspArgSerArgTyrProSerMetProProGluCys275280285LysArgAspArgAspIle290<210>7<211>891<212>DNA<213>玉蜀黍(Zeamays)<400>7atggcgacggcgcggtggctgcaggttgccgccatggcggtggcgctgctggcggagtgg60gcgacggcggcggcgcctcggaagcccgtggacgtgccgttccagaagaactacgtgccg120acgtgggcgcaggaccacatccactacatcgacggcgggcgggaggtgcagctgtacctg180gacaagtccaccggcacgggcttccagacgcggggctcctacctcttcggccacttcagc240atgcacatgaagctcgtcggcggcgactccgcgggcaccgtcacggccttctacctgtcg300tcgcagaactcggagcacgacgagatcgacttcgagttcctgggcaaccggacggggcag360ccctacatcctgcagaccaacgtgttcaccggcggcaagggcgaccgcgagcagcggatc420tacctctggttcgaccccaccaaggagtaccactcctactccgtcctctggaacctctac480atgatcgcgttcttcgtggacgacgtgccgatccgggtgttcaagaacacgagcgccgac540ctgggcgtgcggtacccgttcagccagccgatgaagctctactccagcctgtggaacgcc600gacgactgggcgacccgcggcgggcgcgagaagacggactggtccaacgcgcccttcgtc660gcctcctaccgcggcttccacgtcgacggctgcgaggcctccgcggaggcgcgcttctgc720gccacccagggcgcgcgctggtgggaccagccggagttcagggacctcgacgccgcgcag780taccgccgcctggccgaggtcaggcgccgctacaccatctacaactactgcaccgatcgc840gaccgctacggcgccgccgtgccgcccgagtgcgcccgcgaccgcgacgtc891<210>8<211>297<212>PRT<213>玉蜀黍(Zeamays)<400>8MetAlaThrAlaArgTrpLeuGlnValAlaAlaMetAlaValAlaLeu151015LeuAlaGluTrpAlaThrAlaAlaAlaProArgLysProValAspVal202530ProPheGlnLysAsnTyrValProThrTrpAlaGlnAspHisIleHis354045TyrIleAspGlyGlyArgGluValGlnLeuTyrLeuAspLysSerThr505560GlyThrGlyPheGlnThrArgGlySerTyrLeuPheGlyHisPheSer65707580MetHisMetLysLeuValGlyGlyAspSerAlaGlyThrValThrAla859095PheTyrLeuSerSerGlnAsnSerGluHisAspGluIleAspPheGlu100105110PheLeuGlyAsnArgThrGlyGlnProTyrIleLeuGlnThrAsnVal115120125PheThrGlyGlyLysGlyAspArgGluGlnArgIleTyrLeuTrpPhe130135140AspProThrLysGluTyrHisSerTyrSerValLeuTrpAsnLeuTyr145150155160MetIleAlaPhePheValAspAspValProIleArgValPheLysAsn165170175ThrSerAlaAspLeuGlyValArgTyrProPheSerGlnProMetLys180185190LeuTyrSerSerLeuTrpAsnAlaAspAspTrpAlaThrArgGlyGly195200205ArgGluLysThrAspTrpSerAsnAlaProPheValAlaSerTyrArg210215220GlyPheHisValAspGlyCysGluAlaSerAlaGluAlaArgPheCys225230235240AlaThrGlnGlyAlaArgTrpTrpAspGlnProGluPheArgAspLeu245250255AspAlaAlaGlnTyrArgArgLeuAlaGluValArgArgArgTyrThr260265270IleTyrAsnTyrCysThrAspArgAspArgTyrGlyAlaAlaValPro275280285ProGluCysAlaArgAspArgAspVal290295<210>9<211>882<212>DNA<213>大麦(Hordeumvulgare)<400>9atgaaggctaccgcgggggccctcctcgccgtggtggccacggtgctactgcgaggcatc60gcggcagcgccgccccggaagccggtggacgtgccattcgagaagaactacgtcccgaca120tgggcggaggaccacatccactacgtgaacggcggacgggaggtgcagctgtccctcgac180aagaccaccggcactggcttccagacccggggctcctacctcttcggccacttcagcatg240cacatcaagctcgtcggcggcgactccgccggcacagtcaccgccttctacctgtcgtcg300cagaactcggagcacgacgagatcgacttcgagttcttggggaacaggacggggcagccg360tacatcctgcagacgaacgtgttctccggcgggaagggcgaccgggaacagaggatctac420ctctggttcgacccaaccaaggactaccactcctactccgtcctctggaacctctacatg480atcgcgttctttgtggacgacacgccgatccgggtgttcaagaacagcaaggacctcggc540gtgcggtaccccttcgaccagcctatgaagctctactcgagcctgtggaacgcggacgac600tgggcgactcggggagggcgggagaagacggactggtccaaggcgcccttcgtcgcctcc660taccggggcttccacgtcgacggctgcgaggcgtcggcggaggccaagttgtgcgccacc720cagggcgcccgctggtgggatcagcccgagttccaggacctggacgccgcgcagtaccgc780cgcctcgcctgggtcaggaaggagcacaccatctacaactactgcacagaccgcgaacga840tacgccgccatgtcgcccgagtgcaaacgcgaccgcgacgtc882<210>10<211>294<212>PRT<213>大麦(Hordeumvulgare)<400>10MetLysAlaThrAlaGlyAlaLeuLeuAlaValValAlaThrValLeu151015LeuArgGlyIleAlaAlaAlaProProArgLysProValAspValPro202530PheGluLysAsnTyrValProThrTrpAlaGluAspHisIleHisTyr354045ValAsnGlyGlyArgGluValGlnLeuSerLeuAspLysThrThrGly505560ThrGlyPheGlnThrArgGlySerTyrLeuPheGlyHisPheSerMet65707580HisIleLysLeuValGlyGlyAspSerAlaGlyThrValThrAlaPhe859095TyrLeuSerSerGlnAsnSerGluHisAspGluIleAspPheGluPhe100105110LeuGlyAsnArgThrGlyGlnProTyrIleLeuGlnThrAsnValPhe115120125SerGlyGlyLysGlyAspArgGluGlnArgIleTyrLeuTrpPheAsp130135140ProThrLysAspTyrHisSerTyrSerValLeuTrpAsnLeuTyrMet145150155160IleAlaPhePheValAspAspThrProIleArgValPheLysAsnSer165170175LysAspLeuGlyValArgTyrProPheAspGlnProMetLysLeuTyr180185190SerSerLeuTrpAsnAlaAspAspTrpAlaThrArgGlyGlyArgGlu195200205LysThrAspTrpSerLysAlaProPheValAlaSerTyrArgGlyPhe210215220HisValAspGlyCysGluAlaSerAlaGluAlaLysLeuCysAlaThr225230235240GlnGlyAlaArgTrpTrpAspGlnProGluPheGlnAspLeuAspAla245250255AlaGlnTyrArgArgLeuAlaTrpValArgLysGluHisThrIleTyr260265270AsnTyrCysThrAspArgGluArgTyrAlaAlaMetSerProGluCys275280285LysArgAspArgAspVal290<210>11<211>855<212>DNA<213>大麦(Hordeumvulgare)<400>11atggcgtccggtcccagtagaacagtcccgtgctctgtgctgccactgctgctgctgctc60gccggcgtggcccgcgcggccggcaacttctaccaggacgtggacatcacgtggggcgac120gggcgcggcaagatcctcggcggcggcgacctcctcacgctgtcgctcgacagggcctcc180ggctccgggttccagtccaagaaccagtacctgtacggccgcttcgacatgcagatcaag240ctcgtccccggcgactccgccggcaccgtcgccactttctacctgtcgtcgcaggggtcg300gcgcacgacgagatcgacttcgagttcctgggcaacgcgagcgggcagccctacacggtg360cacaccaacgtgtacagccagggcaagggcggccgggagcagcagttccgcatgtggttc420gaccccaccgccgacttccacacctactccgtcctctggaaccccacacacatcctgttc480tacgtggacgggacgccgatacgggagcaccgcaaccgggaggcggcgacgggggttccc540tacctgcggagccaggcgatgagggtgtacgcgagcgtgtgggacgcggaagagtgggcg600acgcagggcgggcgggtgaggacggactggtcgcgggcgccgttcgtggcgtcgtacaag660gggctcgccgcgagcgggtgcgcgtcgcaggacgcggcggcgtgcgccaactccaacggc720gcgtggatgtaccaggagctggacgccacggcgttggaccgcctccagtgggtgcagaag780aactacatgatctacaactactgcacggacacgtggaggttcaaggacggcgccccgccc840gagtgcgccagcaag855<210>12<211>285<212>PRT<213>大麦(Hordeumvulgare)<400>12MetAlaSerGlyProSerArgThrValProCysSerValLeuProLeu151015LeuLeuLeuLeuAlaGlyValAlaArgAlaAlaGlyAsnPheTyrGln202530AspValAspIleThrTrpGlyAspGlyArgGlyLysIleLeuGlyGly354045GlyAspLeuLeuThrLeuSerLeuAspArgAlaSerGlySerGlyPhe505560GlnSerLysAsnGlnTyrLeuTyrGlyArgPheAspMetGlnIleLys65707580LeuValProGlyAspSerAlaGlyThrValAlaThrPheTyrLeuSer859095SerGlnGlySerAlaHisAspGluIleAspPheGluPheLeuGlyAsn100105110AlaSerGlyGlnProTyrThrValHisThrAsnValTyrSerGlnGly115120125LysGlyGlyArgGluGlnGlnPheArgMetTrpPheAspProThrAla130135140AspPheHisThrTyrSerValLeuTrpAsnProThrHisIleLeuPhe145150155160TyrValAspGlyThrProIleArgGluHisArgAsnArgGluAlaAla165170175ThrGlyValProTyrLeuArgSerGlnAlaMetArgValTyrAlaSer180185190ValTrpAspAlaGluGluTrpAlaThrGlnGlyGlyArgValArgThr195200205AspTrpSerArgAlaProPheValAlaSerTyrLysGlyLeuAlaAla210215220SerGlyCysAlaSerGlnAspAlaAlaAlaCysAlaAsnSerAsnGly225230235240AlaTrpMetTyrGlnGluLeuAspAlaThrAlaLeuAspArgLeuGln245250255TrpValGlnLysAsnTyrMetIleTyrAsnTyrCysThrAspThrTrp260265270ArgPheLysAspGlyAlaProProGluCysAlaSerLys275280285<210>13<211>969<212>DNA<213>大麦(Hordeumvulgare)<400>13atggcagcagccagcgctcttctcatggctgctctcgcggtcttcgccgtggccgccgcc60gcggcgctggacacgtcgccggtgccgttcgacgccgggtacgcgcctctcttcggcggc120gacaacctcgtccggtcggcggacggccggagcgtcacgctcaagctggaccgatacacc180gggtctgggttcgtgtcaaagtcggcctaccgccatggcttcttcggcgcttccattaag240ctgcccggcgactataccgccggcgtcgtcgtcgccttctacctatcgaactgggacgag300taccccaagaaccacgacgagctggacttcgagctgctgggcaaccggcgcggccacggc360tggcgtgtccagaccaacatgtacggcaacggcagcaccgcccgcggccgagaggagcgt420taccatctccccgtcgaacctaccgtcaccggcgtccaccgctatgccattgcctggacc480cccaacaacatcgtcttctacctcgacggcgttcccatccgtgaggttgtccgcgttccc540tccatgggcggcgacttcccctccaagcccatgtccgtgtacgccaccatctgggacggc600tccgcctgggcgaccgacggcggcaagtacaaggtcgactacgcctacgcgcccttcgct660gccgagttttccgacctcgtcctgagcggctgcggcgcgggcaacgtcgccgaccccgag720gggtgccaggtcgacctgctcacgcacgacgtcgcagtcatggcaccggccaagcgcgcc780gccatgagagggttccgcgagcagcacctaacctacacggcgtgccgcgacagggtgcga840tacaagaccaccgtcttccccgagtgtgacgacctcgccgacggcgactccagcttccac900ctctggggggagtccaagaagaggcgccgccggtcgtcctcgccgctgcagtactcttcc960agcatgcag969<210>14<211>323<212>PRT<213>大麦(Hordeumvulgare)<400>14MetAlaAlaAlaSerAlaLeuLeuMetAlaAlaLeuAlaValPheAla151015ValAlaAlaAlaAlaAlaLeuAspThrSerProValProPheAspAla202530GlyTyrAlaProLeuPheGlyGlyAspAsnLeuValArgSerAlaAsp354045GlyArgSerValThrLeuLysLeuAspArgTyrThrGlySerGlyPhe505560ValSerLysSerAlaTyrArgHisGlyPhePheGlyAlaSerIleLys65707580LeuProGlyAspTyrThrAlaGlyValValValAlaPheTyrLeuSer859095AsnTrpAspGluTyrProLysAsnHisAspGluLeuAspPheGluLeu100105110LeuGlyAsnArgArgGlyHisGlyTrpArgValGlnThrAsnMetTyr115120125GlyAsnGlySerThrAlaArgGlyArgGluGluArgTyrHisLeuPro130135140ValGluProThrValThrGlyValHisArgTyrAlaIleAlaTrpThr145150155160ProAsnAsnIleValPheTyrLeuAspGlyValProIleArgGluVal165170175ValArgValProSerMetGlyGlyAspPheProSerLysProMetSer180185190ValTyrAlaThrIleTrpAspGlySerAlaTrpAlaThrAspGlyGly195200205LysTyrLysValAspTyrAlaTyrAlaProPheAlaAlaGluPheSer210215220AspLeuValLeuSerGlyCysGlyAlaGlyAsnValAlaAspProGlu225230235240GlyCysGlnValAspLeuLeuThrHisAspValAlaValMetAlaPro245250255AlaLysArgAlaAlaMetArgGlyPheArgGluGlnHisLeuThrTyr260265270ThrAlaCysArgAspArgValArgTyrLysThrThrValPheProGlu275280285CysAspAspLeuAlaAspGlyAspSerSerPheHisLeuTrpGlyGlu290295300SerLysLysArgArgArgArgSerSerSerProLeuGlnTyrSerSer305310315320SerMetGln<210>15<211>897<212>DNA<213>水稻(Oryzasativa)<400>15atgcgtggtggcgcctctctccggttgaggtggccggcggctctcgtcgccgtcgtagct60gcggcagtgacggcggcggcggcggcggggcatggcgaccacaatttccaccgggacttt120gacgccgtgtgggggaagggcaatgcgaggttccgggacggcgggcggatggtggagctg180acgctggacgagcagacgggggcgcggctgcagtccaaggagcggttcttgtttgggagg240ttcgatctcgagatcaagctcgtcaggggagagtctgcagggaccatcacttccttctat300atctgcagcggcggcgcgcggcacgacgaggtggacttcgagttcctgggcaacgtgagc360ggcgagccgtacctgctgcacaccaacatcttcagcgacggcaagggcgagcgtgagcag420cagttcgtgctctggttcgaccccaccgccgacttccacacctactccatcctctggaac480ccacacaacatcatactgtacattgacgggacgccgatcagggtgttcaagaacaacgag540gcgtacggggtgccattcccgacgcggcagccagtgcacgtgttcgccagcatctggaac600gcggaggagtgggcgacgcagggcggccgcgtcaagacggactggtcgcgcgcgccgttc660gtcgccacgtaccgccgctacaacgtctccaacgcctgcgtctgggacgccgccggcgcc720ggcgcctcccggtgcgccggcggcggcggcgggtggatgaggcggaggatggactggtgg780agctggatgacgctcaactgggtgcggatgaactacatggcctacgactactgcgccgac840aggaagcgattcccgcaccgtttccccgccgagtgcatcatccccatcggtagaacg897<210>16<211>299<212>PRT<213>水稻(Oryzasativa)<400>16MetArgGlyGlyAlaSerLeuArgLeuArgTrpProAlaAlaLeuVal151015AlaValValAlaAlaAlaValThrAlaAlaAlaAlaAlaGlyHisGly202530AspHisAsnPheHisArgAspPheAspAlaValTrpGlyLysGlyAsn354045AlaArgPheArgAspGlyGlyArgMetValGluLeuThrLeuAspGlu505560GlnThrGlyAlaArgLeuGlnSerLysGluArgPheLeuPheGlyArg65707580PheAspLeuGluIleLysLeuValArgGlyGluSerAlaGlyThrIle859095ThrSerPheTyrIleCysSerGlyGlyAlaArgHisAspGluValAsp100105110PheGluPheLeuGlyAsnValSerGlyGluProTyrLeuLeuHisThr115120125AsnIlePheSerAspGlyLysGlyGluArgGluGlnGlnPheValLeu130135140TrpPheAspProThrAlaAspPheHisThrTyrSerIleLeuTrpAsn145150155160ProHisAsnIleIleLeuTyrIleAspGlyThrProIleArgValPhe165170175LysAsnAsnGluAlaTyrGlyValProPheProThrArgGlnProVal180185190HisValPheAlaSerIleTrpAsnAlaGluGluTrpAlaThrGlnGly195200205GlyArgValLysThrAspTrpSerArgAlaProPheValAlaThrTyr210215220ArgArgTyrAsnValSerAsnAlaCysValTrpAspAlaAlaGlyAla225230235240GlyAlaSerArgCysAlaGlyGlyGlyGlyGlyTrpMetArgArgArg245250255MetAspTrpTrpSerTrpMetThrLeuAsnTrpValArgMetAsnTyr260265270MetAlaTyrAspTyrCysAlaAspArgLysArgPheProHisArgPhe275280285ProAlaGluCysIleIleProIleGlyArgThr290295<210>17<211>825<212>DNA<213>番茄(Lycopersiconesculentum)<400>17atgatcaaaacatcaagttgtatatttactttctttcttctaatatgtttcttcgtagtg60gtggcttttggtggtactttcgaccaagaatttgatgttacatggggttatgggagggtg120aaaatactcgaaaacgggcaacttcttactctttccctcgatagaagttctggctctgga180tttaagtctaaacaacaatatatgtttgcaaagattgacatgaagatcaaacttgtccct240ggtaattctgctggcaccgctactacatactatttatcttcggtggggtcagctcatgat300gagattgactttgagtttcttgggaatgtaagtggagaaccatatactcttcatacaaat360gtgtatgcacaaggcaagggagatagagagcaacaatttcatctttggtttgaccctact420aaggatttccacacctactctattctttggaatcctcgaaatatcatatttttggtagat480gggacaccaataagacaatacaagaatcttgaagcaacaaatggaataccttacccaaag540aaccaaccaatgtggttatactcaagtttatggaatgctgaggaatgggcaacaagaggt600ggccttgtaaggactgattggagtaaagccccttttattgcttcttatagaaacttcaat660gcccaaacttccaagaatcccacagccaattcttggttaactcaatcattggacaatgtt720gggttaacaaggatgaaatgggtgcaaaagaactatatgatatacaattattgcactgat780actaaacgtttccctcaagggttccctcatgaatgcactctaaat825<210>18<211>275<212>PRT<213>番茄(Lycopersiconesculentum)<400>18MetIleLysThrSerSerCysIlePheThrPhePheLeuLeuIleCys151015PhePheValValValAlaPheGlyGlyThrPheAspGlnGluPheAsp202530ValThrTrpGlyTyrGlyArgValLysIleLeuGluAsnGlyGlnLeu354045LeuThrLeuSerLeuAspArgSerSerGlySerGlyPheLysSerLys505560GlnGlnTyrMetPheAlaLysIleAspMetLysIleLysLeuValPro65707580GlyAsnSerAlaGlyThrAlaThrThrTyrTyrLeuSerSerValGly859095SerAlaHisAspGluIleAspPheGluPheLeuGlyAsnValSerGly100105110GluProTyrThrLeuHisThrAsnValTyrAlaGlnGlyLysGlyAsp115120125ArgGluGlnGlnPheHisLeuTrpPheAspProThrLysAspPheHis130135140ThrTyrSerIleLeuTrpAsnProArgAsnIleIlePheLeuValAsp145150155160GlyThrProIleArgGlnTyrLysAsnLeuGluAlaThrAsnGlyIle165170175ProTyrProLysAsnGlnProMetTrpLeuTyrSerSerLeuTrpAsn180185190AlaGluGluTrpAlaThrArgGlyGlyLeuValArgThrAspTrpSer195200205LysAlaProPheIleAlaSerTyrArgAsnPheAsnAlaGlnThrSer210215220LysAsnProThrAlaAsnSerTrpLeuThrGlnSerLeuAspAsnVal225230235240GlyLeuThrArgMetLysTrpValGlnLysAsnTyrMetIleTyrAsn245250255TyrCysThrAspThrLysArgPheProGlnGlyPheProHisGluCys260265270ThrLeuAsn275<210>19<211>990<212>DNA<213>胡萝卜(Daucuscarota)<400>19atggtgaacttgtatgtgaatttgtttgtaatgtgttctgttgttgttttggctcatggg60tcatcaagaaatcttccaattgtttcatttgaagaagggtactcacagctctttggtgat120agtaatctcatgattcttcaagatggaaaatcagctcacctttctcttgatgaaagaaca180ggctctggatttgtatctcatgacctctataaacatggattctttagtgcttcaattaag240ttaccagcagattacacagctggggtggttgttgcattctatatgtcgaatgtagacatg300tttgcaaagaaccatgatgagattgattttgagtttttggggaatattagaggcaaagag360tggagacttcagaccaatgtttatggaaatggaagcacaggtgcaggcagagaagaaaga420tacggtctctggtttgacccttctgatgattttcatcagtacagtatattgtggagcaaa480gatagaattatcttttacatagacaatgttcctataagagaggtcaagaagacagaagca540atgggtggggacttcccttccaagccaatgagcttgtatgctactatttgggatggatct600aattgggctacaaatggaggtaaatacaaggtcaattataaatattccccttacattgcc660gagttctccgatttcgtgcttcacggttgtgccgttgatcccatcgagatgtcgacctca720tgtgatactgcgcccaagtctcaatctgttcctacaggtacaacaaaagaaagtagaact780aaaatgcagaacctgaggaaaaaatacatgcagtactcctattgttatgatacaactcga840taccaggttccaccttctgagtgtgtaatcgatccacttgagtccgagagacttagagga900ttcgacccggtaacatttggtacaagccaccgcgggcatggtaaaagacatcacaaccgc960cgatcataccgtcatggaataaactctgtt990<210>20<211>330<212>PRT<213>胡萝卜(Daucuscarota)<400>20MetValAsnLeuTyrValAsnLeuPheValMetCysSerValValVal151015LeuAlaHisGlySerSerArgAsnLeuProIleValSerPheGluGlu202530GlyTyrSerGlnLeuPheGlyAspSerAsnLeuMetIleLeuGlnAsp354045GlyLysSerAlaHisLeuSerLeuAspGluArgThrGlySerGlyPhe505560ValSerHisAspLeuTyrLysHisGlyPhePheSerAlaSerIleLys65707580LeuProAlaAspTyrThrAlaGlyValValValAlaPheTyrMetSer859095AsnValAspMetPheAlaLysAsnHisAspGluIleAspPheGluPhe100105110LeuGlyAsnIleArgGlyLysGluTrpArgLeuGlnThrAsnValTyr115120125GlyAsnGlySerThrGlyAlaGlyArgGluGluArgTyrGlyLeuTrp130135140PheAspProSerAspAspPheHisGlnTyrSerIleLeuTrpSerLys145150155160AspArgIleIlePheTyrIleAspAsnValProIleArgGluValLys165170175LysThrGluAlaMetGlyGlyAspPheProSerLysProMetSerLeu180185190TyrAlaThrIleTrpAspGlySerAsnTrpAlaThrAsnGlyGlyLys195200205TyrLysValAsnTyrLysTyrSerProTyrIleAlaGluPheSerAsp210215220PheValLeuHisGlyCysAlaValAspProIleGluMetSerThrSer225230235240CysAspThrAlaProLysSerGlnSerValProThrGlyThrThrLys245250255GluSerArgThrLysMetGlnAsnLeuArgLysLysTyrMetGlnTyr260265270SerTyrCysTyrAspThrThrArgTyrGlnValProProSerGluCys275280285ValIleAspProLeuGluSerGluArgLeuArgGlyPheAspProVal290295300ThrPheGlyThrSerHisArgGlyHisGlyLysArgHisHisAsnArg305310315320ArgSerTyrArgHisGlyIleAsnSerVal325330<210>21<211>891<212>DNA<213>番木瓜(Caricapapaya)<400>21atggcaaagcttagttgtgtgttaatggctccactactattctcttttctgctcattctc60atggtttcttctcagggtcctccctcaccagggtactatcctacatccagattcccgtct120ttaggttttgatcaaggattcagaaatcgttggggtcctcagcaccagagagtagaacag180ggtgtcctaacaatctggcttgattctacctcaggaagtgggttcaaatcacttcataca240tatcaatctggttattttggagctgccattaaacttcaacccggttatactgctggagta300attacatctttctatctttcaaataatgaagattatccaggaaatcatgatgaaattgac360attgaatttctgggtacaactccaggggaaccctacactttgcagacaaatgttttcata420agaggaagtggagatagaaatatcgtcggcagggaagtcaaatttcatctctggtttgat480ccaacacaagacttccataactatgctattctctggaccccaagtgagatcgtatttttt540gtggatgatgtgcctattcggaggtatccaaggaagagtgatgccacatttcccttaaga600accatgtgggtttatgggtcaatttgggatgcttcatcttgggctactgacgatggaaaa660tacaaagctgactaccaataccaaccctttgttggtaggtataagaatttcaaaatagcc720gcgtgcagagccgacgggcaagcttcctgtaggcccccttctgtgtctccatctggcttt780ggcgtgttgagcccacaacaagaatcggccatggaatgggcacagagaaattctttggtt840tataactattgtttagaccctaagagagatcataccccaacgcccgagtgt891<210>22<211>297<212>PRT<213>番木瓜(Caricapapaya)<400>22MetAlaLysLeuSerCysValLeuMetAlaProLeuLeuPheSerPhe151015LeuLeuIleLeuMetValSerSerGlnGlyProProSerProGlyTyr202530TyrProThrSerArgPheProSerLeuGlyPheAspGlnGlyPheArg354045AsnArgTrpGlyProGlnHisGlnArgValGluGlnGlyValLeuThr505560IleTrpLeuAspSerThrSerGlySerGlyPheLysSerLeuHisThr65707580TyrGlnSerGlyTyrPheGlyAlaAlaIleLysLeuGlnProGlyTyr859095ThrAlaGlyValIleThrSerPheTyrLeuSerAsnAsnGluAspTyr100105110ProGlyAsnHisAspGluIleAspIleGluPheLeuGlyThrThrPro115120125GlyGluProTyrThrLeuGlnThrAsnValPheIleArgGlySerGly130135140AspArgAsnIleValGlyArgGluValLysPheHisLeuTrpPheAsp145150155160ProThrGlnAspPheHisAsnTyrAlaIleLeuTrpThrProSerGlu165170175IleValPhePheValAspAspValProIleArgArgTyrProArgLys180185190SerAspAlaThrPheProLeuArgThrMetTrpValTyrGlySerIle195200205TrpAspAlaSerSerTrpAlaThrAspAspGlyLysTyrLysAlaAsp210215220TyrGlnTyrGlnProPheValGlyArgTyrLysAsnPheLysIleAla225230235240AlaCysArgAlaAspGlyGlnAlaSerCysArgProProSerValSer245250255ProSerGlyPheGlyValLeuSerProGlnGlnGluSerAlaMetGlu260265270TrpAlaGlnArgAsnSerLeuValTyrAsnTyrCysLeuAspProLys275280285ArgAspHisThrProThrProGluCys290295<210>23<211>864<212>DNA<213>草地羊茅(Festucapratensis)<400>23atggggaagcctgcagcgttgattccggtgctagctcttcttgcctgcttggcttttggg60cttgtgtccggcggcaacttcaacgacgattgcgacgcgacctgggagccccagaactgc120tgggtttatgatgctggcaatagcctctccctcgccctcgtcagcaactcctcaggctcg180atgatccggtccaagaggcagttcatctttgggaccgtctccacatggatccaagcttcg240tcaagggcaactcttgcccggcaccgtcacaacatattacctatcctcggcggagacaat300catgacgaaatcgacttcgagttcctggggaacgagaccggacagccctacaccctgcac360accaacgtctacgccgacggcgtcggcaacaaggagatgcagttcgtgccctggttcgac420cccaccggcggttaccacaagtacaccatctcctggacgccctgcatgatcgtctggtac480gtcgacgacctccccatccgggtcttccgcaattaccgccagagcaacggcgtggccttc540ccgacgagccgccccatgtacgcctactccagcatctggtccgccgacgactgggccacg600cagggcggccgcgtcaagaccgactggacccacgcgcccttcgtcgccaactaccacggc660atcgacctcgacatctgcgagtgctacggcggcgactgcatctacggctgcgccggcgcc720tacggccagccctactgcggcgggctcaccgacgagcagcgctggaagatgcagtgggta780caggagagatacaggatctacgactactgcgtggactacaaggacggcaaggtgcccggc840gtcgagtgcagcctgccccagtac864<210>24<211>288<212>PRT<213>草地羊茅(Festucapratensis)<400>24MetGlyLysProAlaAlaLeuIleProValLeuAlaLeuLeuAlaCys151015LeuAlaPheGlyLeuValSerGlyGlyAsnPheAsnAspAspCysAsp202530AlaThrTrpGluProGlnAsnCysTrpValTyrAspAlaGlyAsnSer354045LeuSerLeuAlaLeuValSerAsnSerSerGlySerMetIleArgSer505560LysArgGlnPheIlePheGlyThrValSerThrTrpIleGlnAlaSer65707580SerArgAlaThrLeuAlaArgHisArgHisAsnIleLeuProIleLeu859095GlyGlyAspAsnHisAspGluIleAspPheGluPheLeuGlyAsnGlu100105110ThrGlyGlnProTyrThrLeuHisThrAsnValTyrAlaAspGlyVal115120125GlyAsnLysGluMetGlnPheValProTrpPheAspProThrGlyGly130135140TyrHisLysTyrThrIleSerTrpThrProCysMetIleValTrpTyr145150155160ValAspAspLeuProIleArgValPheArgAsnTyrArgGlnSerAsn165170175GlyValAlaPheProThrSerArgProMetTyrAlaTyrSerSerIle180185190TrpSerAlaAspAspTrpAlaThrGlnGlyGlyArgValLysThrAsp195200205TrpThrHisAlaProPheValAlaAsnTyrHisGlyIleAspLeuAsp210215220IleCysGluCysTyrGlyGlyAspCysIleTyrGlyCysAlaGlyAla225230235240TyrGlyGlnProTyrCysGlyGlyLeuThrAspGluGlnArgTrpLys245250255MetGlnTrpValGlnGluArgTyrArgIleTyrAspTyrCysValAsp260265270TyrLysAspGlyLysValProGlyValGluCysSerLeuProGlnTyr275280285<210>25<211>933<212>DNA<213>水稻(Oryzasativa)<400>25atggcgaaggctctcctcgccgtcgtcgtcgtcgccgtggcggcggtgctcgagctgggg60ctcgtcggagccaacttccaggatcagtgtgacatcacgtgggagccacagaacgccaag120atgacggagggaggagaccacctcacgctctcccttgtcagcaactcctctggttgtatg180ctccggaccaagaagcagttcatctacggcagcgtgtcgactcgcatccagcttgtgaag240ggcaactcggccggcaccgtcaccacctactacacatcgtccatcggcgacaaacacgac300gagatcgacttcgagttcctgggcaactccagcggcctcccctacaccttccacaccaac360gtcttcgccgacggcgtcggcagccgcgagatgcagttccgcccctggttcgaccccacc420gacggctaccacaactacaccatcttctggaacccctgcatgatcgtgtggttcgtggac480agcatcccaatcagggtgttccggaaccacgagaaggaaggggtgccgttcccgacaaag540cggccgatgtacgccttctccagcatctgggcggcggaggattgggccacgcagggtggc600cgcgtcaagacggactggaccaaggcccctttcgtcgccgagtaccgtgacatcggcctc660aacatctgtgagtgccccggctcaggctctggctccagctccagcttcagctccagctcc720agctctacctccggcgatgccgaggacccagcgtgcgcgcagcggtgcgcgacgtcggac780cactggtacgcggcggaggggctgtgccagctgagcgacaagcagctgcggcagatgaag840gcggtgcagctgggctacaccatctacgactactgcgccgacgcccaggccaagggacgc900cccgtgccgccggagtgcagcatgccgcagtac933<210>26<211>311<212>PRT<213>水稻(Oryzasativa)<400>26MetAlaLysAlaLeuLeuAlaValValValValAlaValAlaAlaVal151015LeuGluLeuGlyLeuValGlyAlaAsnPheGlnAspGlnCysAspIle202530ThrTrpGluProGlnAsnAlaLysMetThrGluGlyGlyAspHisLeu354045ThrLeuSerLeuValSerAsnSerSerGlyCysMetLeuArgThrLys505560LysGlnPheIleTyrGlySerValSerThrArgIleGlnLeuValLys65707580GlyAsnSerAlaGlyThrValThrThrTyrTyrThrSerSerIleGly859095AspLysHisAspGluIleAspPheGluPheLeuGlyAsnSerSerGly100105110LeuProTyrThrPheHisThrAsnValPheAlaAspGlyValGlySer115120125ArgGluMetGlnPheArgProTrpPheAspProThrAspGlyTyrHis130135140AsnTyrThrIlePheTrpAsnProCysMetIleValTrpPheValAsp145150155160SerIleProIleArgValPheArgAsnHisGluLysGluGlyValPro165170175PheProThrLysArgProMetTyrAlaPheSerSerIleTrpAlaAla180185190GluAspTrpAlaThrGlnGlyGlyArgValLysThrAspTrpThrLys195200205AlaProPheValAlaGluTyrArgAspIleGlyLeuAsnIleCysGlu210215220CysProGlySerGlySerGlySerSerSerSerPheSerSerSerSer225230235240SerSerThrSerGlyAspAlaGluAspProAlaCysAlaGlnArgCys245250255AlaThrSerAspHisTrpTyrAlaAlaGluGlyLeuCysGlnLeuSer260265270AspLysGlnLeuArgGlnMetLysAlaValGlnLeuGlyTyrThrIle275280285TyrAspTyrCysAlaAspAlaGlnAlaLysGlyArgProValProPro290295300GluCysSerMetProGlnTyr305310<210>27<211>873<212>DNA<213>高粱(Sorghumbicolor)<400>27atggcgcggccggcggcgtggactccgcctgctctgtcgctcgcggtcgccgtcctctgc60ctcgtcggccggtccacctccgcggagttctggctcgacgagttcaccacggacggcgac120gtccgcaaagactacgacggttctggccggcaggtggcgtcgctggtcctcgaccagagt180tctggcgctggcttcaactccacccgcaagtacctcttcggcgagttcagcgtcgagatg240aagctcgtcgccggcaactccgccggcactgtcacctccttctacctgacgtccggcgag300ggcgacgagcacgacgagatcgacatggagttcatgggcaactccagcgggtcgccgacg360gtgctcaacaccaacgtgtgggccagcggcgacggcaagaaggagcaccagttctacctg420tggttcgacccggccgccgacttccacaagtacaagatcacctggaacgacaagaacatc480atcttccaggtggacgacgtgacggtgcgcgtcttcaagcgctacgccgacctgccgtac540ccgagctccaagcccatggcggtgcacgccacgctgtgggacggcagctactgggccacc600gagaagggcaaggtgcccatcgactggtcccgcgcgcccttcgtcgtctcctaccgagca660tacacggccaacgcctgcgccgtcggcggcggcggcgggggctcgtcgtcgtcgtgcccc720gacgccgccggtgggaacgagtggatggacaggcagcccgacgacaccgaccggctcacc780gtggagtgggcgcgccggaactgcttgcagtacaactactgcgaggacgggtggcgcttc840ccgcaggggttccccggcgagtgcgcccgcaac873<210>28<211>291<212>PRT<213>高粱(Sorghumbicolor)<400>28MetAlaArgProAlaAlaTrpThrProProAlaLeuSerLeuAlaVal151015AlaValLeuCysLeuValGlyArgSerThrSerAlaGluPheTrpLeu202530AspGluPheThrThrAspGlyAspValArgLysAspTyrAspGlySer354045GlyArgGlnValAlaSerLeuValLeuAspGlnSerSerGlyAlaGly505560PheAsnSerThrArgLysTyrLeuPheGlyGluPheSerValGluMet65707580LysLeuValAlaGlyAsnSerAlaGlyThrValThrSerPheTyrLeu859095ThrSerGlyGluGlyAspGluHisAspGluIleAspMetGluPheMet100105110GlyAsnSerSerGlySerProThrValLeuAsnThrAsnValTrpAla115120125SerGlyAspGlyLysLysGluHisGlnPheTyrLeuTrpPheAspPro130135140AlaAlaAspPheHisLysTyrLysIleThrTrpAsnAspLysAsnIle145150155160IlePheGlnValAspAspValThrValArgValPheLysArgTyrAla165170175AspLeuProTyrProSerSerLysProMetAlaValHisAlaThrLeu180185190TrpAspGlySerTyrTrpAlaThrGluLysGlyLysValProIleAsp195200205TrpSerArgAlaProPheValValSerTyrArgAlaTyrThrAlaAsn210215220AlaCysAlaValGlyGlyGlyGlyGlyGlySerSerSerSerCysPro225230235240AspAlaAlaGlyGlyAsnGluTrpMetAspArgGlnProAspAspThr245250255AspArgLeuThrValGluTrpAlaArgArgAsnCysLeuGlnTyrAsn260265270TyrCysGluAspGlyTrpArgPheProGlnGlyPheProGlyGluCys275280285AlaArgAsn290<210>29<211>840<212>DNA<213>高粱(Sorghumbicolor)<400>29atggcgcggcggtctctggcccttctgctcgcgtcgtcgctggccctcgtgatgatggcg60gccgtcgcgtcggcggactcgtggctctacgagaagttcagcacggacggcaccgtccgc120accaactacgacgagtccggcgcgcaggtggcgatgctcaacctcgaccgcagctccggc180gccggcttcaactccaaggagcagtacctctacggccagttcagcgtccagatgaagctc240atccccggcaactccgccggcaccgtctcctgcttctacctttcttctggcgacggcgac300gggcatgacgagatcgacatggagttcatgggcaacgccacggggcagccggtggtgctc360aacacaaacgtgtgggccaacggcgacggcaagaaggagcaccagttcgacctgtggttc420gacccggccgccgactaccacacctacaccatcatctggaacccgaccaacatcctcttc480aaggtggacgacaacgtgatccgcgtcttcaagcgctacgccgacctggcctacccgagc540tccaagcccatgacgctgcacgccacgctgtgggacggcagctactgggccaccgagaag600ggcaaggtgcccatcgactggtccggcgcgcccttcgtcgtctcctacaaaggctactcc660gccgacgcctgcgtcagcggcggcgcctgcgccgccggcagcggcgcgtggatgaacaag720cagcctgacagcgccgagtggggcaccgtcaagtgggccgagagcaactacatgcgctac780aactactgcgacgacggctggaggttcccgcaggggctgccggcggagtgcagccgcagc840<210>30<211>280<212>PRT<213>高粱(Sorghumbicolor)<400>30MetAlaArgArgSerLeuAlaLeuLeuLeuAlaSerSerLeuAlaLeu151015ValMetMetAlaAlaValAlaSerAlaAspSerTrpLeuTyrGluLys202530PheSerThrAspGlyThrValArgThrAsnTyrAspGluSerGlyAla354045GlnValAlaMetLeuAsnLeuAspArgSerSerGlyAlaGlyPheAsn505560SerLysGluGlnTyrLeuTyrGlyGlnPheSerValGlnMetLysLeu65707580IleProGlyAsnSerAlaGlyThrValSerCysPheTyrLeuSerSer859095GlyAspGlyAspGlyHisAspGluIleAspMetGluPheMetGlyAsn100105110AlaThrGlyGlnProValValLeuAsnThrAsnValTrpAlaAsnGly115120125AspGlyLysLysGluHisGlnPheAspLeuTrpPheAspProAlaAla130135140AspTyrHisThrTyrThrIleIleTrpAsnProThrAsnIleLeuPhe145150155160LysValAspAspAsnValIleArgValPheLysArgTyrAlaAspLeu165170175AlaTyrProSerSerLysProMetThrLeuHisAlaThrLeuTrpAsp180185190GlySerTyrTrpAlaThrGluLysGlyLysValProIleAspTrpSer195200205GlyAlaProPheValValSerTyrLysGlyTyrSerAlaAspAlaCys210215220ValSerGlyGlyAlaCysAlaAlaGlySerGlyAlaTrpMetAsnLys225230235240GlnProAspSerAlaGluTrpGlyThrValLysTrpAlaGluSerAsn245250255TyrMetArgTyrAsnTyrCysAspAspGlyTrpArgPheProGlnGly260265270LeuProAlaGluCysSerArgSer275280<210>31<211>870<212>DNA<213>拟南芥(Arabidopsisthaliana)<400>31atggactatatgagaatctttagtgtttttgtagtaacgttatggatcataagggttgat60gctagggttttcggaggacgaggaatcgaaaaatttgtaactttcggtcagaattatatt120gttacgtggggacaaagtcatgtttctacactccattctggcgaagaagttgatctttac180atggatcaatcttcaggtggtgggttcgaatccaaggacgcctacggatcaggtctcttt240gaaatgagaatcaaagtgccttcaggaaatactggcggcatcgtcaccgctttttacttg300acgtcgaaaggaggtggtcacgacgagattgacttcgagtttctagggaacaacaacgga360aaaccggtaacgttacagacaaatctgtttctaaatggagaaggaaacagagaagagagg420tttttgctttggttcaatccaaccaaacactaccacacttatgggcttctttggaaccct480taccaaattgtgttttacgtggacaacatcccaataagagtatacaaaaacgaaaacggc540gtaagttatccatcaaagcctatgcaagtcgaggctagtctctggaacggtgacgattgg600gcgactgatggtggtcggactaaggttaactggtcatactctcctttcattgcacatttc660cgagacttcgccttatccggctgcaacatagatggtcggagtaataatgtcggcgcttgt720gaatcctccaactactggtggaatgcaggcaactatcaaagattaagcggaaacgaacag780aaactttatgaacatgtgaggagcaagtacatgaactatgactattgtactgatcgctct840aagtaccaaactcctcctagagaatgttat870<210>32<211>290<212>PRT<213>拟南芥(Arabidopsisthaliana)<400>32MetAspTyrMetArgIlePheSerValPheValValThrLeuTrpIle151015IleArgValAspAlaArgValPheGlyGlyArgGlyIleGluLysPhe202530ValThrPheGlyGlnAsnTyrIleValThrTrpGlyGlnSerHisVal354045SerThrLeuHisSerGlyGluGluValAspLeuTyrMetAspGlnSer505560SerGlyGlyGlyPheGluSerLysAspAlaTyrGlySerGlyLeuPhe65707580GluMetArgIleLysValProSerGlyAsnThrGlyGlyIleValThr859095AlaPheTyrLeuThrSerLysGlyGlyGlyHisAspGluIleAspPhe100105110GluPheLeuGlyAsnAsnAsnGlyLysProValThrLeuGlnThrAsn115120125LeuPheLeuAsnGlyGluGlyAsnArgGluGluArgPheLeuLeuTrp130135140PheAsnProThrLysHisTyrHisThrTyrGlyLeuLeuTrpAsnPro145150155160TyrGlnIleValPheTyrValAspAsnIleProIleArgValTyrLys165170175AsnGluAsnGlyValSerTyrProSerLysProMetGlnValGluAla180185190SerLeuTrpAsnGlyAspAspTrpAlaThrAspGlyGlyArgThrLys195200205ValAsnTrpSerTyrSerProPheIleAlaHisPheArgAspPheAla210215220LeuSerGlyCysAsnIleAspGlyArgSerAsnAsnValGlyAlaCys225230235240GluSerSerAsnTyrTrpTrpAsnAlaGlyAsnTyrGlnArgLeuSer245250255GlyAsnGluGlnLysLeuTyrGluHisValArgSerLysTyrMetAsn260265270TyrAspTyrCysThrAspArgSerLysTyrGlnThrProProArgGlu275280285CysTyr290<210>33<211>906<212>DNA<213>大豆(Glycinemax)<400>33atggatgcaagggcatcatcttcttcaatgtccatgattcttttcattacagctattgtg60gctgcagcttattttcattcatcagaagcagctgtgtcaaaaggatcttttgaggacaat120ttcagcataatgtggtctgaggaccattttactacctctaaagatggacagatctggtat180ctctcactagacaaagacacaggatgtgggtttcaaacaaaacaacgctacagattcggg240tggttcagtatgaagctgaagttggtagcaggtgactctgcaggtgttgtgacagcttat300tatatgtgctcagaaaatggtgcagggccggagagggatgagcttgattttgagtttttg360ggaaacagaactggggagccttatttgattcagactaatgtgtacaagaatgggactgga420gggcgtgagatgaggcatatgctatggtttgaccccacagaggactaccacacttattcc480attctctggaacaaccaccagatagtgttttttgtggatagagttccagtgagggtgttc540aagaacaatggggaaccaaacaacttcttcccgaatgagaagcccatgtacttgttctcc600agcatatggaatgcagatgagtgggccacaagaggtgggcttgagaagacaaactggaaa660ttagccccatttgtgtcatcctacaaagacttcagtgtggatggttgccagtgggaagac720ccataccctgcctgtgtctcaaccacaaccaaaaattggtgggatcagtatgatgcttgg780cacctttctgatgatcagaaaaaggattatgcttgggttcagaggaaccttgttatctat840gactactgccaggattctgcacgttatccaactaccccagaggagtgttcattgagtcca900tgggat906<210>34<211>302<212>PRT<213>大豆(Glycinemax)<400>34MetAspAlaArgAlaSerSerSerSerMetSerMetIleLeuPheIle151015ThrAlaIleValAlaAlaAlaTyrPheHisSerSerGluAlaAlaVal202530SerLysGlySerPheGluAspAsnPheSerIleMetTrpSerGluAsp354045HisPheThrThrSerLysAspGlyGlnIleTrpTyrLeuSerLeuAsp505560LysAspThrGlyCysGlyPheGlnThrLysGlnArgTyrArgPheGly65707580TrpPheSerMetLysLeuLysLeuValAlaGlyAspSerAlaGlyVal859095ValThrAlaTyrTyrMetCysSerGluAsnGlyAlaGlyProGluArg100105110AspGluLeuAspPheGluPheLeuGlyAsnArgThrGlyGluProTyr115120125LeuIleGlnThrAsnValTyrLysAsnGlyThrGlyGlyArgGluMet130135140ArgHisMetLeuTrpPheAspProThrGluAspTyrHisThrTyrSer145150155160IleLeuTrpAsnAsnHisGlnIleValPhePheValAspArgValPro165170175ValArgValPheLysAsnAsnGlyGluProAsnAsnPhePheProAsn180185190GluLysProMetTyrLeuPheSerSerIleTrpAsnAlaAspGluTrp195200205AlaThrArgGlyGlyLeuGluLysThrAsnTrpLysLeuAlaProPhe210215220ValSerSerTyrLysAspPheSerValAspGlyCysGlnTrpGluAsp225230235240ProTyrProAlaCysValSerThrThrThrLysAsnTrpTrpAspGln245250255TyrAspAlaTrpHisLeuSerAspAspGlnLysLysAspTyrAlaTrp260265270ValGlnArgAsnLeuValIleTyrAspTyrCysGlnAspSerAlaArg275280285TyrProThrThrProGluGluCysSerLeuSerProTrpAsp290295300<210>35<211>873<212>DNA<213>蒺藜状苜蓿(Medicagotruncatula)<400>35atggccatattttacccctttaaaaacaatggtgtcttttttatgttgttattgtggatt60gtcgtatcatctgtttggggaagaccagcaacttttaatcaagattttcatgtcacgtgg120tcagaaccccatatcaagcaaattgatcaaggcagaactatccaacttaccctagaccaa180ggctctggttgcgggtttgcatcaaaggtgaagtatttgtttggtcgtgtaagcatgaag240atcaaacttgtccctggagattctgctggtactgttactgcattttatatgaactctgac300actgacagtgtacgtgatgagttggattttgagttcttgggaaaccgtactggacaacct360tacacagttcaaacaaacatttatgctcatggaaagggtgatagagaacaaagagttaat420ctttggtttgatccttctgctgatttccacacttactccattctttggaatcatcaccat480attgtgttctacgttgatgaagttcccattagagtatacaagaacaatgaagcaaaaggg540ataccataccccaaaatgcaagccatgggagtattttcaacattatgggaagctgataat600tgggcaacaagaggtggattagagaaaattaattggagtaaagcacctttttatgcttat660tacaaggattttgatattgaaggatgtgcaatttcaggacccaatacatgttccactaac720cccaaaaattggtggggaggggttgaatatcaagcttttagtgccattgaagctagaagg780tataggtgggtttgtatgaaccatgtgatctatgattattgtcaagataaatcaaggtac840ccaatgaccccacatgagtgtctttcaggcatt873<210>36<211>291<212>PRT<213>蒺藜状苜蓿(Medicagotruncatula)<400>36MetAlaIlePheTyrProPheLysAsnAsnGlyValPhePheMetLeu151015LeuLeuTrpIleValValSerSerValTrpGlyArgProAlaThrPhe202530AsnGlnAspPheHisValThrTrpSerGluProHisIleLysGlnIle354045AspGlnGlyArgThrIleGlnLeuThrLeuAspGlnGlySerGlyCys505560GlyPheAlaSerLysValLysTyrLeuPheGlyArgValSerMetLys65707580IleLysLeuValProGlyAspSerAlaGlyThrValThrAlaPheTyr859095MetAsnSerAspThrAspSerValArgAspGluLeuAspPheGluPhe100105110LeuGlyAsnArgThrGlyGlnProTyrThrValGlnThrAsnIleTyr115120125AlaHisGlyLysGlyAspArgGluGlnArgValAsnLeuTrpPheAsp130135140ProSerAlaAspPheHisThrTyrSerIleLeuTrpAsnHisHisHis145150155160IleValPheTyrValAspGluValProIleArgValTyrLysAsnAsn165170175GluAlaLysGlyIleProTyrProLysMetGlnAlaMetGlyValPhe180185190SerThrLeuTrpGluAlaAspAsnTrpAlaThrArgGlyGlyLeuGlu195200205LysIleAsnTrpSerLysAlaProPheTyrAlaTyrTyrLysAspPhe210215220AspIleGluGlyCysAlaIleSerGlyProAsnThrCysSerThrAsn225230235240ProLysAsnTrpTrpGlyGlyValGluTyrGlnAlaPheSerAlaIle245250255GluAlaArgArgTyrArgTrpValCysMetAsnHisValIleTyrAsp260265270TyrCysGlnAspLysSerArgTyrProMetThrProHisGluCysLeu275280285SerGlyIle290<210>37<211>897<212>DNA<213>拟南芥(Arabidopsisthaliana)<400>37atgactctaataaatcgctcaaaaccatttgtacttctcgtaggcttctcaattatctca60agtttgctattatgggtctcacaagcttccgttgtttcatcaggagattttaacaaagac120ttctttgtgacatggtctcctacccacgttaacacctccaatgatggtcgatcaagaacc180cttaaactcgatcaagaatctggagcttctttttcttccattcagacgttcttgttcggc240caaatagatatgaagatcaaacttatccgtggaagttctcaaggaaccgtggttgcttat300tatatgtcctcagatcagcctaaccgggatgagattgactttgagttcctcggaaacgtt360aatgggcagccttacattctccagacaaatgtctatgccgagggacttgataaccgcgaa420gaaaggatccatctctggttcgacccagccaaggactttcacacttactctatcttgtgg480aacattcaccaaattgtatttatggtggatcagattccgataaggttgtatagaaaccat540ggggagaaaggcgtagcctacccaaggttgcaacctatgagtgtacaggcgagtctatgg600aacggtgagagctgggctacgcgcggtggacatgacaagattgattggtcaaagggtcca660tttgtggcttcctttggggattacaagatagacgcttgtatttggataggcaatacaagt720ttttgtaatggagagagcactgaaaactggtggaacaagaatgagttcagttctttgaca780agagtgcaaaagagatggtttaaatgggtgagaaaatatcacttgatttatgattattgc840caagattatggaaggttcaataacaagctaccgaaggaatgttctcttcctaaatat897<210>38<211>299<212>PRT<213>拟南芥(Arabidopsisthaliana)<400>38MetThrLeuIleAsnArgSerLysProPheValLeuLeuValGlyPhe151015SerIleIleSerSerLeuLeuLeuTrpValSerGlnAlaSerValVal202530SerSerGlyAspPheAsnLysAspPhePheValThrTrpSerProThr354045HisValAsnThrSerAsnAspGlyArgSerArgThrLeuLysLeuAsp505560GlnGluSerGlyAlaSerPheSerSerIleGlnThrPheLeuPheGly65707580GlnIleAspMetLysIleLysLeuIleArgGlySerSerGlnGlyThr859095ValValAlaTyrTyrMetSerSerAspGlnProAsnArgAspGluIle100105110AspPheGluPheLeuGlyAsnValAsnGlyGlnProTyrIleLeuGln115120125ThrAsnValTyrAlaGluGlyLeuAspAsnArgGluGluArgIleHis130135140LeuTrpPheAspProAlaLysAspPheHisThrTyrSerIleLeuTrp145150155160AsnIleHisGlnIleValPheMetValAspGlnIleProIleArgLeu165170175TyrArgAsnHisGlyGluLysGlyValAlaTyrProArgLeuGlnPro180185190MetSerValGlnAlaSerLeuTrpAsnGlyGluSerTrpAlaThrArg195200205GlyGlyHisAspLysIleAspTrpSerLysGlyProPheValAlaSer210215220PheGlyAspTyrLysIleAspAlaCysIleTrpIleGlyAsnThrSer225230235240PheCysAsnGlyGluSerThrGluAsnTrpTrpAsnLysAsnGluPhe245250255SerSerLeuThrArgValGlnLysArgTrpPheLysTrpValArgLys260265270TyrHisLeuIleTyrAspTyrCysGlnAspTyrGlyArgPheAsnAsn275280285LysLeuProLysGluCysSerLeuProLysTyr290295<210>39<211>870<212>DNA<213>拟南芥(Arabidopsisthaliana)<400>39atggtcggtatggatttgttcaaatgtgtaatgatgataatggtgttggttgtctcttgt60ggtgaagctgtttctggagctaagttcgatgagctttatcggtcaagctgggctatggat120cattgtgtcaacgaaggcgaagtcacaaaactcaagcttgacaattactctggagctggg180tttgaatcaagaagcaaatacttgtttggtaaagtctctatccagattaagcttgttgag240ggtgattcagcaggaacagtcactgctttctacatgtcttcagatggtccgaaccacaac300gaattcgattttgagttcttagggaacactaccggcgagccttacatagtccagaccaat360atctacgtgaacggagttggaaacagagaacaaagactcaacctttggttcgatcccacc420actgagtttcacacttactcaatcctctggagtaaacgcagtgttgtattcatggtagac480gaaacaccgattcgagttcagaagaatcttgaagagaaaggtatcccatttgctaaagat540caagcaatgggtgtttacagctccatttggaatgcagatgattgggctacacaaggaggt600cttgttaaaactgattggagtcacgctcctttcgttgcttcttacaaagaattccaaatc660gatgcatgtgagattccaacaactactgatctaagcaagtgtaatggagaccagaagttt720tggtgggatgaaccaactgtctctgagcttagccttcatcagaatcatcagcttatttgg780gttcgagctaatcatatgatttatgattattgttttgatgctactaggtttcctgttact840cctcttgagtgccaacatcatcgtcatttg870<210>40<211>290<212>PRT<213>拟南芥(Arabidopsisthaliana)<400>40MetValGlyMetAspLeuPheLysCysValMetMetIleMetValLeu151015ValValSerCysGlyGluAlaValSerGlyAlaLysPheAspGluLeu202530TyrArgSerSerTrpAlaMetAspHisCysValAsnGluGlyGluVal354045ThrLysLeuLysLeuAspAsnTyrSerGlyAlaGlyPheGluSerArg505560SerLysTyrLeuPheGlyLysValSerIleGlnIleLysLeuValGlu65707580GlyAspSerAlaGlyThrValThrAlaPheTyrMetSerSerAspGly859095ProAsnHisAsnGluPheAspPheGluPheLeuGlyAsnThrThrGly100105110GluProTyrIleValGlnThrAsnIleTyrValAsnGlyValGlyAsn115120125ArgGluGlnArgLeuAsnLeuTrpPheAspProThrThrGluPheHis130135140ThrTyrSerIleLeuTrpSerLysArgSerValValPheMetValAsp145150155160GluThrProIleArgValGlnLysAsnLeuGluGluLysGlyIlePro165170175PheAlaLysAspGlnAlaMetGlyValTyrSerSerIleTrpAsnAla180185190AspAspTrpAlaThrGlnGlyGlyLeuValLysThrAspTrpSerHis195200205AlaProPheValAlaSerTyrLysGluPheGlnIleAspAlaCysGlu210215220IleProThrThrThrAspLeuSerLysCysAsnGlyAspGlnLysPhe225230235240TrpTrpAspGluProThrValSerGluLeuSerLeuHisGlnAsnHis245250255GlnLeuIleTrpValArgAlaAsnHisMetIleTyrAspTyrCysPhe260265270AspAlaThrArgPheProValThrProLeuGluCysGlnHisHisArg275280285HisLeu290<210>41<211>912<212>DNA<213>矮慈姑(Sagittariapygmaea)<400>41atggctctccgaccgtctttacataaacagttcgctctcctagtggcagcgtctgtgctg60gccttttgctgctcggcagcaggccgcagcgtcggcctcgccccggcctcgccctccagc120ttcaacaacgatttcgacatcacctggggtgatggccgggggaagatcctaaacaacggc180cagctcctcagcctctccctggacaaaacctccggctcagggtttcaatctaaaaaccag240tacctctttggcaacatcgacatgcaactgaagctcgtccccggcaactcggctggaacg300gtcaccgcctattacctatcctcacagggccctacccacgacgagatcgactttgagttc360ctcgggaacgtctccggccagccctacaccctgcacaccaacgttttctcccaggggaaa420ggcaacagggagcaacagttccggctttggttcgaccccaccaaggacttccacacctat480tcgatcctctggaaccccagccacatcgtcttctacgtcgacggcacccccatcagagac540ttccgcaataacgaggccagaggagttcccttccccaaggcccagccgatgcggatctat600tccagtctttggaacgccgacgactgggctacccaaggaggccggatcaagacagactgg660acgcaggcgccattcacggcgtcctacaggggattcaaggcggacgcatgtgccgttgga720tcaactggatcctcctccaaatgcgccgccttgcagaccaccgccggcaacaccaactgg780tggatccggcaggagctcgatgccaccggccaggagcgcatgcgctgggtgcagcgcaac840cacatgatctacaactactgcacagacgtcaaccgcttccctcaggggttacccactgag900tgctccgttgcc912<210>42<211>304<212>PRT<213>矮慈姑(Sagittariapygmaea)<400>42MetAlaLeuArgProSerLeuHisLysGlnPheAlaLeuLeuValAla151015AlaSerValLeuAlaPheCysCysSerAlaAlaGlyArgSerValGly202530LeuAlaProAlaSerProSerSerPheAsnAsnAspPheAspIleThr354045TrpGlyAspGlyArgGlyLysIleLeuAsnAsnGlyGlnLeuLeuSer505560LeuSerLeuAspLysThrSerGlySerGlyPheGlnSerLysAsnGln65707580TyrLeuPheGlyAsnIleAspMetGlnLeuLysLeuValProGlyAsn859095SerAlaGlyThrValThrAlaTyrTyrLeuSerSerGlnGlyProThr100105110HisAspGluIleAspPheGluPheLeuGlyAsnValSerGlyGlnPro115120125TyrThrLeuHisThrAsnValPheSerGlnGlyLysGlyAsnArgGlu130135140GlnGlnPheArgLeuTrpPheAspProThrLysAspPheHisThrTyr145150155160SerIleLeuTrpAsnProSerHisIleValPheTyrValAspGlyThr165170175ProIleArgAspPheArgAsnAsnGluAlaArgGlyValProPhePro180185190LysAlaGlnProMetArgIleTyrSerSerLeuTrpAsnAlaAspAsp195200205TrpAlaThrGlnGlyGlyArgIleLysThrAspTrpThrGlnAlaPro210215220PheThrAlaSerTyrArgGlyPheLysAlaAspAlaCysAlaValGly225230235240SerThrGlySerSerSerLysCysAlaAlaLeuGlnThrThrAlaGly245250255AsnThrAsnTrpTrpIleArgGlnGluLeuAspAlaThrGlyGlnGlu260265270ArgMetArgTrpValGlnArgAsnHisMetIleTyrAsnTyrCysThr275280285AspValAsnArgPheProGlnGlyLeuProThrGluCysSerValAla290295300<210>43<211>876<212>DNA<213>大麦(Hordeumvulgare)<400>43atggctcgcatgggggcgtcggtgctggtgatcctgctcgcctcttgtgccctggcggcg60gcgagcttcgacaaggagttcgacgttacctggggtgacgggcgcggcaagatcctcaac120aatggccagctgctgatgctggggctggacaaggtctccggctccgggttccagtccaag180cgcgagtacctcttcggcaagatcgacatgcagctcaagctcgtccccggcaactccgcc240ggcaccgtcaccgcatattacctgtcgtcgcagggtccgacgcacgacgagatcgacttc300gagttcctgggcaacgtcaccggcgagccatacacgctgcacaccaacgtgttcacgcag360gggcagggccagcgggagcagcagttccgcctctggttcgatcctaccaacgacttccac420acctactccatcctctggaacccgaagcacatcatcttcttggttgacgacatgccgatc480agggacttcaggaacatggagggaaaggggatcgccttccccaagaaccagcctatgcgg540ctgtactccagcctctggaacgccgacgactgggcgacacagggtggccgcgtcaagacc600gactggtcccacgctccgttttccgcctcctaccgcggcttcaaggccgacgcgtgcgtg660gtgaccgtaggtggccggccgcgctgcggcgccagcatcggcacggacgcggccccaggg720accggcggcgcggccgcggtcggcgactggtacaaccaagagctggatcttacgcggcag780cagcgcatgcgttgggtgcagagcaattacatgatctacaactactgcactgaccccaag840cgcgtcgccaagggcgtccctgccgagtgctccatg876<210>44<211>292<212>PRT<213>大麦(Hordeumvulgare)<400>44MetAlaArgMetGlyAlaSerValLeuValIleLeuLeuAlaSerCys151015AlaLeuAlaAlaAlaSerPheAspLysGluPheAspValThrTrpGly202530AspGlyArgGlyLysIleLeuAsnAsnGlyGlnLeuLeuMetLeuGly354045LeuAspLysValSerGlySerGlyPheGlnSerLysArgGluTyrLeu505560PheGlyLysIleAspMetGlnLeuLysLeuValProGlyAsnSerAla65707580GlyThrValThrAlaTyrTyrLeuSerSerGlnGlyProThrHisAsp859095GluIleAspPheGluPheLeuGlyAsnValThrGlyGluProTyrThr100105110LeuHisThrAsnValPheThrGlnGlyGlnGlyGlnArgGluGlnGln115120125PheArgLeuTrpPheAspProThrAsnAspPheHisThrTyrSerIle130135140LeuTrpAsnProLysHisIleIlePheLeuValAspAspMetProIle145150155160ArgAspPheArgAsnMetGluGlyLysGlyIleAlaPheProLysAsn165170175GlnProMetArgLeuTyrSerSerLeuTrpAsnAlaAspAspTrpAla180185190ThrGlnGlyGlyArgValLysThrAspTrpSerHisAlaProPheSer195200205AlaSerTyrArgGlyPheLysAlaAspAlaCysValValThrValGly210215220GlyArgProArgCysGlyAlaSerIleGlyThrAspAlaAlaProGly225230235240ThrGlyGlyAlaAlaAlaValGlyAspTrpTyrAsnGlnGluLeuAsp245250255LeuThrArgGlnGlnArgMetArgTrpValGlnSerAsnTyrMetIle260265270TyrAsnTyrCysThrAspProLysArgValAlaLysGlyValProAla275280285GluCysSerMet290<210>45<211>849<212>DNA<213>大豆(Glycinemax)<400>45atggctcctagttctgctcacaacaatgggttctatgtgcttatgctagttgggatagtg60gttagcactatggttgctacctgtgctggtagcttctaccaagactttgatctaacatgg120ggtggtgaccgtgctaagatattcaatggtggccagcttctatcactttccctagacaaa180gtctctggctctggcttcaaatcaaagaaagaatacctatttgggaggattgatatgcag240ctcaagctcgttgccggcaactctgctggcactgtcactgcttactacttgtcatcccaa300gggccaacacatgatgagattgatttcgagtttttgggaaacctaagtggggacccttat360attctccacacaaacatcttcacccaaggcaaaggcaacagggagcaacagttctatctc420tggttcgaccccaccagaaacttccacacttactctatcatttggaagccccagcacatc480atattcttggttgataacacacccataagggtattcaagaatgctgaacctcttggtgtt540ccttttccaaagaaccagcccatgagaatctattctagcctctggaatgctgatgactgg600gccaccagaggaggattggtgaaaactgattggtccaaagcaccctttacagcatactac660cgcaatttcaaggccattgagttctcatccaagtcttccatttcaaattctggggctgaa720tatgaggcaaatgagcttgatgcttatagcagaagaagactgagatgggttcagaagtac780ttcatgatctataactactgcagtgatctcaagcgattcccacaaggtcttcctgctgaa840tgtaaacgt849<210>46<211>283<212>PRT<213>大豆(Glycinemax)<400>46MetAlaProSerSerAlaHisAsnAsnGlyPheTyrValLeuMetLeu151015ValGlyIleValValSerThrMetValAlaThrCysAlaGlySerPhe202530TyrGlnAspPheAspLeuThrTrpGlyGlyAspArgAlaLysIlePhe354045AsnGlyGlyGlnLeuLeuSerLeuSerLeuAspLysValSerGlySer505560GlyPheLysSerLysLysGluTyrLeuPheGlyArgIleAspMetGln65707580LeuLysLeuValAlaGlyAsnSerAlaGlyThrValThrAlaTyrTyr859095LeuSerSerGlnGlyProThrHisAspGluIleAspPheGluPheLeu100105110GlyAsnLeuSerGlyAspProTyrIleLeuHisThrAsnIlePheThr115120125GlnGlyLysGlyAsnArgGluGlnGlnPheTyrLeuTrpPheAspPro130135140ThrArgAsnPheHisThrTyrSerIleIleTrpLysProGlnHisIle145150155160IlePheLeuValAspAsnThrProIleArgValPheLysAsnAlaGlu165170175ProLeuGlyValProPheProLysAsnGlnProMetArgIleTyrSer180185190SerLeuTrpAsnAlaAspAspTrpAlaThrArgGlyGlyLeuValLys195200205ThrAspTrpSerLysAlaProPheThrAlaTyrTyrArgAsnPheLys210215220AlaIleGluPheSerSerLysSerSerIleSerAsnSerGlyAlaGlu225230235240TyrGluAlaAsnGluLeuAspAlaTyrSerArgArgArgLeuArgTrp245250255ValGlnLysTyrPheMetIleTyrAsnTyrCysSerAspLeuLysArg260265270PheProGlnGlyLeuProAlaGluCysLysArg275280<210>47<211>849<212>DNA<213>黄瓜(Cucumissativus)<400>47atggcttccacaactctgtttctcacgcttcttttctctgcttccctaattgccatctct60tcggctaacttcaaccaagattttcaaatcacttggggagatggtcgagcaaagattctc120aacaatggtgaacttctaactctgtcccttgacaaggcctctggctctggcttccagtct180caaaacgagtacttgtttggaaagattgatatgcaactcaagcttgtggctggcaattct240gctggcactgtcactgcttattatttgtcttcgaaaggatctacatgggatgagattgac300tttgagttcttggggaatttgagtggtgacccttacactctccataccaatgtgtttagc360caagggaaaggcaacagagaacaacagttccatctttggtttgaccccactgctgatttc420cacacctattccattctttggaaccccaatcgtattatattttcggtggacggtactccg480ataagagaattcaagaactgggaatcgaacggcgtgccatttccgaaggaccagccaatg540agaatatactcgagcttatggaacgcggacgattgggcaacaagaggagggctggtgaag600acggactggacaaaggctcctttcactgcttcctacaaaaacttcaaagccgaagcctgc660gtttggtccggggggaaatcttcttgcagcccctcgccggcggggtcgagcaactcatgg720ctgtcccaagagatggactctgcaagtcaagagagactaaaatgggtgcaaaagaattat780atgatttacaactattgtacggacaccaagaggtttcctcaaggcctccctacagagtgc840aacttgcca849<210>48<211>283<212>PRT<213>黄瓜(Cucumissativus)<400>48MetAlaSerThrThrLeuPheLeuThrLeuLeuPheSerAlaSerLeu151015IleAlaIleSerSerAlaAsnPheAsnGlnAspPheGlnIleThrTrp202530GlyAspGlyArgAlaLysIleLeuAsnAsnGlyGluLeuLeuThrLeu354045SerLeuAspLysAlaSerGlySerGlyPheGlnSerGlnAsnGluTyr505560LeuPheGlyLysIleAspMetGlnLeuLysLeuValAlaGlyAsnSer65707580AlaGlyThrValThrAlaTyrTyrLeuSerSerLysGlySerThrTrp859095AspGluIleAspPheGluPheLeuGlyAsnLeuSerGlyAspProTyr100105110ThrLeuHisThrAsnValPheSerGlnGlyLysGlyAsnArgGluGln115120125GlnPheHisLeuTrpPheAspProThrAlaAspPheHisThrTyrSer130135140IleLeuTrpAsnProAsnArgIleIlePheSerValAspGlyThrPro145150155160IleArgGluPheLysAsnTrpGluSerAsnGlyValProPheProLys165170175AspGlnProMetArgIleTyrSerSerLeuTrpAsnAlaAspAspTrp180185190AlaThrArgGlyGlyLeuValLysThrAspTrpThrLysAlaProPhe195200205ThrAlaSerTyrLysAsnPheLysAlaGluAlaCysValTrpSerGly210215220GlyLysSerSerCysSerProSerProAlaGlySerSerAsnSerTrp225230235240LeuSerGlnGluMetAspSerAlaSerGlnGluArgLeuLysTrpVal245250255GlnLysAsnTyrMetIleTyrAsnTyrCysThrAspThrLysArgPhe260265270ProGlnGlyLeuProThrGluCysAsnLeuPro275280<210>49<211>870<212>DNA<213>水稻(Oryzasativa)<400>49atggggcaacctagggctcaactcctaccctccatgtccatggcagctttgtatctcatc60ctagccacctccccagtaatcagcgacatgaccgacagccttgacatgctgtggggcaat120acacaggtgctctatgacagcactggccaccaaattgtatcactgtcccttgaccgctgg180actacttctgcattccgctcaaagactaagtatctctttgcgaggattgacatggacatc240aagcttgttgccaaggactcagccggcactgtcaccacattatatatgataactgagggg300ctgtgggacatacatgatgagattgatctcgagttcttggggaacaccaccggtgagccc360tacaccttgcacactaacatctatgccagaggaacaggtggccgagaaaagcagtaccgg420ctttggtttgatccaacagaagacttccatacctacaccatcatctggaacccgcagatg480atcttaatacttgttgatggcacaccaattcggcagatgaagaatcagctaaggaatgat540atacccttcccgctatatcaaccaatgaggctgtacgccagcatctgggatgccgatgac600tgggcaacgcagggtgggcgtatcaaaaccgactggtctcaagcaccatttaccgcattc660ttccggaactaccaagctaacgcctgcattccgtacaaaactgcctggatttgcagccaa720gggtctaatgacagcagctggtttacccaggatttggatgaggaagggaagcagaaactt780aaggacgtagatgataactacaagatttatgattactgcactgactcaaggaggtaccca840aatgggtatcccccagagtgcgggtcacag870<210>50<211>290<212>PRT<213>水稻(Oryzasativa)<400>50MetGlyGlnProArgAlaGlnLeuLeuProSerMetSerMetAlaAla151015LeuTyrLeuIleLeuAlaThrSerProValIleSerAspMetThrAsp202530SerLeuAspMetLeuTrpGlyAsnThrGlnValLeuTyrAspSerThr354045GlyHisGlnIleValSerLeuSerLeuAspArgTrpThrThrSerAla505560PheArgSerLysThrLysTyrLeuPheAlaArgIleAspMetAspIle65707580LysLeuValAlaLysAspSerAlaGlyThrValThrThrLeuTyrMet859095IleThrGluGlyLeuTrpAspIleHisAspGluIleAspLeuGluPhe100105110LeuGlyAsnThrThrGlyGluProTyrThrLeuHisThrAsnIleTyr115120125AlaArgGlyThrGlyGlyArgGluLysGlnTyrArgLeuTrpPheAsp130135140ProThrGluAspPheHisThrTyrThrIleIleTrpAsnProGlnMet145150155160IleLeuIleLeuValAspGlyThrProIleArgGlnMetLysAsnGln165170175LeuArgAsnAspIleProPheProLeuTyrGlnProMetArgLeuTyr180185190AlaSerIleTrpAspAlaAspAspTrpAlaThrGlnGlyGlyArgIle195200205LysThrAspTrpSerGlnAlaProPheThrAlaPhePheArgAsnTyr210215220GlnAlaAsnAlaCysIleProTyrLysThrAlaTrpIleCysSerGln225230235240GlySerAsnAspSerSerTrpPheThrGlnAspLeuAspGluGluGly245250255LysGlnLysLeuLysAspValAspAspAsnTyrLysIleTyrAspTyr260265270CysThrAspSerArgArgTyrProAsnGlyTyrProProGluCysGly275280285SerGln290<210>51<211>876<212>DNA<213>人工序列<220><223>人工DNA序列<400>51atgggctcgtccctctggacttgtttgatcctcctctccttggcatcggcatccttcgca60gcgaaccctcgaactccgatcgatgtgcctttcggacggaactacgtgccgacatgggca120ttcgaccacattaagtatttgaacggaggctcggagatccagttgcatctcgacaagtac180accggcactggtttccagtcgaagggctcctacttgttcggacatttctccatgtacatc240aaattggtgcctggtgactcggcaggaactgtcaccgcattctacctctcgtcgacaaac300gcagagcatgacgaaatcgacttcgagttcctcggcaacaggacaggacagccgtacatc360ctccagaccaacgtcttcacaggaggcaaaggtgatcgggaacagcggatctacttgtgg420ttcgatcccacaacccagtaccataggtactcggtgctctggaacatgtatcagatcgtc480ttctacgtcgacgattatccgatccgagtgttcaagaactccaacgacttgggcgtcaaa540ttccccttcaaccagcccatgaagatttacaactcgttgtggaacgccgacgattgggca600accaggggtggtctcgagaagacagattggtcgaaagcacctttcatcgcgtcgtacaag660ggtttccacatcgacggatgtgaagcctccgtgaacgccaagttctgtgacacccagggc720aaacgatggtgggatcagccggaattccgggatttggatgcagcccagtggcagaagctc780gcgtgggtcaggaacaagtacaccatctataactactgtaccgatcggaaacgatattcg840caggtgcctcccgagtgtacacgcgatagggacatc876<210>52<211>36<212>DNA<213>人工序列<220><223>人工DNA引物<400>52acatgtctttgataagctagcgggccgcatcatgta36<210>53<211>36<212>DNA<213>人工序列<220><223>人工DNA引物<400>53tacatgatgcggcccgctagcttatcaaagacatgt36<210>54<211>41<212>DNA<213>人工序列<220><223>人工DNA引物<400>54ttaatcgccttgcagcacaccgcttcctcgctcactgactc41<210>55<211>47<212>DNA<213>人工序列<220><223>人工DNA引物<400>55acaataaccctgataaatgcggaacaacactcaaccctatctcggtc47<210>56<211>53<212>DNA<213>人工序列<220><223>人工DNA引物<400>56agatagggttgagtgttgttccgcatttatcagggttattgtctcatgagcgg53<210>57<211>42<212>DNA<213>人工序列<220><223>人工DNA引物<400>57ttctacacgaaggaaagaggaggagagagttgaacctggacg42<210>58<211>47<212>DNA<213>人工序列<220><223>人工DNA引物<400>58aggttcaactctctcctcctctttccttcgtgtagaagaccagacag47<210>59<211>43<212>DNA<213>人工序列<220><223>人工DNA引物<400>59tcagtgagcgaggaagcggtgtgctgcaaggcgattaagttgg43<210>60<211>50<212>DNA<213>人工序列<220><223>人工DNA引物<400>60ttcctcaatcctctatatacacaactggccatgggctcgtccctctggac50<210>61<211>45<212>DNA<213>人工序列<220><223>人工DNA引物<400>61agctcgctagagtcgacctagatgtccctatcgcgtgtacactcg45<210>62<211>27<212>DNA<213>人工序列<220><223>人工DNA引物<400>62taggtcgactctagcgagctcgagatc27<210>63<211>40<212>DNA<213>人工序列<220><223>人工DNA引物<400>63catggccagttgtgtatatagaggattgaggaaggaagag40<210>64<211>11101<212>DNA<213>人工序列<220><223>人工DNA序列<400>64ttgaagttcctattccgagttcctattctctagaaagtataggaacttcagtacccgggt60ataagctagcttccgttaaattgccgtcgtcagccgttaaattaccgattaatcccgata120aatttccgagatctccgttaaattgccgttcgcagccgttaaattaccggggacgaccga180taaatttccgcgatgaattcatggtgttttgatcattttaaatttttatatggcgggtgg240tgggcaactcgcttgcgcgggcaactcgcttaccgattacgttagggctgatatttacgt300aaaaatcgtcaagggatgcaagaccaaaccgttaaatttccggagtcaacagcatccaag360cccaagtccttcacggagaaaccccagcgtccacatcacgagcgaaggaccacctctagg420catcggacgcaccatccaattagaagcagcaaagcgaaacagcccaagaaaaaggtcggc480ccgtcggccttttctgcaacgctgatcacgggcagcgatccaaccaacaccctccagagt540gactaggggcggaaatttatcgggattaatttccactcaaccacaaatcacagtcgtccc600cggtaatttaacggctgcagacggcaatttaacggcttctgcgaatcgcttggattcccc660gcccctggccgtagagcttaaagtatgtcccttgtcgatgcgatgtatcacaacatataa720atactggcaagggatgccatgcttggagtttccaactcaatttacctctatccacacttc780tcttccttcctcaatcctctatatacacaactggggatccaccatgttctcggcaggcca840caagattaagggtacagtcgtcctcatgcctaaaaacgagttggaagtgaaccccgatgg900ctccgcagtcgataacctcaacgcattcctcggacgttcggtgtcgctccagctcatctc960cgcgaccaaagccgacgcccacggtaagggaaaggtgggcaaggacacgttcttggaagg1020tatcaacacttcgctccctaccttgggagcaggagagtccgcattcaacattcacttcga1080gtgggacggttcgatgggcattcccggagcgttctatatcaagaactatatgcaggtgga1140gttcttcttgaagtccttgaccttggaggcaatctcgaaccagggtaccatccgtttcgt1200gtgtaactcgtgggtctacaacaccaagctctacaaatccgtgcggatcttcttcgcgaa1260ccacacttacgtcccttcggagacacctgcccctttggtgtcgtaccgcgaggaggaatt1320gaagtccctccgtggtaacggtactggagaaaggaaggagtatgataggatctacgacta1380cgacgtctataacgatttgggtaaccccgacaaatcggaaaagttggcacgtcctgtgtt1440gggaggctcctccaccttcccctaccctcgacgcggccgcacgggacgcggtcccactgt1500caccgatccgaacacagagaagcagggcgaagtcttctacgtgcccagggacgaaaacct1560cggccacttgaagtcgaaggatgcattggagattggaaccaagtccctctcccagatcgt1620ccagcctgcattcgaatcggcgttcgatttgaaatcgacgcccatcgagttccactcgtt1680ccaggacgtccatgacttgtatgaaggtggtatcaaattgcctcgggacgtcatctccac1740cattatccccctccccgtgatcaaggaattgtaccgcaccgacggccagcatattctcaa1800attcccccagccgcacgtcgtccaggtctcgcagtccgcatggatgacagatgaggaatt1860cgcgagggaaatgattgcaggtgtcaacccgtgtgtcatccgaggcttggaggagttccc1920tcctaagtccaacctcgatcctgccatctatggagaccagtcctccaagattacagccga1980ttccctcgatctcgacggttatactatggatgaagcactcggttccaggcgattgttcat2040gctcgattatcatgatatcttcatgccctatgtgcgccagatcaaccagttgaactcggc2100aaaaacatatgcaacgaggacgatcctcttcctccgagaagacggcacactcaagcctgt2160ggcaatcgagctctcgctcccccattccgcaggcgatctctccgcagccgtgtcgcaggt2220ggtgttgcctgcaaaagaaggagtggagtcgaccatctggctcttggccaaagcatatgt2280gattgtgaacgattcctgttatcaccagctcatgtcgcattggctcaacactcacgcggc2340aatggaacccttcgtgatcgccacgcaccggcacctctcggtgctccacccgatctacaa2400gctcctcactccccactaccgtaacaacatgaacattaacgccttggcacggcagtcgtt2460gatcaacgcgaacggcatcattgagacaacgttcctcccctccaagtactccgtcgaaat2520gtcgtccgcagtctacaaaaactgggtcttcaccgaccaggcgttgcctgccgacttgat2580caaacgaggcgtcgcaatcaaagatccctccactcctcatggcgtccgcctcttgatcga2640ggactacccctacgcagcggacggattggaaatctgggcagccatcaagacctgggtgca2700ggaatacgtccctttgtactatgcgagggacgatgatgtcaaaaacgactcggaactcca2760gcattggtggaaggaggcagtggaaaagggccatggagatctcaaggataaaccctggtg2820gcctaagctccagaccttggaggacctcgtcgaagtgtgtttgatcattatctggatcgc2880atccgcgttgcatgcagccgtgaacttcggacagtatccctatggaggcctcatcatgaa2940ccgtcccaccgcatccaggaggctcctccccgaaaaaggaacacccgaatacgaagaaat3000gatcaacaaccacgaaaaggcatacctccggaccatcacttccaaactcccgaccttgat3060ctcgctctccgtgatcgagattttgtcgacacatgcgtcggacgaggtctatttgggtca3120gcgggataacccgcactggacatccgattccaaggccctccaggcgttccagaagttcgg3180caacaagctcaaggagatcgaggagaaactcgtgaggcggaacaacgacccttccctcca3240gggaaaccggttgggacctgtccagctcccgtatacgttgctctacccctcctcggaaga3300aggcctcactttcaggggtatccccaactcgatttccatctgactcgagatctagagggt3360gactgacacctggcggtagacaatcaatccatttcgctatagttaaaggatggggatgag3420ggcaattggttatatgatcatgtatgtagtgggtgtgcataatagtagtgaaatggaagc3480caagtcatgtgattgtaatcgaccgacggaattgaggatatccggaaatacagacaccgt3540gaaagccatggtctttccttcgtgtagaagaccagacagacagtccctgatttacccttg3600cacaaagcactagaaaattagcattccatccttctctgcttgctctgctgatatcactgt3660cattcaatgcatagccatgagctcatcttagatccaagcacgtaattccatagccgaggt3720ccacagtggagcagcaacattccccatcattgctttccccaggggcctcccaacgactaa3780atcaagagtatatctctaccgtccaatagatcgtcttcgcttcaaaatctttgacaattc3840caagagggtccccatccatcaaacccagttcaataatagccgagatgcatggtggagtca3900attaggcagtattgctggaatgtcggggccagttggccgggtggtcattggccgcctgtg3960atgccatctgccactaaatccgatcattgatccaccgcccacgaggcgcgtctttgcttt4020ttgcgcggcgtccaggttcaactctctcttaattaaatagcgacaagccgaacggcaccg4080gcaggtacaatggttcgctgtacttgcttgcgcaagcgggtctttggggattgagcgcat4140ttggtgttgcaaaggatttgatgtaaatgtagtcgacatcttagcacagaggggagagtt4200gataaaatgtggtctgtttgaatgatagtcgggttcgtgacctatattcgtgatagtgga4260gataggtctgcgcctatcttatcgggccggagcaaaaattccaccgcagcggggtgagtt4320ttcgttatacagccatcccacttccagcttcaaattgtcagtttaatccagcccaattca4380atcattggagaaccggttttatgtcttcgaagtcccacctcccctacgcaattcgcgcaa4440ccaaccatcccaaccctttaacatctaaactcttctccatcgccgaggagaagaaaacca4500acgtcaccgtctccgcagacgttactacttccgccgagctcctcgatcttgctgaccgcc4560taggcccctatatcgcagttctgaaaacccacatcgacatcctcaccgatctcaccccgt4620cgaccctttcctcgctccaatccctcgcgacaaagcacaacttcctcatctttgaggacc4680gcaagttcatcgacatcggcaacaccgtgcaaaagcagtaccacggtggcgctctccgca4740tctccgaatgggcacacatcatcaactgcgccatcctgccgggcgaagggatcgtcgagg4800ccctcgcacagacaaccaagtctcctgactttaaagacgcgaatcaacgaggtctcctga4860ttcttgccgagatgacgagtaagggatctcttgcgacaggggagtacacggcacgctcgg4920ttgagtacgcgcggaagtataaggggtttgtgatgggattcgtgagtacaagggcgttga4980gtgaggtgctgcccgaacagaaagaggagagcgaggattttgtcgtctttacgactgggg5040tgaatctgtcggataagggggataagctggggcagcagtatcagacacctgggtcggcgg5100ttgggcgaggtgcggactttatcattgcgggtaggggcatctataaggcggacgatccag5160tcgaggcggttcagaggtaccgggaggaaggctggaaagcttacgagaaaagagttggac5220tttgagggtgactgacacctggcggtagacaatcaatccatttcgctatagttaaaggat5280ggggatgagggcaattggttatatgatcatgtatgtagtgggtgtgcataatagtagtga5340aatggaagccaagtcatgtgattgtaatcgaccgacggaattgaggatatccggaaatac5400agacaccgtgaaagccatggtctttccttcgtgtagaagaccagacagacagtccctgat5460ttacccttgcacaaagcactagaaaattagcattccatccttctctgcttgctctgctga5520tatcactgtcattcaatgcatagccatgagctcatcttagatccaagcacgtaattccat5580agccgaggtccacagtggagcagcaacattccccatcattgctttccccaggggcctccc5640aacgactaaatcaagagtatatctctaccgtccaatagatcgtcttcgcttcaaaatctt5700tgacaattccaagagggtccccatccatcaaacccagttcaataatagccgagatgcatg5760gtggagtcaattaggcagtattgctggaatgtcggggccagttggccgggtggtcattgg5820ccgcctgtgatgccatctgccactaaatccgatcattgatccaccgcccacgaggcgcgt5880ctttgctttttgcgcggcgtccaggttcaactctctcctctaggttgaagttcctattcc5940gagttcctattcttcaaatagtataggaacttcaactagctagtgcatgcgtacgatttt6000gacatttgctccattgtcgaggatggatggaacgagcggcgtgcgccacgaaagtgaggc6060tattgcctatcagctctttgctacattccggaaacaaacatccctttttgtgaattatct6120acgcaacttagatggcgtgaacgcatcttcaaagtctttcggcaggtccggcacgacttt6180tgcatccagagaagcgcctacatgtgtattcgaccacctcctagcgcgcttggatatgag6240gaaatattactgagagtcgaaaacaagctccaccgcaccagctcttcttggagttttata6300ttaaagaatattcccagctcgttgtattattctttttctaccgtgctaatgtatcaagga6360ctttggtacctattaacgttattattcgtgtgctattcccaaacataaccctgtatatgt6420ttcgaacgccgttatgacccatgtcttacatactcattaagtcattcccttggataatct6480cgactcagatgcggcggttgatgtaggaggagaggtaatcgaggacctcctgggagatga6540tgccgttccaggcggggtagcggatggagccctcggcggagcccttgagctgctcgatat6600gctgccactcctcgatggggttggtctcatccttgagggcgatcatctccttggagatgg6660gatcgtaggcgtagtagcgggagactagtgcgaagtaatgatcggggatggcggtgatct6720gatgggtgtaggtggtgcgggcgacggcggaggcgcgcttatcggaccagttgccgacga6780cgttggtgagctcggtgaggcccttcatggagaggaaggaggtcatgagatggcggccga6840tatgggacttggggccgttcttgatggcgaagatggagtagggggcgttcttcttgaggg6900ccttgttgtaggagcggacgaggttatccttgaggagctggtactcctgcttgttggagg6960aggagttgccggtgcggttgacgcgcttgaggacgggctcggagttgcggaggaactcat7020cgaggtagacgaggggatcgatgcggccgcgggcggagaagaagtagatatggcgggaga7080cggaggtcttggtctcggtgacgaggcactggatgatgacgccgaggtacttgttctgga7140cgagcttgaaggacttgggatcgacgttcttgatatcggagaagcggccgcagttgatga7200aggtggcgaggaagaggaactggtagagggtcttggtcttggtgaagcgggaggtgtact7260cgaaggagttgaggatcttctcggtgatctcccagatggactcgccctcggagaggaggg7320ccttgagcatcttcttggaatgggagttgcccttatcggcctcctcggaggactcgaact7380ggagctggagggaggagacgatatcggtgatatcggactgatgcttctggccgtagtagg7440ggatgatggtgaactcccaggcggggatgagcttcttgagggaggcctccaggatggtgg7500ccttctgggtcttgtacttgaactggagggacttgttgacgatatcgaaggagagggagt7560tggagatgatggtgttgtaggacatgaaggtggcgcgcttgatggcggtgccgttatggg7620tgatcatccagcagaggtaggtgagctcggcggcgcagagggcgatcttctcgccggagg7680ggcgctcgaagcgctcgacgaactggcggacgaggaccttggggggggtcttgcagagga7740tatcgaactggggcatggtgctcagatactacggctgatcgcgtagaggtactgagcaaa7800acagatgtcagtaaggagaagagttgaatgaatggaagaagagtaggaaaggaggtatgg7860gggaaagatatacgtactgatgcggacgaagagagaaagaaggaaaaaagttgtgggagg7920ggaaggagggggaatccttatatggaggggcaagcgagaaggcgaattagtgggcgggct7980taagccctcgaccgccgcccttatcattggacatggaggggtaatgcccccaccacgcat8040gtgcgggaccgacgcagaatctgcacggcggagtctcttccagactgttgacttttgggc8100gatgactcttgttgctgcggccttttgggtacaccaacctcgttgatcttgtttccttgg8160ttctctttcgctcggagacccgaccatgaccccaccatcagtcactatcctgcctcgtcg8220ataaaaattttttcttccctctgattgttacatagtatgtttccacctttccggtggatt8280tcggacagtcaaactgggcatcaacgcagtggtgggctgcttcgtttgctgcgtgttgta8340cttgtttgcatttgaaccccgcggtcgttcgagtccttaattggtccgctcccggtcaac8400acccaagcagctgtggcccggccgagtggcgcctgtctggtccacagtaagcttggcgta8460atcatggtcatagctgtttcctgtgtgaaattgttatccgctcacaattccacacaacat8520acgagccggaagcataaagtgtaaagcctggggtgcctaatgagtgagctaactcacatt8580aattgcgttgcgctcactgcccgctttccagtcgggaaacctgtcgtgccagctgcatta8640atgaatcggccaacgcgcggggagaggcggtttgcgtattgggcgctcttccgcttcctc8700gctcactgactcgctgcgctcggtcgttcggctgcggcgagcggtatcagctcactcaaa8760ggcggtaatacggttatccacagaatcaggggataacgcaggaaagaacatgtgagcaaa8820aggccagcaaaaggccaggaaccgtaaaaaggccgcgttgctggcgtttttccataggct8880ccgcccccctgacgagcatcacaaaaatcgacgctcaagtcagaggtggcgaaacccgac8940aggactataaagataccaggcgtttccccctggaagctccctcgtgcgctctcctgttcc9000gaccctgccgcttaccggatacctgtccgcctttttcccttcgggaagcgtggcgctttc9060tcatagctcacgctgtaggtatctcagttcggtgtaggtcgttcgctccaagctgggctg9120tgtgcacgaaccccccgttcagcccgaccgctgcgccttatccggtaactatcgtcttga9180gtccaacccggtaagacacgacttatcgccactggcagcagccactggtaacaggattag9240cagagcgaggtatgtaggcggtgctacagagttcttgaagtggtggcctaactacggcta9300cactagaagaacagtatttggtatctgcgctctgctgaagccagttaccttcggaaaaag9360agttggtagctcttgatccggcaaacaaaccaccgctggtagcggtggtttttttgtttg9420caagcagcagattacgcgcagaaaaaaaggatctcaagaagatcctttgatcttttctac9480ggggtctgacgctcagtggaacgaaaactcacgttaagggattttggtcatgagattatc9540aaaaaggatcttcacctagatccttttaaattaaaaatgaagttttaaatcaatctaaag9600tatatatgagtaaacttggtctgacagttaccaatgcttaatcagtgaggcacctatctc9660agcgatctgtctatttcgttcatccatagttgcctgactccccgtcgtgtagataactac9720gatacgggagggcttaccatctggccccagtgctgcaatgataccgcgagacccacgctc9780accggctccagatttatcagcaataaaccagccagccggaagggccgagcgcagaagtgg9840tcctgcaactttatccgcctccatccagtctattaattgttgccgggaagctagagtaag9900tagttcgccagttaatagtttgcgcaacgttgttgccattgctacaggcatcgtggtgtc9960acgctcgtcgtttggtatggcttcattcagctccggttcccaacgatcaaggcgagttac10020atgatcccccatgttgtgcaaaaaagcggttagctccttcggtcctccgatcgttgtcag10080aagtaagttggccgcagtgttatcactcatggttatggcagcactgcataattctcttac10140tgtcatgccatccgtaagatgcttttctgtgactggtgagtactcaaccaagtcattctg10200agaatagtgtatgcggcgaccgagttgctcttgcccggcgtcaatacgggataataccgc10260gccacatagcagaactttaaaagtgctcatcattggaaaacgttcttcggggcgaaaact10320ctcaaggatcttaccgctgttgagatccagttcgatgtaacccactcgtgcacccaactg10380atcttcagcatcttttactttcaccagcgtttctgggtgagcaaaaacaggaaggcaaaa10440tgccgcaaaaaagggaataagggcgacacggaaatgttgaatactcatactcttcctttt10500tcaatattattgaagcatttatcagggttattgtctcatgagcggatacatatttgaatg10560tatttagaaaaataaacaaataggggttccgcgcacatttccccgaaaagtgccacctga10620cgtctaagaaaccattattatcatgacattaacctataaaaataggcgtatcacgaggcc10680ctttcgtctcgcgcgtttcggtgatgacggtgaaaacctctgacacatgcagctcccgga10740gacggtcacagcttgtctgtaagcggatgccgggagcagacaagcccgtcagggcgcgtc10800agcgggtgttggcgggtgtcggggctggcttaactatgcggcatcagagcagattgtact10860gagagtgcaccatatgcggtgtgaaataccgcacagatgcgtaaggagaaaataccgcat10920caggcgccattcgccattcaggctgcgcaactgttgggaagggcgatcggtgcgggcctc10980ttcgctattacgccagctggcgaaagggggatgtgctgcaaggcgattaagttgggtaac11040gccagggttttcccagtcacgacgttgtaaaacgacggccagtgaattcgagctcggtac11100c11101<210>65<211>50<212>DNA<213>人工序列<220><223>人工DNA引物<400>65accgggaggaaggctggaaagcttacgagaaaagagttggactttgaggg50<210>66<211>40<212>DNA<213>人工序列<220><223>人工DNA引物<400>66tgagcgaggaagcggaagagcgcccaatacgcaaaccgcc40<210>67<211>40<212>DNA<213>人工序列<220><223>人工DNA引物<400>67tgcgtattgggcgctcttccgcttcctcgctcactgactc40<210>68<211>68<212>DNA<213>人工序列<220><223>人工DNA引物<400>68tatactttctagagaataggaactcggaataggaacttcaaggaacaacactcaacccta60tctcggtc68<210>69<211>67<212>DNA<213>人工序列<220><223>人工DNA引物<400>69tccgagttcctattctctagaaagtataggaacttcgcatttatcagggttattgtctca60tgagcgg67<210>70<211>41<212>DNA<213>人工序列<220><223>人工DNA引物<400>70tctagatctcgagtcagatgtccctatcgcgtgtacactcg41<210>71<211>45<212>DNA<213>人工序列<220><223>人工DNA引物<400>71acacgcgatagggacatctgactcgagatctagagggtgactgac45<210>72<211>40<212>DNA<213>人工序列<220><223>人工DNA引物<400>72aactcttttctcgtaagctttccagccttcctcccggtac40当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1