一种基于Cpf1的DNA体外拼接方法与流程
本发明属于生物技术领域,具体地,本发明涉及一种基于cpf1的dna体外拼接方法。本发明方法可以产生预定粘端,并可用于体外dna无缝拼接。
背景技术:
2010年,j.craigventer实验室完成了蕈状支原体“synthia”的人工构建,掀起了一场合成生物学研究风暴。相对于传统的限制性内切酶酶切-连接酶连接克隆方法,无缝拼接因其不引入额外序列,具有设计方便,兼容性好等多个优点,在合成生物发展过程发挥越来越重要的作用。
目前已有的无缝拼接技术主要包括基于typeiis限制性内切酶的拼接方法、基于特定碱基修饰的拼接方法和基于同源序列的拼接方法,这些方法的存在极大的方便了dna的拼接组装,但同时它们都有各自的局限,有待于进一步的改进。goldengate利用typeiis限制性内切酶切割位点位于识别位点的外部的特点,通过人为设计不同的粘端实现无缝的体外拼接,对于短片段的体外拼接,尤其是短的重复序列拼接非常有效,但在大片段的拼接过程中由于片段内部的酶切位点的限制而使其应用受到限制。userfusion克隆方法和master(methylation-assistedtailorableendsrational)ligation克隆方法很好的避开了goldengate的这一限制,但这两种方法在使用过程中因为要合成是含有尿嘧啶或者含有甲基化的引物,因此成本较高。slic(sequenceandligation-independentcloning)是一个方便高效的体外拼接方法,因为其仅通过片段两端的同源序列,通过3'-5'外切酶的消化或者特定的化学修饰而形成同源的粘端,不涉及酶切连接过程,因此在设计的时候非常方便,但slic克隆在拼接5个以上的片段以及10kb以上片段时要求同源片段长度较长(大于40bp),而且效率会急剧下降。gibsonassembly拼接方法在slic的基础上进行改造,在体系中引入5'-3'聚合酶以及连接酶,因此拼接的效率提高了很多,可以拼接达100kb以上的片段。基于酵母重组的酵母体内拼接可以进一步提高拼接片段大小的上限,但与slic、gibson拼接一样,它们都是基于同源序列的拼接,对于存在重复序列的片段拼接无能为力。
crispr系统是原核生物中与其获得性免疫相关的防御系统,因其可以被改造为基因组编辑以及相关应用而被人们广泛关注[1-3]。cpf1是typeⅴ类的一员,其在对应的crrna介导下切割双链dna,在双链dna的18位和23位切割dna,形成5'端突出的粘端[4]。
综上所述,本领域需要提供一种高通用性、高特异性、高效地进行体外核酸拼接方法。
技术实现要素:
本发明的目的在于提供一种高通用性、高特异性、高效地进行体外核酸拼接方法。
本发明另一目的在于提供一种在体外简便快速的预定粘端生成的方法。
在本发明的第一方面,提供了一种核酸构建物(或核酸构建物组合),所述的核酸构建物包括:
(a)第一核酸构建物,所述第一核酸构建物为双链dna构建物,并且所述第一核酸构建物的序列中含有至少一个式i所示的cpf1识别切割元件;
d1-d2-d3(i)
式中,
d1为前间区序列邻近基序pam;
d2为长度n2个核苷酸的cpf1识别区,其中,n2为正整数14、15、16或17;
d3为长度为n3个核苷酸的cpf1切割区,其中,n3为4-10的正整数;
以及
(b)第二核酸构建物,所述第二核酸构建物为rna构建物,并且所述第二核酸构建物为结构如式ii所示的crrna元件;
r1-r2-r3(ii)
式中,
r1为5'的发夹区;
r2为长度m2个核苷酸的、与d2互补的cpf1识别引导区,其中m2为正整数14、15、16或17;
r3为无、或长度为m3个核苷酸的切割定位区,其中m3为1-20的正整数;
并且,所述的d3的序列与所述r3的序列是不匹配的。
在另一优选例中,所述的“不匹配”指,沿5'-3'方向,d3序列的第一碱基与r3的序列的第一碱基是不互补的。
在另一优选例中,所述的“不匹配”指,沿5'-3'方向,d3序列的前p个碱基与r3的序列的前p个碱基是不互补的,其中p为2、3、4、5、6或7。
在另一优选例中,所述的核酸构建物,所述的核酸构建物还包括:
(c)第三核酸构建物,所述第三核酸构建物为dna构建物,并且所述第三核酸构建物的序列中含有至少一个式iii所示的cpf1识别切割元件;
e1-e2-e3(iii)
式中,
e1为前间区序列邻近基序pam;
e2为长度n2个核苷酸的cpf1识别区,其中,n2为正整数14、15、16或17;
e3为长度为n3个核苷酸的cpf1切割区,其中,n3为4-10的正整数。
在另一优选例中,所述的核酸构建物,所述的构建物还包括:
(d)第四核酸构建物,所述第四核酸构建物为rna构建物,并且所述第四核酸构建物为结构如式iv所示的crrna元件;
s1-s2-s3(iv)
式中,
s1为5'的发夹区;
s2为长度m2个核苷酸的、与e2互补的cpf1识别引导区,其中m2为正整数14、15、16或17;
s3为无、或长度为m3个核苷酸的切割定位区,其中m3为1-20的正整数;
并且,所述的e3的序列与所述s3的序列是不匹配的。
在另一优选例中,所述的“不匹配”指,沿5'-3'方向,e3序列的第一碱基与s3的序列的第一碱基是不互补的。
在另一优选例中,所述的“不匹配”指,沿5'-3'方向,e3序列的前p个碱基与s3的序列的前p个碱基是不互补的,其中p为2、3、4、5、6或7。
在另一优选例中,所述的核酸构建物,所述的式i所示的cpf1识别切割元件与式iii所示的cpf1识别切割元件是相同的;和/或
所述的如式ii所示的crrna元件与式iv所示的crrna元件是相同的。
在另一优选例中,所述的核酸构建物,所述的第一核酸构建物中含有2个或多个式i所示的cpf1识别切割元件;和/或
所述的第三核酸构建物中含有2个或多个式iii所示的cpf1识别切割元件。
在另一优选例中,所述的第三核酸构建物中含有2个或多个式i所示的cpf1识别切割元件。
在另一优选例中,所述的第一核酸构建物包括表达载体、核酸片段、质粒、染色体片段。
在另一优选例中,所述的第三核酸构建物包括核酸片段、质粒。
在另一优选例中,所述的第一核酸构建物包括一种或多种第一核酸构建物。
在另一优选例中,所述的第三核酸构建物包括一种或多种第三核酸构建物。
在另一优选例中,所述的r1的长度为m1个核苷酸,m1为20-32的正整数。
在另一优选例中,n2=m2。
在另一优选例中,n2为15、16或17。
在另一优选例中,n2为16或17。
在另一优选例中,n2为17。
在另一优选例中,n3为5-8,较佳地为5-6,更佳地为5。
在另一优选例中,所述的式i和式ii的结构为5'至3'的结构。
在本发明的第二方面,提供了一种反应体系,包括:
(i)本发明第一方面中所述的核酸构建物;和
(ii)cpf1酶。
在另一优选例中,所述的反应体系为液态。
在另一优选例中,所述的反应体系还包括一种或多种选自下组的组分:
(c1)缓冲液;
(c2)taqdna连接酶;
(c3)去磷酸酶fastap。
在本发明的第三方面,提供了一种试剂组合,包括:
(i)本发明第一方面所述的核酸构建物;和
(ii)cpf1酶。
在另一优选例中,在所述的试剂组合中,所述的组分(i)和(ii)是独立的或混合在一起的。
在另一优选例中,在所述的试剂组合中,所述的第一核酸构建物、第二核酸构建物、任选的第三核酸构建物、以及任选的第四核酸构建物是独立的、部分混合、或全部混合的。
在本发明的第四方面,提供了一种试剂盒,所述的试剂盒包括:
(h1)任选的第a1容器,以及位于第a1容器的第一核酸构建物,所述第一核酸构建物为dna构建物,并且所述第一核酸构建物的序列中含有至少一个式i所示的cpf1识别切割元件;
d1-d2-d3(i)
式中,
d1为前间区序列邻近基序pam;
d2为长度n2个核苷酸的cpf1识别区,其中,n2为正整数14、15、16或17;
d3为长度为n3个核苷酸的cpf1切割区,其中,n3为4-10的正整数;
(h2)第a2容器,以及位于所述第a2容器的第二核酸构建物,所述第二核酸构建物为rna构建物,并且所述第二核酸构建物为结构如式ii所示的crrna元件;
r1-r2-r3(ii)
式中,
r1为5'的发夹区;
r2为长度m2个核苷酸的、与d2互补的cpf1识别引导区,其中m2为正整数14、15、16或17;
r3为无、或长度为m3个核苷酸的切割定位区,其中m3为1-20的正整数;
并且,所述的d3的序列与所述r3的序列是不匹配的;
(h3)第b1容器,以及位于所述第b1容器的cpf1酶。
在另一优选例中,所述的试剂盒还包括:
(h4)第a3容器,以及位于所述第a3容器的第三核酸构建物;
(h5)第a4容器,以及位于所述第a4容器的第四核酸构建物。
在另一优选例中,所述的试剂盒还包括选自下组的一种或多种组分:
(h6)用于酶切反应的试剂;
(h7)用于连接反应的试剂;
(h8)缓冲组分;
(h9)使用说明书。
在本发明的第五方面,提供了一种体外的、用于产生预定的粘性末端的酶切方法,包括步骤:
(i)提供一反应体系,所述反应体系中含有本发明第一方面所述的核酸构建物以及cpf1酶;和
(ii)在所述第二核酸构建物的引导下,用所述的cpf1酶对所述第一核酸构建物中的式i所示的cpf1识别切割元件进行切割,从而产生具有预定的粘性末端的酶切产物。
在另一优选例中,所述的预定的粘性末端为5bp-8bp的粘性末端。
在本发明的第六方面,提供了一种体外的、核酸拼接方法,包括步骤:
(a)在所述第二核酸构建物的引导下,用cpf1酶对所述第一核酸构建物中的式i所示的cpf1识别切割元件进行切割,从而产生具有预定的第一粘性末端的第一酶切产物;其中所述的第一核酸构建物和第二核酸构建物如上所述;
以及提供一待拼接的核酸拼接元件,所述核酸拼接元件具有第二粘性末端,所述的第一粘性末端和第二粘性末端是互补的;
和(b)对所述的第一酶切产物和所述的待拼接的核酸拼接元件,通过所述的第一粘性末端和第二粘性末端进行粘性末端连接,从而形成经拼接的核酸产物。
在另一优选例中,所述的方法是非诊断性和非治疗性的。
在另一优选例中,所述的待拼接的核酸拼接元件是用以下方法制备的:
在所述第四核酸构建物的引导下,用cpf1酶对所述第三核酸构建物中的式iii所示的cpf1识别切割元件进行切割,从而产生具有预定的第二粘性末端的第二酶切产物,作为待拼接的核酸拼接元件;
其中,所述的第三核酸构建物以及第四核酸构建物如上所述。
应理解,在本发明范围内中,本发明的上述各技术特征和在下文(如实施例)中具体描述的各技术特征之间都可以互相组合,从而构成新的或优选的技术方案。限于篇幅,在此不再一一累述。
附图说明
图1显示了本发明一个实例中,利用cpf1进行一步法无缝拼接的示意图。其中载体(vector)与外源插入片段(insert)由pcr扩增制得,通过引物引入cpf1识别位点(17bp)和切割位点(5bp),crrna序列包括5'端发卡结构和3'端与靶标dna互补配对的17-nt识别序列和7-nt非互补的辅助序列。cpf1在包含17-nt配对区加7-nt辅助序列的crrna介导下,以一定地比例在靶标dna的17位和22位进行切割(pam下游第一个碱基定义为1位,下同),形成5'突出粘性末端,连接酶将互补配对的粘性末端连接成一条完整的dna片段,反应时在同一ep管中加入dna片段、cpf1-crrna复合体、连接酶30℃反应1h,65℃灭活后直接转化dh10b。
图2显示克隆阳性率验证结果,包括菌落pcr和sanger测序验证。阳性克隆扩增条带约1.7kb。
图3显示利用cpf1进行放线紫红素表达载体中调控基因actii-orf4启动子元件无缝替换示意图。在放线紫红素表达载体中,蓝色方框actii-orf4启动子元件,紫色方框和棕色方框分别表示其上下游序列,红线表示离actii-orf4启动子元件最近的pam,橙色方框表示红霉素启动子。crrna2、crrna3分别包含5'端发卡结构和3'端与e1元件上下游序列配对的17-bp序列。cpf1在17-nt配对的crrna介导下在靶标dna的14位和22位进行切割,形成5'突出粘性末端,通过taqdna连接酶催化的连接反应可以将具有相同粘性末端的dna产物连接在一起(a)。
图4显示了通过cpf1将放线紫红素生物合成基因簇中途径特异性调控因子actⅱ-orf4启动子(orf4p)替换成组成型表达的红霉素启动子(emp)。将替换前和替换后的放线紫红素生物合成基因簇接合转移至高温链霉菌4f,比较它们在30℃培养温度下放线紫红素产量。
具体实施方式
本发明人经过广泛而深入的研究,通过大量筛选,首次开发了一种可基于cpf1的、高通用性、高特异性、并对识别位点与切割位点进行有效分开的体外核酸切割和拼接方法。具体地,基于本发明方法,不仅可以使得产生例如长度为5-8nt的预定序列的粘性末端,从而提供后续拼接的特异性和效率,并且可以有效解决长片段(如≥1kb、≥2kb、或≥5kb)dna片段在切割和拼接时缺乏合适的特异性切割位点的问题,因此特别适用于长片段核酸(如dna分子)的切割和拼接。在此基础上完成了本发明。
具体地,本发明人通过对不同长度crrna介导cpf1切割靶标dna的特点进行系统的改造,并通过改造设计特定crrna,采用特定长度的核苷酸的cpf1识别区,从而使得cpf1的一个或两个切割位点能够移动到cpf1识别区之外,从而显著增加了这类crrna介导cpf1切割反应的灵活性。在此基础上,还开发了一种体外dna无缝拼接的技术方案。实验结果表明,采用上述技术方案成功地完成了阿泊拉抗性基因(apr)无缝拼接和放线紫红素生物合成基因簇中途径特异性调控因子actⅱ-orf4启动子的替换。
术语
术语“crispr”是指成簇的、规律间隔的短回文重复序列(clusteredregularlyinterspacedshortpalindromicrepeats),与原核生物的获得性免疫有关。
术语“crrna”是指crisprrna,是短的引导cpf1到靶向dna序列的rna。
术语“元件”是指具有一定结构特征的生物模块,是构成生命体复杂生物活性的基本组成单元。
术语“pam”是指前间区序列邻近基序(protospacer-adjacentmotif),是cpf1切割所必须,fncpf1的pam为ttn序列。
如本文所用,术语“大片段dna”指大于5kb的环形质粒或者线性片段。
cpf1酶
术语“cpf1”是指crrna依赖的内切酶,它是crispr系统分类中v型(typev)的酶。cpf1是typeⅴ类的一员,其在对应的crrna介导下切割双链dna,在双链dna的18位和23位切割dna,形成5'端突出的粘端。
在本发明中,所述的cpf1酶可以是野生型的或突变型的。此外,可以分离的,也可以是重组的。此外,可用于本发明的cpf1酶可以来自不同物种。一种代表性的优选的本发明中的cpf1为土拉弗朗西斯菌(francisellatularensis)的cpf1。
一种典型的fncpf1的氨基酸序列如seqidno.:1所示。
研究表明,通常cpf1酶可以在24-ntcrrna介导下切割双链靶标dna的18位与24位(两个切割位点均位于crrna介导的识别区内),形成5-nt粘性末端。然而,采用本发明特定结构的如式ii所示的crrna元件时,出乎意料地,cpf1酶不仅仍可保留特异性地切割活性,而且可以形成至少一个位于所述crrna介导的识别区内之外的切割位点。显然,当一个或两个(优选两个)切割位点位于识别区内之外时,可以极大地提高本发明所述方法的通用性。
本发明的cpf1识别切割元件
在本发明中,“本发明的cpf1识别切割元件”指可以被cpf1特异性识别并特异性切割的核苷酸构建物。
典型地,本发明的cpf1识别切割元件包括:上述的第一核酸构建物、上述的第三核酸构建物、或其组合。
在本发明中,第一核酸构建物和第三核酸构建物可以是相同的,也可以是不同的。此外,虽然第一核酸构建物和第三核酸构建物的序列是不同的,但是它们可以具有相同的cpf1识别切割元件。或者,虽然第一核酸构建物和第三核酸构建物具有不同的cpf1识别切割元件,但是其经切割后可形成相同或互补的粘性末端。
典型地,所述的第一核酸构建物为双链dna构建物,并且所述第一核酸构建物的序列中含有至少一个式i所示的cpf1识别切割元件;
d1-d2-d3(i)
式中,
d1为前间区序列邻近基序pam;
d2为长度n2个核苷酸的cpf1识别区,其中,n2为正整数14、15、16或17;
d3为长度为n3个核苷酸的cpf1切割区,其中,n3为4-10的正整数。
典型地,所述第三核酸构建物为dna构建物,并且所述第三核酸构建物的序列中含有至少一个式iii所示的cpf1识别切割元件;
e1-e2-e3(iii)
式中,
e1为前间区序列邻近基序pam;
e2为长度n2个核苷酸的cpf1识别区,其中,n2为正整数14、15、16或17;
e3为长度为n3个核苷酸的cpf1切割区,其中,n3为4-10的正整数。
在本发明中,d3和e3是cpf1切割区,这意味着,cpf1特异性切割所形成的两个切割位点中有至少一个(或优选地两个)位于所述的cpf1切割区。
在一个优选例中,所述的切割区包括二个位于识别区外的切割位点,例如切割位点位于第17和22位(此时,两个切割位点相距5-nt),其中以cpf1识别区的第一个碱基为第1位。
在另一优选例中,所述切割区仅包括一个位于所述的d3和/或e3的切割位点,还包括另一个位于识别区内且靠近所述切割区的切割位点(此时,两个切割位点相距8-nt),例如切割位点位于第14位(位于识别区)和22位(位于切割区),其中以cpf1识别区的第一个碱基为第1位。
酶切方法
采用本发明所述的具有特定结构的核酸构建物(或其组合),通过第一核酸构建物中式i所示的cpf1识别切割元件和第二核酸构建物中式ii所示的crrna元件,可以利用cpf1酶,奇妙地形成至少一个位于所述识别区之外的切割位点,从而不仅克服了现有技术中的诸多不便,例如缺乏一定长度(≥10nt)的特异性识别区,难以形成≥4nt的粘性末端,切割位点全部位于识别区导致应用面狭窄,而且具有高通用性、高特异性等多种优点。例如,在本发明方法中,由于具有较长的识别序列(如17nt),而且识别序列可以根据需要任意设计,因此可以极其方便和有效地解决大片段拼接过程中片段内部存在识别位点的问题。
参见图1。在一个实施例中,当采用例如17nt配对+7nt不配对的crrna作为向导rna进行靶向切割dna时,易于形成二个位于识别区外的切割位点,例如切割位点位于第17和22位,其中以cpf1识别区的第一个碱基为第1位。在这种情况下,两个切割位点相距5-nt。
在一个优选实施例中,本发明人利用fncpf1,以质粒或其他大片段dna为底物,在17-nt配对(即n2为17)加上7-nt辅助序列(未配对)(即n3为7)的crrna介导下,从而高效地切割靶标dna(第一核苷酸构建物)的17位与22位。
在另一实施例中,当采用例如17-nt配对的crrna作为向导rna进行靶向切割dna时,易于形成一个位于所述的d3和/或e3的切割位点,以及另一个位于识别区内的切割位点,例如切割位点位于第14位(位于识别区)和22位(位于切割区),其中以cpf1识别区的第一个碱基为第1位。在这种情况下,两个切割位点相距8-nt。
拼接方法
本发明不仅提供了一种有效的高特异性地对大片段核酸进行酶切的方法,还提供了基于所述酶切方法的拼接方法。
在本发明中,由于可将cpf1的识别位点与切割位点分开,因此可以在进行体外实现dna无缝拼接。
一种典型的方法是基于本发明所述的cpf1酶切,以及常规的连接方法(如t4dna连接酶或者taq连接酶连接的方法)的组合。
本发明还提供了通过上述酶切和连接方法,实现大片段中元件的无缝替换。在一个实施例中,通过fncpf1在17-nt配对的crrna介导下,对第一核酸构建物进行特异切割(如位于14位与22位),形成8-nt长粘端。同样,可以对第三核酸构建物进行特异切割(如位于14位与22位),也形成8-nt长粘端。当两个长粘端互补时,可以方便地对经切割的第一核酸构建物和第三核酸构建物进行连接反应。
通过合理设计替换元件的两端序列,可以实现大片段dna的无缝替换。随着合成生物学元件库的丰富和合成通路不断构建,更换元件库中各元件以达到最佳合成效果成为合成生物学的发展趋势,因此本发明中涉及的大片段元件无缝替换在合成生物学中具有巨大的应用前景。
本发明的主要优点在于:
(1)本发明中的cpf1核酸酶,相对于传统的限制性内切酶,其识别序列要长很多(17bp),而且cpf1切割需要pam(ttn),这两个因素结合一起大大降低了目标序列中存在识别位点的可能性,此外cpf1的识别序列可以根据需要任意调整,进一步增加了此方法的可操作性。
(2)本发明中的拼接方法均为无缝拼接,dna拼接过程不会引入额外的不需要序列,设计方便,兼容性好。
(3)本发明中的大片段元件替换只需要简单的酶切连接,操作简便,设计简单,应用性强。
下面结合具体实施例,进一步阐述本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。下列实施例中未注明具体条件的实验方法,通常按照常规条件如sambrook等人,分子克隆:实验室手册(newyork:coldspringharborlaboratorypress,1989)中所述的条件,或按照制造厂商所建议的条件。除非另外说明,否则百分比和份数按重量计算。本发明中所涉及的实验材料如无特殊说明均可从市售渠道获得。
材料
1.大肠杆菌dh10b菌株购自takara公司;dna限制性内切酶,t4dna连接酶,t4polynucleotidekinase,等购自newenglandbiolabs公司;fastap,t7rna聚合酶购自thermo公司;
2.培养基配方:液体lb(1%tryptone,0.5%yeastextract,1%nacl),配置固体lb时,只需要在液体lb中添加1%的琼脂即可。
序列
实施例中涉及的序列如下:
>fncpf1(seqidno.1)
>puc18(seqidno.2)
>apr(seqidno.3)
>crrna1(seqidno.4)
aauuucuacuguuguagaugagaagucauuuaauaacugaacu
>crrna2(seqidno.5)
aauuucuacuguuguagaugagaagucauuuaauaa
>crrna3(seqidno.6)
aauuucuacuguuguagauaacuuauugggacgugu
>actii-orf4p(seqidno.7)
>emp(seqidno.8)
>phiw(seqidno.9)
>peasy-blunt(seqidno.10)
>crrna3(seqidno.11)
aauuucuacuguuguagauaucagugcagcgagcug
>crrna4(seqidno.12)
aauuucuacuguuguagauaacuuauugggacgugu
>puc18-cf(seqidno.13)
ggatcccgggatcctttcgagaagtcatttaataagccaccgggtaccgagctcgaattcgtaatc
>puc18-cr(seqidno.14)
ggatcccgggatcctttcgagaagtcatttaataacgatggggatcctctagagtcgacctgcag
>apr-cf(seqidno.15)
ggatcccgggatcctttcgagaagtcatttaataacatcgtgatgccgtatttgcagtaccagcg
>apr-cr(seqidno.16)
ggatcccgggatcctttcgagaagtcatttaataagtggcagctatttacccgcaggacatatcc
>emp-pf(seqidno.17)
cagtgcagctcgctgcactgattaaagcccgacccgagcacgcgc
>emp-pr(seqidno.18)
catggacacgtcccaataagttgaatctcaccgctggatcctaccaaccggc
实施例1阿泊拉抗性基因无缝拼接克隆至puc18中。
1)以pbc-am作为模板,用引物apr-cf(seqidno.14)和apr-cr(seqidno.15)pcr扩增阿泊拉抗性基因apr(seqidno.3),并通过引物在两端引入17-bpcpf1识别序列和pam(ttn)。
2)以puc18(seqidno.2)作为模板,用引物puc18-cf(seqidno.12)和puc18-cr(seqidno.13)pcr扩增线性载体,并通过引物在两端引入17-bpcpf1识别序列和pam(ttn)以及与apr(seqidno.3)产生相同粘性末端的5-bp接头序列。
3)回收纯化上述pcr产物,并以等摩尔比混合在一起,加入fncpf1(seqidno.1)、crrna1(seqidno.4)、t4dna连接酶,30℃反应1h。
4)上述反应产物65℃灭活20min,立即插冰上5min,转化dh10b。
5)通过菌落pcr、sanger法进行测序验证克隆正确率。
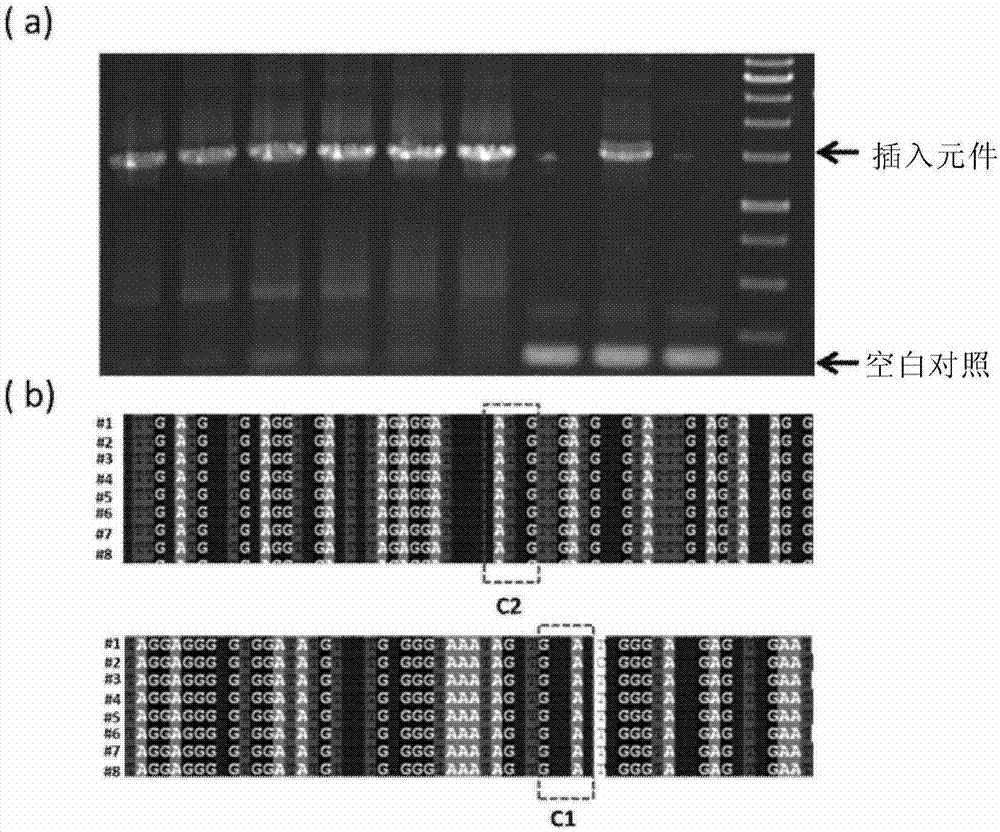
图2显示阿泊拉抗性基因无缝拼接的菌落pcr以及sanger测序验证结果。
实施例2放线紫红素生物合成基因簇中途径特异性调控因子actⅱ-orf4启动子替换为组成型表达的红霉素启动子。
如图3所示,通过17-ntcrrna1和crrna2的介导,fncpf1分别在hiw质粒和peasy-emp质粒上形成两端适配的粘性末端,通过taqdna连接酶将actⅱ-orf4(seqidno.7)启动子替换为红霉素启动子(seqidno.8)。具体实施方式如下:
1)以pbs-emp作为模板,用引物emp-pf(seqidno.17)和emp-pr(seqidno.18)pcr扩增得到红霉素启动子(emp)(seqidno.8),并克隆至peasy-blunt(seqidno.10)中得到亚克隆peasy-emp,并进行sanger测序验证。通过引物在两端引入pam(ttn)、17-bp的fncpf1识别序列以及待替换元件与识别序列之前的上下游同源序列。
2)放线紫红素表达质粒phiw(seqidno.9)与peasy-emp分别用crrna2(seqidno.5),crrna3(seqidno.6)介导fncpf1于37℃酶切30min,65℃灭活20min。
3)上述酶切后的phiw(seqidno.9)用fastap于37℃去磷30min,65℃灭活20min。
4)酶切完peasy-emp以及去磷后的phiw(seqidno.9)回收纯化,并以摩尔比10:1的比例用taqdna连接酶进行连接反应,分别于不同温度,不同反应时间测试连接效率。
5)连接完成后直接转化dh10b,通过菌落pcr以及sanger测序验证正确率。
表1:无缝替换阳性率统计
注:表中数字表示菌落pcr验证正确的克隆数/菌落pcr验证的总克隆数,括号里为平皿上得到的总克隆数,“-”表示没有测试。
表1显示actⅱ-orf4启动子替换克隆验证结果。最适反应条件45℃,10min,阳性率达到75%。。
图4显示替换红霉素启动子后质粒phiw-emp接合转移至高温链霉菌4f中的放线紫红素产量分析结果。替换启动子后放线紫红素产量提高两倍,并且放线紫红素生成时间明显提前。
在本发明提及的所有文献都在本申请中引用作为参考,就如同每一篇文献被单独引用作为参考那样。此外应理解,在阅读了本发明的上述讲授内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本申请所附权利要求书所限定的范围。
参考文献:
1.sampson,t.r.,etal.,acrispr/cassystemmediatesbacterialinnateimmuneevasionandvirulence.nature,2013.497(7448):p.254-7.
2.sternberg,s.h.,etal.,dnainterrogationbythecrisprrna-guidedendonucleasecas9.nature,2014.507(7490):p.62-7.
3.jinek,m.,etal.,aprogrammabledual-rna-guideddnaendonucleaseinadaptivebacterialimmunity.science,2012.337(6096):p.816-21.
4.zetsche,b.,etal.,cpf1isasinglerna-guidedendonucleaseofaclass2crispr-cassystem.cell,2015.163(3):p.759-771.
- 还没有人留言评论。精彩留言会获得点赞!