一种高效提取干燥植物DNA的方法与流程
本发明涉及一种植物基因组DNA的提取方法,具体涉及一种高效提取干燥植物DNA的方法。
技术背景
从植物组织中提取DNA是进行分子生物学方面研究的第一步。高质量的基因组DNA是进行如SSR、ISSR分子标记的基础(张立荣,2002)。迄今为止,人们提出了诸多提取DNA的方法,目前最常用的提取方法是SDS法、CTAB法(Murry MG,1980)。
远距离采样一般无法对材料即刻提取DNA,通常采用硅胶脱水干燥保存。有部分研究表明,硅胶脱水干燥的样品中DNA有不同程度的降解,其提取质量远不如鲜样中提取效果(陈亮,2002;王经源,2002)。尤其枯萎的植物样品中DNA易被核酸内源酶降解,硅胶干燥后DNA降解程度更加严重,提取质量下降。因此,亟待开发一种能够适用于临近枯萎植物干燥样品的基因组DNA的通用方法以适应研究要求。
技术实现要素:
本发明的目的在于提供一种高效提取干燥植物样品DNA的方法,该方法通用、高效、安全和简便。
实现本发明目的的技术方案如下:
一种高效提取干燥植物DNA的方法,包括如下步骤:
取干燥植物材料,粉碎,加入CTAB裂解缓冲液和一定体积的RnaseA,涡旋振荡;于65℃水浴30-50min,加入等体积的氯仿-异戊醇(24:1),颠倒混匀,离心,取上清,加入异丙醇沉淀DNA,离心,弃上清,用EB溶解沉淀,加入等体积的氯仿-异戊醇(24:1),颠倒混匀,离心,取上清;加入等体积的BB1,颠倒混匀,将混合液转移至干净离心柱,离心;弃液体,加入适量的CB1,静置一定时间,离心;弃液体,加入适量的WB1,离心;弃液体,再加入适量的WB1,离心;弃液体,离心,彻底去除残留的WB1;用预热至60℃-70℃的EB洗涤离心柱;离心,收集所得离心液,即得植物基因组DNA。
本发明所述干燥植物材料包括叶片、根、茎等,优选为叶片。
本发明所述干燥植物材料的含水率低于5%。
优选地,本发明所述干燥植物(材料)包括山莨菪(Anisodus tanguticus)、
桃儿七(Sinopodophyllum hexandrum(Royle)Ying)、四数獐牙菜(Swertia tetraptera Maxim.)、椭圆叶花锚(Halenia elliptica)。优选地,将粉碎后的植物材料置于无菌离心管总,再加入CTAB裂解缓冲液进行裂解。
本发明所述CTAB裂解缓冲液与本领域常规配方相同;具体如下:CTAB 20g/L,Nacl 1.4mol/L,EDTA 10mmol/L,Tris 100mmol/L,调节pH至8.0。
优选地,所述干燥植物材料与CTAB裂解缓冲液的质量体积比为30-50mg/1mL;进一步优选为30mg/1mL。
本发明在水浴反应过程中,每隔5-8min上下颠倒混合反应液。
本发明在采用异丙醇沉淀DNA时,是用预冷至-20℃的异丙醇;优选地,所述异丙醇的用量为混合液离心后上清液总体积的2/3。
本发明所述离心转速为10000-12000rpm,温度为4-15℃,离心时间为1-10min。
具体地,所述高效提取干燥植物DNA的方法,包括如下步骤:
(1)取粉碎的干燥植物材料30-50mg,加入已预热至65℃的CTAB裂解缓冲液1mL,再加入15ul RnaseA,于65℃水浴30min,每隔5min上下颠倒混匀;
(2)加入等体积的氯仿-异戊醇(24:1),轻颠倒混匀,15℃,12000rpm,离心10min;
(3)转移上清至干净的离心管中,加入等体积的氯仿-异戊醇(24:1),轻颠倒混匀,15℃,12000rpm,离心10min;
(4)转移上清至干净的离心管中,加入上清液2/3体积的预冷至-20℃的异丙醇,轻颠倒混匀,于-20℃静置30min;
(5)于15℃,12000rpm,离心10min,弃上清,加入400ul EB溶解沉淀,加入等体积氯仿-异戊醇(24:1),轻颠倒混匀,15℃,12000rpm,离心10min;
(6)转移上清至干净的离心管中,加入等体积BB1,轻颠倒混匀,将混合液转移至干净离心柱,15℃,12000rpm,离心5min;
(7)弃液体,加入500ul CB1,静置5min,15℃,12000rpm,离心5min;
(8)弃液体,加入500ul WB1,15℃,12000rpm,离心5min;重复本步骤至少一次;
(9)弃液体,15℃,12000rpm,离心5min,彻底去除残留的WB1;
(10)弃离心管,将离心柱放置在干净滤纸上风干5min;
(11)将离心柱转移到干净的离心管,向离心柱中心膜上加入40ul预热至60℃-70℃的EB,静置2min,15℃,12000rpm,离心2min;重复本步骤至少一次;
(12)弃离心柱,收集所得离心液,即得植物基因组DNA。
本发明所述EB、BB1,CB1、WB1、RnaseA均来自于北京全式金生物技术有限公司提供的DNA提取试剂盒(EasyPure Plant Genomic DNA Kit,目录号:EE111)。根据该试剂盒说明书的记载,EB、BB1,CB1、WB1分别为Elution Buffer,Binding Buffer 1,Clean Buffer 1,Wash Buffer 1。
与现有技术相对,本发明具有以下优点:
(1)利用本发明方法从干燥植物样品组织中获得的DNA量显著高于普通提取方法获得的DNA,且DNA纯度符合下游应用的要求。
(2)经PCR和酶切试验证明最终获得的DNA能够达到一般分子生物学研究的要求。
(3)本发明提取方法具有通用、高效和简便的特点。
附图说明
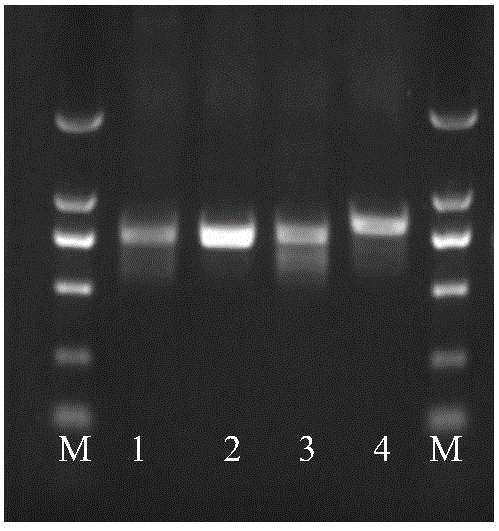
图1为本发明实施例1-4中提取的4种干燥植物叶片总DNA的电泳结果,图1中从左到右依次为:DNA maker(DL2000);1:山莨菪;2:桃儿七;3:四数獐牙菜;4:椭圆叶花锚。
图2为本发明实施例1-4中提取的4种干燥植物DNA PCR电泳结果,图2中从左到右依次为:DNA maker(DL2000);1:山莨菪;2:桃儿七;3:四数獐牙菜;4:椭圆叶花锚。
图3为本发明实施例1-4中提取的4种干燥植物DNA EcoRⅠ限制性酶切电泳结果,图3中从左到右依次为:DNA maker(Trans 2K plus II);1:山莨菪;2:桃儿七;3:四数獐牙菜;4:椭圆叶花锚。
图4为本发明实验例3-4中提取的4种干燥植物叶片总DNA的电泳效果。图4中从左到右依次为:DNA maker(Trans 2K plus II);1:山莨菪(CTAB法);2:桃儿七(CTAB法);3:四数獐牙菜(CTAB法);4:椭圆叶花锚(CTAB法);5:山莨菪(EE111);6:桃儿七(EE111);7:四数獐牙菜(EE111);8:椭圆叶花锚(EE111)。
具体实施方式
下面结合实施例,对本发明的具体实施方式做进一步详细描述,但并不局限于本发明,仅作示例说明。下列实施例中未注明具体实验的条件方法,通常按照常规条件,或者制造厂商所建议的条件。
本发明采用的氯仿、异戊醇和异丙醇优购于北京宏业图成科技有限公司。
本发明所述EB、BB1,CB1、WB1、RnaseA、RB1、BB1均来自于北京全式金生物技术有限公司提供的DNA提取试剂盒(EasyPure Plant Genomic DNA Kit,目录号:EE111)。
本发明中采用的缩略词语为:CTAB为溴化十六烷基三甲基铵(cetyltrimethylammonium bromide)、EDTA为乙二胺四乙酸(Ethylenediaminetetraacetic acid)
实施例1
本实施提供的山莨菪干燥叶片的DNA的提取过程如下:
(1)以干燥山莨菪叶片(含水率低于5%)为原料,采用植物组织研磨仪将叶片研磨成粉末,每个样品称取约30-50mg。余下的组织粉末保存于-80℃冰箱备用。
(2)称取30mg研磨后的样品,加入已预热至65℃的CTAB裂解缓冲液1mL,再加入15ul RnaseA,65℃水浴30min(每隔5min上下颠倒混匀)。
(3)加入等体积的氯仿-异戊醇(24:1),轻颠倒混匀,15℃,12000rpm,离心10min。
(4)转移上清至干净的离心管中,加入等体积的氯仿-异戊醇(24:1),轻颠倒混匀,15℃,12000rpm,离心10min。
(5)转移上清至干净的离心管中,加入上清液2/3体积的(-20℃预冷的)异丙醇,轻颠倒混匀,于-20℃静置30min。
(6)于15℃,12000rpm,离心10min,弃上清,加入400ul EB溶解沉淀,加入等体积氯仿-异戊醇(24:1),轻颠倒混匀,15℃,12000rpm,离心10min。
(7)转移上清至干净的离心管中,加入等体积BB1,轻颠倒混匀,将混合液转移至干净离心柱,15℃,12000rpm,离心5min。
(8)弃液体,加入500ul CB1,静置5min,15℃,12000rpm,离心5min。
(9)弃液体,加入500ul WB1,15℃,12000rpm,离心5min;重复本步骤一次。
(10)弃液体,15℃,12000rpm,离心5min,彻底去除残留的WB1。
(11)弃离心管,将离心柱放置在干净滤纸上风干5min。
(12)将离心柱转移到干净的离心管,向离心柱中心膜上加入40ul 60℃预热的EB,静置2min,15℃,12000rpm,离心2min,重复本步骤一次。
(13)弃离心柱,收集所得离心液,即得样品山莨菪基因组DNA。
实施例2
与实施例1的区别仅在于将植物原料替换为桃儿七的叶片(含水率低于5%),制得样品桃儿七基因组DNA。
实施例3
与实施例1的区别仅在于将植物原料替换为四数獐牙菜的叶片(含水率低于5%),制得样品四数獐牙菜基因组DNA。
实施例4
与实施例1的区别仅在于将植物原料替换为椭圆叶花锚的叶片(含水率为低于5%),制得样品椭圆叶花锚基因组DNA。
上述实施例1-4提取的干燥枯萎植物基因组DNA的产率和ISSR
实验例1本发明方法提取4种干燥植物样品叶片DNA质量比较
采用紫外分光光度计(Merinton,SMA4000)分别测量DNA溶液在230nm、260nm、280nm的光吸收峰度值。通过计算OD260/OD280和OD230/OD260比值分别获得DNA溶液中蛋白、多糖、酚类以及RNA的污染度,从而估算DNA浓度。公式为:DNA样品浓度(ug/ul)=OD260×稀释倍数×50
比较不同方法提取DNA的产率,琼脂糖凝胶电泳时每个样品上样3ulDNA溶液。1%琼脂糖(Invitrogen,California,美国),1×TBE(上海生工),GoldView(北京赛百盛基因技术有限公司)染色,100V,30min电泳,检测DNA。利用UPV紫外成像系统(EC3system,美国)扫描成像。
采用以上实施例1-4方法提取四种枯萎干燥植物叶片基因组DNA:山莨菪(Anisodus tanguticus)、桃儿七(Sinopodophyllum hexandrum(Royle)Ying)、四数獐牙菜(Swertia tetraptera Maxim.)、椭圆叶花锚(Halenia elliptica)。从下表1和图1可以看到,本发明方法能够从上述样品中成功提取出DNA,紫外分光光度计检测结果表明提取获得的DNA纯度符合要求。
表1本发明方法提取4种枯萎干燥植物叶片DNA的质量比较
实验例2本发明方法提取的4种植物DNA的PCR验证和酶切实验
DNA的提取是分子生物学研究中至关重要的第一步。因此,DNA提取方法的试用性还需要下游实验的验证。本发明中,通过PCR和酶切进一步验证本发明提取方法所获得的DNA的质量。
通过PCR反应验证本发明方法所获得的DNA的质量。方法如下:25μLPCR反应体系包含有:12.5μL 2×Taq PCR Master Mix(Transgene),上下游引物各0.5ul,2ul基因组DNA(浓度约为40ng/ul),9.5ul ddH2O。用于扩增ITS序列的一对通用引物的序列:ITS1(5’-AGAAGTCGTAACAAGGTTTC-3’)ITS4(5’-TCCTCCGCTTATTTATATGC-3’)PCR扩增程序为:,95℃,5min预变性;95℃,变性30s;52℃,退火45s;72℃,延伸1min40s;35个循环,72℃,终延伸10min;15℃,停止反应。
20ul酶切体系包括:1ug DNA模板,2ul 10×FlyCutTMBuffer,0.5ul EcoRⅠ酶(20unit/ul)(TransGene)。37℃,10min,65℃,20min。
利用ITS引物以本发明实施例1-4提取获得的上述4种植物基因组DNA为模板,进行PCR扩增,结果如图2所示。电泳结果显示从所有四个样品中都扩增出清晰的目的条带。PCR实验结果证明本发明提取方法获得的植物基因组DNA符合下游实验的要求。EcoRⅠ限制性酶切电泳结果如附图3所示,酶切获得的条带在凝胶上成均匀的弥散状分布,这表明本发明所获得的DNA纯度良好,符合酶切试验及下游实验的要求。
更为重要的是,本发明运用改良的CTAB法与全式金植物基因组DNA提取试剂盒相结合的方法,能够有效去除多酚和糖类物质对DNA提取的干扰,尤其适应于临近干燥或枯萎的植物干燥叶片,能够有效地富集DNA,并消除颜色对DNA的影响。PCR实验结果证明本发明提取方法获得的DNA能够达到下游实验的要求。
以上各实施例中采集的材料为临近枯萎的植物干燥叶片,另外,以上述植物或其他植物的根、茎为原料,采用上述本发明提取方法,同样可以获得符合要求的植物基因组DNA,此处不再一一举例说明。
实验例3
采用传统CTAB法提取山莨菪、桃儿七、四数獐牙菜以及椭圆叶花锚干燥叶片的DNA,山莨菪、桃儿七、四数獐牙菜以及椭圆叶花锚的干燥叶片分别与实施例1-4相同。
具体提取过程如下:
(1)分别以上述四种干燥植物叶片(含水率低于5%)为原料,采用植物组织研磨仪将叶片研磨成粉末。
(2)称取30mg研磨后的样品,加入已预热至65℃的CTAB裂解缓冲液1mL,20ulβ-巯基乙醇,15ul RnaseA,65℃水浴30min(每隔5min上下颠倒混匀)。
(3)加入等体积的酚-氯仿-异戊醇(25:24:1),轻颠倒混匀,12000rpm,15℃,10min
(4)转移上清至干净的离心管中,加入等体积的氯仿/异戊醇(24:1),轻颠倒混匀,12000rpm,15℃,10min
(5)转移上清至干净的离心管中,加入上清液2/3体积的(-20℃预冷的)异丙醇,轻颠倒混匀,-20℃,静置30min。
(6)于15℃,12000rpm,离心10min,弃上清,400ul去离子水溶解沉淀,加入等体积氯仿-异戊醇(24:1),轻颠倒混匀,12000rpm,15℃,离心10min。
(7)转移上清至干净的离心管中,加入0.1倍体积的NaAC,2.5倍体积的无水乙醇(-20℃预冷),上下颠倒,轻柔混匀,-20℃,沉降30min。
(8)12000rpm,15℃,离心10min,弃上清。
(9)75%乙醇漂洗沉淀,12000rpm,15℃,离心5min,弃上清。
(10)重复步骤(9)
(11)弃上清,将离心管放置超净台中吹干。
(12)50ul去离子水溶解沉淀,此溶液即为DNA,-20℃保存待用。
取3ul DNA溶液进行1%琼脂糖凝胶电泳检测,100V,30min。并用紫外分光光度计测定其浓度。结果见表2和图4。
实验例4
采用全式金植物基因组DNA提取试剂盒(目录号:EE111)提取山莨菪、桃儿七、四数獐牙菜以及椭圆叶花锚干燥叶片的DNA,山莨菪、桃儿七、四数獐牙菜以及椭圆叶花锚的干燥叶片分别与实施例1-4相同。
具体提取过程如下:
(1)分别以四种干燥植物叶片(含水率低于5%)为原料,采用植物组织研
磨仪将叶片研磨成粉末,每个样品称取约20mg。
(2)加入250μl溶液RB1,15μl RNase A充分混匀。
(3)55℃水浴孵育15min。
(4)15℃,12000rpm,离心5min。转移上清至新的离心管中。
(5)加入100ul溶液PB1,充分混匀,冰浴5min,15℃,12000rpm,离心5min。
(6)转移上清至一新的离心管中,加入375ul溶液BB1,充分混匀。
(7)吸取全部混合液体加入离心柱中,15℃,12000rpm,离心30s,弃液体。
(8)加入500ul溶液CB1,15℃,12000rpm,离心30s,弃液体。
(9)重复步骤(8)。
(10)加入500ul溶液WB1,15℃,12000rpm,离心30s,弃液体。
(11)重复步骤(10)。
(12)15℃,12000rpm,离心2min,彻底去除残留的WB1。
(13)将离心柱置于新的1.5ml离心管中,在柱的中央加入50ul EB缓冲液。室温静置1min,15℃,12000rpm,离心2min,洗脱DNA。
(13)为了得到浓度更高的DNA,进行第二次洗脱,将洗脱下来的DNA溶液重新上柱加入柱中央,室温静置1min,15℃,12000rpm,离心2min,洗脱DNA。
取3ul DNA溶液使用1%琼脂糖凝胶进行电泳检测,100V,30min。并用紫外分光光度计测定其浓度。结果见表2和图4。
从表2和图4可以看到,传统的CTAB方法以及全式金植物基因组DNA提取试剂盒(EE111)均不能从上述样品中成功提取出DNA,紫外分光光度计检测结果表明提取获得的DNA纯度较差,不符合下游实验要求。
表2 CTAB方法与EE111试剂盒提取4种枯萎干燥植物叶片DNA的质量比较
参考文献
1.张立荣,徐大庆,刘大群.SSR和ISSR分子标记及其在植物遗传育种研究中的应用[J].河北农业大学学报,2002,25(1):90-94.
2.Murray M G,Thompson W F.Rapid isolation of high molecular weight plant DNA[J].Nucleic acids research,1980,8(19):4321-4326.
3.陈亮.茶组植物的分子系统学研究[D].杭州:浙江大学博士论文,2002.
4.王经源,黄儒珠.用于DNA提取的刺桫椤叶片组织保存方法研究[J].福建师范大学学报:自然科学版,2002,18(1):82-85.
虽然,上文中已经用一般性说明及具体实施方案对本发明作了详尽的描述,但在本发明基础上,可以对之作一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
- 还没有人留言评论。精彩留言会获得点赞!