用于早期鉴别木瓜性别及性别相关性状的分子标记及其应用的制作方法
本发明是有关于一种分子标记及其应用,特别是有关于一种分子标记及其用于早期鉴别木瓜性别及性别相关性状的方法和系统。
背景技术:
番木瓜(Carica papayaL.)俗称木瓜,属番木瓜科(Caricaceae),番木瓜属(Carica)植物。番木瓜一年四季都能结果,其含丰富的营养价值,为热带地区的重要作物。番木瓜同时具有雌雄异株异花(dioecious),分别是雌株(Female,F)、雄株(Male,M)和雌雄同株同花的两性株(Hermaphrodite,H)。
一般而言,雄株不结果,不具经济价值。雌株需人工授粉才能结果。两性株可自花授粉,经济价值高,因此商业栽培者比较希望种植纯两性株。
番木瓜从生长至花蕾形成前,不论是叶型、茎干颜色、株型等各种外部型态,都尚未发现与植株性别连锁的外部特征,无法利用外部型态判断植株性别。农民为得到较具商业价值的两性株,需在每一植穴中种植2-3株番木瓜苗,等生长6-8个月待植株花蕾形成后,才能由花朵构造确认其性别,此时再选留一棵两性株,其余植株砍除,以达到全园生产长椭圆形的两性果。然而这样的栽培方式不仅造成番木瓜幼苗期的养分竞争与浪费,也造成栽培者大量的劳力支出,增加苗木费用与管理费用。此外,部分植穴可能因无栽植到两性株而造成缺株情形,不易控制正常株距。
为了生产同一性别的番木瓜种苗,番木瓜种苗业者经常实行无性繁殖法,如利用组织培养、扦插、嫁接等技术。在这些方式中,组织培养繁殖的商业生产,其繁殖倍数高且所有植株均为两性株是其优点,但是这种生产方式的技术门坎高,生产成本相对提高,致使组织培养苗的售价约为实生苗的7-15倍。嫁接苗因需要分别培养母穗及砧木,生产步骤多,需要较大的生产空间及加湿设备,且苗木生产速度较慢。至于扦插苗的繁殖倍率虽然较嫁接苗高,但是因为与组织培养苗同样没有种子根,部分农民认为其对逆境环境的忍受性较差而不喜欢种植。因此,对于多数农民而言,全两性株的种子苗被认为是较具发展价值的种苗生产方式。
近年来,生物科技相关技术进展迅速,目前分子标记已证明在生物学上可以进行早期检测,解决许多问题,具有极佳的应用价值。常见的分子标记检测种类可例如简单重复序列(Simple Sequence Repeat,SSR)、简单重复序列间多态性(Inter-Simple Sequence Repeats,ISSR)、随机扩增多态性DNA(Random Amplified Polymorphic DNA,RAPD)、扩增片段长度多态性(Amplified Fragment Length Polymorphism,AFLP)、限制性片段长度多态性(Restriction Fragment Length Polymorphism,RFLP)、特异性序列扩增区域(Sequence Characterized Amplified Region,SCAR)等。
举例而言,在过去研究中,上述SSR可用于区别石斛兰品种鉴定以及番木瓜的亲源鉴定等。ISSR可应用于番木瓜以及茄子种内鉴定分析。RAPD、AFLP及RFLP可应用于区分番木瓜性别。SCAR则是由RAPD衍生而来,可作为区分番木瓜性别的分子标记。
目前发展出一些分子标记,在番木瓜幼苗株无法以目视辨识性别的情况下,可用于鉴别性别。然而,目前大多数的木瓜性别相关基因的分子标记,单一种分子标记仅供鉴别单一种性别,无法在同时鉴别三种性别。其次,木瓜幼株在不同环境培养下,花性会不稳定,尤其两性株的花朵在不合适的环境或营养缺乏的情况下,甚至有退化、畸形的现象产生,影响性别相关性状,这也会影响鉴别性别及性别相关性状的准确率。
有鉴于此,亟需开发一种早期鉴别木瓜性别及性别相关性状的分子标记,以改善习知分子标记的种种缺点。
技术实现要素:
因此,本发明的一个方面是提供一种用于早期鉴别木瓜性别及性别相关性状的分子标记。
本发明的另一个方面是提供一种早期鉴别木瓜性别及性别相关性状的方法,其是由包含多个单核苷酸多态性位点、基因组序列及转录组序列的分子标记与多个性别相关基因建立相关性模式,以便在木瓜样本的花器发育早期,同时且准确鉴别其性别及性别相关性状。
本发明的又一个方面是提供一种提出一种早期鉴别木瓜性别及性别相关性状的系统,其包含上述分子标记。
本发明的再一个方面是提供一种重组载体,其包含上述的分子标记。
本发明又另一个方面是提供一种转基因植物细胞,其包含上述的重组载体。
根据本发明的上述方面,提出一种用于早期鉴别木瓜性别及性别相关性状的分子标记,包含至少一个序列,所述序列选自由SEQ ID NO:1~64以及与SEQ ID NO:1~51、53、55、57、59、61、63互补的序列所组成的群组。
根据本发明的另一个方面,提出一种早期鉴别木瓜性别及性别相关性状的方法,其是通过包含多个单核苷酸多态性位点、基因组序列及转录组序列的分子标记与多个性别相关基因建立相关性模式,以便在木瓜样本的花器发育早期,同时且准确鉴别其性别及性别相关性状。
在上述实施例中,建立相关性模式的步骤可包含但不限于由多个木瓜的多个花器获得多个核酸样本。接着,由所述核酸样本获得多个分子标记,其中所述分子标记包括至少一个序列,所述序列选自由SEQ ID NO:1~64以及与SEQ ID NO:1~51、53、55、57、59、61、63互补的序列所组成的群组。之后,定义所述性别相关基因的任一个与所述分子标记的至少一个的对应关系,以建立相关性模式。
在上述实施例中,所述获得核酸样本的步骤中,所述花器的平均长度不超过0.2cm,且所述核酸样本包含多个基因组序列以及多个转录组序列。
在上述实施例中,所述利用相关性模式鉴别木瓜样本的性别以及多个性别相关基因的步骤可包含但不限于由木瓜样本的待测花器获得待测核酸样本,由所述待测核酸样本获得多个性别分型数据,将所述性别分型数据输入所述相关性模式,以比对所述性别分型数据是否与所述分子标记的至少一个相符,并获得分子标记的至少一个的对应关系的相关正确率。当所述性别分型数据的至少一个与所述分子标记的至少一个相符,且相关正确率为100%时,判断此木瓜样本具有对应关系的性别以及所述性别相关基因的至少一个。
依据本发明的一个实施例,所述木瓜以及木瓜样本可包含但不限于番木瓜的佛罗里达种、日升种、吉隆坡种、日升13号种、日升23号种、日升与台农7号杂交种。
依据本发明的一个实施例,当上述第二性别分型数据包含SEQ ID NO:1~16、18、20~21、25~26、30~31、35~36、40~41、45~46、49~50、53~58、63~64以及与SEQ ID NO:1~16、18、20~21、25~26、30~31、35~36、40~41、45~46、49~50、53~58、63~64互补的序列的至少一个时,判断木瓜样本的性别为二性。
依据本发明的一个实施例,当上述第二性别分型数据包含SEQ ID NO:1~17、19、23~24、28~29、33~34、38~39、43~44、48、51~52、61~62以及与SEQ ID NO:1~17、19、23~24、28~29、33~34、38~39、43~44、48、51~52、61~62互补的序列的至少一个时,判断木瓜样本的性别为雄性。
依据本发明的一个实施例,当上述第二性别分型数据包含SEQ ID NO:1~16、22、27、32、37、42、47、59~60以及与SEQ ID NO:1~16、22、27、32、37、42、47、59~60互补的序列的至少一个时,判断木瓜样本的性别为雌性。
依据本发明的一个实施例,上述这些性别相关性状基因与多个性别相关性状相关,且这些性别相关性状包含多个花器型态及/或多个果实形状。
根据本发明的又一个方面,提出一种早期鉴别木瓜性别及性别相关性状的系统,其包含上述早期鉴别木瓜性别及性别相关性状的分子标记。
根据本发明的再一个方面,提供一种重组载体,其包含上述的分子标记。
根据本发明又另一个方面,提供一种转基因植物细胞,其包含上述的重组载体。
应用本发明的用于早期鉴别木瓜性别及性别相关性状的分子标记及其应用,其利用特定的分子标记与性别相关性状基因的任一个建立相关性模式,可于木瓜花器早期发育时,鉴别所述木瓜样本的性别以及性别相关性状,以有效、快速且准确获得木瓜两性株,并有助于减少不结实花及畸形花果的产生,进而多方应用,例如用于早期鉴别木瓜性别及性别相关性状的系统、制得包含上述的分子标记重组载体以及含此重组载体的转基因植物细胞等。
附图说明
为让本发明的上述和其他目的、特征、优点与实施例能更明显易懂,所附图式的详细说明如下:
(图1)为绘示根据本发明的一个实施例的早期鉴别木瓜性别及性别相关性状的方法的部分流程图。
(图2A)至(图2B)为绘示根据本发明的一个实施例的Yh染色体BAC71E16分析CpSVP-LIKE于番木瓜不同性型表现的cDNA序列(图2A)及内含子/外显子在BAC位置(图2B)的差异性。
(图3A)至(图3B)为绘示根据本发明的一个实施例的Yh染色体BAC71E16分析CpSERK于番木瓜不同性型表现的cDNA序列(图3A)及内含子/外显子在BAC位置(图3B)的差异性。
(图4A)至(图4E)为绘示根据本发明数个实施例以不同SNP基因型作为番木瓜性别及性别相关性状基因的分子标记的HRM分析结果,其中图4A为利用CpSVP-LIKE 88引物对的HRM分析结果,图4B为利用CpSERK 30704引物对的HRM分析结果,图4C为利用CpSERK 34072引物对的HRM分析结果,图4D为利用CpSERK 34760引物对的HRM分析结果,图4E为利用CpSERK 34787引物对的HRM分析结果,F代表雌性,M代表雄性,H代表两性。
(图5A)至(图5B)为绘示根据本发明数个实施例以不同SNP基因型作为番木瓜性别及性别相关性状基因的分子标记的PCR-RFLP分析结果,其中F代表雌性,M代表雄性,H代表两性,左边的M代表DNA标记(Marker),H代表两性木瓜株,FM代表佛罗里达种雄性木瓜株。
(图6)为绘示根据本发明的一个实施例的番木瓜性别及性别相关性状基因的分子标记的三性型基因连锁图及其表现模式,其中F代表雌性,M代表雄性,H代表两性。
具体实施方式
依照以前所述的,本发明提供一种用于早期鉴别木瓜性别及性别相关性状基因的分子标记及其应用,其利用包含多个特定的单核酸多态性位点、基因组序列及转录组序列的分子标记与性别相关性状基因建立相关性模式,以便于木瓜样本的花器发育早期,同时且准确鉴别木瓜样本的性别及性别相关性状。
展开来讲,本发明此处所称的木瓜以及木瓜样本,是指番木瓜,其品种并无特别限制,可包含但不限于番木瓜的佛罗里达种、日升种、吉隆坡种、日升13号种、日升23号种、日升与台农7号杂交种等。
本发明此处所称的木瓜性别,是指雄性、雌性及两性。依据Storey在1938年及1953年提出的理论,番木瓜性别(雄株、雌株及两性株)分别由3个等位基因控制:M1(雄,显性)、M2(两性,显性)及m(雌性,隐性),而显性等位基因(MM,MMh,MhMh)的所有组合都是胚致死。通常两性株自交,后代发生两性株及雌性株的比例为2:1,两性株与雌性株杂交后代发生两性株与雌性株的比例为1:1。
本发明此处所称的性别相关性状,是指其性别相关性状基因表现及/或性状表现与性别相关。在一个例示中,上述性别相关性状包含多个花器型态和/或多个果实形状或与上述性状相关的基因表现。
本发明此处所称的早期鉴别木瓜性别及性别相关性状的分子标记,包括至少一个序列,所述序列选自由SEQ ID NO:1~64以及与SEQ ID NO:1~51、53、55、57、59、61、63互补的序列所组成的群组。在一个实施例中,上述分子标记包含上述序列本身、或者上述序列衍生的产物且含有上述序列的序列,例如PCR产物、HRM的产物、RFLP的产物、由上述序列转录的RNA(或mRNA)、由上述序列转译的多肽等。
本发明此处所称的早期发育,是指木瓜花器的平均长度不超过0.2cm。一般而言,平均长度不超过0.2cm的花器无法以目视辨识性别,但使用本发明的分子标记可以有效、快速且准确区分番木瓜性别以及性别相关性状。
展开来讲,在一个实施例中,上述分子标记可用于早期鉴别木瓜性别及性别相关性状的方法,其通过分子标记与多个性别相关基因建立相关性模式,以在木瓜样本的花器发育早期,同时且准确鉴别其性别及性别相关性状。在此实施例中,上述分子标记可包含多个单核苷酸多态性位点、基因组序列、转录组序列及多肽的分子标记。
在一个例示中,本发明此处所称用于鉴别木瓜性别及性别相关性状的单核苷酸多态性(Single Nucleotide Polymorphism,SNP)位点,是指与性别具有显著相关性,并足够鉴别不同性别的SNP位点。前述位点可以座落于性别相关性状基因的外显子(exon)或内含子(intron),例如SEQ ID NO:17~51、53、55、57、59、61、63以及与SEQ ID NO:17~51、53、55、57、59、61、63互补的序列的至少一个。
上述用于鉴别木瓜性别及性别相关性状的基因组序列指与性别具有显著相关性,且为性别相关性状基因的外显子及/或内含子的部分或全部,例如SEQ ID NO:17~50以及与SEQ ID NO:17~50互补的序列的至少一个。
上述用于鉴别木瓜性别及性别相关性状的转录组序列,是指对应于上述性别相关性状基因所能转录出的所有RNA的总和,例如SEQIDNO:51、53、55、57、59、61、63以及与SEQ ID NO:51、53、55、57、59、61、63互补的序列的至少一个。在其他例子中,上述用于鉴别木瓜性别及性别相关性状的分子标记也可包含由上述转录组序列所转译的所有多肽的总和,例如SEQ ID NO:52、54、56、58、60、62、64的至少一个。
请参照表1,其是显示根据本发明的一个实施例的部分的分子标记、引物对序列及其座落的性别相关性状基因的比对表。
表1
举例而言,本发明适用的SNP分子标记可包含例如表1所列的SNP位点,包含CpSVP-LIKE基因内含子3第8511个核苷酸的SNP位点(SNP#88)、CpSERK基因内含子1第122453个核苷酸的SNP位点(SNP#30704)、CpSERK基因内含子7第119290、119295个核苷酸的SNP位点(SNPs#34072a、34072b)、CpSERK基因外显子9第118602个核苷酸的SNP位点(SNP#34760)以及CpSERK基因外显子9第118575个核苷酸的SNP位点(SNP#34787)。
在一些实施例中,上述分子标记可以是探针、引物对、基因组序列或转录组序列,其长度并无特别限制,只要涵盖上述SNP位点即可。然而在一些实施例中,当上述分子标记是探针或引物对时,其长度可例如15至30个碱基对、或如表1所列、抑或更短或更长,以便检测上述SNP位点的至少一个,进而同时鉴别性别及性别相关性状。
在其他实施例中,当上述分子标记是基因组序列或转录组序列本身时,其长度可例如200至400个碱基对,以检测上述SNP位点并检测其上下游约200至400个碱基对的序列,以便提高检测的准确性。
上述分子标记可应用于早期鉴别木瓜性别及性别相关性状的方法。请参照图1,其为绘示根据本发明的一个实施例的早期鉴别木瓜性别及性别相关性状的方法100的部分流程图。
在一个实施例中,此方法100可如步骤120所示,首先由多个木瓜的多个花器获得多个核酸样本,其中上述花器的平均长度不超过0.2cm,且所述核酸样本包含多个基因组序列以及多个转录组序列。接着,如步骤130所示,由核酸样本获得多个分子标记,以建立基因组数据库及转录组数据库,其中分子标记包括至少一个序列,所述序列选自由SEQ ID NO:1~64以及与SEQ ID NO:1~51、53、55、57、59、61、63互补的序列所组成的群组。之后,如步骤140所示,定义性别相关基因的任一个与分子标记的至少一个的对应关系,以建立相关性模式。
上述性别分型数据可包含单核苷酸多态性位点、基因组序列、转录组序列及多肽,其可利用例如高分辨率熔解分析(High Resolution Melting,HRM)、PCR-RFLP和/或常规解析氨基酸序列的方式获得。以HRM为例,HRM为近来DNA分子标记技术发展出的方式,通过高分辨率的熔解曲线,以分辨序列中单一核苷酸差异。由于HRM具有精确、快速、方便以及低成本等优点,过去常用于基因序列性状上差异性分析,例如桃树品种间性状差异性序列的品种差异性分析、西红柿诱变育种特定基因的扫描分析以及羽扇豆遗传连锁图谱分析等。在其他实施例中,本发明也可使用其他常规方式,例如聚合酶链反应-限制性片段长度多态性(PCR-RFLP)分析法和/或其他习知方法,取得性别分型数据,且其结果也可与前述HRM分析结果再核对。前述所述的建立基因组数据库及转录组数据库、HRM、PCR-RFLP和/或其他常规方法为参考常规方法建立,应为本发明所属技术领域中任何具有通常知识的人所熟知,此处不再赘述。
在一个实施例中,本发明此处所称的对应关系,是指利用常规商业统计软件,将所得的性别分型数据进行相关性分析,以定义性别相关基因的任一个与分子标记的至少一个的对应关系。
在建立相关性模式之后,在一个例示中,上述利用相关性模式鉴别木瓜样本的性别以及多个性别相关基因的步骤150可包括由木瓜样本的待测花器获得待测核酸样本,如步骤160所示。在一个例示中,上述花器的平均长度不超过0.2cm,且所述核酸样本也包含多个待测基因组序列以及多个待测转录组序列。
然后,如步骤170所示,由待测核酸样本获得多个性别分型数据,其中性别分型数据可包括例如多个待测单核苷酸多态性位点、待测单核苷酸多态性位点对应的待测基因组序列以及待测基因组序列对应的待测转录组序列。上述性别分型数据利用例如HRM分析法、PCR-RFLP分析法和/或其他常规方法获得,此处不再赘述。
接下来,如步骤180所示,将性别分型数据输入上述相关性模式,以比对性别分型数据是否与分子标记的至少一个相符,并获得分子标记的至少一个的对应关系的相关正确率。当性别分型数据的至少一个与分子标记的至少一个相符且相关正确率为100%时,判断木瓜样本具有对应关系的性别的一个以及性别相关基因的至少一个,如步骤190所示。
举例而言,当待测核酸样本利用HRM所得的性别分型数据,与上述相关性模式比对后,与分子标记中的SNP#88(T)相符,则判断木瓜样本为两性株,且具有对应CpSVP-LIKE基因的内含子(intron)3。CpSVP-LIKE基因在两性株中具有(Alternative Splicing,AS)的多态性,详见以后说明。
本发明的技术特征之一,在于定义前述分子标记与性别相关性状基因之间的对应关系,以建立相关性模式。值得留意的是,倘若使用上述以外的分子标记和/或上述以外的相关性模式鉴别木瓜株,无法预期能于花器早期发育时同时且准确鉴别出木瓜性别及特定的性别相关性状。
在上述实施例中,前述利用相关性模式鉴别木瓜样本的性别以及多个性别相关基因的步骤可包含但不限于由木瓜样本的待测花器获得待测核酸样本,由所述待测核酸样本获得多个性别分型数据,将这些性别分型数据输入所述相关性模式,以比对这些性别分型数据是否与这些分子标记的至少一个相符,并获得所述分子标记的至少一个的对应关系的相关正确率。当这些性别分型数据的至少一个与这些分子标记的至少一个相符,且相关正确率为100%时,判断该木瓜样本具有所述对应关系的这些性别以及这些性别相关基因的至少一个。
依据Storey在1938年及1953年所提出番木瓜性别(雄株、雌株及两性株)遗传假说分别由3个等位基因控制:M1(雄)、M2(两性)及m(雌性),而显性等位基因(MM,MMh,MhMh)的所有组合都是胚致死。通常两性株自交,后代发生两性株及雌性株的比例为2:1。两性株与雌性株杂交,后代发生两性株与雌性株的比例为1:1。
在此说明的是,本发明的分子标记不仅可同时鉴别木瓜株的三种性别,也可由转录组的选择性剪接(AS)的多态性,判断木瓜花器的性别相关性状是否受影响而有退化、畸形的可能,以便根据进行早期检测的结果及早应对。
举例而言,当上述第二性别分型数据包含SEQ ID NO:1~16、18、20~21、25~26、30~31、35~36、40~41、45~46、49~50、53~58、63~64以及与SEQ ID NO:1~16、18、20~21、25~26、30~31、35~36、40~41、45~46、49~50、53~58、63~64互补的序列的至少一个时,判断木瓜样本的性别为二性。
当上述第二性别分型数据报含SEQ ID NO:1~17、19、23~24、28~29、33~34、38~39、43~44、48、51~52、61~62以及与SEQ ID NO:1~17、19、23~24、28~29、33~34、38~39、43~44、48、51~52、61~62互补的序列的至少一个时,判断木瓜样本的性别为雄性。
当上述第二性别分型数据报含SEQ ID NO:1~16、22、27、32、37、42、47、59~60以及与SEQ ID NO:1~16、22、27、32、37、42、47、59~60互补的序列的至少一个时,判断木瓜样本的性别为雌性。
此外,上述分子标记更可多方应用,例如用于早期鉴别木瓜性别及性别相关性状的系统、包含上述的分子标记重组载体以及含所述重组载体的转基因植物细胞等。
以下利用数个实施例以说明本发明的应用,然其并非用以限定本发明,本领域技术人员在不脱离本发明的精神和范围内,当可作各种更动与润饰。
实施例1.建立番木瓜基因组数据库及转录组数据库
此实施例是由多个木瓜株建立番木瓜基因组数据库(或称基因组BAC库)及转录组数据库(或称转录组BAC库)。
首先,取番木瓜不同性别植株与花器,包含雌花4个、雄花4个及两性花4个,共12个样品,经萃取其基因组及转录组后,进行高通量定序(High-throughput Sequencing),以获得多个基因组序列及转录组序列。
前述基因组数据库及转录组数据库是参考常会方法建立。简言之,在NCBI上搜寻番木瓜细菌人工染色体(Bacterial Artificial Chromosome,BAC)基因库,以BAC序列为模板,将上述转录组所表达的基因序列组装到模板上,并依据不同性别表达量的不同,选出15个番木瓜BAC(图未绘示)。
实施例2.番木瓜性别决定基因的筛选
在上述15个番木瓜BAC中,初步筛选出性别相关基因共117个,其中有27个基因可于二种性别株中表现,有28个基因可于三种性别株中表现,合计55个基因。
将上述所得55个性别相关基因进行核酸及氨基酸多重序列比对,其为利用市售统计分析软件,例如excel比对,根据外显子跳跃(Exon Jump)、内含子跳跃(Intron Jump)、氨基酸短少(AA Short)及氨基酸无改变(None AA Change)等氨基酸不同变化,筛选出RNA具有选择性剪接(Alternative RNA splice)的性别相关基因。上述所得55个基因中,番木瓜BAC-71E16的基因中产生氨基酸变化者,如表2所示。
表2
将上述选出的基因利用商业软件,例如ClustalW2(网址为http://www.ebi.ac.uk/Tools/msa/clustalw2/)以及基因核酸与氨基酸的多重序列比对后,依照基因表现序列区分为type_1(单一性别不表现,另二个性别的氨基酸表现序列有差异)、type_2(三个性别均表现,且氨基酸表现序列均有差异)以及type_3(三个性别均有表现,其中二个性别的氨基酸表现序列无差异)等三种区分方式,以由番木瓜BAC-71E16中筛选出RNA具有选择性剪接(Alternative RNA splice)的性别相关基因。将选出的基因利用商业软件,例如SMART(网址为http://smart.embl-heidelberg.de/smart/change_mode.pl),预测蛋白质功能域,其结果如表3所示。
表3
由表3比对结果可知,在由番木瓜BAC-71E16中,type_1基因有2个,type_2基因有1个,而type_3基因有1个。
实施例3.番木瓜不同性别间CpSVP-LIKE及CpSERK的基因结构分析
此实施例利用SNP-HRM以及PCR-RFLP等方式,评估分子标记用于检测并分析性别与性别性状基因的相关性。
1.利用SNP-HRM分子标记,检测并分析性别与性别性状基因的相关性
经由基因组数据分析性别相关的单核苷酸多态性(SNP)位置,利用BeaconDesignerTM软件设计SNP-HRM分析引物,两基因共设计5组引物。将64个已知性别的番木瓜样品DNA(包含亲本与子代),进行SNP-HRM检测,每一HRM反应试剂包含:Precision melt supermix(Bio-Rad):6.34μl DDH2O,10μl Precision melt supermix,0.4μl 10μM正向引物,0.4μl 10μM反向引物,3μl(1.5ng/μl)DNA样品,总共15μl/孔,于96孔盘(BIOplastics)封膜(BIOplastics),利用实时定量聚合酶链式反应仪(Bio-Rad CFX Connect)配合软件(Bio-Rad CFX Manager)Real-time Polymerase chain reaction(PCR)program。
上述PCR温度程序设定条件如下:DNA双股解离(Denaturation):95℃进行2min。接着,进行39个循环的聚合酶链式反应,包括DNA双股解离(Denaturation):95℃10sec;以及引物配对黏合与单股DNA加长(Annealing and Elongation):57℃30sec。上述PCR产物荧光解离曲线温度程序的设定条件如下:DNA双股解离(Denaturation):95℃30sec,60℃1min;以及DNA双股荧光检测:65℃至90℃,每10秒增加0.2℃。
上述Real-timePCR结果利用软件(Bio-Rad Precision Melt Analysis)进行聚类分析(cluster analysis),并计算与番木瓜性别的相关正确率(correlection%)。将所得相关正确率达100%的SNP-HRM分子标记,于两性亲本与雄性亲本杂交子代族群中选出96个番木瓜盲样,计算SNP-HRM分析的性别数量,进行卡方分析了解子代族群SNP-HRM分子标记的性别性状分离率是否符合理论值。
2.利用PCR-RFLP分子标记,检测并分析性别与性别性状基因的相关性
依据SNP序列设计PCR-RFLP引物其PCR产物大小约200bp,PCR条件为:34μl DDH2O,5μl DMSO(Dimethyl sulfoxide),5μl 10xPC2 buffer(500mM Tris-HCl pH=9.1,160mM(NH4)2SO4,35mM MgCl2,1.5mg/ml BSA),1μl 10mM dNTP,1μl 10mM正向引物,1μl 10mM反向引物,1μl Taq(2U/μl),2μl DNA样品,总共50μl。分装于0.2ml eppendorf经聚合酶链式反应仪(BIOER-GenePro)PCR温度程序设定:Denaturation:94℃40sec;35个循环反应:引物配对黏合(Annealing):61℃40sec,单股DNA加长(Elongation):72℃30sec。所得PCR产物用纯化试剂组(Geneaid),进行纯化后,检测PCR产物浓度。取400ng的PCR产物和酶作用分析,其中:MADS-BOXgene的PCR产物,以NdeI酶配合buffer分析;SERKgene的PCR产物,以AseI酶配合buffer分析,均于37℃作用8小时,将酶作用前与作用后的PCR产物,分别进行电泳分析并UV数字照相。
经上述HRM与PCR-RFLP筛选后,发现位于Yh chromosome BAC 71E16距离约26000bp的短营养生长期(Short vegetative phase,CpSVP-LIKE)及体细胞胚胎发育受体激酶(Somatic Embryogenesis Receptor Kinase,CpSERK),此二基因均与性别相关。其次,番木瓜(Carica papayaL.,Cp)CpSVP-LIKE基因与CpSERK基因与酿酒葡萄(Vitisvinifera,Vv)VvSVP及VvSERK的氨基酸序列比对后,分别可达72%及96%相似度(数据未显示)。
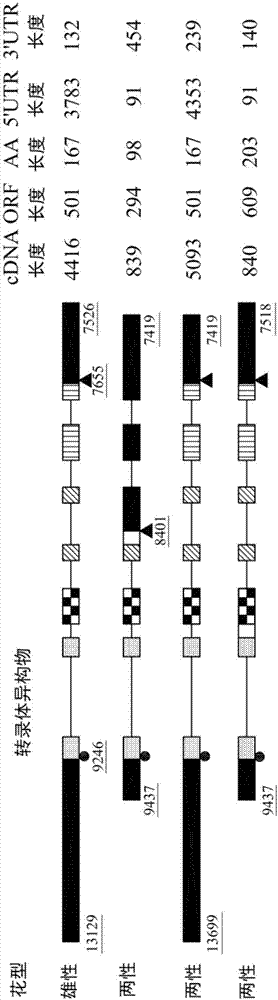
请参照图2A及图2B。图2A至图2B为绘示根据本发明的一个实施例的Yh染色体BAC71E16分析CpSVP-LIKE于番木瓜不同性型表现的cDNA序列(图2A)及内含子/外显子在BAC位置(图2B)的差异性。图2A至图2B的双底线的数字代表BAC上的位置,F代表雌性,M代表雄性,H代表两性,圆型图号代表起始密码,三角型图号代表终止密码,方格代表外显子,黑色方格代表无转译区,直条纹方格代表非特定区,方格E1/E2代表MADS-BOX区(GenBank序列编号:BAR64349.1,其氨基酸序列如SEQ ID NO:65所示),方格E3/E4/E5代表K-BOX区,横线代表内含子。每种结构的右侧标示其核酸、氨基酸数目。ORF代表开放阅读框(open reading frame),AA代表氨基酸(amino acid),UTR代表非转译区(untranslated regions),单底线数字为核酸数,结构下方标有primer位置,h代表HRM引物,dc代表PCR-RFLP引物,箭头表示SNP位置。
图2A结果指出,CpSVP-LIKE基因推测蛋白质序列长度而其推测蛋白质序列包含有K-BOX domain,含有6个内含子以及7个外显子。所述基因于番木瓜雌性0.2cm花苞不表现;雄性0.2cm花苞具有一种转录表现型推测转译长度167氨基酸(cDNA:4416bp;ORF:501bp);而两性0.2cm花苞则是3种转录表现型,两性的三种推测转译长度分别为98个氨基酸(cDNA:839bp;ORF:294bp;4th intron alternative splicing)、167个氨基酸(cDNA:5093bp;ORF:501bp)以及203个氨基酸(cDNA:840bp;ORF:609bp;2nd intron alternative splicing)。经由性别性状SNP genotyping分析,发现位于intron3第8511个核酸(MSY Yh chromosome BAC 71E16为参考位置)SNP G(M)→T(H)具有性别性状的相关性,而且仅有CpSVP-LIKE基因在两性株中具有AS的多态性,确实具有性别相关性,如图2A所示。因此,依据SNP位置设计SNP-HRM引物,及PCR-RFLP引物,分别命名为:SVP_HRM_88及SVP_RFLP_NdeI,如表1及SEQ ID NO:1~4所示。
请参照图3A及图3B,其绘示根据本发明的一个实施例的Yh染色体BAC71E16分析CpSERK于番木瓜不同性型表现的cDNA序列(图3A)及内含子/外显子在BAC位置(图3B)的差异性。
图3A至图3B的双底线的数字代表BAC上的位置,F代表雌性,M代表雄性,H代表两性,圆型图号代表起始密码,三角型图号代表终止密码,方格代表外显子,黑色方格代表无转译区,方格E1代表信号肽(Signal peptide,SP域),方格E2/E3/E4/E5代表富含亮胺酸重复序列(Leucine-rich repeats,或称LRR域),方格E7代表穿膜区(Transmembrane region,或称TM域)(E7),方格E9/E10/E11代表丝胺酸/苏胺酸蛋白激酶(Serine/Threonine protein kinases,或称Kinase域),横线代表内含子。每种结构的右侧标示其核酸、氨基酸数目。ORF代表开放阅读框(open reading frame),AA代表氨基酸(amino acid),UTR代表非转译区(untranslated regions),单底线数字为核酸数,结构下方标有primer位置,h代表HRM引物,dc代表PCR-RFLP引物,箭头表示SNP位置。
图3A、图3B、表3及表4的结果指出,CpSERK基因蛋白质序列包含有信号肽(SP域)、富含亮胺酸重复序列(LRR域)、穿膜区(TM域)与丝胺酸/苏胺酸蛋白激酶(Kinase域)的C端域“C-terminal(S_TKC-like)domain”,含有10个内含子以及11个外显子,而且番木瓜三种性别都不同:雌性593个氨基酸(cDNA:2183bp;ORF:1779bp)、雄性523个氨基酸(cDNA:1952bp;ORF:1569bp;10th intron alternative splicing)以及两性628个氨基酸(cDNA:2467bp;ORF:1884bp)。经由性别性状SNP genotyping分析,发现intron 1第122453个核酸的SNP若为A则为雌性,若为G则为雄性及两性;intron 7第119295个核酸的SNP若为T则为雌性,若为G则为雄性及两性;以及第119290个核酸的SNP若为A则为雌性及雄性,若为T则为两性;Exon9第118602个核酸的SNP若为C则为雌性,若为T则为雄性及两性;以及第118575个核酸的SNP若为G则为雌性,若为A则为雄性及两性。上述SNP均具有性别相关的变化,可用于鉴别木瓜性别。依据SNP位置分别设计SNP-HRM及PCR-RFLP引物对,依序命名为:SERK_HRM_30704、SERK_HRM_34072、SERK_HRM_34760、SERK_HRM_34787、SERK_RFLP_DdeI与SERK_RFLP_SpeI,如表1及SEQ ID NO:1~16所示。
请参照表4,其绘示根据本发明的一个实施例的三性CpSERK与Yh染色体BAC 71E16氨基酸序列的比对。
表4
表4的结果指出,上述2个基因(CpSVP-LIKE及CpSERK)确实具有性别相关性。另外,上述2个基因(CpSVP-LIKE及CpSERK)仅有CpSVP-LIKE基因在两性株中具有AS的多态性,如图2A所示。
实施例4.番木瓜性别分子标记检测与遗传分析
此实施例系利用上述分子标记,进行40个盲样族群测试,以评估上述分子标记用于早期鉴别木瓜性别及性别相关性状的相关正确率。
首先,在蛋白质编码的基因组范围进行性别性状SNP genotyping分析,发现CpSVP-LIKE及CpSERK基因有性别相关的基因型SNP分布,如图2A至图3B及表5所示。
表5
其次,在CpSVP-LIKE基因中,SVP_HRM_88引物检测结果发现三性之间具有明显的DNA解离荧光曲线的差异(图4A,CpSVP-LIKE88引物对),性别性状遗传分析结果发现24个亲本与40个子代样品测试,三性间SVP-HRM_88检测的相关性达100%。再者,40个盲样族群测试中,SVP_HRM_88引物检测三性别的比值,经卡方检测符合1:1:1理论值,如表5的结果所示。经由PCR-RFLP分析结果则发现SVP_RFLP_NdeI引物组针对不同性别进行PCR,雌性不会产生的PCR产物,而雄性与两性的的PCR产物,经NdeI酶处理后,所述SNP可以区别雄性与两性,如图5A的结果所示。
在CpSERK基因中,SERK_HRM_34072、SERK_HRM_30704、SERK_HRM_34760以及SERK_HRM_34787四组引物对,检测都能区别雌性,如图4B至图4E的结果所示。而SERK_HRM_34072则是发现三性之间具有明显的DNA解离荧光曲线的差异,如图4B的结果所示。所述遗传分析中亲本与子代的SERK_HRM_34072、SERK_HRM_34760与SERK_HRM_34787的分析结果发现,雌性与两性的SNP-HRM检测的相关性达100%,40个盲样族群测试该三组检测雌性与其他性别的比值,经卡方检测符合1:2理论值,如表5的结果所示。而PCR-RFLP分析第119737核酸,利用SERK_RFLP_SpeI引物组针对不同性别进行PCR,其产物不同性别之间并无差异,而经SpeI酶处理后,所述SNP可以区别两性与其他性别,如图5B的结果所示。
上述实施例中,每组数据的样本数至少为3个(n≧3)。
请参照图6,绘示根据本发明的一个实施例的番木瓜性别及性别相关性状基因的分子标记的三性型基因连锁图及其表现模式,其中F代表雌性,M代表雄性,H代表两性。举例而言,当上述第二性别分型数据报含SEQ ID NO:1~16、22、27、32、37、42、47、59~60以及与SEQ ID NO:1~16、22、27、32、37、42、47、59~60互补的序列的至少一个但不含SEQIDNO:51~58时,判断木瓜样本的花器型态及/或果实形状为稳定的雌性型。当上述第二性别分型数据包含SEQ ID NO:1~17、19、23~24、28~29、33~34、38~39、43~44、48、51~52、61~62以及与SEQ ID NO:1~17、19、23~24、28~29、33~34、38~39、43~44、48、51~52、61~62互补的序列的至少一个时,判断木瓜样本的花器型态和/或果实形状为稳定的雄性型。当上述第二性别分型数据包含SEQ ID NO:1~16、18、20~21、25~26、30~31、35~36、40~41、45~46、49~50、53~58、63~64以及与SEQ ID NO:1~16、18、20~21、25~26、30~31、35~36、40~41、45~46、49~50、53~58、63~64互补的序列的至少一个时,判断木瓜样本的花器型态和/或果实形状为不稳定的两性型。
总之,由上述数个实施例证实,本发明的用于早期鉴别木瓜性别及性别相关性状的分子标记,确实可在木瓜样本的花器发育早期,鉴别其性别及性别相关性状,且经实验证实,由此所得的鉴别结果确实能于早期同时且准确鉴别木瓜性别及性别相关性状。
展开来说,上述数个实施例证实,在基因组方面,CpSVP-LIKE在番木瓜具有X、Ym与Yh基因组上有着可以区分三性型的差异,且CpSVP-LIKE与CpSERK基因则是在X基因组紧密连锁着相同雌性型的基因型变化。在转录组方面,所述基因组上CpSVP-LIKE在两性花器上的表现序列型可达三种,而雄性的表现序列只有一种,不仅可解释所述基因组在三性型X、Ym与Yh基因组的差异可能造成CpSVP-LIKE在不同性别表现上的差异,特别是两性株CpSVP-LIKE表现序列的多态性与番木瓜两性花器畸型变化的现象可能具有关联性(如图6所示)。
其次,上述数个实施例也证实,利用HRM分析盲样基因组数据,SNP#34072、SNP#34760及SNP#34787可达到100%雌性相关;而利用PCR-RFLP分析盲样CpSVP-LIKE基因的SNP#88及CpSERK基因的SNP#34072均可区别三性及雌性别。另外,利用HRM分析盲样转录组数据,CpSVP-LIKE基因在雌株无法表现,于雄性则是单一表现序列,而两性基因表现具有选择性RNA剪接(alternative RNA splicing)的三种表现序列;CpSERK基因表现序列在不同性别间的转录组序列也具有差异性。由上述结果可判断木瓜花器的性别与性别相关性状是否受影响而有退化、畸形的可能,以便根据进行早期检测的结果及早应对。
再者,上述数个实施例更证实,本发明的上述分子标记可多方应用,例如用于早期鉴别木瓜性别及性别相关性状的系统、包含上述的分子标记重组载体以及含所述重组载体的转基因植物细胞等。
需补充的是,本发明虽以特定的品种、特定的分子标记、特定的性别相关性状基因或特定的评估方式作为示例,说明本发明的用于早期鉴别木瓜性别及性别相关性状的分子标记及其应用,然而本领域技术人员可知,本发明并不限于此,在不脱离本发明的精神和范围内,本发明的用于早期鉴别木瓜性别及性别相关性状的分子标记及其应用,也可使用其他的品种、其他的分子标记、其他的性别相关性状基因或其他的评估方式进行。
由上述实施例可知,本发明的用于早期鉴别木瓜性别及性别相关性状的分子标记及其应用,其利用特定的分子标记与性别相关性状基因的任一个建立相关性模式,可于木瓜花器早期发育时,鉴别此木瓜样本的性别以及性别相关性状,以便有效、快速且准确获得木瓜两性株,并有助于减少不结实花及畸形花果的产生。
虽然本发明已以数个实施例公开如上,然而其并非用以限定本发明,对本领域技术人员来说,在不脱离本发明的精神和范围内,可以作出各种更动与润饰,且本发明的保护范围应当以所附的权利要求所界定的为准。
【序列表】
<110> 国立屏东科技大学
<120> 用于早期鉴别木瓜性别及性别相关性状的分子标记及其应用
<130> TWLB7102-16P1
<160> 65
<210> 1
<211> 27
<212> DNA
<213> 人工序列
<220>
<223> SVP_HRM_88的正向引物
<400> 1
aataaaatgc ataattacat agcacag 27
<210> 2
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> SVP_HRM_88的反向引物
<400> 2
agaaaggtca gtttaccaca taa 23
<210> 3
<211> 27
<212> DNA
<213> 人工序列
<220>
<223> SVP_RFLP_Nde I的正向引物
<400> 3
ctgcaaagta atgttcttta agcatat 27
<210> 4
<211> 27
<212> DNA
<213> 人工序列
<220>
<223> SVP_RFLP_Nde I的反向引物
<400> 4
aataaaatgc ataattacat agcacag 27
<210> 5
<211> 19
<212> DNA
<213> 人工序列
<220>
<223> SERK_HRM_30704的正向引物
<400> 5
ttcattgaga tgcgtattg 19
<210> 6
<211> 19
<212> DNA
<213> 人工序列
<220>
<223> SERK_HRM_30704的反向引物
<400> 6
gctcatcaca agtaaagtc 19
<210> 7
<211> 18
<212> DNA
<213> 人工序列
<220>
<223> SERK_HRM_34072的正向引物 (检测第119290个碱基对)
<400> 7
CTCCTCCACC ATTTGTTC 18
<210> 8
<211> 19
<212> DNA
<213> 人工序列
<220>
<223> SERK_HRM_34072的反向引物(检测第119290个碱基对)
<400> 8
gctattgatg tgttgctac 19
<210> 9
<211> 18
<212> DNA
<213> 人工序列
<220>
<223> SERK_HRM_34072的正向引物 (检测第119295个碱基对)
<400> 9
ctcctccacc atttgttc 18
<210> 10
<211> 19
<212> DNA
<213> 人工序列
<220>
<223> SERK_HRM_34072的反向引物(检测第119295个碱基对)
<400> 10
gctattgatg tgttgctac 19
<210> 11
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> SERK_HRM_34760的正向引物
<400> 11
ttttgggtag aggaggattt 20
<210> 12
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> SERK_HRM_34760的反向引物
<400> 12
agctactagt gtaccatcag 20
<210> 13
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> SERK_HRM_34787的正向引物
<400> 13
tagctgatgg tacactagta 20
<210> 14
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> SERK_HRM_34787的反向引物
<400> 14
tctacttctg tctgaaactg 20
<210> 15
<211> 26
<212> DNA
<213> 人工序列
<220>
<223> SERK_RFLP_Spe I的正向引物
<400> 15
tgttgctaca aaattcaata tactag 26
<210> 16
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> SERK_RFLP_Spe I的反向引物
<400> 16
ttaggttatt ccgtgactat c 21
<210> 17
<211> 231
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SVP_HRM_88引物对(SNP# 88)的雄性株PCR产物
<400> 17
agaaaggtca gtttaccaca taatatttgt attctgcaaa gtaatgttct ttaagcttat 60
gacatataag ttgaaattgg aatagtccct aattatctat atttttcttc tgcatctcag 120
ggtcaaaata taataaatga gatcagtgat cttcaaagga aggtcagaca ctaaaaaccc 180
ataaattttc acagatattt atatctgtgc tatgtaatta tgcattttat t 231
<210> 18
<211> 4416
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SVP_HRM_88引物对(SNP# 88)的两性株PCR产物
<400> 18
agaaaggtca gtttaccaca taatatttgt attctgcaaa gtaatgttct ttaagcttat 60
tacatataag ttgaaattgg aatagtccct aattatctat atttttcttc tgcatctcag 120
ggtcaaaata taataaatga gatcagtgat cttcaaagga aggtcagaca ctaaaaaccc 180
ataaattttc acagatattt atatctgtgc tatgtaatta tgcattttat t 231
<210> 19
<211> 88
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SVP_RFLP_Nde I引物对(SNP# 88)的雌性株PCR产物
<400> 19
ttcattgaga tgcgtattga tcttttaaag tacacaacag agaaaaatct acaaagttgt 60
acccgttatg actttacttg tgatgagc 88
<210> 20
<211> 88
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SVP_RFLP_Nde I引物对(SNP# 88)的雄性株PCR产物
<400> 20
ttcattgaga tgcgtattga tcttttaaag tacacaacag agagaaatct acaaagttgt 60
acccgttatg actttacttg tgatgagc 88
<210> 21
<211> 88
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SVP_RFLP_Nde I引物对(SNP# 88)的雄性株PCR产物
<400> 21
ttcattgaga tgcgtattga tcttttaaag tacacaacag agaaaaatct acaaagttgt 60
acccgttatg actttacttg tgatgagc 88
<210> 22
<211> 88
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_30704引物对(SNP# 30704)的雌性株PCR产物
<400> 22
ttcattgaga tgcgtattga tcttttaaag tacacaacag agaaaaatct acaaagttgt 60
acccgttatg actttacttg tgatgagc 88
<210> 23
<211> 4416
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_30704引物对(SNP# 30704)的雄性株PCR产物
<400> 23
ttcattgaga tgcgtattga tcttttaaag tacacaacag agagaaatct acaaagttgt 60
acccgttatg actttacttg tgatgagc 88
<210> 24
<211> 4416
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_30704引物对(SNP# 30704)的雄性株PCR产物
<400> 24
ttcattgaga tgcgtattga tcttttaaag tacacaacag agaaaaatct acaaagttgt 60
acccgttatg actttacttg tgatgagc 88
<210> 25
<211> 88
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_30704引物对(SNP# 30704)的两性株PCR产物
<400> 25
ttcattgaga tgcgtattga tcttttaaag tacacaacag agagaaatct acaaagttgt 60
acccgttatg actttacttg tgatgagc 88
<210> 26
<211> 88
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_30704引物对(SNP# 30704)的两性株PCR产物
<400> 26
ttcattgaga tgcgtattga tcttttaaag tacacaacag agaaaaatct acaaagttgt 60
acccgttatg actttacttg tgatgagc 88
<210> 27
<211> 92
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34072引物对(SNP# 34072a)的雌性株PCR产物
<400> 27
gctattgatg tgttgctaca aaattcaata taatagtagt ccgattacct ggggaagaa 60
attggaggtg ggggaacaa atggtggagg ag 92
<210> 28
<211> 92
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34072引物对(SNP# 34072a)的雄性株PCR产物
<400> 28
gctattgatg tgttgctaca aaattcaata taatagtagt ccgattacct ggggaagaa 60
attggaggtg ggggaacaaa tggtggagg ag 92
<210> 29
<211> 92
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34072引物对(SNP# 34072a)的雄性株PCR产物
<400> 29
gctattgatg tgttgctaca aaattcaata taatagcagt ccgattacct ggggaagaa 60
attggaggtg ggggaacaaa tggtggagg ag 92
<210> 30
<211> 92
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34072引物对(SNP# 34072a)的两性株PCR产物
<400> 30
gctattgatg tgttgctaca aaattcaata taatagcagt ccgattacct ggggaagaa 60
attggaggtg ggggaacaa atggtggagg ag 92
<210> 31
<211> 92
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34072引物对(SNP# 34072a)的两性株PCR产物
<400> 31
gctattgatg tgttgctaca aaattcaata taatagtagt ccgattacct ggggaagaa 60
attggaggtg ggggaacaa atggtggagg ag 92
<210> 32
<211> 90
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34072引物对(SNP# 34072b)的雌性株PCR产物
<400> 32
gctattgatg tgttgctaca aaattcaata taatagtagt cagattacct ggggaagaaa 60
ttggaggtgg gggaacaaat ggtggaggag 90
<210> 33
<211> 92
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34072引物对(SNP# 34072b)的雄性株PCR产物
<400> 33
gctattgatg tgttgctaca aaattcaata taatagtagt ccgattacct ggggaagaa 60
attggaggtg ggggaacaa atggtggagg ag 92
<210> 34
<211> 92
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34072引物对(SNP# 34072b)的雄性株PCR产物
<400> 34
gctattgatg tgttgctaca aaattcaata taatagtagt cagattacct ggggaagaa 60
attggaggtg ggggaacaa atggtggagg ag 92
<210> 35
<211> 90
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34072引物对(SNP# 34072b)的两性株PCR产物
<400> 35
gctattgatg tgttgctaca aaattcaata taatagtagt ccgattacct ggggaagaaa 60
ttggaggtgg gggaacaaat ggtggaggag 90
<210> 36
<211> 90
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34072引物对(SNP# 34072b)的两性株PCR产物
<400> 36
gctattgatg tgttgctaca aaattcaata taatagtagt cagattacct ggggaagaaa 60
ttggaggtgg gggaacaaat ggtggaggag 90
<210> 37
<211> 65
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34760引物对(SNP# 34760)的雌性株PCR产物
<400> 37
ttttgggtag aggaggattt ggtaaggttt acaaaggtcg cctagctgat ggtacactag 60
tagct 65
<210> 38
<211> 65
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34760引物对(SNP# 34760)的雄性株PCR产物
<400> 38
ttttgggtag aggaggattt ggtaaggttt acaaaggtcg tctagctgat ggtacactag 60
tagct 65
<210> 39
<211> 65
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34760引物对(SNP# 34760)的雄性株PCR产物
<400> 39
ttttgggtag aggaggattt ggtaaggttt acaaaggtcg cctagctgat ggtacactag 60
tagct 65
<210> 40
<211> 65
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34760引物对(SNP# 34760)的两性株PCR产物
<400> 40
ttttgggtag aggaggattt ggtaaggttt acaaaggtcg tctagctgat ggtacactag 60
tagct 65
<210> 41
<211> 65
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34760引物对(SNP# 34760)的两性株PCR产物
<400> 41
ttttgggtag aggaggattt ggtaaggttt acaaaggtcg cctagctgat ggtacactag 60
tagct 65
<210> 42
<211> 85
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34787引物对(SNP# 34787)的雌性株PCR产物
<400> 42
tagctgatgg tacactagta gctgtgaaaa gactgaaaga agagcgtact cctggtgggg 60
agctgcagtt tcagacagaa gtaga 85
<210> 43
<211> 85
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34787引物对(SNP# 34787)的雄性株PCR产物
<400> 43
tagctgatgg tacactagta gctgtaaaaa gactgaaaga agagcgtact cctggtgggg 60
agctgcagtt tcagacagaa gtaga 85
=
<210> 44
<211> 85
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34787引物对(SNP# 34787)的雄性株PCR产物
<400> 44
tagctgatgg tacactagta gctgtgaaaa gactgaaaga agagcgtact cctggtgggg 60
agctgcagtt tcagacagaa gtaga 85
<210> 45
<211> 4416
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34787引物对(SNP# 34787)的两性株PCR产物
<400> 45
tagctgatgg tacactagta gctgtaaaaa gactgaaaga agagcgtact cctggtgggg 60
agctgcagtt tcagacagaa gtaga 85
<210> 46
<211> 4416
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_34787引物对(SNP# 34787)的两性株PCR产物
<400> 46
tagctgatgg tacactagta gctgtgaaaa gactgaaaga agagcgtact cctggtgggg 60
agctgcagtt tcagacagaa gtaga 85
<210> 47
<211> 238
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_RFLP_Spe I引物对(SNP# 34072a)的雌性株PCR产物
<400> 47
tgttgctaca aaattcaata taatagtagt ccgattacct ggggaagaaa ttggaggtgg 60
gggaacaaat ggtggaggag gagaaaatgg tggagatcct gggcaggggc gtccagtaac 120
cggaccacat agatccaagt tgttggcaaa actgcagatg cgaataaaac atttatgctt 180
catgttccaa accagtcagt aaacaaaata taaaccggat agtcacggaa taacctaa 238
<210> 48
<211> 238
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_RFLP_Spe I引物对(SNP# 34072a)的雄性株PCR产物
<400> 48
tgttgctaca aaattcaata taatagtagt ccgattacct ggggaagaaa ttggaggtgg 60
gggaacaaat ggtggaggag gagaaaatgg tggagatcct gggcaggggc gtccagtaac 120
cggaccacat agatccaagt tgttggcaaa actgcagatg cgaataaaac atttatgctt 180
catgttccaa accagtcagt aaacaaaata taaaccggat agtcacggaa taacctaa 238
<210> 49
<211> 248
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_RFLP_Spe I引物对(SNP# 34072a)的两性株PCR产物
<400> 49
gctattgatg tgttgctaca aaattcaata taatagcagt ccgattacct ggggaagaaa 60
ttggaggtgg gggaacaaat ggtggaggag gagaaaatgg tggagatcct gggcaggggc 120
gtccagtaac cggaccacat agatccaagt tgttggcaaa actgcagatg cgaataaaac 180
atttatgctt catgttccaa accagtcagt aaacaaaata taaaccggat agtcacggaa 240
taacctaa 248
<210> 50
<211> 248
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_RFLP_Spe I引物对(SNP# 34072a)的两性株PCR产物
<400> 50
gctattgatg tgttgctaca aaattcaata taatagtagt ccgattacct ggggaagaaa 60
ttggaggtgg gggaacaaat ggtggaggag gagaaaatgg tggagatcct gggcaggggc 120
gtccagtaac cggaccacat agatccaagt tgttggcaaa actgcagatg cgaataaaac 180
atttatgctt catgttccaa accagtcagt aaacaaaata taaaccggat agtcacggaa 240
taacctaa 248
<210> 51
<211> 4416
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SVP_HRM_88引物对对应的雄性株CpSVP-LIKE基因的转录组cDNA序列
<400> 51
atgggttttg accctcaaag atgaaaaatc tccatcttcg tcattggggc ccccagtgtg 60
ttattctgtg ggttttgtgt actatttttc ccaacttcac gctgtcttgg gtttagaaga 120
atacctcgtt catgggtttg gtagggaaga gaatgaagcg ttggtcaatc tgcactttac 180
tctagtcctc atttaataca tctttgccat aaatctgaca gtttcacgtc gtccagttgc 240
tgccattttt gttgttcgtt tactaaagca tttcccttgt tatttggcca tcgctacaat 300
atttgtaaac ggctcgacgg atttgtacag cttgatacca agttgttagg agcctttgag 360
atgtacacca ataacaattt aggttgtgcg aatagatgaa gtaataaatg agagaaattg 420
ataaaaggaa gagagataaa aaaggtaaga ggagagaatg agagagatac tcagcaagag 480
ctgctgaatt agataagttt ttgataacat taatcataca ttagaaatca agctcatagt 540
taatatattc ctctaccaga ctctggcaag aaaatgtaaa catataaaat ggttataaca 600
gaaaagggaa tccataacta atttctataa atcagacaat tcgttttatt ccctactcag 660
acctggacca caagccaaca tcagattggt gtcagtaggg gctttgttta ctttatcctc 720
aagctcatct ttcttatttt gagccttaat tgtggcattt cttggctttc tatatcatga 780
ttattctttt cttgtagttc agtagataaa gtcataaaag agcaacattt aacagggtta 840
atatacagct gtttcaaact actgattttt ttttctataa tgattttttt cccccagtgt 900
aagaaagaac tttattgcat aaagaaacat gtacgtacct gaaatgttac ttatataatt 960
tgcaaccaaa cacagaacac aacaaaggca tgaaaacgcc agcacaacct atgatacaag 1020
atgatagcag aaacagatgc aggaactgca accaaagctt aataccttct cttttaagtg 1080
ttagattatg atatcctaat atcattaagt gttagtttct ttttctgtca tggttaaatt 1140
atgatatctt aaatatcttt tcttttaagt cttaggtttc aaattgaatc tcaattgttt 1200
gatagatttt caataagaag atttactttg actgattgat gatttagtgt gattcgaaaa 1260
tgtcttgatc tcgaaccatg gtacaacatt cataaactgt ctgatagatt ttcatttttt 1320
ttttcttttt agaaatgaaa acaggtaaaa aaagtcctac caacttagta acctctcttc 1380
aaattggtat gattatatga attgatgtct agtttaagct atctttatta tctaagaaaa 1440
attgcgagta cataatttgc actcatgaga tgcgcgttct tttaagcatc aaacaattga 1500
atattttgtt gttcctttct tttcatttcc tttattccta acatagctga tgcagaaccc 1560
agaatcaata tatgtgataa aaaaccctag atatatatca atagtcatat tctaaagctc 1620
tcaagaaatt ctccgtactt gacctacatt caacgtgtga atatcaacca agataattga 1680
gatgaggatc tgaattgact aaatcctacg agaattatat cttgataagt aaaattaact 1740
caagggagat tgacaaagcg ccataagaac ttattttgat aagttgaatc aacctgttag 1800
tcagcaagat cgttgaagac accctcttac agattacaca caattagcaa agaggtaaaa 1860
acaaactcga atattttatt gaacttgaac ttgtgaatta cgaagggggt ggtggcattc 1920
ttatatagat ttccatccac ttattctact agaaaacgaa ttaaataatt ctaatcaaac 1980
tcttaatcct acttaaaaag aaatacgaac ttcctaattg aacacttaat ccaaatacaa 2040
ttagaaaact aaaaacttaa aaaattaaag acttacatta aaagattaaa ggatttctct 2100
tgggtggctt ttcaagattc ctatttggat tcaagctttt gtcttcctct cggttttagc 2160
ttctctttca tttaatgatt attttgctgc agatttcgga taggatttgt agacttcttg 2220
cggatttgta gtagaaaatg tgaagggaaa tttgataaga gtagtcttct cttggccggt 2280
tgcttcatgc ttctaattct ttccttctct tgatgatttc atcctgcgcc ttgaattcta 2340
gtttctacat catgtaggag tctgaattca acctgagaaa aagaaaagaa aagaagaaat 2400
acacacaaga aagacttgca gtaggagagg gaatccgatt ccctcttgga taaggtgtcc 2460
tgcgcggaat cctcctccaa ttcttcttct cgtgaattgg ctttatttta ggaccctttt 2520
gccgacttct tcttggacgt ttatttcttg taaaccgatc ttgaatcata gtaggaattc 2580
tgtcaaacct ttccaaagag aaaggacttt gcatttctta agaggtgaag gaacctgtgc 2640
gggcgggtct tgaatccctt cccttcgctc agccatgggc ccctacggtc aaaagcatgt 2700
gtggccgggc tgtatctccc tttcttggac cgattcttgg tagatcttgg actgctagac 2760
acgattcttt gttgaaatac tttagggcca agtgtttggg agcttggggg ctccctttga 2820
gtcagattct tgggtattga ccaaaagggc cttgaatctt tggttcttga ttcttggatg 2880
gatcttgagt gtaaaccggc ttgggacagt ttcttggctt gacaatccat tccttggagt 2940
gatggaaatc cgaaagatgg actccaaatt cccacaggat tgggatttcc cgtgagcatg 3000
agattttctt cgcgtggtct ctgcaggtgc gtgcgtgctg tgcgcgctca cgggtgtggg 3060
tgattccttg cccgcgggcc tgcttttctt gtctctccgg gacttgttgt tttccttatc 3120
taggatcaca tcaatagcgt ggaaagaggt ttaatacatg actacaaatg cacttatgac 3180
atatttatag tagaagcatc ttctctatct gaaacaggta aaagctcgga caactaccag 3240
aacaagtttg atcttgttaa cttctgagaa taaatgctct attaccactt gaactaccat 3300
tttagtagtt tatgtgtgat ctgtgatgga ttggactttt tgctttgaaa tattatgttg 3360
gactgttatt tgggttatgt ttgggaaaac ataaatttgt tgtattcatt tgaaaatttt 3420
atattatttt aatatgaaag agttaaagat cccacgtaac tacataagat gataaaattg 3480
acttgtatat tagacagttt aagtattcac tataatttat tttaatgata gccttaatct 3540
cttaattaat cctcatcatt tatataatga aaagaaattg gaattgggag ttgccttcat 3600
gtatttaatt tctgttacta ggtgggaata acatttatac tttctttgat ctctagaatt 3660
tatgtatcca aggatggtgt tgtatgaata cttctaatta attaaggtta tcttatcttc 3720
caacaattat atatatatgc attaatgtgg tagtgacttt gtgctctttg agcctttcga 3780
agcatgaagg aaatacttga aaggcatatg ttgcacgaaa agaaccttga gaagctggaa 3840
cagccatctc ttgaactaaa gctagttgag aatggcgacc gctccagatt gtgcaacgaa 3900
attgctcaga gaagccatca cttgaagcaa atgagggggg aggagctcca aggattaagc 3960
atagagcaac tgcagcagtt ggaaaaattc cttgaagtgg gattgcgccg tgttatagag 4020
agaaagggtc aaaatataat aaatgagatc agtgatcttc aaaggaaggg aatacaattg 4080
atggaagaga atgagaaact gaggcaacaa ttgttgcaga tatctaacaa aggaaaacaa 4140
ggtggtgcag agtcggagaa tgtagcgtct gaagagggcc agtcatcaga gtctgtaacc 4200
aatctctgca actctaatgg tccaccacac gactatgaaa gctcagatac ctctctaaag 4260
cttgggttac cctattccgg ctaaatcagg ggtaatgcca atgtcagaga gggtatcaaa 4320
agaagaaaaa gaaacatatg aatctgtatt aagttcacta tgttcatgtt tgcaagtaca 4380
gagtttgcca ttgaactaga gatacttatt tgtaat 4416
<210> 52
<211> 166
<212> PRT
<213> 番木瓜(Carica papaya L.)
<220>
<223> SVP_HRM_88引物对对应的雄性株CpSVP-LIKE基因的转录组氨基酸序列
<400> 52
Met Lys Glu Ile Leu Glu Arg His Met Leu His Glu Lys Asn Leu Glu
1 5 10 15
Lys Leu Glu Gln Pro Ser Leu Glu Leu Lys Leu Val Glu Asn Gly Asp
20 25 30
Arg Ser Arg Leu Cys Asn Glu Ile Ala Gln Arg Ser His His Leu Lys
35 40 45
Gln Met Arg Gly Glu Glu Leu Gln Gly Leu Ser Ile Glu Gln Leu Gln
50 55 60
Gln Leu Glu Lys Phe Leu Glu Val Gly Leu Arg Arg Val Ile Glu Arg
65 70 75 80
Lys Gly Gln Asn Ile Ile Asn Glu Ile Ser Asp Leu Gln Arg Lys Gly
85 90 95
Ile Gln Leu Met Glu Glu Asn Glu Lys Leu Arg Gln Gln Leu Leu Gln
100 105 110
Ile Ser Asn Lys Gly Lys Gln Gly Gly Ala Glu Ser Glu Asn Val Ala
115 120 125
Ser Glu Glu Gly Gln Ser Ser Glu Ser Val Thr Asn Leu Cys Asn Ser
130 135 140
Asn Gly Pro Pro His Asp Tyr Glu Ser Ser Asp Thr Ser Leu Lys Leu
145 150 155 160
Gly Leu Pro Tyr Ser Gly
165
<210> 53
<211> 840
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SVP_HRM_88引物对对应的两性株H1 CpSVP-LIKE基因的转录组cDNA序列
<400> 53
tctaattaat taaggttatc ttatcttcca acaattatat atatatgcat taatgtggta 60
gtgactttgt gctctttgag cctttcgaag catgaaggaa atacttgaaa ggcatatgtt 120
gcacgaaaag aaccttgaga agctggaaca gccatctctt gaactaaagc tagttgagaa 180
tggcgaccgc tccagattgt gcaacgaaat tgctcagaga agccatcact tgaagtataa 240
taagcttctt cagttttgtt tattaagtgg tttacatcta tttagtatga atatgtatga 300
caatgataat tatcacataa taaacaataa taaacgaaat aggcaaatga ggggggagga 360
gctccaagga ttaagcatag agcaactgca gcagttggaa aaattccttg aagtgggatt 420
gcgccgtgtt atagagagaa agggtcaaaa tataataaat gagatcagtg atcttcaaag 480
gaagggaata caattgatgg aagagaatga gaaactgagg caacaattgt tgcagatatc 540
taacaaagga aaacaaggtg gtgcagagtc ggagaatgta gcgtctgaag agggccagtc 600
atcagagtct gtaaccaatc tctgcaactc taatggtcca ccacacgact atgaaagctc 660
agatacctct ctaaagcttg ggttacccta ttccggctaa atcaggggta atgccaatgt 720
cagagagggt atcaaaagaa gaaaaagaaa catatgaatc tgtattaagt tcactatgtt 780
catgtttgca agtacagagt ttgccattga actagagata cttatttgta attattaatg 840
<210> 54
<211> 202
<212> PRT
<213> 番木瓜(Carica papaya L.)
<220>
<223> SVP_HRM_88引物对对应的两性株H1 CpSVP-LIKE基因的转录组氨基酸序列
<400> 54
Met Lys Glu Ile Leu Glu Arg His Met Leu His Glu Lys Asn Leu Glu
1 5 10 15
Lys Leu Glu Gln Pro Ser Leu Glu Leu Lys Leu Val Glu Asn Gly Asp
20 25 30
Arg Ser Arg Leu Cys Asn Glu Ile Ala Gln Arg Ser His His Leu Lys
35 40 45
Tyr Asn Lys Leu Leu Gln Phe Cys Leu Leu Ser Gly Leu His Leu Phe
50 55 60
Ser Met Asn Met Tyr Asp Asn Asp Asn Tyr His Ile Ile Asn Asn Asn
65 70 75 80
Lys Arg Asn Arg Gln Met Arg Gly Glu Glu Leu Gln Gly Leu Ser Ile
85 90 95
Glu Gln Leu Gln Gln Leu Glu Lys Phe Leu Glu Val Gly Leu Arg Arg
100 105 110
Val Ile Glu Arg Lys Gly Gln Asn Ile Ile Asn Glu Ile Ser Asp Leu
115 120 125
Gln Arg Lys Gly Ile Gln Leu Met Glu Glu Asn Glu Lys Leu Arg Gln
130 135 140
Gln Leu Leu Gln Ile Ser Asn Lys Gly Lys Gln Gly Gly Ala Glu Ser
145 150 155 160
Glu Asn Val Ala Ser Glu Glu Gly Gln Ser Ser Glu Ser Val Thr Asn
165 170 175
Leu Cys Asn Ser Asn Gly Pro Pro His Asp Tyr Glu Ser Ser Asp Thr
180 185 190
Ser Leu Lys Leu Gly Leu Pro Tyr Ser Gly
195 200
<210> 55
<211> 5093
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SVP_HRM_88引物对对应的两性株H2 CpSVP-LIKE基因的转录组cDNA序列
<400> 55
gttaatttat ggtgttagtc atgaaggtgt tggtgtggat atggagaatt tctgggactg 60
gttggtgaat aatgtacttt tggcctgtgg gcaggcgaag attggtggcc gactaatctg 120
gtgtgatcca ttgccttcgt ggattgttct tgtaacaaga gcagctcgaa gacatggttt 180
agggtgagaa gtttatgcat tctcaatgtg ggttgcaact cttccttcaa cctactgatg 240
aaggtagaaa taaaatctct ctcggttaat ctaggatttt caataagcaa caataatttc 300
atctcctcaa aattttcctg gtaattaagt gctgaatcct cttacctaag tttatttaat 360
tcttccacca catcaatttc tgccaaccct ccaaatcttc ttcctagttc ctccataaac 420
atatcccaat ctacttgtcc aggggggttc agctacccat gaaaccaaac ttagacctta 480
tctcctaagt acatggctgc tgtttctact ttgttctctt cagctatgcc aagacctgga 540
aatatttcaa acaatgtttg atataagttc atgggttttg accctcaaag atgaaaaatc 600
tccatcttcg tcattggggc ccccagtgtg ttattctgtg ggttttgtgt actatttttc 660
ccaacttcac gctgtcttgg gtttagaaga atacctcgtt catgggtttg gtagggaaga 720
gaatgaagcg ttggtcaatc tgcactttac tctagtcctc atttaataca tctttgccat 780
aaatctgaca gtttcacgtc gtccagttgc tgccattttt gttgttcgtt tactaaagca 840
tttcccttgt tatttggcca tcgctacaat atttgtaaac ggctcgacgg atttgtacag 900
cttgatacca agttgttagg agcctttgag atgtacacca ataacaattt aggttgtgcg 960
aatagatgaa gtaataaatg agagaaattg ataaaaggaa gagagataaa aaaggtaaga 1020
ggagagaatg agagagatac tcagcaagag ctgctgaatt agataagttt ttgataacat 1080
taatcataca ttagaaatca agctcatagt taatatattc ctctaccaga ctctggcaag 1140
aaaatgtaaa catataaaat ggttataaca gaaaagggaa tccataacta atttctataa 1200
atcagacaat tcgttttatt ccctactcag acctggacca caagccaaca tcagattggt 1260
gtcagtaggg gctttgttta ctttatcctc aagctcatct ttcttatttt gagccttaat 1320
tgtggcattt cttggctttc tatatcatga ttattctttt cttgtagttc agtagataaa 1380
gtcataaaag agcaacattt aacagggtta atatacagct gtttcaaact actgattttt 1440
ttttctataa tgattttttt cccccagtgt aagaaagaac tttattgcat aaagaaacat 1500
gtacgtacct gaaatgttac ttatataatt tgcaaccaaa cacagaacac aacaaaggca 1560
tgaaaacgcc agcacaacct atgatacaag atgatagcag aaacagatgc aggaactgca 1620
accaaagctt aataccttct cttttaagtg ttagattatg atatcctaat atcattaagt 1680
gttagtttct ttttctgtca tggttaaatt atgatatctt aaatatcttt tcttttaagt 1740
cttaggtttc aaattgaatc tcaattgttt gatagatttt caataagaag atttactttg 1800
actgattgat gatttagtgt gattcgaaaa tgtcttgatc tcgaaccatg gtacaacatt 1860
cataaactgt ctgatagatt ttcatttttt ttttcttttt agaaatgaaa acaggtaaaa 1920
aaagtcctac caacttagta acctctcttc aaattggtat gattatatga attgatgtct 1980
agtttaagct atctttatta tctaagaaaa attgcgagta cataatttgc actcatgaga 2040
tgcgcgttct tttaagcatc aaacaattga atattttgtt gttcctttct tttcatttcc 2100
tttattccta acatagctga tgcagaaccc agaatcaata tatgtgataa aaaaccctag 2160
atatatatca atagtcatat tctaaagctc tcaagaaatt ctccgtactt gacctacatt 2220
caacgtgtga atatcaacca agataattga gatgaggatc tgaattgact aaatcctacg 2280
agaattatat cttgataagt aaaattaact caagggagat tgacaaagcg ccataagaac 2340
ttattttgat aagttgaatc aacctgttag tcagcaagat cgttgaagac accctcttac 2400
agattacaca caattagcaa agaggtaaaa acaaactcga atattttatt gaacttgaac 2460
ttgtgaatta cgaagggggt ggtggcattc ttatatagat ttccatccac ttattctact 2520
agaaaacgaa ttaaataatt ctaatcaaac tcttaatcct acttaaaaag aaatacgaac 2580
ttcctaattg aacacttaat ccaaatacaa ttagaaaact aaaaacttaa aaaattaaag 2640
acttacatta aaagattaaa ggatttctct tgggtggctt ttcaagattc ctatttggat 2700
tcaagctttt gtcttcctct cggttttagc ttctctttca tttaatgatt attttgctgc 2760
agatttcgga taggatttgt agacttcttg cggatttgta gtagaaaatg tgaagggaaa 2820
tttgataaga gtagtcttct cttggccggt tgcttcatgc ttctaattct ttccttctct 2880
tgatgatttc atcctgcgcc ttgaattcta gtttctacat catgtaggag tctgaattca 2940
acctgagaaa aagaaaagaa aagaagaaat acacacaaga aagacttgca gtaggagagg 3000
gaatccgatt ccctcttgga taaggtgtcc tgcgcggaat cctcctccaa ttcttcttct 3060
cgtgaattgg ctttatttta ggaccctttt gccgacttct tcttggacgt ttatttcttg 3120
taaaccgatc ttgaatcata gtaggaattc tgtcaaacct ttccaaagag aaaggacttt 3180
gcatttctta agaggtgaag gaacctgtgc gggcgggtct tgaatccctt cccttcgctc 3240
agccatgggc ccctacggtc aaaagcatgt gtggccgggc tgtatctccc tttcttggac 3300
cgattcttgg tagatcttgg actgctagac acgattcttt gttgaaatac tttagggcca 3360
agtgtttggg agcttggggg ctccctttga gtcagattct tgggtattga ccaaaagggc 3420
cttgaatctt tggttcttga ttcttggatg gatcttgagt gtaaaccggc ttgggacagt 3480
ttcttggctt gacaatccat tccttggagt gatggaaatc cgaaagatgg actccaaatt 3540
cccacaggat tgggatttcc cgtgagcatg agattttctt cgcgtggtct ctgcaggtgc 3600
gtgcgtgctg tgcgcgctca cgggtgtggg tgattccttg cccgcgggcc tgcttttctt 3660
gtctctccgg gacttgttgt tttccttatc taggatcaca tcaatagcgt ggaaagaggt 3720
ttaatacatg actacaaatg cacttatgac atatttatag tagaagcatc ttctctatct 3780
gaaacaggta aaagctcgga caactaccag aacaagtttg atcttgttaa cttctgagaa 3840
taaatgctct attaccactt gaactaccat tttagtagtt tatgtgtgat ctgtgatgga 3900
ttggactttt tgctttgaaa tattatgttg gactgttatt tgggttatgt ttgggaaaac 3960
ataaatttgt tgtattcatt tgaaaatttt atattatttt aatatgaaag agttaaagat 4020
cccacgtaac tacataagat gataaaattg acttgtatat tagacagttt aagtattcac 4080
tataatttat tttaatgata gccttaatct cttaattaat cctcatcatt tatataatga 4140
aaagaaattg gaattgggag ttgccttcat gtatttaatt tctgttacta ggtgggaata 4200
acatttatac tttctttgat ctctagaatt tatgtatcca aggatggtgt tgtatgaata 4260
cttctaatta attaaggtta tcttatcttc caacaattat atatatatgc attaatgtgg 4320
tagtgacttt gtgctctttg agcctttcga agcatgaagg aaatacttga aaggcatatg 4380
ttgcacgaaa agaaccttga gaagctggaa cagccatctc ttgaactaaa gctagttgag 4440
aatggcgacc gctccagatt gtgcaacgaa attgctcaga gaagccatca cttgaagcaa 4500
atgagggggg aggagctcca aggattaagc atagagcaac tgcagcagtt ggaaaaattc 4560
cttgaagtgg gattgcgccg tgttatagag agaaagggtc aaaatataat aaatgagatc 4620
agtgatcttc aaaggaaggg aatacaattg atggaagaga atgagaaact gaggcaacaa 4680
ttgttgcaga tatctaacaa aggaaaacaa ggtggtgcag agtcggagaa tgtagcgtct 4740
gaagagggcc agtcatcaga gtctgtaacc aatctctgca actctaatgg tccaccacac 4800
gactatgaaa gctcagatac ctctctaaag cttgggttac cctattccgg ctaaatcagg 4860
ggtaatgcca atgtcagaga gggtatcaaa agaagaaaaa gaaacatatg aatctgtatt 4920
aagttcacta tgttcatgtt tgcaagtaca gagtttgcca ttgaactaga gatacttatt 4980
tgtaattatt aatgttcaaa tctttaattt atggtctgaa ttttattaat aaaacgattc 5040
aaatcttaac ccaatcttat gggttaatcg taatttatta taattcataa aca 5093
<210> 56
<211> 166
<212> PRT
<213> 番木瓜(Carica papaya L.)
<220>
<223> SVP_HRM_88引物对对应的两性株H2 CpSVP-LIKE基因的转录组氨基酸序列
<400> 56
Met Lys Glu Ile Leu Glu Arg His Met Leu His Glu Lys Asn Leu Glu
1 5 10 15
Lys Leu Glu Gln Pro Ser Leu Glu Leu Lys Leu Val Glu Asn Gly Asp
20 25 30
Arg Ser Arg Leu Cys Asn Glu Ile Ala Gln Arg Ser His His Leu Lys
35 40 45
Gln Met Arg Gly Glu Glu Leu Gln Gly Leu Ser Ile Glu Gln Leu Gln
50 55 60
Gln Leu Glu Lys Phe Leu Glu Val Gly Leu Arg Arg Val Ile Glu Arg
65 70 75 80
Lys Gly Gln Asn Ile Ile Asn Glu Ile Ser Asp Leu Gln Arg Lys Gly
85 90 95
Ile Gln Leu Met Glu Glu Asn Glu Lys Leu Arg Gln Gln Leu Leu Gln
100 105 110
Ile Ser Asn Lys Gly Lys Gln Gly Gly Ala Glu Ser Glu Asn Val Ala
115 120 125
Ser Glu Glu Gly Gln Ser Ser Glu Ser Val Thr Asn Leu Cys Asn Ser
130 135 140
Asn Gly Pro Pro His Asp Tyr Glu Ser Ser Asp Thr Ser Leu Lys Leu
145 150 155 160
Gly Leu Pro Tyr Ser Gly
165
<210> 57
<211> 843
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SVP_HRM_88引物对对应的两性株H3 CpSVP-LIKE基因的转录组cDNA序列
<400> 57
tctaattaat taaggttatc ttatcttcca acaattatat atatatgcat taatgtggta 60
gtgactttgt gctctttgag cctttcgaag catgaaggaa atacttgaaa ggcatatgtt 120
gcacgaaaag aaccttgaga agctggaaca gccatctctt gaactaaagc tagttgagaa 180
tggcgaccgc tccagattgt gcaacgaaat tgctcagaga agccatcact tgaagcaaat 240
gaggggggag gagctccaag gattaagcat agagcaactg cagcagttgg aaaaattcct 300
tgaagtggga ttgcgccgtg ttatagagag aaagggtcaa aatataataa atgagatcag 360
tgatcttcaa aggaaggtca gacactaagg aatacaattg atggaagaga atgagaaact 420
gaggcaacaa ttgttgcaga tatctaacaa aggaaaacaa ggtggtgcag agtcggagaa 480
tgtagcgtct gaagagggcc agtcatcaga gtctgtaacc aatctctgca actctaatgg 540
tccaccacac gactatgaaa gctcagatac ctctctaaag cttgggttac cctattccgg 600
ctaaatcagg ggtaatgcca atgtcagaga gggtatcaaa agaagaaaaa gaaacatatg 660
aatctgtatt aagttcacta tgttcatgtt tgcaagtaca gagtttgcca ttgaactaga 720
gatacttatt tgtaattatt aatgttcaaa tctttaattt atggtctgaa ttttattaat 780
aaaacgattc aaatcttaac ccaatcttat gggttaatcg taatttatta taattcataa 840
aca 843
<210> 58
<211> 98
<212> PRT
<213> 番木瓜(Carica papaya L.)
<220>
<223> SVP_HRM_88引物对对应的两性株H3 CpSVP-LIKE基因的转录组氨基酸序列
<400> 58
Met Lys Glu Ile Leu Glu Arg His Met Leu His Glu Lys Asn Leu Glu
1 5 10 15
Lys Leu Glu Gln Pro Ser Leu Glu Leu Lys Leu Val Glu Asn Gly Asp
20 25 30
Arg Ser Arg Leu Cys Asn Glu Ile Ala Gln Arg Ser His His Leu Lys
35 40 45
Gln Met Arg Gly Glu Glu Leu Gln Gly Leu Ser Ile Glu Gln Leu Gln
50 55 60
Gln Leu Glu Lys Phe Leu Glu Val Gly Leu Arg Arg Val Ile Glu Arg
65 70 75 80
Lys Gly Gln Asn Ile Ile Asn Glu Ile Ser Asp Leu Gln Arg Lys Val
85 90 95
Arg His
<210> 59
<211> 2183
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_30702引物对对应的雌性株CpSERK基因的转录组cDNA序列
<400> 59
ccgagttatt aagagcacac acaaaggaga agtaaaattt ttttgttgac aaaaagcagc 60
agggagagaa aagagagaag ggagaagaga gaagtggagt tcaaacgaga aaatgttttg 120
agcaaaaaaa aaaaaatcat tccccgaatt ttgttctggt ttgtcatttt tctttttcca 180
ctgctgcgcg tgatattgat tttttttttg cttacctctt agcttcgtgt tgttaatgag 240
agagatcagg gggatgctgt gattgaggtg attttcgggc gataatggtg ggatttggga 300
gctagggttt tgctgctgtt ggtcatgttg atgttgggat gtatcttgtt gcttggatct 360
gagctgattg gagagaaaga gaagctgaca gtgtagtgta gtatgtcgga tatggaggcc 420
agagttgggg cttttagttt agccttcttg atcttcctgc tgcatccatt atggcttggc 480
tctgctaaca tggaaggtga tgcactacat agcctaagga ccaacctcaa tgatccaaac 540
aatgtcttac aaagttggga tcccactctt gtcaatcctt gcacatggtt ccatgttaca 600
tgcaacaatg ataatagtgt catacgagtt gatctaggga atgcggctct gtctggccaa 660
ttggttccac agcttggcct gcttaaaaat ttgcagtatt tggaactcta cagtaacaac 720
ataagtggac ctattccaag tgatcttggg aatctgacaa gcttggtgag cctggatctt 780
tacttgaaca gtttcagtgg tcccatcccg gattcattgg gaaaactctc gaaattacgc 840
tttcttcggc ttaacaacaa cagtctgaca ggtccaattc ccatgtcatt gactaatatc 900
tcatccctcc aagtactgga tctatcaaac aaccatctct ctggagtagt tcctgataat 960
ggctcctttt cactgttcac ccccattagt tttgccaaca acttggatct atgtggtccg 1020
gttactggac gcccctgccc aggatctcca ccattttctc ctcctccacc atttgttccc 1080
ccacctccaa tttcttcccc aggtgggaat agtgccactg gtgcaattgc tggaggagtt 1140
gctgctggtg ctgccttgct gtttgctgcc cctgccatag ctttcgcatg gtggcgtagg 1200
aggaaacctc aagaattctt ttttgatgta cctgctgaag aggatccaga agtccatctg 1260
gggcagctta agaggttttc attgcgggag ttacaagtcg caacagacag ttttagcaac 1320
aaaaacattt tgggtagagg aggatttggt aaggtttaca aaggtcgtct agctgatggt 1380
acactagtag ctgtaaaaag actgaaagaa gagcgtactc ctggtgggga gctgcagttt 1440
cagacagaag tagaaatgat aagcatggct gtccatcgaa atcttctccg attacgtgga 1500
ttttgtatga caccaactga gagattgctt gtttatccat atatggctaa tggcagtgtt 1560
gcatcatgtt taagagaacg tcctccatca caacctccac ttgattggcc aacgagaaag 1620
cggattgctt taggatctgc tagaggtctt tcttacttgc atgatcactg tgatccaaag 1680
atcattcacc gggacgtgaa agctgcaaac attttgttgg atgaggaatt tgaggctgtt 1740
gttggggact ttggcttggc taaacttatg gactacaaag atactcatgt tactactgct 1800
gtgcgtggta caattgggca tatagctccc gagtatctct caactggcaa atcctcagag 1860
aaaacagatg tttttgggta tggaatcatg cttcttgagc taatcactgg gcagagggca 1920
tttgatcttg cacggcttgc aaatgacgat gatgtcatgt tacttgattg ggtgaaaggg 1980
cttcttaagg agaaaaagtt agaaatgcta gttgatcctg acctccagag aaactacata 2040
gatgctgagg tagagcaatt aatccaggtt gcattgctat gcacacaagg ctccccaatg 2100
gatcgtccaa agatgtcaga agtggtcaga atgcttgaag gtgatgggtt ggccgagcga 2160
tgggacgagt ggcaaaaggt gga 2183
<210> 60
<211> 593
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_30702引物对对应的雌性株CpSERK基因的转录组氨基酸序列
<400> 60
Met Ser Asp Met Glu Ala Arg Val Gly Ala Phe Ser Leu Ala Phe Leu
1 5 10 15
Ile Phe Leu Leu His Pro Leu Trp Leu Gly Ser Ala Asn Met Glu Gly
20 25 30
Asp Ala Leu His Ser Leu Arg Thr Asn Leu Asn Asp Pro Asn Asn Val
35 40 45
Leu Gln Ser Trp Asp Pro Thr Leu Val Asn Pro Cys Thr Trp Phe His
50 55 60
Val Thr Cys Asn Asn Asp Asn Ser Val Ile Arg Val Asp Leu Gly Asn
65 70 75 80
Ala Ala Leu Ser Gly Gln Leu Val Pro Gln Leu Gly Leu Leu Lys Asn
85 90 95
Leu Gln Tyr Leu Glu Leu Tyr Ser Asn Asn Ile Ser Gly Pro Ile Pro
100 105 110
Ser Asp Leu Gly Asn Leu Thr Ser Leu Val Ser Leu Asp Leu Tyr Leu
115 120 125
Asn Ser Phe Ser Gly Pro Ile Pro Asp Ser Leu Gly Lys Leu Ser Lys
130 135 140
Leu Arg Phe Leu Arg Leu Asn Asn Asn Ser Leu Thr Gly Pro Ile Pro
145 150 155 160
Met Ser Leu Thr Asn Ile Ser Ser Leu Gln Val Leu Asp Leu Ser Asn
165 170 175
Asn His Leu Ser Gly Val Val Pro Asp Asn Gly Ser Phe Ser Leu Phe
180 185 190
Thr Pro Ile Ser Phe Ala Asn Asn Leu Asp Leu Cys Gly Pro Val Thr
195 200 205
Gly Arg Pro Cys Pro Gly Ser Pro Pro Phe Ser Pro Pro Pro Pro Phe
210 215 220
Val Pro Pro Pro Pro Ile Ser Ser Pro Gly Gly Asn Ser Ala Thr Gly
225 230 235 240
Ala Ile Ala Gly Gly Val Ala Ala Gly Ala Ala Leu Leu Phe Ala Ala
245 250 255
Pro Ala Ile Ala Phe Ala Trp Trp Arg Arg Arg Lys Pro Gln Glu Phe
260 265 270
Phe Phe Asp Val Pro Ala Glu Glu Asp Pro Glu Val His Leu Gly Gln
275 280 285
Leu Lys Arg Phe Ser Leu Arg Glu Leu Gln Val Ala Thr Asp Ser Phe
290 295 300
Ser Asn Lys Asn Ile Leu Gly Arg Gly Gly Phe Gly Lys Val Tyr Lys
305 310 315 320
Gly Arg Leu Ala Asp Gly Thr Leu Val Ala Val Lys Arg Leu Lys Glu
325 330 335
Glu Arg Thr Pro Gly Gly Glu Leu Gln Phe Gln Thr Glu Val Glu Met
340 345 350
Ile Ser Met Ala Val His Arg Asn Leu Leu Arg Leu Arg Gly Phe Cys
355 360 365
Met Thr Pro Thr Glu Arg Leu Leu Val Tyr Pro Tyr Met Ala Asn Gly
370 375 380
Ser Val Ala Ser Cys Leu Arg Glu Arg Pro Pro Ser Gln Pro Pro Leu
385 390 395 400
Asp Trp Pro Thr Arg Lys Arg Ile Ala Leu Gly Ser Ala Arg Gly Leu
405 410 415
Ser Tyr Leu His Asp His Cys Asp Pro Lys Ile Ile His Arg Asp Val
420 425 430
Lys Ala Ala Asn Ile Leu Leu Asp Glu Glu Phe Glu Ala Val Val Gly
435 440 445
Asp Phe Gly Leu Ala Lys Leu Met Asp Tyr Lys Asp Thr His Val Thr
450 455 460
Thr Ala Val Arg Gly Thr Ile Gly His Ile Ala Pro Glu Tyr Leu Ser
465 470 475 480
Thr Gly Lys Ser Ser Glu Lys Thr Asp Val Phe Gly Tyr Gly Ile Met
485 490 495
Leu Leu Glu Leu Ile Thr Gly Gln Arg Ala Phe Asp Leu Ala Arg Leu
500 505 510
Ala Asn Asp Asp Asp Val Met Leu Leu Asp Trp Val Lys Gly Leu Leu
515 520 525
Lys Glu Lys Lys Leu Glu Met Leu Val Asp Pro Asp Leu Gln Arg Asn
530 535 540
Tyr Ile Asp Ala Glu Val Glu Gln Leu Ile Gln Val Ala Leu Leu Cys
545 550 555 560
Thr Gln Gly Ser Pro Met Asp Arg Pro Lys Met Ser Glu Val Val Arg
565 570 575
Met Leu Glu Gly Asp Gly Leu Ala Glu Arg Trp Asp Glu Trp Gln Lys
580 585 590
Val
<210> 61
<211> 1952
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_30702引物对对应的雄性株CpSERK基因的转录组cDNA序列
<400> 61
caaaggagaa gtaaaatttt tttgttgaca aaaagcagca gggagagaaa agagagaagg 60
gagaagagag aagtggagtt caaacgagaa aatgttttga gcaaaaaaaa aaaaatcatt 120
ccccgaattt tgttctggtt tgtcattttt ctttttccac tgctgcgcgt gatattgatt 180
ttttttttgc ttacctctta gcttcgtgtt gttaatgaga gagatcaggg ggatgctgtg 240
attgaggtga ttttcgggcg ataatggtgg gatttgggag ctagggtttt gctgctgttg 300
gtcatgttga tgttgggatg tatcttgttg cttggatctg agctgattgg agagaaagag 360
aagctgacag tgtagtgtag tatgtcggat atggaggcca gagttggggc ttttagttta 420
gccttcttga tcttcctgct gcatccatta tggcttggct ctgctaacat ggaaggtgat 480
gcactacata gcctaaggac caacctcaat gatccaaaca atgtcttaca aagttgggat 540
cccactcttg tcaatccttg cacatggttc catgttacat gcaacaatga taatagtgtc 600
atacgagttg atctagggaa tgcggctctg tctggccaat tggttccaca gcttggcctg 660
cttaaaaatt tgcagtattt ggaactctac agtaacaaca taagtggacc tattccaagt 720
gatcttggga atctgacaag cttggtgagc ctggatcttt acttgaacag tttcagtggt 780
cccatcccgg attcattggg aaaactctcg aaattacgct ttcttcggct taacaacaac 840
agtctgacag gtccaattcc catgtcattg actaatatct catccctcca agtactggat 900
ctatcaaaca accatctctc tggagtagtt cctgataatg gctccttttc actgttcacc 960
cccattagtt ttgccaacaa cttggatcta tgtggtccgg ttactggacg cccctgccca 1020
ggatctccac cattttctcc tcctccacca tttgttcccc cacctccaat ttcttcccca 1080
ggtgggaata gtgccactgg tgcaattgct ggaggagttg ctgctggtgc tgccttgctg 1140
tttgctgccc ctgccatagc tttcgcatgg tggcgtagga ggaaacctca agaattcttt 1200
tttgatgtac ctgctgaaga ggatccagaa gtccatctgg ggcagcttaa gaggttttca 1260
ttgcgggagt tacaagtcgc aacagacagt tttagcaaca aaaacatttt gggtagagga 1320
ggatttggta aggtttacaa aggtcgtcta gctgatggta cactagtagc tgtaaaaaga 1380
ctgaaagaag agcgtactcc tggtggggag ctgcagtttc agacagaagt agaaatgata 1440
agcatggctg tccatcgaaa tcttctccga ttacgtggat tttgtatgac accaactgag 1500
agattgcttg tttatccata tatggctaat ggcagtgttg catcatgttt aagagaacgt 1560
cctccatcac aacctccact tgattggcca acgagaaagc ggattgcttt aggatctgct 1620
agaggtcttt cttacttgca tgatcactgt gatccaaaga tcattcaccg ggacgtgaaa 1680
gctgcaaaca ttttgttgga tgaggaattt gaggctgttg ttggggactt tggcttggct 1740
aaacttatgg actacaaaga tactcatgtt actactgctg tgcgtggtac aattgggcat 1800
atagctcccg agtatctctc aactggcaaa tcctcagaga aaacagatgt ttttgggtat 1860
ggaatcatgc ttcttgagct aatcactggg cagagggcat ttgatcttgc acggcttgca 1920
aatgacgatg atgtcatgtt acttgattgg gt 1952
<210> 62
<211> 523
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_30702引物对对应的雄性株CpSERK基因的转录组氨基酸序列
<400> 62
Met Ser Asp Met Glu Ala Arg Val Gly Ala Phe Ser Leu Ala Phe Leu
1 5 10 15
Ile Phe Leu Leu His Pro Leu Trp Leu Gly Ser Ala Asn Met Glu Gly
20 25 30
Asp Ala Leu His Ser Leu Arg Thr Asn Leu Asn Asp Pro Asn Asn Val
35 40 45
Leu Gln Ser Trp Asp Pro Thr Leu Val Asn Pro Cys Thr Trp Phe His
50 55 60
Val Thr Cys Asn Asn Asp Asn Ser Val Ile Arg Val Asp Leu Gly Asn
65 70 75 80
Ala Ala Leu Ser Gly Gln Leu Val Pro Gln Leu Gly Leu Leu Lys Asn
85 90 95
Leu Gln Tyr Leu Glu Leu Tyr Ser Asn Asn Ile Ser Gly Pro Ile Pro
100 105 110
Ser Asp Leu Gly Asn Leu Thr Ser Leu Val Ser Leu Asp Leu Tyr Leu
115 120 125
Asn Ser Phe Ser Gly Pro Ile Pro Asp Ser Leu Gly Lys Leu Ser Lys
130 135 140
Leu Arg Phe Leu Arg Leu Asn Asn Asn Ser Leu Thr Gly Pro Ile Pro
145 150 155 160
Met Ser Leu Thr Asn Ile Ser Ser Leu Gln Val Leu Asp Leu Ser Asn
165 170 175
Asn His Leu Ser Gly Val Val Pro Asp Asn Gly Ser Phe Ser Leu Phe
180 185 190
Thr Pro Ile Ser Phe Ala Asn Asn Leu Asp Leu Cys Gly Pro Val Thr
195 200 205
Gly Arg Pro Cys Pro Gly Ser Pro Pro Phe Ser Pro Pro Pro Pro Phe
210 215 220
Val Pro Pro Pro Pro Ile Ser Ser Pro Gly Gly Asn Ser Ala Thr Gly
225 230 235 240
Ala Ile Ala Gly Gly Val Ala Ala Gly Ala Ala Leu Leu Phe Ala Ala
245 250 255
Pro Ala Ile Ala Phe Ala Trp Trp Arg Arg Arg Lys Pro Gln Glu Phe
260 265 270
Phe Phe Asp Val Pro Ala Glu Glu Asp Pro Glu Val His Leu Gly Gln
275 280 285
Leu Lys Arg Phe Ser Leu Arg Glu Leu Gln Val Ala Thr Asp Ser Phe
290 295 300
Ser Asn Lys Asn Ile Leu Gly Arg Gly Gly Phe Gly Lys Val Tyr Lys
305 310 315 320
Gly Arg Leu Ala Asp Gly Thr Leu Val Ala Val Lys Arg Leu Lys Glu
325 330 335
Glu Arg Thr Pro Gly Gly Glu Leu Gln Phe Gln Thr Glu Val Glu Met
340 345 350
Ile Ser Met Ala Val His Arg Asn Leu Leu Arg Leu Arg Gly Phe Cys
355 360 365
Met Thr Pro Thr Glu Arg Leu Leu Val Tyr Pro Tyr Met Ala Asn Gly
370 375 380
Ser Val Ala Ser Cys Leu Arg Glu Arg Pro Pro Ser Gln Pro Pro Leu
385 390 395 400
Asp Trp Pro Thr Arg Lys Arg Ile Ala Leu Gly Ser Ala Arg Gly Leu
405 410 415
Ser Tyr Leu His Asp His Cys Asp Pro Lys Ile Ile His Arg Asp Val
420 425 430
Lys Ala Ala Asn Ile Leu Leu Asp Glu Glu Phe Glu Ala Val Val Gly
435 440 445
Asp Phe Gly Leu Ala Lys Leu Met Asp Tyr Lys Asp Thr His Val Thr
450 455 460
Thr Ala Val Arg Gly Thr Ile Gly His Ile Ala Pro Glu Tyr Leu Ser
465 470 475 480
Thr Gly Lys Ser Ser Glu Lys Thr Asp Val Phe Gly Tyr Gly Ile Met
485 490 495
Leu Leu Glu Leu Ile Thr Gly Gln Arg Ala Phe Asp Leu Ala Arg Leu
500 505 510
Ala Asn Asp Asp Asp Val Met Leu Leu Asp Trp
515 520
<210> 63
<211> 2392
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_30702引物对对应的两性株CpSERK基因的转录组cDNA序列
<400> 63
ggagaagtaa aatttttttg ttgacaaaaa gcagcaggga gagaaaagag agaagggaga 60
agagagaagt ggagttcaaa cgagaaaatg ttttgagcaa aaaaaaaaaa atcattcccc 120
gaattttgtt ctggtttgtc atttttcttt ttccactgct gcgcgtgata ttgatttttt 180
ttttgcttac ctcttagctt cgtgttgtta atgagagaga tcagggggat gctgtgattg 240
aggtgatttt cgggcgataa tggtgggatt tgggagctag ggttttgctg ctgttggtca 300
tgttgatgtt gggatgtatc ttgttgcttg gatctgagct gattggagag aaagagaagc 360
tgacagtgta gtgtagtatg tcggatatgg aggccagagt tggggctttt agtttagcct 420
tcttgatctt cctgctgcat ccattatggc ttggctctgc taacatggaa ggtgatgcac 480
tacatagcct aaggaccaac ctcaatgatc caaacaatgt cttacaaagt tgggatccca 540
ctcttgtcaa tccttgcaca tggttccatg ttacatgcaa caatgataat agtgtcatac 600
gagttgatct agggaatgcg gctctgtctg gccaattggt tccacagctt ggcctgctta 660
aaaatttgca gtatttggaa ctctacagta acaacataag tggacctatt ccaagtgatc 720
ttgggaatct gacaagcttg gtgagcctgg atctttactt gaacagtttc agtggtccca 780
tcccggattc attgggaaaa ctctcgaaat tacgctttct tcggcttaac aacaacagtc 840
tgacaggtcc aattcccatg tcattgacta atatctcatc cctccaagta ctggatctat 900
caaacaacca tctctctgga gtagttcctg ataatggctc cttttcactg ttcaccccca 960
ttagttttgc caacaacttg gatctatgtg gtccggttac tggacgcccc tgcccaggat 1020
ctccaccatt ttctcctcct ccaccatttg ttcccccacc tccaatttct tccccaggtg 1080
ggaatagtgc cactggtgca attgctggag gagttgctgc tggtgctgcc ttgctgtttg 1140
ctgcccctgc catagctttc gcatggtggc gtaggaggaa acctcaagaa ttcttttttg 1200
atgtacctgc tgaagaggat ccagaagtcc atctggggca gcttaagagg ttttcattgc 1260
gggagttaca agtcgcaaca gacagtttta gcaacaaaaa cattttgggt agaggaggat 1320
ttggtaaggt ttacaaaggt cgtctagctg atggtacact agtagctgta aaaagactga 1380
aagaagagcg tactcctggt ggggagctgc agtttcagac agaagtagaa atgataagca 1440
tggctgtcca tcgaaatctt ctccgattac gtggattttg tatgacacca actgagagat 1500
tgcttgttta tccatatatg gctaatggca gtgttgcatc atgtttaaga gaacgtcctc 1560
catcacaacc tccacttgat tggccaacga gaaagcggat tgctttagga tctgctagag 1620
gtctttctta cttgcatgat cactgtgatc caaagatcat tcaccgggac gtgaaagctg 1680
caaacatttt gttggatgag gaatttgagg ctgttgttgg ggactttggc ttggctaaac 1740
ttatggacta caaagatact catgttacta ctgctgtgcg tggtacaatt gggcatatag 1800
ctcccgagta tctctcaact ggcaaatcct cagagaaaac agatgttttt gggtatggaa 1860
tcatgcttct tgagctaatc actgggcaga gggcatttga tcttgcacgg cttgcaaatg 1920
acgatgatgt catgttactt gattgggtga aagggcttct taaggagaaa aagttagaaa 1980
tgctagttga tcctgacctc cagagaaact acatagatgc tgaggtagag caattaatcc 2040
aggttgcatt gctatgcaca caaggctccc caatggatcg tccaaagatg tcagaagtgg 2100
tcagaatgct tgaaggtgat gggttggccg agcgatggga cgagtggcaa aaggtggagg 2160
ttcttcgtca ggaggtggaa cttgctcctc atcctaattc tgattggatt gttgactcaa 2220
cagaaaatct acatgcagtt gagttatctg gtccaaggtg acctagcaca gctacagtaa 2280
aaagagaaga aaggtttact attttttttt tctcatctta attttcctcc ctttttcttt 2340
taaattaaat gaccactcat tttgtctcct gtaagcttag cttgcattgt at 2392
<210> 64
<211> 627
<212> DNA
<213> 番木瓜(Carica papaya L.)
<220>
<223> SERK_HRM_30702引物对对应的两性株CpSERK基因的转录组氨基酸序列
<400> 64
Met Ser Asp Met Glu Ala Arg Val Gly Ala Phe Ser Leu Ala Phe Leu
1 5 10 15
Ile Phe Leu Leu His Pro Leu Trp Leu Gly Ser Ala Asn Met Glu Gly
20 25 30
Asp Ala Leu His Ser Leu Arg Thr Asn Leu Asn Asp Pro Asn Asn Val
35 40 45
Leu Gln Ser Trp Asp Pro Thr Leu Val Asn Pro Cys Thr Trp Phe His
50 55 60
Val Thr Cys Asn Asn Asp Asn Ser Val Ile Arg Val Asp Leu Gly Asn
65 70 75 80
Ala Ala Leu Ser Gly Gln Leu Val Pro Gln Leu Gly Leu Leu Lys Asn
85 90 95
Leu Gln Tyr Leu Glu Leu Tyr Ser Asn Asn Ile Ser Gly Pro Ile Pro
100 105 110
Ser Asp Leu Gly Asn Leu Thr Ser Leu Val Ser Leu Asp Leu Tyr Leu
115 120 125
Asn Ser Phe Ser Gly Pro Ile Pro Asp Ser Leu Gly Lys Leu Ser Lys
130 135 140
Leu Arg Phe Leu Arg Leu Asn Asn Asn Ser Leu Thr Gly Pro Ile Pro
145 150 155 160
Met Ser Leu Thr Asn Ile Ser Ser Leu Gln Val Leu Asp Leu Ser Asn
165 170 175
Asn His Leu Ser Gly Val Val Pro Asp Asn Gly Ser Phe Ser Leu Phe
180 185 190
Thr Pro Ile Ser Phe Ala Asn Asn Leu Asp Leu Cys Gly Pro Val Thr
195 200 205
Gly Arg Pro Cys Pro Gly Ser Pro Pro Phe Ser Pro Pro Pro Pro Phe
210 215 220
Val Pro Pro Pro Pro Ile Ser Ser Pro Gly Gly Asn Ser Ala Thr Gly
225 230 235 240
Ala Ile Ala Gly Gly Val Ala Ala Gly Ala Ala Leu Leu Phe Ala Ala
245 250 255
Pro Ala Ile Ala Phe Ala Trp Trp Arg Arg Arg Lys Pro Gln Glu Phe
260 265 270
Phe Phe Asp Val Pro Ala Glu Glu Asp Pro Glu Val His Leu Gly Gln
275 280 285
Leu Lys Arg Phe Ser Leu Arg Glu Leu Gln Val Ala Thr Asp Ser Phe
290 295 300
Ser Asn Lys Asn Ile Leu Gly Arg Gly Gly Phe Gly Lys Val Tyr Lys
305 310 315 320
Gly Arg Leu Ala Asp Gly Thr Leu Val Ala Val Lys Arg Leu Lys Glu
325 330 335
Glu Arg Thr Pro Gly Gly Glu Leu Gln Phe Gln Thr Glu Val Glu Met
340 345 350
Ile Ser Met Ala Val His Arg Asn Leu Leu Arg Leu Arg Gly Phe Cys
355 360 365
Met Thr Pro Thr Glu Arg Leu Leu Val Tyr Pro Tyr Met Ala Asn Gly
370 375 380
Ser Val Ala Ser Cys Leu Arg Glu Arg Pro Pro Ser Gln Pro Pro Leu
385 390 395 400
Asp Trp Pro Thr Arg Lys Arg Ile Ala Leu Gly Ser Ala Arg Gly Leu
405 410 415
Ser Tyr Leu His Asp His Cys Asp Pro Lys Ile Ile His Arg Asp Val
420 425 430
Lys Ala Ala Asn Ile Leu Leu Asp Glu Glu Phe Glu Ala Val Val Gly
435 440 445
Asp Phe Gly Leu Ala Lys Leu Met Asp Tyr Lys Asp Thr His Val Thr
450 455 460
Thr Ala Val Arg Gly Thr Ile Gly His Ile Ala Pro Glu Tyr Leu Ser
465 470 475 480
Thr Gly Lys Ser Ser Glu Lys Thr Asp Val Phe Gly Tyr Gly Ile Met
485 490 495
Leu Leu Glu Leu Ile Thr Gly Gln Arg Ala Phe Asp Leu Ala Arg Leu
500 505 510
Ala Asn Asp Asp Asp Val Met Leu Leu Asp Trp Val Lys Gly Leu Leu
515 520 525
Lys Glu Lys Lys Leu Glu Met Leu Val Asp Pro Asp Leu Gln Arg Asn
530 535 540
Tyr Ile Asp Ala Glu Val Glu Gln Leu Ile Gln Val Ala Leu Leu Cys
545 550 555 560
Thr Gln Gly Ser Pro Met Asp Arg Pro Lys Met Ser Glu Val Val Arg
565 570 575
Met Leu Glu Gly Asp Gly Leu Ala Glu Arg Trp Asp Glu Trp Gln Lys
580 585 590
Val Glu Val Leu Arg Gln Glu Val Glu Leu Ala Pro His Pro Asn Ser
595 600 605
Asp Trp Ile Val Asp Ser Thr Glu Asn Leu His Ala Val Glu Leu Ser
610 615 620
Gly Pro Arg
625
<210> 65
<211> 266
<212> PRT
<213> 番木瓜(Carica papaya)
<220>
<223> MADS-box蛋白
<400> 65
Met Phe Gln Ser Arg Leu Ile Phe Asn Asn Phe Leu Leu Leu Leu Leu
1 5 10 15
Phe Glu Ile Tyr Arg Tyr Ile Tyr Thr Tyr Thr Gln Thr Thr Lys Gly
20 25 30
Lys Arg Arg Arg Glu Glu Arg Met Ala Arg Glu Lys Ile Gln Ile Lys
35 40 45
Lys Ile Asp Asn Thr Ser Ala Arg Gln Val Thr Phe Ser Lys Arg Arg
50 55 60
Arg Gly Leu Phe Lys Lys Ala Glu Glu Leu Ser Val Leu Cys Asp Ala
65 70 75 80
Asp Val Ala Leu Ile Val Phe Ser Ser Thr Gly Lys Leu Phe Glu Tyr
85 90 95
Ser Ser Ser Ser Met Lys Glu Ile Leu Glu Arg His Met Leu His Glu
100 105 110
Lys Asn Leu Glu Lys Leu Glu Gln Pro Ser Leu Glu Leu Lys Leu Val
115 120 125
Glu Asn Gly Asp Arg Ser Arg Leu Cys Asn Glu Ile Ala Gln Arg Ser
130 135 140
His His Leu Lys Gln Met Arg Gly Glu Glu Leu Gln Gly Leu Ser Ile
145 150 155 160
Glu Gln Leu Gln Gln Leu Glu Lys Phe Leu Glu Val Gly Leu Arg Arg
165 170 175
Val Ile Glu Arg Lys Gly Gln Asn Ile Ile Asn Glu Ile Ser Asp Leu
180 185 190
Gln Arg Lys Gly Ile Gln Leu Met Glu Glu Asn Glu Lys Leu Arg Gln
195 200 205
Gln Leu Leu Gln Ile Ser Asn Lys Gly Lys Gln Gly Gly Ala Glu Ser
210 215 220
Glu Asn Val Ala Ser Glu Glu Gly Gln Ser Ser Glu Ser Val Thr Asn
225 230 235 240
Leu Cys Asn Ser Asn Gly Pro Pro His Asp Tyr Glu Ser Ser Asp Thr
245 250 255
Ser Leu Lys Leu Gly Leu Pro Tyr Ser Gly
260 265
- 还没有人留言评论。精彩留言会获得点赞!