一种与水稻稻瘟病抗性基因紧密连锁的分子标记、引物及其应用的制作方法
本发明涉及生物技术领域,具体地说涉及一种与水稻稻瘟病抗性基因紧密连锁的分子标记、引物及其应用。
背景技术:
基于表型选择的常规育种方法存在选择效率低和育种周期长等缺点,迫切需要注入现代分子技术手段,辅之于高效率地基因型定向选择,才能快速高效地培育出优异水稻新品种。随着分子生物学和基因组学的迅速发展,分子标记技术的应用更加广泛。基于pcr的分子标记如微卫星或ssr(simplesequencerepeat)等具有多态率高、相对稳定、检测方法简便快速及易于操作等特点而被广泛的应用。
由于分子标记辅助选择不易受环境因素影响和性状显隐性干扰等,可以从分子水平定向选择目标性状基因,同时还有可能打破不利基因之间的连锁而高效率地聚合多个优良基因于一体。通常所说的分子标记既包括目标基因的连锁标记也包括目标基因自身功能标记。分子标记辅助多基因聚合育种技术已经成为水稻、玉米等作物育种研究的一种发展趋势,运用该技术能否有效地将优质、多抗和高产等基因聚合到少数骨干品种中,关键在于能否获得与目标性状基因或主效qtl紧密连锁的实用分子标记。
稻瘟病是水稻重要病害之一,可引起大幅度减产,严重时减产40%~50%,甚至颗粒无收。世界各稻区均匀发生。本病在各地均有发生,其中以叶部、节部发生为多,发生后可造成不同程度减产,尤其穗颈瘟或节瘟发生早而重,可造成白穗以致绝产。所以研究水稻茎腐病病原和发病规律,培育抗病品种,减少水稻产量损失有重要意义。
防治稻瘟病主要有两种方法,一是使用化学农药,因成本高、污染环境,不可持续;二是发掘新的抗病资源并选育广谱抗病新品种。据介绍,目前已有25个抗稻瘟病基因被克隆和功能鉴定,但绝大部分抗谱窄,容易丧失抗病性;将多种抗病基因整合到一个品种,不仅难度大周期长,还会导致作物产量和品质降低。
何祖华团队从2002年开始,广泛筛选抗瘟种质,从起源于我国农家品种的育种材料中鉴定了一个广谱抗瘟性新位点pigm。随后他们又用了10年时间,系统解析了这个新位点的功能机制。研究发现,pigm中有2个发挥功能的蛋白pigmr和pigms。pigmr对所有检测的稻瘟病菌小种都具有广谱抗病性,但同时会使水稻种子变小,产量降低;pigms会抑制pigmr的抗病功能,却可以提高水稻产量。pigmr和pigms这对“蛋白兄弟”紧密连锁在染色体的一个小区段内,不能分开,因此选育的品种就既有广谱抗病性又不影响最终的产量(epigeneticregulationofantagonisticreceptorsconfersriceblastresistancewithyieldbalance[j].2017.355(6328):962)。
技术实现要素:
本发明所要解决的技术问题是提供一种与水稻稻瘟病抗性基因紧密连锁的分子标记,扩增该分子标记的引物对以及检测水稻稻瘟病抗性的方法。
为了解决上述技术问题,本发明采用如下技术方案:一种与水稻稻瘟病抗性基因紧密连锁的分子标记,所述分子标记的核苷酸序列如seqidno:1所示或seqidno:2所示。
一种扩增上述分子标记的引物对,包括引物1和引物2,所述引物1的核苷酸序列如seqidno:3所示,所述引物2的核苷酸序列如seqidno:4所示。
一种检测水稻稻瘟病抗性的方法,包括如下步骤:
(1)pcr扩增:以从待测水稻中提取的基因组dna作为dna扩增模板,以seqidno:3所示的核苷酸序列和seqidno:4所示的核苷酸序列组成为扩增引物对进行pcr扩增;
(2)扩增产物的鉴定:若所得pcr扩增产物为272bp碱基片段,则待测水稻为抗稻瘟病品种,若所得pcr扩增产物为252bp碱基片段,则待测水稻为稻瘟病感病品种。
进一步地,步骤(2)中,所述272bp碱基片段为seqidno:1所示的核苷酸序列,所述252bp碱基片段为seqidno:2所示的核苷酸序列。
进一步地,步骤(1)中,从待测水稻中提取的基因组dna的提取方法为碱煮法,具体步骤为:取1/10的水稻籽粒胚乳,加入40ml的0.2mol/l的氢氧化钠水溶液,在100℃条件下水浴3分钟,再加入60ml的0.17mol/l的tris-hcl,在100℃条件下水浴1分钟。
进一步地,步骤(1)中,pcr扩增的反应体系为:含mg2+的10xbuffer2μl,10mm的dntp0.4μl,5μm的如seqidno:3所示的核苷酸序列和5μm的如seqidno:4所示的核苷酸序列各2μl,从待测水稻中提取的基因组dna1μl,5u/μl的taq酶0.5μl,其余为超纯水,反应体积为20μl,滴加一滴矿物油覆盖;pcr扩增的反应程序为:94℃5min;94℃60s,53.5℃60s,72℃60s,35个循环;72℃10min。
本发明还提供上述分子标记、引物对以及检测水稻稻瘟病抗性的方法在水稻育种中的应用。
本发明的有益效果体现在:
本发明分子标记位于抗性基因pigm的基因内部,与水稻稻瘟病抗性基因紧密连锁,与非基因内部标记相比,本发明分子标记不会出现分离,可以更有效地提高育种效率。
本发明引物对可以很好地扩增经过简单处理所得到的从待测水稻中提取的基因组,克服了现有技术中与该基因紧密连锁的其他分子标记无法扩增经过简单处理所得到的从待测水稻中提取的基因组的问题,获得本发明检测水稻稻瘟病抗性的方法,该方法能够对播种前的水稻种粒进行快速基因鉴定筛选,然后有选择性地播种,减少用工用地,缩短育种周期,进一步提高育种效率。
本发明检测水稻稻瘟病抗性的方法克服了常规育种中因表型选择易受环境因素干扰而导致育种效率低等问题,也克服了分子标记辅助育种中,由于分子标记和抗稻瘟病基因有一定的遗传距离,在育种过程中存在分离的可能性的问题,本发明检测水稻稻瘟病抗性的方法利用本发明引物对对水稻稻瘟病抗性基因进行早期世代选择而缩短育种周期,从而较快地筛选出抗稻瘟病的水稻材料。本发明可应用于水稻育种,以提高育种效率和水稻产量水平。
附图说明
图1是用seqidno:5所示的核苷酸序列和seqidno:6所示的核苷酸序列组成的引物对扩增抗病品种谷梅4和不抗品种fd1715后,扩增产物碱基片段对比图。
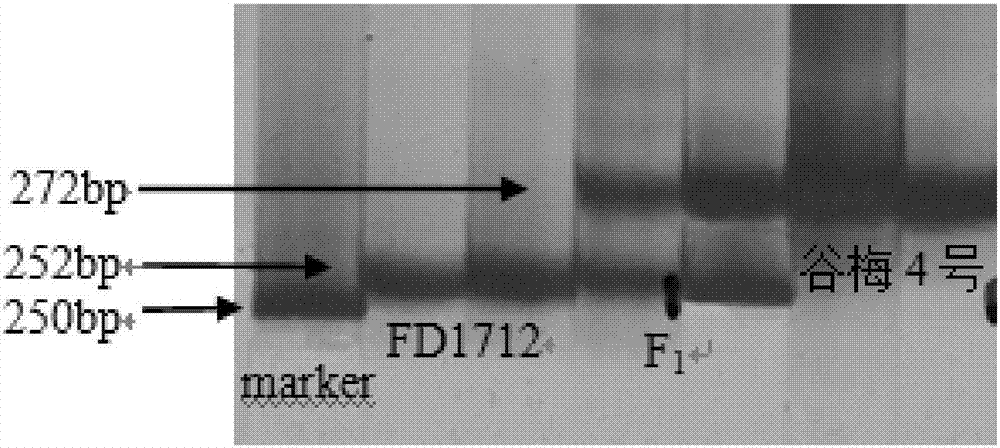
图2是seqidno:3所示的核苷酸序列和seqidno:4所示的核苷酸序列组成的引物对扩增不抗品种fd1715、抗病品种谷梅4和以及两者杂交种的电泳图。
具体实施方式
以下的实施例便于更好地理解本发明,但并不限定本发明。下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的试验材料,如无特殊说明,均由常规生化试剂公司购买得到。
实施例1
稻瘟病抗性性状鉴定专用引物对seqidno:3所示的核苷酸序列和seqidno:4所示的核苷酸序列的获得
1.1双亲基因组扩增
用于创建水稻重组自交系(recombinantinbredline,ril)群体的双亲分别是谷梅4和安徽丰大种业股份有限公司选育的fd1715。fd1715为母本,不抗稻瘟病;谷梅4为父本,高抗稻瘟病。
采用ctab法提取双亲叶片的基因组dna,以yiwendeng(yiwendeng,etal.epigeneticregulationofantagonisticreceptorsconfersriceblastresistancewithyieldbalance[j].2017.355(6328):962)公布的序列,设计引物,由seqidno:5所示的核苷酸序列和seqidno:6所示的核苷酸序列组成,委托北京梓熙生物公司合成后,进行pcr扩增实验。pcr扩增的反应体系为:10xbuffer2μl(含mg2+),dntp0.4μl(10mm),seqidno:5所示的核苷酸序列和seqidno:6所示的核苷酸序列各2μl(5μm),基因组dna1μl(40ng/μl),taq酶0.5μl(5υ/μl),其余为超纯水,反应体积为20μl,滴加一滴矿物油覆盖。pcr扩增的反应程序为:94℃5min;94℃60s,53.5℃60s,72℃60s,35个循环;72℃10min。
pcr扩增产物送去测序,测序公司为北京梓熙生物公司,测序结果在ncbi网站的在线比对结果见图1。图中虚线区域表示20bp的缺失,其中query是谷梅4父本,高抗稻瘟病的序列;sbjct是fd1715母本,不抗稻瘟病的序列。
1.2引物合成和验证
根据图1的测序比对结果,在跨越两个序列有20bp插入缺失差异区域设计引物,上游引物为seqidno:3所示的核苷酸序列,下游引物为seqidno:4的所示核苷酸序列。委托北京梓熙生物公司合成。上游引物和下游引物(将该引物对命名为qk1)位于基因pigm内部功能区域。
利用新合成的引物扩增谷梅4和fd1715,以及两者的杂交种。采用碱煮法从待测水稻中提取基因组dna,具体步骤为:取1/10的水稻籽粒胚乳,加入40ml的0.2mol/l的氢氧化钠水溶液,在100℃条件下水浴3分钟,再加入60ml的0.17mol/l的tris-hcl,在100℃条件下水浴1分钟,即提取得到待测水稻的基因组dna,4℃冰箱冷却备用。
pcr扩增的反应体系为:10xbuffer2μl(含mg2+),dntp0.4μl(10mm),上游引物和下游引物各2μl(5μm),从待测水稻中提取的基因组dna1μl,taq酶0.5μl(5u/μl),其余为超纯水,反应体积为20μl,滴加一滴矿物油覆盖。pcr扩增的反应程序为:94℃5min;94℃60s,53.5℃60s,72℃60s,35个循环;72℃10min。
pcr扩增产物用6%聚丙烯酰胺凝胶电泳分析,快速银染法染色并观察拍照,见图2,图中fd1715、谷梅4和杂交种每个材料各2个重复,marker大小为250bp。对pcr扩增产物进行测序,从扩增产物的电泳图可以清晰地看出,谷梅4有一条电泳带,经测序所述电泳带对应的扩增产物为272bp碱基片段,为seqidno:1所示的核苷酸序列;fd1715有一条电泳带,经测序所述电泳带对应的扩增产物为252bp碱基片段,为seqidno:2所示的核苷酸序列;杂交品种有二条电泳带,经测序所述电泳带对应的扩增产物为272bp和252bp碱基片段,为seqidno:1所示的核苷酸序列和seqidno:2所示核苷酸序列。
实施例2
qk1的扩增产物与水稻稻瘟病抗性性状的相关性验证
以高抗稻瘟病材料谷梅4和不抗稻瘟病材料fd1715做亲本,两者杂交后的432个f2代材料为试验对象。采用碱煮法从待测水稻中提取基因组dna,具体步骤同与实施例1。进行pcr扩增实验。pcr反应体系为:10xbuffer2μl(含mg2+),dntp0.4μl(10mm),seqidno:3所示的核苷酸序列和seqidno:4所示的核苷酸序列各2μl(5μm),从待测水稻中提取的基因组dna1μl,taq酶0.5μl(5u/μl),其余为超纯水,反应体积为20μl,滴加一滴矿物油覆盖。pcr扩增的反应程序为:94℃5min;94℃60s,53.5℃60s,72℃60s,35个循环;72℃10min。
pcr扩增产物用6%聚丙烯酰胺凝胶电泳分析,快速银染法染色并观察拍照记录,其中纯合的272bp的105株,纯合252bp的97株,杂合带型的220株。
室内接种材料包括fd1715、谷梅4号、两者的杂交株。种子浸种催芽后分别播种于32孔塑料连版中,育苗基质为盆栽花卉培养基,幼苗在26℃-28℃的人工气候室内12h光照、12h黑暗培养至三到四叶期。采用单个生理小种约1×105/ml浓度的稻瘟菌孢子悬浮液活体喷雾接种,于26℃黑暗保湿24h后,继续保持光照高湿5-7d,参照bonman的0-5级标准调查病情(0-2级为抗病,3-5级为感病)。
分析如下:如下表1所示,供试水稻样本中存在纯合272bp的105株,其中102株表现为抗病,有3株感病;而不带有272bp片段的97个材料中92株感病,有5株抗病;杂合的220株,216株表现为抗病,4株感病;两者的一致性达到97.1%。因此,可以得出结论,seqidno:3所示的核苷酸序列和seqidno:4所示的核苷酸序列组成的qk1引物对是与稻瘟病抗性性状相关位点相紧密连锁的分子标记,可用于水稻分子辅助育种。
表1qk1扩增产物鉴定结果与田间稻瘟病接种试验对照
应当理解本文所述的例子和实施方式仅为了说明,并不用于限制本发明,本领域技术人员可根据它做出各种修改或变化,凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
sequencelisting
序列表
<110>安徽丰大种业股份有限公司
<120>一种与水稻稻瘟病抗性基因紧密连锁的分子标记、引物及其应用
<160>6
<170>patentinversion3.1
<210>1
<211>272
<212>dna
<213>水稻(zeamaysl.)
<220>
<221>gene
<222>(1)..(272)
<223>
<400>1
ggacctaaccgatgcaacacggattagatatgataatctaatactcgatgagccaataga60
tctgtctaatgtgatggatataacaaatctatttataacagcattgcgattgtagagata120
tatcggctaagacagaatatcagacctaactaaaccgatgcgtctctaaacacaatgcaa180
ttaattagagatataattgagatatcagctaggcaaatatatcaaccaaactggagcgat240
ccaagagatcggagcaatgcagccttgaacaa272
<210>2
<211>480
<212>dna
<213>水稻(zeamaysl.)
<220>
<221>gene
<222>(1)..(252)
<223>
<400>2
ggacctaaccgatgcaacacggattagatatgataatctaatactcgatgagccaataga60
tctgtctaatgtgatggatataacaaatctatttataacagcattgcgaagacagaatat120
cagacctaactaatgggatgcgtctctaaacacaatgcaattaattagagatataattga180
gatatcagctaggcaaatatatcaaccaaactggagcgatccaagagatcggagcaatgc240
agccttgaacaa252
<210>3
<211>20
<212>dna
<213>人工序列
<220>
<221>primer_bind
<222>(1)..(20)
<223>
<400>3
ggacctaaccgatgcaacac20
<210>4
<211>20
<212>dna
<213>人工序列
<220>
<221>primer_bind
<222>(1)..(20)
<234>
<400>4
ttgttcaaggctgcattgct20
<210>5
<211>20
<212>dna
<213>人工序列
<220>
<221>primer_bind
<222>(1)..(20)
<223>
<400>5
gccgatgccgattctagccg20
<210>6
<211>22
<212>dna
<213>人工序列
<220>
<221>primer_bind
<222>(1)..(20)
<234>
<400>6
gagttcaaggaggatacgac20
- 还没有人留言评论。精彩留言会获得点赞!
- 水稻稻瘟病抗性基因Pi2‑1的分子标记方法及应用与流程
- 一种稻瘟病抗性基因Piz‑t功能特异性分子标记及其检测方法和应用与流程
- 一种接种架及稻瘟病菌接种系统的制作方法与工艺
- 谷梅4号抗稻瘟病基因Pigm的分子标记及其应用的制作方法与工艺
- 水稻稻瘟病抗性基因Pita、Pib分子标记方法与流程
- 一种基于猪非典型瘟病毒E2基因的荧光定量RT‑PCR检测试剂盒的制作方法与工艺
- 稻瘟病菌LAMP检测引物组及应用的制造方法与工艺
- 一种制备稻瘟病菌促分裂原活化蛋白激酶MoMps1晶体的方法与流程
- 水稻抗稻瘟病Pi9基因的共显性功能分子标记Pi9InDel1及其应用的制造方法与工艺
- 水稻抗稻瘟病Pi9基因的共显性功能分子标记Pi9InDel2及其应用的制造方法与工艺
- 小麦白粉病抗性基因PmR2及其克隆与应用的制造方法与工艺
- 一种受茉莉酸甲酯和白叶枯病菌诱导的启动子、其制备方法和重组表达载体与流程
- MALDI‑TOFMS技术鉴定临床感染猪肠球菌的方法与流程
- 一种稻瘟病抗性基因Pi2的功能性分子标记及其应用的制造方法与工艺
- 一种与白叶枯病抗性基因Xa7紧密连锁的SNP分子标记及其应用的制造方法与工艺
- 一种与白叶枯病抗性基因Xa7紧密连锁的SNP分子标记及其应用的制造方法与工艺
- 一种水稻穗瘟抗性基因的分子标记方法与制造工艺
- OsSAPK9蛋白及其编码基因在提高水稻白叶枯病抗性中的应用的制造方法与工艺
- 一种硫链丝菌素‑庆大霉素抗性基因体系及还有其的抗性表达盒和重组质粒的制造方法与工艺
- 含抗PD‑1基因和聚亚精胺的复合物及其在治疗肿瘤中的应用的制造方法与工艺