一种单核苷酸多态性位点在辅助鉴定大豆花叶病毒抗性中的应用的制作方法
本发明涉及大豆抗病育种和分子生物学技术领域,具体涉及一种与大豆花叶病毒病相关snp位点的应用及大豆花叶病毒抗性的检测方法。
背景技术:
大豆花叶病毒(soybeanmosaicvirus,smv)是一种世界性大豆病害,对大豆生产影响最大的病毒之一,严重影响大豆的产量和品质。南京农业大学将我国的smv株系划分为sc1-sc21,其中sc3和sc7是黄淮和长江流域大豆产区最流行的株系。大豆花叶病毒分布广、危害大,化学药剂难以防治,培育和筛选抗病品种是解决大豆病毒性的最有效经济途径。
单核苷酸多态性(singlenucleotidepolymorphism,snp),是指在基因组水平上由单个核苷酸的变异所引起的dna序列多态性,snp所表现的多态性只涉及到单个碱基的变异,这种变异可由单个碱基的转换(transition)或颠换(transversion)所引起,也可由碱基的插入或缺失所致,但通常所说的snp并不包括后两种情况。
大豆基因组虽然早在2010年已经完成测序,但各个基因的功能并没有完全阐明。如果能从中找出与大豆花叶病毒抗性相关的位点,并利用该位点开发分子标记,利用分子标记辅助育种,将能大大提高育种效率。近年,单核苷酸多态性(snp)越来越受到科研人员的重视,同时也发现许多snp与作物的重要农艺性状和抗性具有一定的关联性,程浩(2008)博士毕业论文(大豆异黄酮合酶,黄烷酮3-羟化酶基因snps与种子异黄酮含量及抗逆性的关联分析)报道称大豆ifs1,ifs2和f3h基因中都存在与大豆对花叶病毒抗性显著相关的snps。如ifs1基因中与大豆对smvsc-3与smvsc-7抗性显著相关的snps分别为14和2个;在ifs2基因中发现与大豆对smvsc-3与smvsc-7抗性显著相关的为indel(1bp)。在f3h基因中发现与大豆对smvsc-3和sc-7抗性显著相关的snps为7个和1个。因此,发掘和利用大豆中重要的snp位点具有重要的现实和理论意义。
技术实现要素:
本发明的目的是提供一种glyma.07g250300中与大豆花叶病毒病相关snp位点发掘及应用,本发明的目的可以通过以下技术实现:
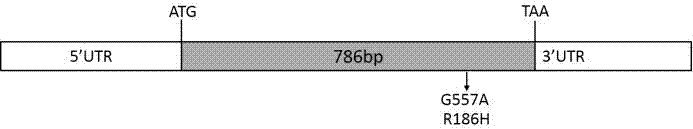
一种单核苷酸多态性位点在辅助鉴定大豆花叶病毒抗性中的应用,该点位于大豆基因组glyma.07g250300基因编码区开放阅读框第557位。
进一步,上述单核苷酸多态性位点的应用是指:对大豆基因组glyma.07g250300基因编码区开放阅读框第557位核苷酸进行基因型鉴定,若第557位核苷酸残基为“a”,则认为大豆材料抗花叶病毒;若第557位核苷酸残基为“g”,则认为大豆材料对花叶病毒敏感。
进一步,上述单核苷酸多态性位点的应用,其具体步骤如下:
1)根据大豆基因组glyma.07g250300基因编码区内开放阅读框(orf)设计上游引物和下游引物,并以大豆基因组dna为模板,进行pcr扩增;
2)对pcr扩增产物进行测序,若glyma.07g250300基因编码区内开放阅读框第557位核苷酸为“a”,则认为大豆材料抗花叶病毒;若第557位核苷酸为“g”,则认为该大豆材料对花叶病毒敏感。
进一步,上述单核苷酸多态性位点的应用中,所述上游引物位于开放阅读框
(5‵→3‵)第71位核苷酸残基开始,第90位核苷酸残基结束;下游引物从开放读框第635位核苷酸残基开始,第657位核苷酸残基结束。
进一步,所述上游引物和下游引物核苷酸序列分别如seqidno.1和seqidno.2所示;
pcr扩增体系:2×phantamaxbuffer25μl;dntpmix(10mmeach)1μl;上游引物4μl(10μm);下游引物4μl(10μm);phantamaxsuper-fidelitydnapolymerase(1u/μl);模板dna4μl(20ng/μl);ddh2o补足到50μl;
pcr扩增程序:95℃预变性3min;然后95℃变性15s,56℃退火45s,72℃延伸30s,35个循环;最后72℃延伸10min,4℃保温;
pcr反应结束后,将所得pcr产物(786bp)进行1%琼脂糖凝胶电泳,然后测序。
一种检测单核苷酸多态性或基因型的物质在制备大豆花叶病毒抗性检测试剂中的应用,所述检测单核苷酸多态性或基因型的物质是指:检测大豆基因组中glyma.07g250300基因编码区开放阅读框第557位单核苷酸多态性或基因型的物质。
进一步,所述检测单核苷酸多态性或基因型的物质是指根据大豆基因组中glyma.07g250300基因编码区开放阅读框所设计的上游引物和下游引物,所述大豆花叶病毒抗性检测试剂是指pcr检测试剂。
进一步,该pcr检测试剂(总体积50μl)包括:2×phantamaxbuffer25μl;dntpmix(10mmeach)1μl;核苷酸序列如seqidno.1所示的上游引物4μl(10μm);核苷酸序列如seqidno.2所示的下游引物4μl(10μm);phantamaxsuper-fidelitydnapolymerase(1u/μl)1μl;模板dna4μl(20ng/μl);ddh2o为11μl。其中模板dna为大豆基因组dna。
进一步,本申请中,所述大豆花叶病毒为sc3株系或sc7株系。
本发明对重测序的302份大豆材料,分析glyma.07g250300基因座的开放阅读框(orf)内含有1个有义突变snp位点,即第557位核苷酸位置有g/a(r/h)两种类型;对9个材料的glyma.07g250300序列进行测定发现bb52和sa101材料中glyma.07g250300基因编码区第557位核苷酸为“a”,其余大豆材料为“g”;利用南方普遍流行的两种病毒株系sc3和sc7分别对所用大豆材料分别接种鉴定,发现bb52和sa101对两种病毒同时表现出抗性;而其余材料则均对两种病毒表现出敏感性,结合snp位点结果,bb52和sa101在第557位a碱基,而对两种病毒表现出抗性,其它材料均为g碱基,而表现出敏感性,为大豆抗病毒病分子标记辅助育种奠定了基础。
在具体应用中,可以根据大豆基因组中glyma.07g250300基因开放阅读框第557位核苷酸设计引物并制备大豆花叶病毒抗性检测试剂(如pcr试剂),或以其他本领域常规方法对该位点的基因型进行检测,若该位点基因型为“g”,则大豆材料表现出敏感性,当位点基因型为“a”时,则大豆材料表现出抗性;通过上述方法,在大豆苗期即可对大豆材料的花叶病毒抗性进行评价,以提高抗病育种效率,在实际育种和生产中具有广阔的应用前景。
附图说明
图1为大豆基因组glyma.07g250300基因编码区snp位点分析结果示意图。
图2大豆基因组glyma.07g250300基因序列在不同大豆材料中的多重比对分析结果示意图。
具体实施方式
下述实施例中所使用的试验方法无特殊说明,均为常规方法。实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
1、实施例所用大豆材料
以下实施例所用大豆材料包括willimas82(对应序列为gmzfp3),野生大豆bb52(gszfp3),sa001,sa002,sa025,sa062,sa101,sa162,sa189,sa242,willimas82,bb52为江苏省农业科学院农业资源与环境研究所盐土农业研究中心保存,其它材料均由南京农业大学大豆研究所提供。
2.以下实施例涉及到生物试剂及引物合成与测序
dna提取试剂盒和dna凝胶回收试剂盒购于axygen公司;
高保真dna聚合酶phantamaxsuper-fidelitydnapolymerase购于vazyme公司;
takaradl2000bpdnamarker购于takara公司;
实施例所使用的pcr仪为lifetechnologies的proflexpcrsystem;所用引物和测序均由南京擎科生物有限公司合成和完成。
实施例涉及的引物序列:
seqidno.1:catgccaaaatcctgaacct;
seqidno.2:tggaacactatcagccaaagat;
实施例1大豆基因glyma.07g250300中snp位点的发掘
glyma.07g250300在大豆基因组数据库(网址
实施例2大豆材料中glyma.07g250300基因编码序列分析
试验大豆材料为:willimas82(对应序列为gmzfp3);野生大豆bb52(gszfp3),sa001,sa002,sa025,sa062,sa101,sa162,sa189,sa242和willimas82。
分别利用ctab法(十六烷基三甲基溴化铵)提取上述10种类大豆(glycinemax(l.)merr.)品种幼苗叶片中基因组dna;分别以该dna为模板,用引物对进行pcr扩增,所用引物序列(用于扩增glyma.07g250300的开放阅读框)为:
正向引物(f)如seqidno.1所示,反向引物如seqidno.2所示;
pcr反应体系:2×phantamaxbuffer25μl;dntpmix(10mmeach)1μl;上下游引物(f/r)4μl(10μm);phantamaxsuper-fidelitydnapolymerase(1u/μl);模板dna4μl(20ng/μl);ddh2o补足到50μl。
pcr扩增程序为95℃预变性3min;然后95℃变性15s,56℃退火45s,72℃延伸30s,35个循环;最后72℃延伸10min,4℃保温。
pcr反应结束后,将所得pcr产物(786bp)进行1%琼脂糖凝胶电泳。片段回收送南京擎科生物有限公司测序,每条序列测定3次。
利用clustalx软件对测序结果进行分析,发现野生大豆bb52和sa101材料中,glyma.07g250300基因编码区第557位核苷酸为“a”,其余大豆材料的glyma.07g250300基因编码区均为“g”(如图2所示)。
实施例3大豆材料的花叶病毒病抗性鉴定
利用南方普遍流行的两种病毒株系sc3和sc7(南京农业大学大豆研究所提供)分别对表1所列大豆材料分别接种鉴定,鉴定方法与评价指标(抗与敏)参照南京农业大学2000年王修强硕士毕业论文“黄淮和长江中下游地区大豆花叶病毒(smv)株系鉴定、抗源筛选及抗性遗传的研究”。
表1大豆花叶病毒鉴定材料与结果
结果表明,bb52和sa101对两种病毒同时表现出抗性,而sa062仅对sc3表现抗性,对sc7为敏感;而其余材料则均对两种病毒表现出敏感性(表1)。结合实施例2的snp位点结果,bb52和sa101在第557位a碱基,而对两种病毒表现出抗性,其它材料均为g碱基,而表现出敏感性;对sa062材料而言,基因序列为g碱基,但对一种病毒表现出了抗性,推断可能大豆中还有其它抗病毒病位点。
由此可以推断,glyma.07g250300基因中的g/asnp位点可能在大豆的抗病毒病过程中起着重要作用。抗性鉴定实验由南京农业大学大豆研究所完成。
以上所述,仅用以说明本发明的技术方案而非限制,本领域普通技术人员对本发明的技术方案所做的其它修改或者等同替换,只要不脱离本发明技术方案的精神和范围,均应涵盖在本发明的权利要求范围当中。
sequencelisting
<110>江苏省农业科学院
<120>一种单核苷酸多态性位点在辅助鉴定大豆花叶病毒抗性中的应用
<130>2
<160>2
<170>patentinversion3.3
<210>1
<211>20
<212>dna
<213>人工合成
<400>1
catgccaaaatcctgaacct20
<210>2
<211>22
<212>dna
<213>人工合成
<400>2
tggaacactatcagccaaagat22
- 还没有人留言评论。精彩留言会获得点赞!
- CYP2C9*3基因多态性快速检测的引物、分子信标、试剂盒及其检测方法与流程
- 一种单核苷酸多态性位点在辅助鉴定大豆花叶病毒抗性中的应用的制造方法与工艺
- DHFR基因多态性检测体系及其试剂盒的制造方法与工艺
- ERCC1基因多态性检测体系及其试剂盒的制造方法与工艺
- 一种判定中国人群rs125055单核苷酸多态性与儿童IBD之间的相关性的方法与制造工艺
- 黄牛ADNCR基因单核苷酸多态性的四引物扩增受阻突变体系PCR检测方法及其应用与制造工艺
- 使用PNA探针的熔解曲线分析、使用熔解曲线分析用于分析核苷酸多态性的方法和试剂盒与制造工艺
- 人KRAS中单核苷酸多态性的检测的制造方法与工艺
- 一种InDel位点基因型分型的方法与制造工艺
- 单核苷酸多态性rs145562243在筛查麻风患者中的应用的制造方法与工艺
- 一种用于评估皮肤防御情况的引物和探针及试剂盒的制作方法
- 一种用于评估皮肤防晒情况的引物和探针及试剂盒的制作方法
- 一种用于评估皮肤抗皱情况的引物和探针及试剂盒的制作方法
- 人PLIN1基因rs2289487位点多态性检测方法及试剂盒的制作方法
- 测定人NEFM基因rs12515位点多态性的方法及试剂盒的制作方法
- 一种人TERF1基因rs3863242位点多态性检测技术的制作方法
- 测定人CCNH基因rs3093816位点多态性的方法及试剂盒的制作方法
- 测定人NEFL基因rs2979704位点多态性的方法及试剂盒的制作方法
- 测定人PON1基因rs854560位点多态性的方法及试剂盒的制作方法
- 测定人PON1基因rs662位点多态性的方法及试剂盒的制作方法
- CYP2C9*3基因多态性快速检测的引物、分子信标、试剂盒及其检测方法与流程
- 一种单核苷酸多态性位点在辅助鉴定大豆花叶病毒抗性中的应用的制造方法与工艺
- DHFR基因多态性检测体系及其试剂盒的制造方法与工艺
- ERCC1基因多态性检测体系及其试剂盒的制造方法与工艺
- 一种判定中国人群rs125055单核苷酸多态性与儿童IBD之间的相关性的方法与制造工艺
- 黄牛ADNCR基因单核苷酸多态性的四引物扩增受阻突变体系PCR检测方法及其应用与制造工艺
- 使用PNA探针的熔解曲线分析、使用熔解曲线分析用于分析核苷酸多态性的方法和试剂盒与制造工艺
- 人KRAS中单核苷酸多态性的检测的制造方法与工艺
- 一种InDel位点基因型分型的方法与制造工艺
- 单核苷酸多态性rs145562243在筛查麻风患者中的应用的制造方法与工艺
- 用于检测人类CYP2D6基因多态性的核酸、试剂盒及方法与流程
- 单核苷酸多态性位点rs2523607在筛查疱疹样皮炎患者中的应用的制作方法与工艺
- 核苷酸多态性检测方法与流程
- 单核苷酸多态性rs146466242在筛查麻风患者中的应用的制作方法与工艺
- 基于信号谱差异的混合样本单核苷酸多态性的检测方法与流程
- 一种检测牛PCAF基因单核苷酸多态性的RFLP方法及试剂盒与流程
- 秦川牛microRNA‑320a‑1基因单核苷酸多态性的检测方法及其应用与流程
- 单核苷酸多态性rs76418789在筛查麻风患者中的应用的制作方法与工艺
- 判定rs4676410和rs2531875单核苷酸多态性与AS之间相关性的方法与流程
- 一种可视化检测基因序列中单核苷酸多态性位点的反向探针、试剂盒及其检测方法与流程