一种合成桧烯的基因工程菌及其构建方法与应用与流程
本发明涉及基因工程技术领域,具体涉及一种合成桧烯的基因工程菌及其构建方法与应用。
背景技术:
桧烯(c10h16)是一种天然的双环单萜,可以从多种植物精油中分离得到。桧烯是一种重要的高密度燃料前体,可以作为航空燃料的添加剂,在医药、食品、能源等领域中,桧烯都具有重要的应用前景,近年来受到人们越来越多的关注。桧烯在植物中的含量非常低,从植物中提取需要耗费大量的植物材料,经济成本较高,既费时费力又破坏环境,远远不能满足市场的需要。
随着合成生物学的不断发展,以微生物作为细胞工厂合成桧烯成为一种可持续、快速、绿色的生产方法。然而,目前,在微生物细胞中生产桧烯并不理想,利用微生物生产桧烯一直缓慢前行,未能实行工业化生产。现有报道,在大肠杆菌(escherichiacoli)中以游离的方式表达桧烯合成酶,利用葡萄糖生产桧烯,产量最高达到82.18mg/l(zhangh,liuq,caoy,fengx,zhengy,zouh,liuh,yangj,xianm.2014.microbialproductionofsabinene—anewterpene-basedprecursorofadvancedbiofuel.microbialcellfactories.13:20)。但是,以质粒形式存在的游离基因在发酵过程中不稳定,易丢失,不利于大规模的发酵生产。
除大肠杆菌外,酿酒酵母是生产萜烯类产品的优良菌株。酿酒酵母具有成熟的真核表达系统,拥有天然的甲羟戊酸(mva)途径,为异源萜类化合物的合成提供了直接前体香叶基焦磷酸(gpp),因此酿酒酵母细胞具有合成异源萜类化合物的天然优势。目前,igneaetal.以半乳糖为碳源在酿酒酵母中生产桧烯,最高产量为17.5mg/l(igneac,pontinim,maffeime,makrisam,kampranissc.2014.engineeringmonoterpeneproductioninyeastusingasyntheticdominantnegativegeranyldiphosphatesynthase.acssynthbiol3(5):298–306)。然而半乳糖价格较高,发酵成本过高,不适于大规模发酵生产,这就限制了其应用。
技术实现要素:
本发明针对上述问题,提供了一种合成桧烯的基因工程菌及其构建方法与应用。
本发明通过crispr-cas9系统对酿酒酵母基因组进行编辑,导入经过密码子优化的桧烯合成酶基因sabs1,使上述基因整合到酵母染色体,通过该基因在酿酒酵母(saccharomycescerevisiae)cen.pk2-1c中异源过量表达,将gpp转化成桧烯从而实现桧烯的生物合成。
一种合成桧烯的基因工程菌,其特征在于,该基因工程菌的出发菌株为酵母菌,所述酵母菌敲除了gal80基因,含有经过密码子优化的桧烯合成酶基因sabs1,genbank登录号为dq785794.1,其序列如seq1所示,所述桧烯合成酶基因sabs1整合在该酵母菌株染色体gal80位点。
优选地:
本发明所述的一种合成桧烯的基因工程菌,其特征在于,所述酵母菌为酿酒酵母(saccharomycescerevisiae),基因型为:cen.pk2-1c(mata;ura3-52;trp1-289;leu2-3_112;his3δ1;mal2-8c;suc2)。
所述酵母菌还含有如下序列:
桧烯合成酶基因sabs1的启动子-gal1启动子,记为pgal1,其序列如seq2所示;
桧烯合成酶基因sabs1的终止子-cyc1终止子,记为tcyc1,其序列如seq3所示;
gal80基因的起始密码子上游的511bp,记为gal80us,其序列如seq4所示;
gal80基因的终止密码子下游的501bp,记为gal80ds,其序列如seq5所示。
本发明所用菌株酿酒酵母(saccharomycescerevisiae),记载在vandijkenetal.,2000.aninterlaboratorycomparisonofphysiologicalandgeneticpropertiesoffoursaccharomycescerevisiaestrains.enzymemicrob.technol.,26(2000),pp.706-714,自申请日起二十年内公众可通过中国科学院青岛生物能源与过程研究所生物基材料实验室获得该菌株。
本发明的另一目的是提供一种合成桧烯的基因工程菌的构建方法。
构建方法如下:
(1)将gal80基因的起始密码子上游的511bp,与终止密码子下游501bp以及pgal1-sabs1-tcyc1片段连接,构建供体dna片段:gal80us-pgal1-sabs1-tcyc1-gal80ds;
(2)将pml04质粒用限制性内切酶swai和bcli进行双酶切,获得线性化的pml104质粒;
(3)以序列taaggctgctgctgaacgt为靶标序列,构建grna的表达片段;
(4)将步骤(1)-步骤(3)所述的供体dna片段、线性化的pml104质粒和grna表达片段同时转入酵母感受态细胞(saccharomycescerevisiae)cen.pk2-1c,得到酵母基因工程菌。
更进一步地,所述构建方法包括如下步骤:
(1)先将经过密码子优化的桧烯合成酶基因sabs1连接到商品质粒pesc-his,得到重组质粒pesc-his-sabs1,通过pcr获得pgal1-sabs1-tcyc1片段;然后将gal80基因的起始密码子上游511bp、pgal1-sabs1-tcyc1片段、终止密码子下游501bp依次连接到商品质粒puc19,得到重组质粒puc19-gal80us-pgal1-sabs1-tcyc1-gal80ds,最后通过pcr获得供体dna片段:gal80us-pgal1-sabs1-tcyc1-gal80ds;
(2)将pml04质粒先用限制性内切酶swai酶切7h,再加入限制性内切酶bcli酶切4h,将酶切产物通过凝胶回收,获得线性化的pml104质粒;
(3)以靶标序列taaggctgctgctgaacgt为中心,分别构建与质粒pml104同源的grna表达片段的上下游两个片段,再通过重叠pcr将两个片段融合到一起获得完整的grna表达片段;
(4)将步骤(1)-步骤(3)所述的供体dna片段、线性化的pml104质粒和grna表达片段同时转入酵母感受态细胞(saccharomycescerevisiae)cen.pk2-1c,培养后获得酵母基因工程菌,其中用量为:供体dna片段400-1000ng、线性化的pml104质粒100-150ng、grna表达片段200-600ng。
本发明利用crispr-cas9系统结合酵母体内同源重组技术,对酵母基因组进行编辑。本发明将桧烯合成酶基因sabs1整合到gal80位点,同时将gal80基因敲除,而不引入其它标记基因;将线性化的含有cas9基因的质粒pml104、含有同源臂的供体dna与grna表达片段共同转入酵母细胞,在酵母中表达cas9蛋白,同时利用5-foa筛选质粒丢失的转化子酵母菌株。
本发明的再一目的是提供上述合成桧烯的基因工程菌在合成桧烯中的应用。
进一步地:
利用本发明所获得的基因工程菌经一级种子培养基培养后,获得的二级种子接种到二级种子培养基中进行发酵得到桧烯。
所述一级种子培养基为ypd培养基:20g/l葡萄糖、10g/l酵母粉、20g/l蛋白胨,其余为水;二级种子培养基的碳源为蔗糖、氮源为酵母粉,ph为5。
二级种子培养基优选为:20g/l蔗糖、10g/l酵母粉、1.5g/lkh2po4、1.0g/lmgso4,其余为水。
所述二级种子初始od600为0.5,振荡培养36h-48h。
本发明的有益效果:
1.本发明利用crispr-cas9系统结合酵母体内同源重组技术,将线性化的含有cas9基因的质粒与含有桧烯合成酶基因sabs1的供体dna片段、grna表达片段共同转入酵母细胞,一步法实现无筛选标记的基因整合及敲除的菌株构建,获得合成桧烯的基因工程菌,并利用该菌株发酵生产桧烯,相比于游离基因以质粒形式表达并生产桧烯,其生产稳定性更强。
2.本发明合成桧烯的方法成本更低,产量更高。现有报道采用价格较高的半乳糖为碳源合成桧烯的方法,合成桧烯最高产量为17.5mg/l,本发明无需使用半乳糖作为碳源,而是以蔗糖为碳源,蔗糖价格低廉,具有可再生性,能够持续、稳定、高效的生产高密度燃料桧烯,合成的桧烯产量可达23.6mg/l,提高了1.3倍。
3.本发明所述的基因工程菌适合于工程酵母菌的中等至大规模培养的任何液体培养基,优选为ypd液体培养基。
4.本发明提供的合成桧烯的生产工艺不涉及高温、高压操作,经济、绿色,获得的产物桧烯组分单一,纯度高,无其他杂质出现,从根本上解决了目前桧烯从植物或精油中分离提取的不足,提供了一条可持续利用的方法合成桧烯,有效缓解高密度燃料行业原料不足的问题。
附图说明
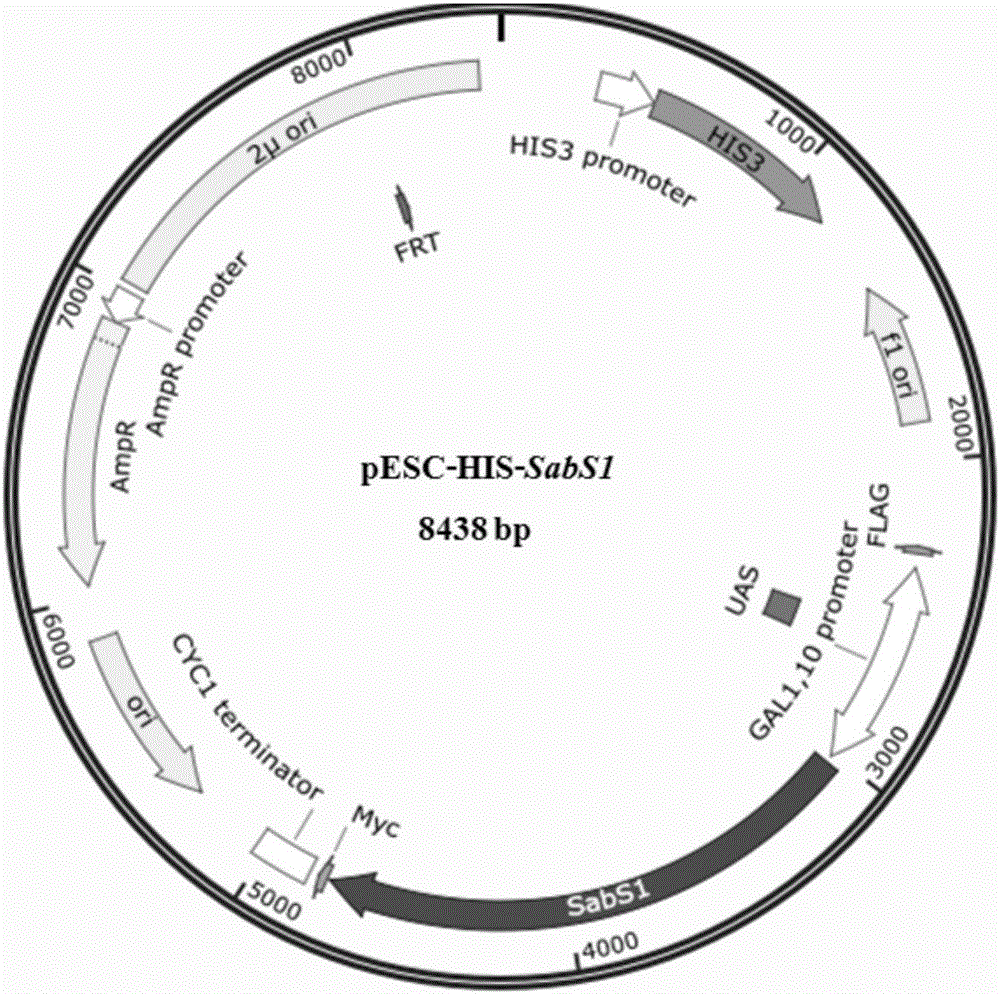
图1为质粒pesc-his-sabs1示意图。
图2为质粒puc19-gal80us-pgal1-sabs1-tcyc1-gal80ds示意图。
图3为质粒pml104示意图。
图4为桧烯生物合成代谢途径示意图。
图5为酵母基因工程菌合成桧烯的气相-质谱检测图,其中a为气相图谱,横坐标为时间,纵坐标pa为皮安,表示检测器的响应值;b为质谱图谱,横坐标为分子质量,纵坐标为m/z的强度。
具体实施方式
下面结合具体实施例对本发明做进一步说明,但本发明不受实施例的限制。实施例中所用质粒pesc-his、质粒pml104、感受态细胞、引物和试剂等均可通过商业途径购买获得或通过本领域技术人员所熟知的常规手段获得。
实施例1.本实施例所述为一种合成桧烯的基因工程菌的构建方法,包括如下步骤:
(1)含有gal80基因的上下游同源臂:启动子为gal1启动子,终止子为cyc1终止子的桧烯合成酶的供体dna片段:gal80us-pgal1-sabs1-tcyc1-gal80ds的构建;
通过分子生物学相关实验操作构建供体dna片段gal80us-pgal1-sabs1-tcyc1-gal80ds,该片段含有如下基因片段:经过密码子优化的桧烯合成酶基因sabs1,该基因来源于鼠尾草(salviapomifera),序列登记号为genbank:dq785794.1,其序列如seq1所示;gal1启动子,其序列如seq2所示;cyc1终止子,其序列如seq3所示;gal80的上游(记为gal80us)511bp,其序列如seq4所示;gal80的下游(记为gal80ds)501bp,其序列如seq5所示,gal80us与gal80ds为同源臂片段。
上述基因片段pcr扩增引物分别为:
经过密码子优化的桧烯合成酶基因sabs1:正向引物为sabs1-bamhi-f,反向引物为sabs1-sali-r;
gal80的上游(gal80us)511bp基因片段:正向引物为gal80us-f,反向引物为gal80us-r;
gal80的下游(gal80ds)501bp基因片段:正向引物为gal80ds-f,反向引物为gal80ds-r;
引物序列见表1所示。
以上基因序列均能够在ncbi数据库中查询获得,因此可以采用基因合成或选择合适的基因组dna作为模板pcr扩增获得,以上方法属于成熟的分子生物学方法,不存在特殊性。
获得相应基因片段后,用限制性内切酶bamhi和sali对质粒pesc-his和pcr产物sabs1基因进行双酶切,通过t4dna连接酶将sabs1基因和pesc-his质粒连接,取适量体积连接液转化大肠杆菌感受态细胞,涂布带有amp抗性的lb平板,37℃过夜培养。
对阳性转化子的酶切验证方法如下:挑取5-10个已长出的细菌菌落,将其转接到新鲜的lb培养基中,添加适当浓度的抗生素,37℃、180rpm振荡培养过夜,利用质粒提取试剂盒提取质粒,分别用不同的限制性内切酶进行单酶切和双酶切鉴定,以验证重组质粒的正确性,得到重组质粒pesc-his-sabs1,该质粒图谱如图1所示。
通过pcr获得pgal1-sabs1-tcyc1片段,正向引物为pgal1-sabs1-tcyc1-f,反向引物为pgal1-sabs1-tcyc1-r,引物序列见表1所示,pcr产物经限制性内切酶ndei和psti双酶切,酶切产物连接到puc19质粒中,再通过ndei和psti将gal80us、gal80ds依次连入pgal1-sabs1-tcyc1的两端,得到重组质粒puc19-gal80us-pgal1-sabs1-tcyc1-gal80ds,质粒图谱如图2所示,最后通过pcr获得供体dna片段:gal80us-pgal1-sabs1-tcyc1-gal80ds,所用引物为正向引物为gal80us-f,反向引物为gal80ds-r。
以上质粒构建方法涉及到pcr基因克隆技术、核酸合成技术、质粒限制性酶切技术、酶切片段回收技术、酶切片段连接技术等,以上方法属于成熟的分子生物学方法,不存在特殊性。
(2)将pml04质粒先用限制性内切酶swai酶切7h,再加入限制性内切酶bcli酶切4h,将酶切产物通过凝胶回收,获得线性化的pml104质粒。
(3)含有19bptaaggctgctgctgaacgt的gal80靶序列的grna表达片段的构建:
通过设计引物时引入19bp的靶标序列taaggctgctgctgaacgt,以该靶标序列为中心,以线性化的pml104为模板,pcr扩增与质粒pml104同源的grna表达片段的上下游各500bpdna序列,两个上下游片段均含有19bp的gal80的靶标序列,再通过重叠pcr将两个片段融合到一起获得完整的grna表达片段,其19bp的靶序列位于grna表达片段的中心。
grna表达片段的上游500bp:正向引物为gal80-grna-us-f,反向引物为gal80-grna-us-r;
grna表达片段的下游500bp:正向引物为gal80-grna-ds-f,反向引物为gal80-grna-ds-r。
引物序列见表1所示,pml104质粒图谱如图3所示。
(4)将步骤(1)-步骤(3)所述的供体dna片段、grna表达片段和线性化的pml104质粒同时转入酿酒酵母感受态细胞(saccharomycescerevisiae)cen.pk2-1c,其中用量为:供体dna片段500ng、grna表达片段300ng,线性化质粒pml104100ng,30℃培养2天后,通过pcr鉴定得到正确菌株,获得酵母基因工程菌。所用pcr鉴定用引物为:test-sabs1-f,test-sabs1-r;test-gal80-f,test-gal80-r,引物序列见表1所示。
所述pcr验证可以通过菌落pcr、菌液pcr或菌落培养后提取基因组pcr等方法验证均可。
本实施例获得的基因工程菌,以酵母为出发菌株,该菌株含有经过密码子优化的桧烯合成酶基因sabs1,该基因整合在该酵母菌株染色体gal80基因位点,genbank登录号为dq785794.1,其序列如seq1所示,同时该菌株敲除了gal80基因。
在本发明所述的重组酵母菌株构建方法中,相关质粒的构建以及感受态细胞的转化方法不受限制,可以采用本领域中已知的常规方法,即利用化学转化法将单个或多个片段转入酿酒酵母感受态细胞中,采用营养缺陷筛选平板对阳性转化子进行筛选。
实施例2.本实施所述为一种合成桧烯的应用。
酵母中含有天然的甲羟戊酸(mva)途径,为异源单萜类化合物的合成提供了直接前体香叶基焦磷酸gpp,gpp进一步转化即获得终产物桧烯,其生物合成代谢途径如图4所示。
本实施例所述的合成桧烯的应用是指利用本发明实施例1中构建的酵母基因工程菌,在含有合适碳源,如以蔗糖为原料、氮源及其他生长因子的条件下,所述酵母基因工程菌株能够转化发酵液中的糖类生产桧烯,利用气相色谱等检测设备,从尾气或发酵液中能够检测到桧烯。
菌种活化:取-80℃保存的菌种,在ypd平板上划线,在30℃培养箱静置培养2天,挑取单菌落接种于液体培养基ypd中30℃、180rpm摇床培养活化菌种,获得一级种子。
本发明所述的合成桧烯的酵母基因工程菌能够适合于工程酵母菌的中等至大规模培养的任何液体培养基,优选为ypd液体培养基,其配方为:20g/l葡萄糖、10g/l酵母粉、20g/l蛋白胨,其余为水。
菌体发酵:选取生长良好的活化菌株转接到含有50ml的ypd液体培养基的三角瓶中,180rpm,30℃培养24h,获得二级种子,按培养基体积5%的菌液接种量,将二级种子接种到发酵培养基内,培养基:20g/l蔗糖、10g/l酵母粉、1.5g/lkh2po4、1g/lmgso4、其余为水,ph=5。控制发酵瓶的装液量不超过瓶总体积的1/10~1/5,以保证菌体的生长处于有氧条件。将发酵瓶转入30℃、160rpm~180rpm的培养箱中振荡培养36~48h,利用气相色谱gc或气相色谱-质谱联用仪gc-ms对产物桧烯进行定性定量检测。
桧烯产物检测:通过气相色谱gc或气相色谱-质谱联用仪gc-ms对发酵产物桧烯进行分析测定。gc检测系统为sp-6890型气相色谱仪,购自山东鲁南瑞虹化工仪器有限公司,毛细管色谱柱agilenthp-innowax,规格为30m×0.25mm×0.25μm。升温程序为:初始柱温为75℃,维持0.5min;以10℃/min升温速度升至100℃,维持5min;检测器温度240℃,气化室温度为220℃。
实施例3.本实施例是对实施例2的进一步限定,本实施例所述的一种合成桧烯的应用,所述菌体发酵:选取生长良好的活化菌株转接到含有50ml的ypd液体培养基的三角瓶中,180rpm,30℃培养24h,获得二级种子,按培养基体积5%的菌液接种比例,将二级种子接种到发酵培养基内,其初始od为0.5,培养基:20g/l蔗糖、10g/l酵母粉、1.5g/lkh2po4、1g/lmgso4、其余为水,ph=5。控制发酵瓶的装液量不超过瓶总体积的1/10,以保证菌体的生长处于有氧条件。将发酵瓶转入30℃、180rpm的培养箱中振荡培养36h,利用气相色谱gc或气相色谱-质谱联用仪gc-ms对产物桧烯进行定性定量检测。
桧烯产物检测:通过气相色谱gc或气相色谱-质谱联用仪gc-ms对发酵产物桧烯进行分析测定。gc检测系统为sp-6890型气相色谱仪,购自山东鲁南瑞虹化工仪器有限公司,毛细管色谱柱agilenthp-innowax,规格为30m×0.25mm×0.25μm。升温程序为:初始柱温为75℃,维持0.5min;以10℃/min升温速度升至100℃,维持5min;检测器温度240℃,气化室温度为220℃。桧烯的产物鉴定结果如图5-a和图5-b所示。
根据图5可知,本实施例利用实施例1获得的酵母基因工程菌,经过发酵培养后,该菌株合成的桧烯,根据气相检测结果,产量为23.6mg/l。
表1引物序列表
虽然本发明已以较佳的实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明精神和范围内,都可以做各种的改动与修饰,因此,本发明的保护范围应该以权利要求书所界定的为准。
sequencelisting
<110>中国科学院青岛生物能源与过程研究所
<120>一种合成桧烯的基因工程菌及其构建方法与应用
<130>
<160>21
<170>patentinversion3.5
<210>1
<211>1746
<212>dna
<213>seq1
<400>1
atgccgctgaactctctgcacaacctggaacgtaaaccgtctaaagcgtggtctacctct60
tgcaccgctccggctgctcgtctccaggctagcttctctctgcaacaggaagaaccgcgt120
cagatccgtcgttctggtgactaccagccgtctctgtgggacttcaactacatccagtct180
ctgaacaccccgtacaaagaacagcgttacgttaaccgtcaggctgaactgatcatgcag240
gttcgtatgctgctgaaagttaaaatggaagctatccagcagctggaactgatcgacgac300
cttcagtacctgggtctgtcttacttcttcccggacgaaatcaaacagatcctgtcttct360
atccacaacgaacaccgttacttccacaacaacgacctgtacctgactgcgctgggcttc420
cgtatcctgcgtcaacacggcttcaacgtaagcgaggacgtattcgactgcttcaagacc480
gaaaaatgctctgacttcaacgctaacctggctcaggacaccaaaggtatgctgcaactg540
tacgaagcgtctttcctgctgcgtgaaggtgaagacaccctggaactggctcgtcgtttc600
tctacccgttctctgcgtgaaaaactggacgaagacggtgacgaaatcgacgaagacctg660
tcttcttggatccgtcactctctggacctgccgctgcactggcgtatccagggtctggaa720
gctcgttggttcctggacgcttacgctcgtcgtccggacatgaacccgctgatcttcaaa780
ctggctaaactgaacttcaacatcgttcaggctacctaccaggaagaactgaaagacgtt840
tctcgttggtggaactcttcttgcctggctgaaaaactgccgttcgttcgtgaccgtatc900
gttgaatgcttcttctgggctatcggtgctttcgaaccgcaccagtattcttaccagcgt960
aaaatggctgctatcatcatcaccttcgttaccatcatagacgatgtgtacgacgtgtac1020
gggaccctggaagaactggaactgttcaccgacatgatccgtcgttgggacaacatctct1080
atctctcagctgccgtactacatgcaggtttgctacctggctctgtacaacttcgtttct1140
gaacgtgcttacgacatcctgaaagaccagcacttcaactctatcccgtaccttcagcgt1200
tcttgggtttctctggttgaaggttacctgaaagaagcgtactggtactacaacggttac1260
aaaccgtctctggaagaatacctgaacaacgctaaaatctctatctctgctccgaccatc1320
atctctcagctgtacttcaccctggctaactctaccgacgaaaccgttatcgaatctctg1380
tacgaataccacaacatcctgtacctgtctggcaccatcctgcgtctggctgacgacctg1440
ggcacctctcagcacgaactggaacgtggtgacgttccgaaagctatccagtgctacatg1500
aaagacaccaacgcttctgaacgtgaagctgttgaacacgttaaattcctgatccgtgaa1560
acctggaaagaaatgaacaccgttaccaccgcttctgactgcccgttcaccgacgacctg1620
gttgctgttgctaccaacctggctcgtgctgctcagttcatctacctggacggtgacggt1680
cacggtgttcagcactctgaaatccaccagcagatgggtggtctgctcttccagccgtac1740
gtctaa1746
<210>2
<211>665
<212>dna
<213>seq2
<400>2
tttcaaaaattcttactttttttttggatggacgcaaagaagtttaataatcatattaca60
tggcattaccaccatatacatatccatatacatatccatatctaatcttacttatatgtt120
gtggaaatgtaaagagccccattatcttagcctaaaaaaaccttctctttggaactttca180
gtaatacgcttaactgctcattgctatattgaagtacggattagaagccgccgagcgggt240
gacagccctccgaaggaagactctcctccgtgcgtcctcgtcttcaccggtcgcgttcct300
gaaacgcagatgtgcctcgcgccgcactgctccgaacaataaagattctacaatactagc360
ttttatggttatgaagaggaaaaattggcagtaacctggccccacaaaccttcaaatgaa420
cgaatcaaattaacaaccataggatgataatgcgattagttttttagccttatttctggg480
gtaattaatcagcgaagcgatgatttttgatctattaacagatatataaatgcaaaaact540
gcataaccactttaactaatactttcaacattttcggtttgtattacttcttattcaaat600
gtaataaaagtatcaacaaaaaattgttaatatacctctatactttaacgtcaaggagaa660
aaaac665
<210>3
<211>190
<212>dna
<213>seq3
<400>3
atccgctctaaccgaaaaggaaggagttagacaacctgaagtctaggtccctatttattt60
ttttatagttatgttagtattaagaacgttatttatatttcaaatttttcttttttttct120
gtacagacgcgtgtacgcatgtaacattatactgaaaaccttgcttgagaaggttttggg180
acgctcgaag190
<210>4
<211>511
<212>dna
<213>seq4
<400>4
gaacctcctccagatggaatcccttccatagagagaaggagcaagcaactgacccaatat60
tgactgccactggacctgaagacatgcaacaaagtgcaagcatagtggggccttcttcca120
atgctaatccggtcactgccactgctgctacggaaaaccaacctaaaggtattaacttct180
tcactataagaaaatcacacgagcgcccggacgatgtctctgtttaaatggcgcaagttt240
tccgctttgtaatatatatttatacccctttcttctctcccctgcaatataatagtttaa300
ttctaatattaataatatcctatattttcttcatttaccggcgcactctcgcccgaacga360
cctcaaaatgtctgctacattcataataaccaaaagctcataacttttttttttgaacct420
gaatatatatacatcacatatcactgctggtccttgccgaccagcgtatacaatctcgat480
agttggtttcccgttctttccactcccgtca511
<210>5
<211>501
<212>dna
<213>seq5
<400>5
aaagcatcttgccctgtgcttggcccccagtgcagcgaacgttataaaaacgaatactga60
gtatatatctatgtaaaacaaccatatcatttcttgttctgaactttgtttacctaacta120
gttttaaatttccctttttcgtgcatgcgggtgttcttatttattagcatactacatttg180
aaatatcaaatttccttagtagaaaagtgagagaaggtgcactgacacaaaaaataaaat240
gctacgtataactgtcaaaactttgcagcagcgggcatccttccatcatagcttcaaaca300
tattagcgttcctgatcttcatacccgtgctcaaaatgatcaaacaaactgttattgcca360
agaaataaacgcaaggctgccttcaaaaactgatccattagatcctcatatcaagcttcc420
tcatagaacgcccaattacaataagcatgttttgctgttatcaccgggtgataggtttgc480
tcaaccatggaaggtagcatg501
<210>6
<211>43
<212>dna
<213>sabs1-bamhⅰ-f
<400>6
aaaaaaccccgggatccgccaccatgccgctgaactctctgca43
<210>7
<211>39
<212>dna
<213>sabs1-salⅰ-r
<400>7
tcaacttctgttccatgtcgacgtacggctggaagagca39
<210>8
<211>20
<212>dna
<213>test-sabs1-f
<400>8
aagcgtggtctacctcttgc20
<210>9
<211>20
<212>dna
<213>test-sabs1-r
<400>9
ggaaccaacgagcttccaga20
<210>10
<211>37
<212>dna
<213>gal80us-f
<400>10
tgagagtgcaccatatggaacctcctccagatggaat37
<210>11
<211>35
<212>dna
<213>gal80us-r
<400>11
atttcacaccgcatatgacgggagtggaaagaacg35
<210>12
<211>41
<212>dna
<213>gal80ds-f
<400>12
ttgggacgctcgaagctgcagaaagcatcttgccctgtgct41
<210>13
<211>41
<212>dna
<213>gal80ds-r
<400>13
gccaagcttgcatgcctgcagcatgctaccttccatggttg41
<210>14
<211>20
<212>dna
<213>test-gal80-f
<400>14
atggactacaacaagagatc20
<210>15
<211>20
<212>dna
<213>test-gal80-r
<400>15
ctataatgcgagatattgct20
<210>16
<211>21
<212>dna
<213>gal80-grna-us-f
<400>16
gctggcacgacaggtttcccg21
<210>17
<211>56
<212>dna
<213>gal80-grna-us-r
<400>17
atttctagctctaaaacacgttcagcagcagccttagatcatttatctttcactgc56
<210>18
<211>57
<212>dna
<213>gal80-grna-ds-f
<400>18
tgatctaaggctgctgctgaacgtgttttagagctagaaatagcaagttaaaataag57
<210>19
<211>21
<212>dna
<213>gal80-grna-ds-r
<400>19
catccaccagagcatcaccgg21
<210>20
<211>37
<212>dna
<213>pgal1-sabs1-tcyc1-f
<400>20
tctagagtcgacctgcaggagcgacctcatgctatac37
<210>21
<211>37
<212>dna
<213>pgal1-sabs1-tcyc1-r
<400>21
atgattacgccaagcttcttcgagcgtcccaaaacct37
- 还没有人留言评论。精彩留言会获得点赞!