一种稻瘟病抗性相关的miRNA及其应用的制作方法
本发明涉及一种mirna,具体涉及一种与稻瘟病抗性相关的mirna,及其在水稻抗稻瘟病中的应用。
背景技术:
水稻(oryzasatival.)是世界上最重要的粮食作物之一,全球约有50%的人口以稻米作为主食。随着人口的增长,粮食需求量将逐年增加。由稻瘟病菌(magnaportheoryzae)引起的稻瘟病是水稻生产的主要限制因子之一,严重威胁着世界的粮食生产。全球每年因稻瘟病造成的产量损失达11%~30%。稻瘟病在水稻全生育期内均可发生,经常在水稻区造成巨大损失。长期的生产实践证明,通过鉴定和利用广谱抗性基因进行水稻抗病育种成为了防治病害的有效、经济的策略。
然而,由于稻瘟病菌小种的遗传复杂性及易变性,新选育的抗病品种常常在推广3-5年后因为能侵染该品种的优势小种的形成而丧失抗性,从而使其失去利用价值。于是人们提出了不同的战略战术来培育具有持久抗性的水稻品种:一是利用分子标记将具有不同抗性谱的抗病基因聚合到一个水稻品种中以防止抗病性的退化,减少单一的选择压力,进一步利用分子标记辅助选择(molecular-assisted-selection,mas)来进行育种。因此,寻找及利用具有广谱、持久抗性的新抗源,结合新技术新方法培育抗性新品种,并结合合理的栽培布局,是解决稻瘟病抗性品种寿命短这一问题的主要途径。
mirna是一类内源性的、19~24个碱基长度的小分子非编码rna,是真核生物基因表达中的一类负调控因子,它可以通过与靶基因mrna分子完全或部分匹配,指导mrna剪切或者抑制翻译等方式来调节植物基因的表达,参与调控植物生长发育各个方面以及逆境胁迫响应等生物学过程。在植物mirna的研究中,较多的mirna已于模式植物拟南芥、水稻中鉴定得到;通过构建mirnacdna文库、大规模测序及生物信息学等,多个与水稻生长发育、逆境胁迫等相关的mirna也已被鉴定出来,mirbase数据库中共有超过700条水稻mirna序列。li等研究发现,mir160a、mir164a和mir168a受稻瘟病菌的显著诱导表达,mir396、mir827和mir1871等表达量下调,mir169a、mir172a和mir398b在抗病材料和感病材料中都表现为上调(liy,luyg,shiy,wul,xuyj,huangf,guoxy,zhangy,fanj,zhaojq,zhanghy,xupz,zhoujm,wuxj,wangprandwangwm.2014.multiplericemicrornasareinvolvedinimmunityagainsttheblastfungusmagnaportheoryzae.plantphysiol.164(2):1077-92)。mirna可通过调控相应的靶基因而在植物的抗病反应表现重要的调控作用。mirna也直接调控植物抗性基因及蛋白,引起抗病防卫基因的表达,使其表达产物抑制病原菌生长。mirna亦可直接靶切nbs-lrr类抗性基因,进而参与r基因介导的eti,如nta-mir6019和nta-mir6020可以通过靶切tir-nb-lrr类n基因参与烟草(nicotianabenthamiana)对烟草花叶病毒(tobaccomosaicvirus,tmv)的抗性;在蒺藜苜蓿(medicagotruncatula)、番茄(solanumlycopersicum)、马铃薯(solanumtuberosum)、大豆(glycinemax)、棉花(gossypiumraimondii)和杨树中,mir482和mir2118通过靶切各类的nbs-lrr基因参与病原菌诱导的防卫反应。
上述研究表明mirna的调控与各个层级的抗病反应途径具有重要关系,mirna能够调控某些抗病途径中的关键靶基因来抵御病菌的入侵。相反,病原菌为了反抗,细菌也会像病毒一样,进化出效应分子蛋白去抑制mirna通路,削弱植物的抗病性。王兴军等发明的“利用人工microrna提高水稻抗黑条矮缩病的方法及其专用双链rna”(中国专利cn102676510a),公开了一种利用人工mirna提高水稻抗黑条矮缩病毒的方法及其专用双链rna,这些研究表明mirna可用于增强植物抗病性。然而,关于mirna水稻三大病害之一的稻瘟病抗病育种中的应用还未见报道。因此,克隆与稻瘟病抗病反应相关的mirna将在不同分子水平上丰富植物抗病机理,且通过改造mirna来进行抗病育种,为解决作物抗病性提供新思路,具有重要的应用价值。
技术实现要素:
为了解决上述技术问题,本发明提供了一种与抗稻瘟病相关的、来源于水稻的mirna;并提供该mirna在培育抗稻瘟病水稻新品种的应用。通过抑制该mirna的表达,提高靶基因pik2-h4的表达,从而提高水稻对稻瘟病菌的抗性。
本发明稻瘟病抗性相关的mirna记为t46,其具有如seqidno:1所示的序列。
本发明还提供了上述稻瘟病抗性相关mirna的前体序列,其具有如seqidno:2所示的序列。
本发明上述mirna或其前体序列可调控水稻抗稻瘟病基因表达。经实验证实,在稻瘟病菌侵染水稻时,本发明所述mirna表达量呈现下调趋势。
本发明上述mirna的靶基因,即所述抗稻瘟病基因为pik2-h4。pik2-h4是一个nbs-lrr抗性基因,它位于pik位点,该位点包含pik1与pik2两个基因,他们对于介导稻瘟病抗性缺一不可。本发明通过5’race实验证实,本发明的mirna可以直接裂解pik2-h4的mrna,从而负调控pik2-h4的表达。
本发明还提供了上述mirna或其前体序列在培育抗稻瘟病水稻中的应用。由于t46的靶基因为pik2-h4,t46的表达量与pik2-h4的表达量呈负相关。r基因在介导抗病反应发生时需要大量的pik2-h4的表达才能使水稻表现抗病。pik2-h4持续高水平表达,水稻表现出自我免疫甚至高抗,因此过表达t46后,pik2的表达水平降低,水稻丧失抗性;相反,如果敲除t46,会使靶基因表达处于较高的水平,表现较高的抗性。在水稻中过表达t46或敲除t46的前体后的转基因植株的抗病表明,过表达t46使水稻更易感病,而敲除t46抑制t46基因的表达能使水稻对稻瘟病具有更好的抗性。
本发明利用荧光定量pcr结果显示,mirna-t46受稻瘟病菌调控,表达量呈现下调趋势。靶基因pik2-h4在稻瘟病菌侵染后则出现上调表达的趋势,两者呈现负相关,进一步证实t46与pik2-h4之间的调控关系,表明t46在水稻抗稻瘟病调控过程中的重要作用。本发明通过构建水稻t46的过表达载体,将该过表达载体质粒转化进土壤农杆菌侵染pik-h4nil的愈伤组织。对获得的转基因植株进行抗病性分析结果表明,过表达t46显著降低水稻对稻瘟病的抗性,进一步表明t46对pik2-h4的重要调控作用,为后续研究奠定了重要基础。
本发明通过构建mirna-t46敲除用的crispr/cas9敲除载体,将该载体质粒转化进土壤农杆菌侵染pik-h4nil的愈伤组织,获得阳性植株。利用荧光定量pcr检测阳性植株及dna测序验证敲除植株。对获得的转基因水稻进行抗病性分析表明,转基因水稻植株能显著提高对稻瘟病的抗性,具有较好的应用前景。
附图说明
图1为为实时荧光定量pcr对mirna-t46受稻瘟病菌侵染后表达模式的分析结果,结果显示mirna-t46在h4中的表达受到明显下调,说明其在抗稻瘟病过程中起着重要作用;
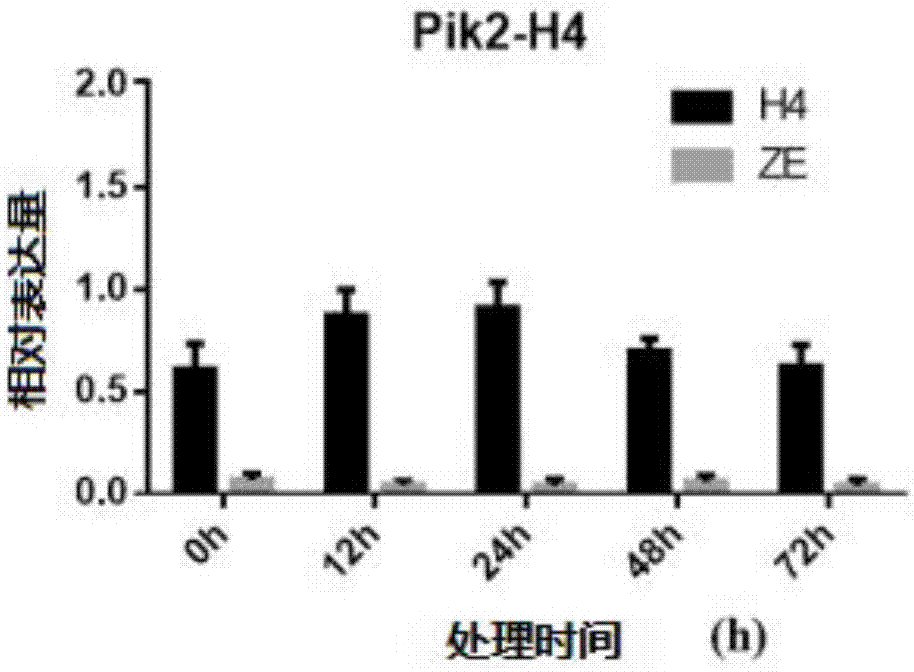
图2为为水稻受稻瘟病菌侵染后靶基因pik2-h4的表达模式,结果显示pik2-h4的表达量变化呈负相关,进一步证明了mirna-t46负向调控靶基因pik2-h4;
图3为mirna-t46对靶基因pik2-h4的剪切及剪切位点示意图,剪切位点位于3’非翻译区域内;
图4为过表达(t46-ox)、缺失t46(t46-ko)转基因材料的抗病性测定,结果显示过表达mirna-t46后促进了稻瘟病菌的侵染,而缺失则具有较高的抗病性。
具体实施方式
本发明提供了一种与稻瘟病抗性相关的mirna-t46,其是从水稻smallrna测序中得到的,序列如seqidno:1所示。
本发明是利用两个抗感水稻材料(h4与中二软占)研究证实:t46的靶基因为pik2(核苷酸序列如seqidno:3所示)。其中h4含有pik2-h4抗性基因,因此t46极有可能靶向调控pik2-h4。为验证这一假设,本发明采用pcr检测接种稻瘟病菌后水稻材料中t46的表达情况,结果表明,t46表达量在h4中总体表现为先升后降的趋势,在接种后24小时表达量达最低,t46在接种后各时间点表达量差异显著,这与靶基因pik2-h4的表达量变化恰恰相反,进一步表明t46与pik2-h4之间是呈负相关。而在中二软占中,虽然t46的表达量也变现为先升后降的趋势,但是接种后各时间点表达量差异不显著。
本发明还提供了上述技术方案所述mirna的前体序列pre-t46,序列如seqidno:2所示,也受稻瘟病诱导表达,且与t46具有类似的表达模式,对pre-t46表达调控的研究在一定程度上反映出t46表达水平的变化,通过pre-t46的研究反映出对应产物mirna的表达情况。
本发明还提供了上述mirna或其前体序列在培育抗稻瘟病水稻中的应用。通过敲除水稻中的mirna-t46或其前体序列,抑制水稻中mirna-t46或其前体序列的表达,进而提高水稻的抗稻瘟病性能,培育出高抗稻瘟病的水稻新品种。
为使本发明的目的、技术方案和优点更加清楚明白,下面结合实施例对本发明进行详细的说明,但是不能把它们理解为对本发明保护范围的限定。
实施例1
实时荧光定量pcr分析mirna-t46在稻瘟病抗性中的作用
1.rna反转录及实时定量pcr的引物设计
根据mirna序列预测的前体序列设计stem-loop引物如下:
(1)rt引物序列(seqidno:4)
5’-ctcaactggtgtcgtggagtcggcaattcagttgagccctcat-3’
(2)mirna-t46正向引物(seqidno:5)
5’-gccgcgaagaactgttacag-3’
(3)mirna-t46反向引物(seqidno:6)
5’-aactggtgtcgtggagt-3’
(4)pik2-h4正向引物(seqidno:7)
5’-tgaacttccacgattggatccac-3’
(5)pik2-h4反向引物(seqidno:8)
5’-acatggttcttgaatacatcatgtc-3’
2.材料培养及稻瘟病菌侵染
试验水稻材料为中二软占(ze)及经过空间搭载诱变的中二软占的突变体h4株系。h4是通过地面选育的广谱、高抗的水稻优质种质资源,对稻瘟病有较高抗性,在文献“水稻抗稻瘟病蛋白pik2-h4基因的克隆及互作蛋白的筛选[j].广东农业科学,2004,(04):156-160.”中已公开。
温室种植h4与中二软占,到三叶期采用高压喷雾法把稻瘟病孢子gd0193的明胶溶液均匀喷施在叶片表面(分生孢子浓度为5×105/ml),接种苗于25℃暗室中保湿24h后,放荫棚下继续保湿,7d后调查发病情况并拍照。
3.水稻总rna的提取,使用trizol试剂,提取方法如下:
取接种之后0h、12h、24h、48h及72h取水稻叶片抽提rna。具体方法如下:取100mg中二软占及h4四叶期水稻幼苗,用液氮冷冻后在研钵中研磨成粉末,转入到装有1mltrizol(invitrogen公司)的1.5ml离心管中,混匀后静置5min,加入200μl氯仿,混匀并静置5min后,4℃10000g离心15min,收集上层水相到新的1.5ml离心管内;加入500μl预冷的异丙醇,混匀室温静置20min后,4℃,10000g离心15min,弃去上清,收集沉淀,用75%的酒精洗去杂质,待酒精挥发后溶于无rna酶的超纯水中,-80℃冻存备用。
4.反转录及反转录体系
普通定量反转录:取-80℃保存的总rna样品1μg,加入1μloligo(dt16)(10mm),用rnafreewater补匀到总体积12μl,混匀置于65℃水浴锅中水浴5min后,立即置于冰上,并向ep管中加入4μl5xrtbuffer,2μldntp(10mm),rnase抑制剂1μl(10u/μl)和1μlrevertraace并混匀。将ep管置于pcr仪中,设置反应程序:30℃10min,42℃60min,99℃5min,25℃5min,得到第一链的单cdna,于-20℃冰箱保存。
mirna的反转录:取0.5μgdnase处理过的rna,加入1.5μl引物混合液(0.25μlrt引物,0.25μldntp和1μldepc。然后稍离心,65℃孵育5min,立即置于冰上3min。将1μlsuperscriptiii配制成混合液,每管中加入1.8μl混合液。离心后,放置于pcr仪上,程序如下16℃30min,50℃30min,85℃5min,25℃1min,将反转录产物置于-20℃过夜,或者立即进行定量分析。
5.实时荧光定量pcr及结果分析:
(1)实时定量pcr反应体系
选用诺唯赞公司的定量试剂对mirna进行定量,反应体系如下:10μlaceqqpcrsybrgreenmastermix,0.8μl10μm引物(检测mirna-t46时用序列5和序列6所示的引物序列;检测pik2-h4时用序列7和序列8所示的引物序列),0.4μlroxreferencedye1,0.2μlcdna与6μl无菌水组成20μl的反应体系,进行定量检测。定量pcr仪设置,95℃5min,95℃10sec,60℃30sec,40个循环。以u6rna作为内参基因,使用2-δδct方法进行相对定量分析。所有反应均为三次重复。
(2)结果分析:利用2-δδct方法进行结果的分析
参看图1、图2,实时荧光定量pcr分析的结果显示,mirna-t46的表达受到稻瘟病菌的调节,稻瘟病菌侵染后mirna-t46的表达量呈现下调趋势,而pik2-h4在稻瘟病菌侵染后则出现上调表达的趋势,两者呈现负相关。这表明在水稻中抑制或过表达t46,将影响水稻对稻瘟病菌的抗性,mirna-t46与pik2-h4之间有调控关系。表明mirna-t46在水稻抗稻瘟病调控过程中的重要作用。
实施例2
mirna-t46靶基因的验证:用5'race方法验证t46靶基因pik2-h4的剪切位点
1.5'race引物设计如下:
reverseprimer:(seqidno:9)
5’–cttcggcactaaaggtgcaaaggattacgccaagctt–3’。
2.rna提取同实施例1。
3.5'race实验
(1)连接rna接头:
选用
(2)反转录为cdna
取上述接头rna加入4.0μl5xfirst-strandbuffer,0.5μldtt(100mm),
1.0μldntps(20mm),0.5μlrnaseinhibitor(40u/μl)和2.0μlsmartscribereversetranscriptase(100u),总反应体系20μl。放置于pcr仪上,程序如下42℃90min,70℃30min,25℃2min,得到第一链cdna。
(3)pcr扩增
pcr体系:15.5μl无菌水,25.0μl2xseqampbuffer,1.0μlseqampdnapolymerase,2.5μlcdna,5μl10xupm,gsp1(10μm)1μl。
其中upm中含有的两条引物序列如下:
5’–ctaatacgactcactatagggcaagcagtggtatcaacgcagagt–3'(seqidno:10);
5’–gcttctgccccatcactaactt-3’(seqidno:11)。
pcr反应为:94℃30sec,68℃30sec,72℃1min,共25cycles。
(4)pcr产物的纯化
根据omega胶纯化回收试剂盒方法进行回收,具体方法如下:取pcr产物进行0.8%凝胶电泳,切下需要回收的目的片段,置于1.5ml离心管中。按照1g/100μl的量加入等体积的结合缓冲液xp2,55℃水浴10min直到凝胶完全溶解。将溶解后的液体全部转移到hibinddna回收柱中,10000g离心1min,倒掉收集管中的废液,将回收柱重新装入收集管中。重新加入300μlxp2,10000g离心1min。加入700μlspw洗液,10000g离心1min,倒掉收集管中的废液,重复2次。13000g离心空的hibinddna回收柱2min,
将hibinddna回收柱重新装入新的1.5ml离心管中,加入30μl洗脱液,室温放置2min,13000g离心2min洗脱dna片段,去除回收柱,-20℃保存。
(5)克隆race产物
按照以下方式配置混合液:1μllineareizedpracevector(smarterrace5’/3’kit提供),7μlpcr纯化产物,2μlin-fusionhdmastermix。race产物与载体的重组反应:50℃水浴15min,然后置于冰上。
取5μl重组反应,加入到50x感受态细胞中,冰浴15min,42℃热激55sec,再次冰浴2min,然后加入450mlsoc,置于37℃摇床中,160rpm摇培1h。取出摇培的ep,短暂离心,挑取过夜培养平板上面的单菌落,送样测序。
(6)5'race结果分析
pcr反应可扩增到250bp左右的特异条带,见图3。克隆该特异产物、测序,结果如下:
结果表明,racer试剂盒的5'接头后的序列为t46和pik2-h4的部分互补结合位点及其3'端序列,该产物为t46切割pik2-h4后的降解产物。在随后挑的克隆测序结果中,切割位点位于mirna第11-12位碱基间,其余克隆位于结合位点其他碱基处。
实施例3
过表达t46、敲除t46的转基因水稻的构建
利用pox为过量表达载体,以ubi启动子驱动mirna-t46的过表达;利用crispr/cas9技术进行mirna-t46的敲除。具体如下:
1、mirna-t46的过表达载体构建
所需引物如下:
pre-t46f(seqidno:12):
5’-cggggtaccataggcatcccaatctttagc-3’,
pre-t46r(seqidno:13):
5’-cgcggatcctgacgagccgttatagatgac-3’,
扩增片段长度为958bp。利用kpni与bamhi将该片段连接到pox载体上,获得pox-t46载体。
2、t46前体的敲除载体构建
构建两个敲除靶点,所需引物如下:
u3f(seqidno:14):5’-ggcattaagaactgttacagatg-3’,
u3r(seqidno:15):5’-aaaccatctgtaacagttcttaa-3’,
u6af(seqidno:16):5’-gccgagatccctaacatctataa-3’,
u6ar(seqidno:17):5’-aaacttatagatgttagggatct-3’。
分别将这两对引物自我融合后,通过巢式pcr的方法,分别连到u3、u6a启动子上,然后利用bsai酶切后回收dna片段后,连接到bsai酶切回收的pylcrispr/cas9-mt(i)载体上,获得含两个敲除t46前体靶点载体pylcrispr/cas9-mt(i)-t46。
3、转基因水稻的构建:
用籼稻品系h4、中二软占的成熟种子诱导愈伤组织,21天后将愈伤进行继代;10天继代一次,继代培养2次后挑选状态良好的胚性愈伤组织用于载体的土壤农杆菌eha105侵染;共培养3天后进入抗性愈伤组织筛选,转基因植株再生通过利用50mg/l潮霉素培养基筛选,每隔2周筛选一次;挑选状态良好的抗性愈伤组织进行2周的预分化培养,再进行2周的分化培养,并将分化苗移至生根培养基,获得转基因植株。经潮霉素基因hpt的pcr检测,并对目的基因进行pcr检测,获得过表达t46或者敲除t46前体的转基因阳性植株。
实施例4
过表达t46、敲除t46的转基因水稻的抗病性分析
对转基因后代阳性植株进行抗病性检测,接种方法参照实施例1。
结果如图4所示,接种稻瘟病菌后,t46过表达转基因植株病斑增多,抗病性明显降低,而敲除植株抗并未见明显侵染病斑,抗性明显增强。
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
序列表
<110>华南农业大学
<120>一种稻瘟病抗性相关的mirna及其应用
<160>17
<170>siposequencelisting1.0
<210>1
<211>21
<212>dna
<213>水稻(oryzasativa)
<400>1
aagaactgttacagatgaggg21
<210>2
<211>114
<212>dna
<213>水稻(oryzasativa)
<400>2
tacatatgacccctcatctgtaatgattcttaatctatagccgttacagatgagatccct60
aacatctataatggttgtagattaagaactgttacagatgaggggtcatctata114
<210>3
<211>4086
<212>dna
<213>水稻(oryzasativa)
<400>3
atggagttggtggtaggtgcttccgaagccaccatgaaatctctcttgggcaagctgggc60
aatcttctagcccaggagtatgctctcatcagcggtatccgtggtgacatccagtacatc120
aatgacgagcttgccagcatgcaggccttcctccgtgatctcagcaacgtgccagagggt180
cacagtcatggccaccggatgaaggactggatgaagcagatccgagacatcgcctatgat240
gttgaggactgtatcgatgactttgcccaccgcctccctcaggattccatcagcgatgcc300
aaatggtccttcctactcacaaaaatctatgaactatggacatggtggccacgtcgtgtg360
attgcttccaacattgcccaactcaaggtacgggcacaacagatcgcagatcgacgtagt420
agatacggagtgaacaacccagaacaccttgacagtagcagcagtgccaggacccgtgct480
gtcaattacgaaattgctgagtatcaggtcacaagccctcagatcattggtataaaggag540
cctgtggggatgaagacggtcatggaggagcttgaggtttggttaactaatcctcaagct600
gaaaatgggcaagctgttctgtccatagtcggttttggaggtgtgggaaagactaccatt660
gccacagcattgtacagaaaagtcagtgataaatttcagtgccgggcatcagtagctgtg720
tctcagaactatgaccaaggcaaagtcctcaatagtattctgagtcaagtcagcaatcag780
gagcagggcagcagcacaacaattagtgagaaaaagaacctcacctcaggcgctaagagc840
atgttgaagacagccctgtcactgctcagaggtaattgtatatgtcagccagaaaatgat900
ggaaaccctgataatacaccaatcaggctgcaggaaacaacggacgatgatcaaaacccc960
agaaaactggaacagctcctggccgaaaagagttatatcctcttgattgatgacatttgg1020
tctgccgaaacatgggagagtatcagatcgattttgcctaaaaataataaaggcggtaga1080
ataatagtgactacaagatttcaagctgttggttcaacatgctcccctcttgaaactgat1140
cgtttgcatacagttgattttctcaccgatgacgagtcccaaaacttattcaatacaagt1200
atttgtgaatcaaagataagaaaagatagcaacaaagtagacgagcaagtccctgaggaa1260
atatggaaaatatgtgggggattgcctttggccatagtcaccatggctggtcttgtcgcc1320
tgcaacccaaggaaagcctgctgcgattggagtaaactttgcaaatcattatttccagag1380
caagaaactcctcttaccctcgatggtgttacaaggatactggattgttgttacaatgat1440
ttgcctgcggatctgaagacttgcttattgtacttgagtatatttccgaagggttggaaa1500
attagtaggaaacgtttgtcccggcgatggatagctgaaggttttgctaatgagaagcaa1560
gggttaacccaggaaagagttgcagaggcatactttaatcaactcacaagaaggaactta1620
gtacgtcccatggagcatggcagcaatgggaaggtaaaaacgtttcaagttcatgacatg1680
gttcttgaatacatcatgtccaaatcaatcgaagagaattttattactgtggttggtgga1740
cactggcagatgactgcaccaagcaataaagtccgtcgactgtcgatgcaaagcagtgga1800
tccaatcgtggaagttcaacaaaaggcctgaacttggctcaagtgagatcactgacggtg1860
tttgggaacctgaaccatatgccattccattcattcaactatgggataatacaggtgctg1920
gatcttgaggactggaagggtttgaaagagagacatatgacggagatatgtcaaatgctt1980
ttactcaagtatttgagcatccgacgaacagaaatttccaaaattccctccaagattcag2040
aaacttgagtacttggaaactcttgacataagggagacatatgtcagggacctgcctaag2100
tcaatagtccagctaaaacggatcattagcatacttggagggaataaaaacacacggaag2160
gggctgaggttgcctcaagaaaaaagtaagaagccaattaaaaacccgtcgcctcaagga2220
aaaacaaaggagcccgcaaagaaaggattcttatcccaagaaaaaggtaaaggcgcaatg2280
aaagcactccgtgtactgtcagggattgagattgttgaggaatcatcagaagtagctgca2340
ggccttcatcagttgacagggctaaggaagcttgccatatacaagctcaatataacaaag2400
ggtggtgataccttcaaacaattacagtcctccattgagtaccttggcagctgtggtctg2460
cagactctggccatcaatgatgagaattctgaatttatcaactcactgggcgacatgccc2520
gcgcctccaagatatcttgtcgcccttgagctgtctggcaagttggagaagctacccaag2580
tggatcaccagcatcactactctcaacaagctaaccatatctgtaacagttcttaggact2640
gaaactttggagatcctccacattttaccttcattgttttccctcaccttcgccttttca2700
cttagtgcagcgaagcaggatcaggacataataaaggacatccttgagaataataaattg2760
gacagtgatggggaaatcgtcattccagctgaaggattcaagagtcttaagctgcttcgc2820
ttctttgcacctttagtgccgaagctcagctttttggacaagaatgcaatgccagcactc2880
gaaatcattgaaatgcggtttaaagacttcgaaggtctatttggcatcgaaatccttgaa2940
aatctccgtgaggtgcatctcaaagttagtgatggggcagaagcaataaccaagttcctt3000
gtaaatgatttgaaggttaatactgagaaaccaaaagtatttgttgatggcatcgtcact3060
gcatgagaagtaaaattgctgcaaatcggagaacttaccaatcatctgaggcttcccctc3120
tattattactctcttagaatatattgttattattgctcaccttgcaaaataaaataggga3180
tggcatagcatattgctacaacgtaccatggttccatcatagttgatttcacttgtcatt3240
acagtgtctgttcagttgtgttttctattaataaaagggagatctccgcaagaaaccatt3300
attatacttatattcggttattgaactctataaatgatgggattgctatatttatggtgc3360
cacaatttccatgagtgcggtatttttttttctagcagttggctggtgttaagatttgtg3420
ctgccattgctcctctattattggtgcccaaaattacgctttgcactatgttcacagttg3480
taaaacttacctaaatttcgtgtattagtactgtaatgttgtgattttcgcgtccagtta3540
tatttttttctttgccagaagtttgatttcaaggtatatctggtaagcttcagccggatt3600
cgttaagtttttagtttaatcagctaaacttttaacagtatgcatgcactagtttctccc3660
gttatcttatccaatataattaatgggctaattggatctatgccattacaaacatggcaa3720
gattcagaaaaatgctactacaatttgtctattcatagctgtgctagtgaaattttacaa3780
aattggaaccgtgctattgatatcacgttttccatccatcttttcttttttttccttttc3840
ttctttatcccgtcttcttcccgcaccgacgagaggcgagtggcggcgggaccggcaagc3900
gatggcaggacctcggcgccctgggtgggcgagtgagggagagctctcccaagtgtctgc3960
gaagctctcatcttcgttttcctcatccccactttgctcctcttccactactgctgttga4020
agctagcggccctctctcaccagatctgggcggagctcaagcgaggcgccaccaccgacg4080
atggag4086
<210>4
<211>43
<212>dna
<213>人工序列(artificialsequence)
<400>4
ctcaactggtgtcgtggagtcggcaattcagttgagccctcat43
<210>5
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>5
gccgcgaagaactgttacag20
<210>6
<211>17
<212>dna
<213>人工序列(artificialsequence)
<400>6
aactggtgtcgtggagt17
<210>7
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>7
tgaacttccacgattggatccac23
<210>8
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>8
acatggttcttgaatacatcatgtc25
<210>9
<211>37
<212>dna
<213>人工序列(artificialsequence)
<400>9
cttcggcactaaaggtgcaaaggattacgccaagctt37
<210>10
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>10
ctaatacgactcactatagggcaagcagtggtatcaacgcagagt45
<210>11
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>11
gcttctgccccatcactaactt22
<210>12
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>12
cggggtaccataggcatcccaatctttagc30
<210>13
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>13
cgcggatcctgacgagccgttatagatgac30
<210>14
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>14
ggcattaagaactgttacagatg23
<210>15
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>15
aaaccatctgtaacagttcttaa23
<210>16
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>16
gccgagatccctaacatctataa23
<210>17
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>17
aaacttatagatgttagggatct23
- 还没有人留言评论。精彩留言会获得点赞!