serpinA3及vitronectin基因在苏姜公猪选育中的应用及其选育方法与流程
本发明涉及基因工程
技术领域:
,具体涉及serpina3及vitronectin基因在苏姜公猪选育中的应用及其选育方法。
背景技术:
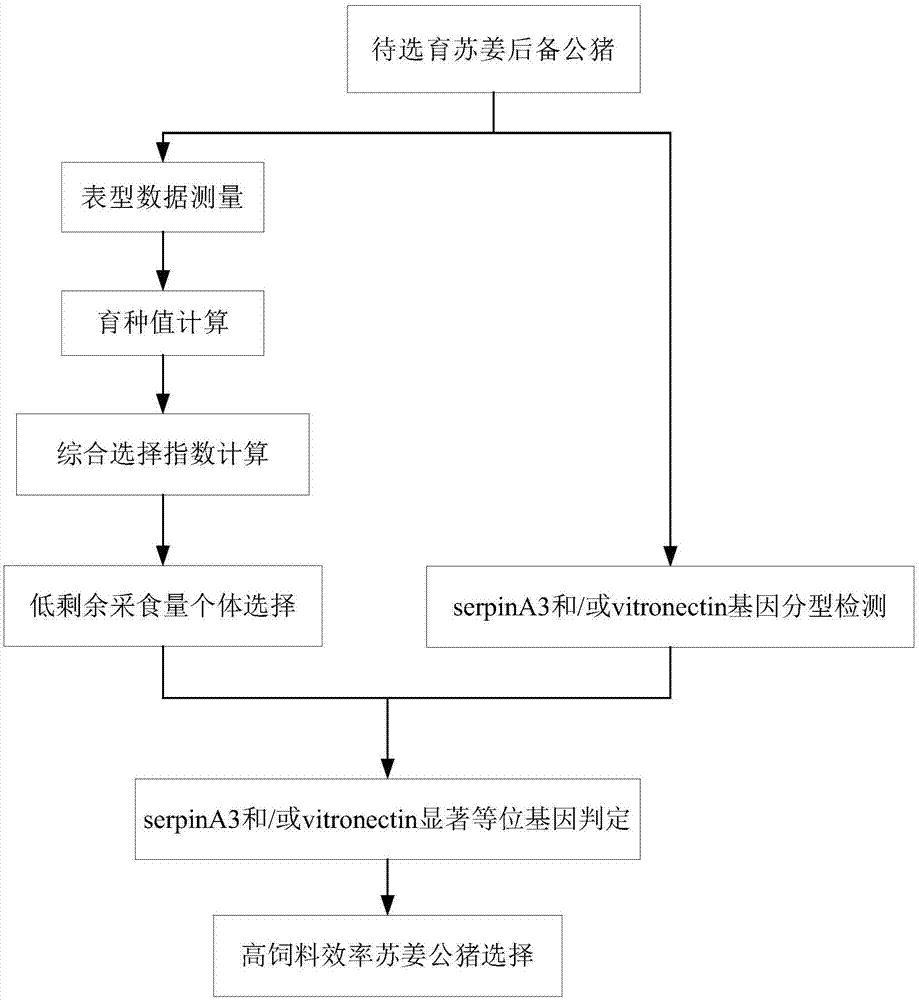
:苏姜猪选育目标为高产仔数、优良肉质及高瘦肉率的新品种猪,经过多个世代的选育,该猪具备良好的体型外貌、繁殖及生产性能[1]。截止目前,饲料成本占现代集约化畜禽养殖生产总成本的比例超过了50%,饲料效率对畜禽规模化养殖的经济效益有重大影响。国内外通常采用料重比(feed/gain,f/g)或重料比(gain/feed,g/f),即饲料转化率(feedconversionrate,fcr)来评定饲料效率。但是,通过f/g或g/f指标评价饲料效率具有如下缺陷:第一,饲料使用量应该是猪的实际采食量,而不是一般的饲料消耗量,如何准确测定采食量是面临的难题,甚至不同的料槽设计都会引起采食量波动[2];第二,体重对饲料效率有影响,因为不同体重的个体维持需要所需能量有差异,维持需要低的个体具有更高的饲料效率[3];第三,日粮能量水平对饲料效率有影响,因为饲料质量(稳定的能量摄入)和日粮中能量测定具有不确定性[4-5]。因此,采用料重比或重料比不能准确反映饲料效率。一般情况下,为了提高饲料效率,可以从外部环境营养调控入手改善动物采食量[6-7],例如注重猪饲料原料的消化率[8]、浓度[9]和能量[10]。但随着世界资源危机的来临,通过选育节粮型猪或应用分子遗传手段提高猪生产性能,从猪内部提高饲料效率是未来发展的必然趋势。回顾过去几十年,猪育种企业对瘦肉率、日增重和背膘厚等性状的选择已近乎达到选择极限,通过提高生长性状间接改善饲料效率已现瓶颈。但猪个体维持基础代谢所需能量有差异这一生理现象为育种者提供了一个新思路,即降低猪个体维持所需能量能够进一步提高饲料效率。维持所需由剩余采食量(residualfeedintake,rfi)指标反映饲料效率。剩余采食量是koch等人于1963年提出的一种估测畜禽饲料效率的指标,是畜禽实际采食量与根据其体型大小和生长速度预期的采食量的差值。换句话说,rfi是畜禽实际采食量与用于维持和增重所需要的预测采食量之差。在实际育种时,选择rfi水平低的个体,在总采食量相同的前提下,个体用于维持所需的采食量越少,则用于生长的采食量越多,饲料效率越高[11-12]。因此,在不影响苏姜猪最初生长、繁殖和肉质育种目标前提下,持续选择低rfi水平个体能够提高饲料效率、降低饲养成本、减少粪便和碳排放,提高养殖企业生产效益的同时减少环境污染,保证经济效益和环境质量协同发展。然而,通过或者仅通过选择低rfi水平个体,即表型选择,具有一定的局限性。例如,通过表型选择筛选出的高rfi水平个体和低rfi水平个体之间的饲料效率数值有差异但不显著。这可能是因为用于选择的个体数目比较少,表型选择误差较大,可能需要多个世代才可以有明显差异,也可能经过多个世代也没有明显变化。已知复杂的代谢通路影响饲料效率,涉及能量代谢[13]、脂类代谢[14]、抗氧化应激[15]、免疫防御[16]和胰岛素形成[17]等生理过程。研究表明,参与肌动蛋白丝组装和免疫应答过程的丝氨酸蛋白酶抑制剂a3蛋白(serineproteaseinhibitora3或serpina3)和参与血液体内平衡及调控皮下脂肪沉积的玻连蛋白(vitronectin)在影响猪rfi的代谢通路中直接发挥重要作用,最终通过调节能量利用并建立体内能量平衡来影响饲料效率。因此,探究serpina3和vitronectin基因与苏姜猪饲料效率之间的关系,挖掘和应用两类基因显著等位基因,同时选育低rif水平苏姜猪,从而为高饲料效率的节粮型苏姜猪新品系培育提供一种有效方法。技术实现要素:如上所述,对具有高饲料效率的苏姜猪的当前选育方法是通过筛选具有低rfi水平个体进行的。然而,该选育方法存在诸多缺陷,例如误差较大,不够准确,选育时间长,可能需要经历多个世代才能完成。因此,本发明期望通过采用分子遗传分析来对苏姜猪进行选育,藉此为高饲料效率的节粮型苏姜猪新品系培育提供了一种有效的方法。本发明人首次意外地发现,serpina3和vitronectin基因与苏姜猪的饲料效率存在显著相关性。更具体地,本发明人发现,具有serpina3基因显著等位基因b1和/或vitronectin基因显著等位基因b2的苏姜猪个体具有低rfi水平,并且其饲料效率可以提高至少约5%以上。基于此发现,本发明人完成了本发明。因此,在第一方面,本发明提供了一种选育苏姜公猪的方法,所述方法包括以下步骤:i.对苏姜公猪的serpina3基因和/或vitronectin基因进行基因分型检测;ii.选择携带有serpina3基因显著等位基因b1和/或vitronectin基因显著等位基因b2的苏姜公猪留为种用。在第二方面,本发明提供了serpina3基因和/或vitronectin基因在选育苏姜公猪中的应用。在第三方面,本发明提供了一种用于选育苏姜公猪的试剂盒,所述试剂盒包括:a)用于扩增serpina3基因的引物对1,和/或用于扩增vitronectin基因的引物对2;以及b)利用本试剂盒选育苏姜公猪的使用说明书。通过本发明,可以对苏姜猪进行更直接、准确和快速的选育,由此为高饲料效率的节粮型苏姜猪新品系培育提供一种有效方法。本发明的选育方法可以选育出具有更高饲料效率的苏姜种公猪,节省养猪周期,提高苏姜猪养猪场的经济效益。附图说明附图用来提供对本发明的进一步理解,并且构成说明书的一部分,与本发明的实施例一起用于解释本发明,并不构成对本发明的限制。在附图中:图1为本发明的技术路线图;图2为引物对1扩增等位基因sscp图;图3为引物对2扩增等位基因sscp图。具体实施方式下面对本发明进行更为详细的描述。应注意,上文的
发明内容部分以及下文的详细描述仅为具体阐释本发明之目的,无意于以任何方式对本发明进行限制。在不背离本发明的精神和主旨的情况下,本发明的范围由随附的权利要求书确定。如上所述,对具有高饲料效率的苏姜猪的当前选育方法是通过筛选具有低rfi水平个体进行的。然而,该选育方法存在诸多缺陷,例如误差较大,不够准确,选育时间长,可能需要经历多个世代才能完成。因此,本发明期望通过采用分子遗传分析来对苏姜猪进行选育,藉此为高饲料效率的节粮型苏姜猪新品系培育提供了一种有效的方法。分子标记辅助选择(mas)是一种通用的鉴定与利用染色体区域数量性状基因座(quantitativetraitlocus,qtl)连锁有利等位基因促进家畜育种的方法,它是目前全基因组关联分析(gwas)的重要辅助方法。显著性等位基因与qtl间有重组事件发生且等位基因的效应可能不同,因此mas实施必须首先确定显性等位基因的正效应或负效应。目前,mas实施更高效的方法是寻找和鉴定功能基因或已知功能基因的新等位基因,评估其对不同表型性状的正负效应,准确性更好,mas是目前科研人员广泛认可的研究单一性状的有效方法。本发明人通过利用mas技术首次意外地在基因水平上发现,serpina3和vitronectin基因与苏姜猪的饲料效率存在显著相关性。更具体地,本发明人发现,具有serpina3基因显著等位基因b1和/或vitronectin基因显著等位基因b2的苏姜猪个体具有低rfi水平,并且其饲料效率可以提高至少约5%以上。基于此发现,本发明人完成了本发明。因此,在第一方面,本发明提供了一种选育苏姜公猪的方法,所述方法包括以下步骤:i对苏姜公猪的serpina3基因和/或vitronectin基因进行基因分型检测;ii选择携带有serpina3基因显著等位基因b1和/或vitronectin基因显著等位基因b2的苏姜公猪留为种用。在本文中述及的“显著等位基因”是指与较低饲料消耗量相关的等位基因,或者说是指与低rfi水平相关的等位基因。换言之,通过本发明方法选育出来的苏姜公猪,由于携带有serpina3基因显著等位基因b1和/或vitronectin基因显著等位基因b2,将具有低rfi水平,由此具有所需的高饲料效率。在本文中,所谓“低rfi水平”是指-0.214kg/d至-0.106kg/d的rfi水平。低rfi水平苏姜公猪具有预期的生长性状,所述生长性状:包括预定体重阶段内的饲料效率、平均日增重和/或背膘厚。更具体地,在预期的生长性状指预定体重阶段内的饲料效率时,是指在预定体重阶段为30-90kg时,饲料效率为2.93:1-3.02:1;在预期的生长性状指预定体重阶段内的平均日增重时,是指在预定体重阶段为30-90kg时,平均日增重650g/d以上;在预期的生长性状是指预定体重阶段内的背膘厚时,是指预定体重阶段为30-90kg时,背膘厚为1.8-3.28cm。如果苏姜公猪的rfi水平不在上述范围内,则不利于节约养猪周期,也不利于提高苏姜猪养猪场的经济效益。在一个实施方案中,serpina3基因显著等位基因b1具有seqidno6所示序列,vitronectin基因显著等位基因b2具有所示序列。通过序列比对可以获知,serpina3基因的等位基因a1和b1之间、以及vitronectin基因的等位基因a2分别与其等位基因b2和c2之间,均仅存在一个碱基差异。更具体地,等位基因a1第123位碱基a在其等位基因b1的对应位置为c;等位基因a2第306位碱基t在其等位基因b2的对应位置为c,第137位碱基g在其等位基因c2的对应位置为a。本领域技术人员应该理解的是,在本发明中,除了利用在本文中专门由seqidno6和表示的序列自身外,还可以利用各自包含上文所述碱基差异、与之分别具有一定序列同源性例如80%-100%同源性(例如81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%、100%)且依然保留其功能性的序列。在此所谓“依然保留其功能性”是指:在苏姜公猪体内不具备seqidno6和/或序列本身而具有其同源序列时,该同源序列的存在仍然能够指示苏姜公猪具有上文所述的低rfi水平。在一个实施方案中,步骤i中所述基因分型采用pcr-sscp分型法。pcr-sscp分型法也称为聚合酶链式反应-单链构象多态性(polymerasechainreaction-singlestrandconformationpolymorphism)分型法,其是在pcr技术基础上发展起来的一种简单、快速、经济地显示在pcr反应产物中单碱基突变(点突变)的方法。该方法已被用做癌基因和抑癌基因突变的筛查检测、遗传病的致病基因分析和基因诊断、基因制图等领域。在sscp测定中,双链dna(dsdna)被变性成为单链dna(ssdna),每一条单链dna都基于它们的内部序列而呈现出一种独有的折叠构象,即使同样长度的dna单链因其碱基顺序不同、甚至单个碱基的不同会形成不同的构象。这些单链dna在非变性条件下,用非变性聚丙烯酰胺凝胶电泳分离,单链dna的迁移率和带型,都取决于其折叠构象和电泳时的温度。相同长度的单链dna因其碱基顺序不同,甚至单个碱基的差异会形成不同的构象从而导致电泳时泳动速度不同。如靶dna中发生碱基替代等改变时就会出现泳动变位,从而鉴别有无基因突变。在一个实施方案中,serpina3基因采用引物对1即seqidno1和seqidno2进行pcr扩增,vitronectin基因采用引物对2即seqidno3和seqidno4进行pcr扩增。但是本领域技术人员可以理解的是,除了上文列出的引物对之外,serpina3基因和vitronectin基因也可以采用本文未述及的其它引物对来进行扩增,只要该引物能够扩增出这两个基因的显著等位基因即可。在一个实施方案中,在进行所述步骤i之前进行以下步骤:a1)测量待选育苏姜公猪的表型数据,所述表型数据包括:初测体重、结测体重、平均日采食量、中间代谢体重、背膘厚、和平均日增重;a2)计算剩余采食量育种值ebvrfi和综合选择指数i;a3)选择综合选择指数i为0.24-1.62的个体继续用于步骤i)至步骤ii)。在步骤a2)中,计算剩余采食量育种值ebvrfi和综合选择指数i可以通过如下方式进行:首先,采用式(1)计算剩余采食量rfi:rfi=adfi-(b1×onbw+b2×offbw+b3×metamidbw+b4×adg+b5×bfa)(1)对于苏姜公猪,具体采用式(2)来计算其剩余采食量rfi:rfi=adfi+0.11323×onbw+0.11673×offbw-0.96405×metamidbw-0.30524×adg-0.02367×bfa)(2)其中,adfi表示平均日采食量,onbw表示初测体重,offbw表示结测体重,metamidbw表示中间代谢体重,adg表示平均日增重,bf表示背膘厚,其中metamidbw=[(onbw+offbw)/2]×0.75。随后,以剩余采食量rfi为目标性状,平均日增重adg和背膘厚bf为约束性状,根据如下模型分别计算rfi育种值ebvrfi、adg育种值ebvadg和bf育种值ebvbf:ebvrfi=adfi-group-year-onage-offbw-adg-bf-e(3)ebvadg=adg-group-year-onage-e(4)ebvbf=bf-group-year-offwt-e(5)其中,adfi、offbw、adg和bf定义同上,onage表示初测年龄,group为测定群体,year为测定年份,e为随机残差。之后,利用计算得到的各育种值根据式(6)计算综合选择指数(i):i=-aebvrfi+bebvadg-cebvbf(6)在本文中,对于目标性状(rfi)和约束性状(adg和bf)的权重,设定ebvrfi占75%,ebvadg占15%,ebvbf占10%。由此,上文用于计算综合选择指数i的公式(6)可以进一步明确为下式(7):i=-0.75ebvrfi+0.15ebvadg-0.1ebvbf(7)若某个选育苏姜公猪个体的i为0.24-1.62时,表示该选育苏姜公猪个体具有所需rfi水平,并因此选择所述个体继续用于步骤i至步骤ii。上述步骤a1)至a3),本质上构成了一种表型选择方法。通过额外地引入这几个步骤,可以将传统的表型选择方法与本发明的分子遗传选择方法有机地结合起来,由此通过先行选择低rfi水平个体,再从中选择携带本文所述显著等位基因的个体,由此可选育出具有预期的生长性状的苏姜公猪。传统的苏姜公猪选育方法,是以饲料效率性状为次要育种目标、同时结合表型测定初步开展苏姜猪选育的。这种方法具有较大的误差,准确率不够高。另外,这种方法所需要的选育时间长,要经过多个世代才会有明显差异,并且有时即便历经多个世代也不会有明显差异。然而,相对于传统的苏姜猪选育方法,本发明方法通过个体是否携带serpina3和/或vitronectin基因的显著等位基因来进行苏姜公猪选育,该方法即便在低rfi个体零世代也能通过对显著等位基因的检测来实现准确的苏姜公猪选育。在此分子遗传分析方法的基础上进一步辅以传统的选育方法,将提供更为完美的苏姜公猪选育方法。在第二方面,本发明涉及serpina3基因和/或vitronectin基因在选育苏姜公猪中的应用。在一个实施方案中,选择携带serpina3基因显著等位基因b1和/或vitronectin基因显著等位基因b2的苏姜公猪留为种用。在一个具体的实施方案中,serpina3基因的显著等位基因b1具有序列seqidno6,vitronectin基因的显著等位基因b2具有序列。通过序列比对可以获知,serpina3基因的等位基因a1和b1之间、以及vitronectin基因的等位基因a2分别与其等位基因b2和c2之间,均仅存在一个碱基差异。更具体地,等位基因a1第123位碱基a在其等位基因b1的对应位置为c;等位基因a2第306位碱基t在其等位基因b2的对应位置为c,第137位碱基g在其等位基因c2的对应位置为a。如本文第一方面所述,本领域技术人员应该理解的是,在本发明中,除了利用在本文中专门由seqidno6和表示的序列自身外,还可以利用各自包含上文所述碱基差异、与之分别具有一定序列同源性例如80%-100%同源性(例如81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%、100%)且依然保留其功能性的序列。在此所谓“依然保留其功能性”是指:在苏姜公猪体内不具备seqidno6和/或序列本身而具有其同源序列时,该同源序列的存在仍然能够指示苏姜公猪具有上文所述的低rfi水平。如上文所述,在利用serpina3和/或vitronectin基因进行苏姜公猪选育所选育出的苏姜公猪,由于携带有serpina3基因显著等位基因b1和/或vitronectin基因显著等位基因b2,将具有低rfi水平,由此具有所需的高饲料效率。此外,该应用还可以与表型选择方法相结合,如本发明第一方面所述。更具体地,在应用serpina3和/或vitronectin基因之前,可以预先进行在本发明第一方面所述及的表型选择方法,其包括步骤a1)-a3)。在第三方面,本发明涉及一种用于选育苏姜公猪的试剂盒,所述试剂盒包括:a)用于扩增serpina3基因的引物对1,和/或用于扩增vitronectin基因的引物对2;以及b)利用本试剂盒选育苏姜公猪的使用说明书。在一个实施方案中,所述引物对1的上游引物序列包含seqidno1,下游引物序列包含seqidno2。在另一个实施方案中,所述引物对2的上游引物序列包含seqidno3,下游引物序列包含seqidno4。在通过本发明第一方面的方法对苏姜猪个体进行选育时,可以采用本发明的试剂盒,由此实现对低rfi水平苏姜猪的快速、准确选育。通过本发明,可以对苏姜猪进行更直接、准确和快速的选育,由此为高饲料效率的节粮型苏姜猪新品系培育提供一种有效方法。本发明的选育方法可以选育出具有更高饲料效率的苏姜种公猪,节省养猪周期,提高苏姜猪养猪场的经济效益。下面结合附图以及实施例对本发明进行更为具体和详细的描述。实施例实施例1.对低rfi水平苏姜公猪的评估一、表型数据测定选择初测体重为30kg的苏姜后备公猪,一共56头,饲喂日粮参照《瘦肉型猪饲养技术》中给出的标准(周光宏.瘦肉型猪饲养技术.北京:金盾出版社,2008),结测体重为90kg。在表型数据收集过程中,利用种猪性能测定系统或人工饲喂测定和记录初测日龄、平均日增重、平均日采食量和饲料消耗量,并利用活体背膘仪来测定背膘厚。测定结果见表1。表1各性状的测定结果二、rfi数据模型拟合和计算基于表型数据测定结果(参见表1),应用多元线性回归方法分析拟合猪的最优rfi模型,最终估计出个体的剩余采食量(rfi),数据模型由式(1)所示:rfi=adfi-(b1×onbw+b2×offbw+b3×metamidbw+b4×adg+b5×bf)(1)其中,adfi为平均日采食量、onbw为初测体重、offbw为结测体重、metamidbw为中间代谢体重、adg为平均日增重、bf为背膘厚,其中metamidbw=[(onbw+offbw)/2]×0.75。对于苏姜猪,rfi数据模型具体由式(2)所示:rfi=adfi+0.11323×onbw+0.11673×offbw-0.96405×metamidbw-0.30524×adg-0.02367×bf)(2)测定结果见表2。表2rfi测定结果三、苏姜猪低rfi水平选择在本发明中,对低rfi水平苏姜猪的选择,不仅仅考虑了rfi水平本身,还考虑了平均日增重和背膘厚这两个因素。为此,本发明人以rfi为目标性状,以平均日增重(adg)和背膘厚(bf)为约束性状,根据如下模型分别计算了rfi育种值(ebvrfi)、adg育种值(ebvadg)和背膘厚育种值(ebvbf):ebvrfi=adfi-group-year-onage-offbw-adg-bf-e(3)ebvadg=adg-group-year-onage-e(4)ebvbf=bf-group-year-offbw-e(5)其中adfi为平均日采食量,offbw为结测体重,onage初测年龄,adg为平均日增重,bf为背膘厚,group为测定群体,year为测定年份,e为随机残差。ebvrfi、ebvadg和ebvbf的计算由gps软件进行,结果见表3。表3育种值结果随后,采用公式(6)来计算综合选择指数(i):i=-aebvrfi+bebvadg-cebvbf(6)在本发明中,对于目标性状(剩余采食量(rfi))和约束性状(平均日增重(adg)和背膘厚(bf))的权重,设定rfi育种值的占75%,平均日增重育种值的占15%,平均背膘厚的占10%。由此,上文用于计算综合选择指数(i)的公式(6)可以进一步明确为下式(7):i=-0.75ebvrfi+0.15ebvadg-0.1ebvbf(7)根据计算得到的数值,选择i值为0.24-1.62的个体,该个体为低rfi水平的个体,其rfi水平为-0.214kg/d至-0.106kg/d。综合选择指数(i)的结果见表4。表4综合选择指数(i)选择结果采用不同综合选择指数(i)选择的个体,其生产性状结果如下,其中低rfi水平个体的饲料消耗量低于高rfi水平个体的饲料消耗量。表5高低rfi个体生产性状低rfi(n=9)高rfi(n=10)饲料消耗量(kg)179.6±2.64185.2±3.27平均日增重(g/d)655.3±4.32671.2±2.83平均背膘厚(cm)2.92±2.12.85±3.3实施例2.对饲料效率有显著影响的等位基因的筛选一、从实施例1中使用到的苏姜猪公猪身上采集血液220份,利用na-oh两步法提取dna,方法如下:fta卡打孔(直径1.2mm)取样,200微升20mmnaoh浸泡血样小点30分钟,吸净变色液体加入200微升1倍te室温放置5分钟,吸净残液待血样小点干燥后进行以下步骤。二、等位基因分型分析pcr扩增所使用引物如下:serpina3基因的扩增引物对(引物对1)为:上游引物(seqidno1):agcctcttcacccttctaggccg下游引物(seqidno2):gtagaggctgaaggcgaagtcavitronectin基因的扩增引物对(引物对2)为:上游引物(seqidno3):aggcaactcctcttctctg下游引物(seqidno4):gcttgcactcggccacgtagt上述两对引物的pcr反应体系为:这两对引物pcr反应条件为:95℃预变性5分钟,94℃变性30秒,退火30秒(退火温度:引物对1为58℃,引物对2为60℃),72℃延伸45秒,最后再72℃延伸10分钟。pcr扩增之后,对经这两对引物扩增得到的pcr产物进行sscp分型,具体方法如下:将20μlpcr产物与60μl变性上样缓冲液(98%去离子甲酰胺、10mmedta、0.025%溴酚蓝、0.025%二甲苯青ff)混合,105℃变性5分钟,立即冰浴冷却10分钟,10%(引物对1)和14%(引物对2)非变性聚丙烯酰胺凝胶在室温10.5℃、冷循环4.2℃、390v电压下电泳19小时,银染后显色拍照。对sscp检测为纯合型和杂合型带型样本分别测序,获得通过这两对引物扩增得到的等位基因,其中:serpina3基因的第一个等位基因(在本文中标注为a1等位基因)的序列如下,其中:加粗下划线部分表示外显子:serpina3基因的第二个等位基因(在本文中标注为b1等位基因)的序列如下,其中:加粗下划线部分表示外显子,并且其相对于a1等位基因仅在第123位有差异,从a改变为c,即下文序列中加框碱基:vitronectin基因的第一个等位基因序列(在本文中标注为a2等位基因)的序列如下,其中:加粗下划线部分表示外显子:vitronectin基因的第二个等位基因序列(在本文中标注为b2等位基因)的序列如下,其中:加粗下划线部分表示外显子,并且其相对于a2基因仅在第306位有差异,从t改变为c,即下文序列中加框碱基:vitronectin基因的第三个等位基因序列(在本文中标注为c2等位基因)的序列如下,其中:加粗下划线部分表示外显子,并且其相对于a2基因仅在第137位有差异,从g改变为a,即下文序列中加框碱基:三、显著等位基因判定显著等位基因判定,具体方法如下:应用一般线性混合效应模型(glmm)评估等位基因存在/缺失对饲料消耗量的影响。针对目标性状饲料消耗量,将等位基因的存在和缺失分别定义为1和0。考虑等位基因效应、性别效应及年龄效应为固定因素,家系效应为随机因素,配合下列模型进行最小二乘方差分析,yijkng=μ+mi+gj+wg+ck+xn+eijkng其中:yijkng为目标性状;μ为群体平均值;mi为基因型,等位基因效应;gj为年龄效应;wg为性别效应;ck为家系效应;xn为二级或二级以上互作效应;eijkng为随机残差效应。分析结果采用“平均数+标准误”,其中p<0.05为显著水平,p<0.1表示有影响趋势,p>0.15表示无影响。分析结果见表6。从该表中可以看出,携带b1等位基因(178±2.48kg,p<0.05)和b2等位基因(176.1±5.38kg,p<0.05)的个体,其饲料消耗量显著较低。表6等位基因对猪饲料消耗量的影响(体重阶段30kg-90kg)由上可知,通过对待测样本进行sscp检测,判断个体等位基因b1和b2sscp带型,可以选择携带等位基因b1和/或b2sscp带型的个体,用于培育。上文已经通过一般性说明及具体实施方案对本发明做了详尽的描述,但在本发明基础上,可以对其做一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。参考文献[1]经荣斌,吉文林,赵旭庭,等.苏姜猪新品种培养及展望[j],猪业科学,2016,33(8):129-132.[2]baxtermr.thedesignofthefeedingenvironmentforthepig[d].phdthesis:universityofaberdeen,1986.[3]saintilanr,merouri,brossardl,etal.geneticsofresidualfeedintakeingrowingpigs:relationshipswithproductiontraits,andnitrogenandphosphorusexcretiontraits[j].journalofanimalscience,2013,91(6):2542-2554.[4]patiencejf,rossoni-seraomc,gutierrezna.areviewoffeedefficiencyinswine:biologyandapplication[j],jouranlofanimalsciencebiotechnology,2015,6(1):33.[5]kochrm,gregoryke,chambersd,etal.efficiencyoffeeduseinbeefcattle[j],journalofanimalscience,1963,22:486-94.[6]dehaer,lcmanddevriesag.feed-intakepatternsofandfeeddigestibilityingrowingpigshousedindividuallyoringroups[j],proceedingscience,1993,33:277-292.[7]vallimontje,dechowcd,daubertjm.heritabilityofgrossfeedefficiencyandassociationswithyield,intake,residualintake,bodyweight,andbodyconditionscorein11commercialpennsylvaniatiestalls[j],journalofdairyscience,2011,94(4):2108-2113.[8]liermannw,berka,boschenv,etal.effectsofdietsdifferinginproteinsourceandtechnicaltreatmentondigestibility,performanceandvisceralandbiochemicalparametersoffatteningpigs[j],archivesofanimalnutrition,2016,70(3):190-208.[9]camaral,berrocosojd,comaj,etal.growthperformanceandcarcassqualityofcrossbredspigsfromtwopietrainsirefinesfedisoproteicdietsvaryinginenergyconcentration[j],meatscience,2016,114:69-74.[10]addahw,dzewurr,alenyoregeb.effectsofdietaryrestrictionfollowedbyhighdietaryenergyorproteinoncompensatorygrowthofashantiblackxlargewhitecrossbredweanerpigs[j],tropicalanimalhealthandproduction,2015,1-6.[11]kennedybw,vanderwerfjhj,meuwissenthe.geneticandstatisticalpropertiesofresidualfeed-intake[j],journalofanimalscience,1993,71:3239-3250.[12]caiw,caseyds,dekkersjcm.selectionresponseandgeneticparametersforresidualfeedintakeinyorkshireswine[j],journalofanimalscience,2008,86:287-298.[13]jegoum,gondretf,vincenta,etal.wholebloodtranscriptomicsisrelevanttoidentifymolecularchangesinresponsetogeneticselectionforfeedefficiencyandnutritionalstatusinthepig[j],plosone,2016,11(1):e0146550.[14]jingl,houy,wuh,etal.transcriptomeanalysisofmrnaandmirnainskeletalmuscleindicatesanimportantnetworkfordifferentialresidualfeedintakeinpigs[j],scientificreports,2014(5):11953.[15]vincenta,louveaui,gondretf,etal.divergentselectionforresidualfeedintakeaffectsthetranscriptomicandproteomicprofilesofpigmuscle[j],journalofanimalscience,2015,93(6):2745-2758.[16]dodn,startheab,ostersent,etal.genome-wideassociationandpathwayanalysisoffeedefficiencyinpigsrevealcandidategenesandpathwaysforresidualfeedintake[j],frontgenetics,2014,(5):307.[17]vigorss,sweeneyt,osheacj,etal.pigsthataredivergentinfeedefficiency,differinintestinalenzymeandnutrienttransportergeneexpression,nutrientdigestibilityandmicrobialactivity[j],animal,2016,10(11):1848-1855.序列表<110>江苏农牧科技职业学院<120>serpina3及vitronectin基因在苏姜公猪选育中的应用及其选育方法<130>cf180506s<160>9<170>siposequencelisting1.0<210>1<211>23<212>dna<213>人工序列(artificialsequence)<400>1agcctcttcacccttctaggccg23<210>2<211>22<212>dna<213>人工序列(artificialsequence)<400>2gtagaggctgaaggcgaagtca22<210>3<211>19<212>dna<213>人工序列(artificialsequence)<400>3aggcaactcctcttctctg19<210>4<211>21<212>dna<213>人工序列(artificialsequence)<400>4gcttgcactcggccacgtagt21<210>5<211>408<212>dna<213>苏姜猪(sujiangsusscrofa)<400>5agcctcttcacccttctaggccgtttcccactgggaaaagcagcagctgttacagtgctt60gctggcaaggctgaagggaagctccgggtataacaatgcacacaggctccccaggagggc120gcatacccatacccatcggaggtcacagagccagctagtggcacagaggaggcagcggga180ggctctcggggcttggtctggccctgcctgctcagatgatctttgcttttcagagtggag240acgatgtcacccctcctggctctggggctcttgttggccgggttctgtcctgctgtcttc300tgccaccctggtggcccaacaaaggccgctgaggacggagacaatgggatgcacgtggac360agcctcagcctggcttccagaaacactgacttcgccttcagcctctac408<210>6<211>408<212>dna<213>苏姜猪(sujiangsusscrofa)<400>6agcctcttcacccttctaggccgtttcccactgggaaaagcagcagctgttacagtgctt60gctggcaaggctgaagggaagctccgggtataacaatgcacacaggctccccaggagggc120gcctacccatacccatcggaggtcacagagccagctagtggcacagaggaggcagcggga180ggctctcggggcttggtctggccctgcctgctcagatgatctttgcttttcagagtggag240acgatgtcacccctcctggctctggggctcttgttggccgggttctgtcctgctgtcttc300tgccaccctggtggcccaacaaaggccgctgaggacggagacaatgggatgcacgtggac360agcctcagcctggcttccagaaacactgacttcgccttcagcctctac408<210>7<211>385<212>dna<213>苏姜猪(sujiangsusscrofa)<400>7aggcaactcctcttctctggttccctcatccatgacctctcatctctctgtcccttcctc60aggcatcagaacctagtttaccagacgtgaggccggaggtgctgccatggcacccctgag120gccccttctgatgctggccctgctggcatgggttgctctggctgaccaaggtgcgggagg180atatggatattggtgaccatttgggtcaatgtagggtgggtaagtgtggcctggcctggg240cggtgccagctgtcatactccctctccacagagtcgtgcaagggccgctgcacagacggc300ttcattgccgaaaggaagtgtcagtgtgacgagctgtgctcttactaccagagctgctgc360gctgactacgtggccgagtgcaagc385<210>8<211>385<212>dna<213>苏姜猪(sujiangsusscrofa)<400>8aggcaactcctcttctctggttccctcatccatgacctctcatctctctgtcccttcctc60aggcatcagaacctagtttaccagacgtgaggccggaggtgctgccatggcacccctgag120gccccttctgatgctggccctgctggcatgggttgctctggctgaccaaggtgcgggagg180atatggatattggtgaccatttgggtcaatgtagggtgggtaagtgtggcctggcctggg240cggtgccagctgtcatactccctctccacagagtcgtgcaagggccgctgcacagacggc300ttcatcgccgaaaggaagtgtcagtgtgacgagctgtgctcttactaccagagctgctgc360gctgactacgtggccgagtgcaagc385<210>9<211>385<212>dna<213>苏姜猪(sujiangsusscrofa)<400>9aggcaactcctcttctctggttccctcatccatgacctctcatctctctgtcccttcctc60aggcatcagaacctagtttaccagacgtgaggccggaggtgctgccatggcacccctgag120gccccttctgatgctgaccctgctggcatgggttgctctggctgaccaaggtgcgggagg180atatggatattggtgaccatttgggtcaatgtagggtgggtaagtgtggcctggcctggg240cggtgccagctgtcatactccctctccacagagtcgtgcaagggccgctgcacagacggc300ttcatcgccgaaaggaagtgtcagtgtgacgagctgtgctcttactaccagagctgctgc360gctgactacgtggccgagtgcaagc385当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1