三七SSR多态性引物、多态性检测方法及SSR标记在总皂苷含量确定上的应用与流程
本发明涉及分子技术领域,具体涉及三七ssr多态性引物、多态性检测方法及ssr标记在总皂苷含量确定上的应用。
背景技术:
三七(panaxnotoginseng(burk.)f.h.chen)是五加科(araliaceae)人参属(panax)多年生草本植物,分布于中国西南地区。三七的主要成分有三七总皂苷(pns)、三七素(三七氨基酸)、黄酮、挥发油、氨基酸、糖类等,具有止血、保护心肌细胞、保护脑组织、降血脂、抗血栓、增强免疫力、抗炎、抗纤维化、抗肿瘤、消除氧自由基、抗氧化等药理作用。三七纺锤根是著名的跌打损伤特效药,止血散瘀、定痛消肿的功效良好,叶、果及根状茎也可入药,是“云南白药”和“片仔癀”的有效成分之一。
三七主要分布于我国北回归线附近的云南、广西的高海拔地区。云南东南部的文山及其周边地区是三七主产区和道地产区,种质面积和产量占全国98%。到目前三七已有400多年的栽培历史,但三七种质混杂现象仍然存在。加之三七生态适应能力弱、地理分布区域狭窄、生长周漫长、繁殖系数低、种子寿命短以及种植过程中,频繁的使用农药和化肥导致三七产生连作障碍,可种植三七的土地面积逐渐减少,原因主要是由于目前对三七遗传多样性了解还不够,遗传资源的利用缺乏理论指导;对形态类型的遗传背景不清楚,不能有针对的进行新品种选育。开发分子标记将为三七种质资源的利用和遗传改良提供依据。
三七分子标记研究:
目前国内外对三七的研究主要集中在化学成分、皂苷含量变、药理药效、三七对重金属敏感性以及连作体系中土壤细菌和真菌群落动态与三七死亡率的相关性。随着dna分子标记技术快速发展,snp、rapd、aflp及est-ssr等标记相继用于人参属植物中。
hongmeiluo等利用二代测序技术从三七的根部获得大量est数据,找到与三萜皂苷生物合成相关酶编码序列的ssr标记。董林林等对三七抗病群体的snp位点进行检测,发现record_519688位点与三七抗病性相关,并对三七群体潜在的抗病性进行系统选育,选育了首个三七抗病新品种苗乡抗七1号;李翠婷等通过对野三七转录组数据进行分析,发现野三七的转录组中存在大量ssr位点,为野三七的遗传多样性分析、遗传图谱构建提供了丰富的候选分子标记;杨云等利用issr、srap两种分子标记,对集团选择四代后的3个改良群体和自然群进行一致性、稳定性和特异性鉴定与评价。结果表明,集团选择的3个群体一致性均大于自然群体;刘丽等利用rapd技术对人参、西洋参以及三七的亲缘关系进行评价,结果表明人参和西洋参亲缘关系较近,和三七的亲缘关系相对较远;张金渝等利用est-ssr标记,以4个不同栽培居群通过3代集团选育的17份外观形状基本一致的选育品系为材料,以屏边三七为对照分析群体间遗传多样性,结果显示,17份选育品系材料和屏边三七被划分为4大类群,其中17份三七选育品系被分为3个类群,屏边三七单独聚为1个类群。
三七遗传多样性分析:
遗传多样性是指生物所携带的遗传信息的总和。广义上是指种内或种间在分子、细胞、个体水平上的遗传变异;狭义上指种内不同群体以及同一群体不同个体间的遗传多态性程度。
从1987年云南省文山州三七研究所建立屏边三七、姜状三七、竹节参、人参、西洋参、峨眉三七、狭叶竹节参等人参属植物种质资源圃以来,三七在栽培育种、生物学、化学成分、病虫害防治以及gap种植等方面已经进行了细致研究,但对其遗传结构和遗传多样性的研究还不够,没有进行系统全面的分析。
遗传变异检测最古老、最简单易形的方法利用形态学或者表型性状,表型性状主要包括符合孟德尔遗传规律的单基因性状和由多基因决定的数量性状。肖慧等对文山州四个地区的10个三七栽培群体33个表型性状进行观测、统计和分析,结果表明三七居群间和居群内多形性明显,表型变异丰富,具有丰富的遗传多样性;张金渝等对文山四个地区10个不同栽培居群的表型性状进行统计分析,结果表明有关质量性状(包括植株鲜重和干重、主根鲜重和干重、剪口鲜重和干重)的变异系数和幅度在所有性状均较大,表明三七栽培居群质量变异丰富。根据表型多样性指数分析显示,数量性状的遗传多样性较丰富。
通过遗传标记中的分子标记找到控制表型性状的基因,以及基因组所处的位置,来实现标记辅助选择育种。dongwang等用荧光aflp对来自文山4个县栽培三七群体的92个植株的遗传多样性进行分析,发现样品之间不仅形态特征变异较大,分子变异分析也显示种群内的遗传多样性高达93.5%;肖慧等通过同工酶标记对三七及其近缘种屏边三七的遗传多样性进行研究,结果显示,遗传变异主要存在于居群间,居群内遗传分化较小;张金渝通过est-ssr对文山6个居群三七和珠子参以及屏边三七的遗传多样性进行分析,结果显示三七居群内遗传多样性丰富,三七和珠子参有较近的遗传关系,和屏边三七遗传距离较远。
以上研究均表明三七在群体内部和群体之间都存在丰富的遗传变异。三七新品种的选育成效很大程度上取决于品种的遗传多样性。通过研究三七的遗传多样性,不仅有利于建立三七品种收集保存的理论依据,还能锁定和改良某些性状,为三七品种改良提供理想的种质资源。
ssr作为一种分子标记,和est-ssr相比较,基因组ssr能够揭示比est-ssr更高的多态性和遗传信息,同时基因组ssr分布贯穿生物基因组,est-ssr只是表达基因的一部分。choyg等在水稻中发现,基因组ssr多态性为83.80%高于est-ssr的多态性(54.00%),eujayi对硬粒小麦的基因组ssr和est-ssr进行比较发现,est-ssr产生高质量的标记,但是揭示的多态性(25%)比基因组ssr低(53.00%)。
前人对三七及其野生近缘种遗传多样性的研究包括同工酶标记、issr、rapd和est-ssr。但是对于三七新品种选育和遗传改良的分子标记还很少,通过est开发的ssr标记,只对基因组序列的一小部分进行分析,因此对三七全基因组ssr标记的分布特点研究较少。
技术实现要素:
针对以上技术问题,本发明提供了三七ssr多态性引物、多态性检测方法及ssr标记在总皂苷含量确定上的应用。
本发明的技术方案:三七ssr多态性引物,包括16组引物对,如序列表中seqidno.1-32所示。
三七ssr多态性检测方法,包括:以三七的基因组dna为模板,采用前面所述的引物进行pcr扩增,将得到的产物吸取2μl和6μl溴酚蓝混合,在1×tbe缓冲液、0.8%含eb的琼脂糖凝胶中进行电泳,电压300v,时间12min,凝胶在jy04s-3c凝胶成像分析系统进行成像分析;
反应体系为15μl,正向引物和反向引物各1μl(10nmol),dna模板1μl(20ng),荧光引物1μl(10nmol),ddh2o4.5μl,其余用水补齐;
反应程序为:94℃预变性5min,94℃变性30s,各引物的最佳退火温度30s,72℃延伸30s,30个循环,72℃延伸10min,4℃保存。
本发明还提供了三七ssr标记在总皂苷含量确定上的用途。
进一步的,三七总皂苷与位点ssr35有关联。
进一步的,三七总皂苷与位点ssr2-57有关联。
与现有技术相比,本发明的有益效果为:
1、本发明提供了三七多态性检测的方法及引物,对三七品种表型的研究提供了有利的技术支持,品种鉴定。
2、对ssr标记与总皂苷的关联性研究,有利于高含量总皂苷三七育种的开展。
附图说明
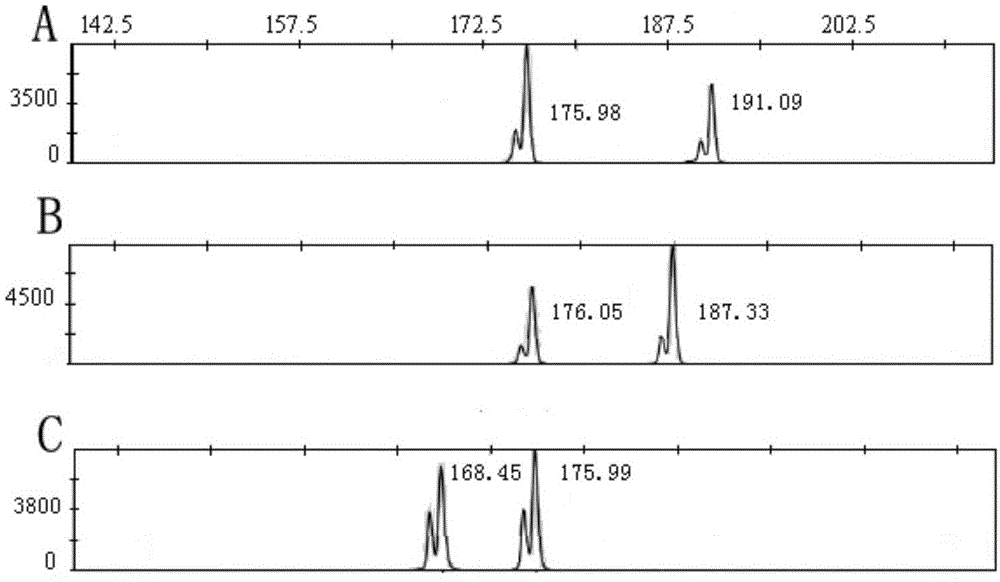
图1为引物psq63部分扩增产物毛线管电泳峰图,图中横坐标是扩增产物大小,纵坐标是信号强度,双峰表示二倍体杂合体植株,a、b、c分别代表对照、紫茎和绿茎三七;
图2为三七群体结构分析lnp(d)值随k值的变化规律;
图3为三七群体结构分析δk值随k值的变化规律;
图4为基于ssr标记的248份三七不同栽培居群遗传结构图,红色表示群体1,绿色表示群体2,图中编号1-28代表zy群体、29-57代表ag群体、58-80代表u6群体、81-92代表lj群体、93-121代表dqhy群体、122-147代表kq群体、148-175代表bq1群体、176-203代表bq2群体、204-231代表zhy群体,232-248代表mxyh群体;
图5为三七群体皂苷含量hplc色图谱,a代表ag,b代表kq,c代表zy。
具体实施方式
以下将结合附图和实施例对本发明技术方案作进一步地详述。
实施例:本发明基于三七基因组测序信息,挖掘三七基因组中ssr位点信息,开发多态性引物,分析三七(p.notoginseng)及其近缘野生种竹节参(p.japonicus),屏边三七(p.stipuleanatus),姜状三七(p.zingiberensis)和变种狭叶竹节参(p.japonicusvar.angustifolius),野三七(p.vietnamensisvar.fuscidiscus),珠子参(p.japonicusvar.major)之间亲缘关系。
本发明采用不同的三七种质资源作为关联分析的材料,主要包括经过集团选育的三七品种;“苗乡三七1号”、抗七1号“”、滇七1号“”;两个选育品系:优6、矮杆;以及分别来自丽江市古城区七河乡、普洱市镇沅县九甲乡、文山州丘北县的5个栽培居群,在分析遗传多样性的基础上,不同软件处理结果相互验证得出群体结构。与此同时,观测三七的表型性状,测定皂苷含量,分析各性状间的相关性,并用tassel软件得到各性状相关联的ssr标记。找到与三七表型关联的ssr标记,为三七遗传改良和新品种选育提供理论依据。
1材料和方法
1.1试验材料:
表1所示材料均为三七,包括经集团选育形成的苗乡三七1号、抗七1号、滇七1号;两个选育品系:优6、矮杆以及分别来自文山州丘北县、砚山县中和营乡、普洱市镇沅县九甲乡、丽江市古城区七河乡的5个不同栽培三七居群。取新鲜幼嫩叶片用于提取三七基因组dna;并对10个三七群体的表型性状进行测量和统计。
表1三七不同栽培群体材料信息
1.2实验仪器设备和试剂
主要实验仪器设备:实验所用主要仪器为游标卡尺、直尺、卷尺、铅笔、电子天平、各量程移液枪、h1650-w台式微量离心机(湖南湘仪实验仪器开发有限公司)、l550台式低速大容量离心机、vortexql-901涡旋混合器、ssw-420-2s型电热恒温水浴槽、thermoq恒温金属浴、2720thermalcyclerpcr仪、3730测序仪、jy300cpower电泳仪、jy04s-3c凝胶成像分析系统、agilent1260infinity液相色谱仪(安捷伦科技有限公司)、sb-5200d超声波清洗机(宁波新芝生物科技股份有限公司)、美国丹佛电子分析天平tp-214、mettler-toledoae240型电子天平(瑞士mettler-toledo公司)、奥利邦1000a型粉碎机(上海赛耐机械有限公司)。
主要试剂:实验用的主要试剂为mix酶、1×溴酚蓝、琼脂糖、gs500liz、荧光染料tamra、hex、fam、marker、eb、0.5×tbe、100%甲醇、无水乙醇、无菌水、昆明硕擎生物科技有限公司提供的trelieftmplantgenomicdnakit试剂盒(rnasea、bufferbl、buffergp1、buffergp2、bufferpw、washbuffer、tebuffer、spincolunms、collectiontube)
1.3实验方法
1.3.1三七基因组中ssr位点信息搜索
利用软件misa(microsatelliteidentificationtool,http://pgrc.ipkgatersleben.de/misa/)对重新组装的2.39gb三七基因组进行ssr位点检测。搜索标准选择重复单元为1、2、3、4、5、6个碱基的基序,最小重复数分别为12、6、5、5、4、4。两个相邻ssr之间的碱基数≦100,则被定义为复合型ssr。
1.3.2三七ssr引物设计与合成
根据ssr位点两端的保守序列,利用primer3(http://primer3.sourceforge.net/)设计引物,引物长度20~23bp,退火温度60℃左右;引物中碱基重复数小于等于4,引物两端不能有2个连续的a/t碱基;pcr产物大小在150和300bp之间。随机选择200对引物,正向引物5’端加上m13f通用引物序列5'-tgtaaaacgacggccagt-3',由昆明硕阳科技有限公司合成,以筛选多态性引物。
1.3.3三七基因组dna的制备与检测
取供试三七材料嫩叶组织液氮保存,用云南昆明硕擎生物科技有限公司提供的trelieftmplantgenomicdnakit试剂盒进行提取,提取步骤如下:
(1)将spincolumn置于collectiontube中,加入250μlbufferbl,12000rpm离心1min,活化硅胶膜。
(2)取三七幼嫩叶,加入液氮充分研磨,研磨后置于1.5ml离心管中,加入400μlbuffergp1,涡旋震荡1min,65℃水浴10-30min,期间取出颠倒混匀以充分裂解。
(3)加入150μlbuffergp2,涡旋震荡1min,冰浴5min。
(4)12000rp离心5min,将上清液转移至新的离心管中。
(5)加入上清液等体积的无水乙醇,立即充分震荡混匀,液体全部转入spincolumn中,12000rp离心30s,弃废液。
(6)向spincolumn中加入500μlbufferpw(使用前先检查是否已加入无水乙醇),12000rp离心30s,弃废液。
(7)向spincolumn中加入500μlwashbuffer(使用前先检查是否已加入无水乙醇),12000rp离心30s,弃废液。
(8)重复操作步骤7。
(8)将spincolumn放回collectiontube中,12000rp离心2min,开盖晾干,1min。
(10)取出spincolumn放入一个干净的离心管中,在吸附膜的中央加入50~100μltebuffer(65℃预热tebuffer洗脱效果更佳)20~25℃放置2min,12000rp离心2min。
dna检测方法。取2μldna样品加6μl溴酚蓝混匀,0.8%的琼脂糖凝胶,电压300v,时间12min,用jy04s-3c凝胶成像分析系统进行dna质量检测。
1.3.4三七ssr-pcr扩增体系
引物序列参照三七基因组序列中ssr位点的侧翼序列,用primer3进行设计,由昆明硕擎生物科技有限公司合成。
ssr-pcr一次扩增反应体系(15μl):
扩增反应程序为:94℃预变性5min→94℃变性30s→各ssr引物的最佳退火温度30s→72℃延伸30s→30个循环,完成最后一个循环后→72℃延伸10min→4℃保存。
第二次扩增反应体系(15μl):
第一次扩增结束后,将得到的pcr产物吸取2μl和6μl溴酚蓝混合,在1×tbe缓冲液、0.8%琼脂糖凝胶(含eb)中进行电泳,电压300v,时间12min,凝胶在jy04s-3c凝胶成像分析系统进行成像分析,产生清晰条带的引物可用于二次扩增。二次扩增程序和一次扩增的反应程序相同,二次扩增后得到的产物在94℃下变性5min,放在-20℃冰箱保存之后用3730测序仪进行测序。
1.3.5ssr多态性引物筛选
随机选取通过primer3设计的ssr引物200对,采集16个单株的新鲜叶片用于多态性引物筛选,其中自然群体6个单株、苗乡三七1号6个单株和滇七1号4个单株。以16个材料的dna为模板,对这200对引物进行pcr扩增,pcr产物用3730测序仪分析,根据测序结果筛选多态性引物。
1.3.6数据分析
用软件genemapperv4.1对测序结果进行位点多态性分析,根据产生的峰图确定核心位点片段大小,选择特异性高,多态性好的位点峰图产生excel数据,利用popgene32软件计算物种水平和种群水平的观测等位基因数(observednumberofalleles,a)有效等位基因数(effectivenumberofalleles,ae)、shannon信息指数(shannon’sinformationindex.i)、观测杂合度(observedheterozygosity,ho)、期望杂合度(expectedheterozygosity,he)、nei’s期望杂合度(nei’sexpectedheterozygosity,h)、遗传分化系数(geneticdifferentiationcoefficient,fst)、基因流(geneflow,nm)及nei’s无偏遗传距离(nei’sunbiasedgeneticdistance,gd)用于评价遗传多样性。
根据popgene32计算的nei’s无偏遗传一距离,运用ntsyspc2.1软件以不加权成对算术平均法(unweightedpair-groupmethodwitharithmeticmean,upgma)进行聚类分析,并绘制各种群聚类分析图。软件piccalc计算每个标记的多态性信息含量,统计分析时将ssr视为共显性标记。
1.3.7主要表型性状观测
表型性状调查:
本发明对三七10个居群300个单株的根、茎、叶等17个性状进行观测,包括:茎色、茎数、茎粗、株高、复叶数、小叶数、复叶柄长、叶型、叶缘锯齿、中叶长、中叶宽、中叶长宽比、主根形状、主根长、主根粗、主根鲜重和主根干重。
三七皂苷含量测定:
将三七主根在60℃条件下恒温干燥至恒重,用粉碎机将其粉碎,用分析天平称取0.6g样品,置于50ml锥形瓶中,加入50ml100%甲醇溶液,用封口膜封住瓶口浸泡30min,超声提取30min,取出静置30min,用0.45μm微孔滤膜过滤,取滤液,对三七皂苷r1、人参皂苷rg1、人参皂苷re、人参皂苷rb1和人参皂苷rd五种皂苷进行测量。
表2流动相梯度洗脱程序
色谱条件:使用安捷伦高效液相色谱仪(algilent1260,usa)对10个三七居群主根皂苷含量进行检测,色谱柱为agilentzorbaxsb-c18(250mm×4.6mm,5μm)流动相为0.2%磷酸水(a)-乙腈(b),流速为1.5mlmin-1,检测波长203nm,柱温30℃,进样体积10μl。
1.3.8表型性状和皂苷含量数据分析
运用spass20.0软件进行供试材料性状表型数据的统计描述和方差分析,计算性状间的pearson相关系数;变异系数cv%的分析采用(cv%=标准差/平均值)×100。
1.3.9三七品质和表型性状的关联分析
群体结构分析:利用软件structure2.2对群体结构进行估计,structure参数设置分别为:k值选取1-10,重复次数设为10;将mcmc(markovchainmontecarlo)开始时的不作数迭代设为10000次(lengthofburninperiod),再将不作数迭代后的mcmc设为100000次,其余参数采用软件默认的设置。根据lnp(d)计算δk,并依据δk选择一个合适的k值,并得到该k值对应的q矩阵(第i材料的基因组遗传变异来源于第k群体的概率)。
连锁不平衡分析:采用软件tassel2.2(http://www.maizegenetics.net/bioinformatics/tassel/)进行分析,首先将原始的0,1数据按照tassel2.2的数据输入格式转换为等位基因形式导入数据,利用data模块将基因型数据进行过滤,然后再analysis模块中使用link.diseq分析各位点间ld值矩阵图,包括r2和p值。
表型和皂苷含量关联分析:首先利用软件tassel2.2对基因型数据生成亲缘关系矩阵(k矩阵),结合基因型数据、性状值、structure软件得出的q矩阵,采用混合线性模型(mlm)进行关联分析,并计算标记位点对表型变异的贡献率,其中,群体q值作为分析过程的协方差。
2结果和分析
2.1三七基因组ssr多态性引物的筛选和验证
利用随机筛选并合成的200对三七ssr引物对样本位点进行多态性检测,将所有pcr扩增产物进行毛细管电泳,利用genemapperv4.1产生的峰图检测样品位点信息。ssr重复基序的重复数目差异以及短的插入缺失事件形成ssr多态性,峰图所显示的信息是带有荧光引物的pcr产物在经过毛细管电泳之后所反馈的产物片段信息。通过分析每一对引物在各个样品中产生的峰图显示的各自特定位点片段大小信息之后,其中有27对引物的峰图大小相同不存在差异,有132对引物没有扩增出片段,最终得到41对引物的产物符合预期大小,将这41对引物峰图用于计算引物多态性信息含量,见附图1。
通过软件popgene32计算41对引物的等位基因观察值,预期等位基因数,shannon指数和多态性信息含量,各个位点等位基因数目范围从2~10,平均数目为3.9。计算了41对引物的pic值发现16对引物具有高度多态性(pic>0.50),23对引物具有中度多态性(0.30<pic<0.50),2对表现低度多态性(pic<0.30),pic平均值为0.46,见表3。
表3用于本试验的41个ssr位点序列及相关信息
2.2三七栽培群体遗传多样性分析
2.2.1三七主要表型性状频数分析
表4三七不同栽培群体7个二元和多态性状频数分析(n=30)
表5三七不同栽培群体11个数量性状的均值(mean±sd,n=30)
通过分析主要表型性状在10个栽培三七居群间的平均值大小发现,株高在群体间的变化范围为28.8~45.3,kq群体株高最高(45.3);茎粗在群体间的变化范围为0.3~0.7,在lj群体最大(0.7cm);中叶长在群体之间的变化范围为9.7~15.4,其中zy、ag、kq、mxyh群体的中叶长差异不显著,lj群体中叶长最大(15.4),dqyh、u6、qb1、qb2、zhy之间中叶长差异不显著;中叶宽、中叶长宽比在群体之间的差异和中叶长在群体之间的差异分布相同;复叶柄长在群体之间的变化范围为7.0~11.4,lj群体复叶柄最长(11.4),ag群体复叶柄长度最短(7.0);主根长度变化范围为3.7~7.6,其中ag群体主根长度最长(7.6),qb1群体主根长度最短(3.7);在lj(2.7)和zy(2.6)群体中主根粗最大且差异不显著;同样的主根鲜重和主根干重在lj和zy群体中也是最大(表5)。
从表6可以看出个体间的差异几乎存在于每一个居群的每一个性状中。相比之下,中叶长宽比(9.6%)、主根粗(11.0%)、茎粗(11.6%)较其他性状稳定,其中叶长和叶宽在一定程度上反映叶型,中叶长宽比在群体之间变异不大也反映了群体间叶型变异较小;根部性状中主根长、主根鲜重和主根干重变异较大且不稳定。从居群层次单个性状看,相同性状的不同居群间变异不同。以株高为例,十个居群中株高变异范围为9.0%~20.5%,其中qb居群变异系数最小(cv=9.0%),kq变异系数最大(cv=20.5%)。同一居群不同性状的变异系数也不同,以砚山县kq为例,10个性状的cv值各不相同,其中茎粗变异系数为0,主根鲜重和主根干重的变异系数最大,cv值分别为50.4%、51.1%。
在各居群中10个性状的平均变异系数范围为14.33%~24.71%。其中mxyh的变异系数变异度最大(24.71%),ag群体的变异系数变异度最小(14.33%);kq和dqyh两个品种的平均变异系数分别为22.82%、18.80%;两个选育品系ag、u6的平均变异系数分别为14.33%、16.55%。
表6三七不同栽培群体11个数量性状的变异系数cv(%)(n=30)
注:茎数sn:stemnumber;小叶数nl:numberofleaflets;复叶数ncl:numberofcompoundleaves;茎粗sd:stemdiameter;株高ph:plantheight;中叶长mll:middelleaflength;中叶宽mlw:middelleafwidth;中叶长宽比lwr:lengthwidthratioofmiddellobe;复叶柄长plcl:petiolelengthofcompoundleaf;主根长mrl:mainrootlength;主根粗mrd:mainrootdiameter;主根鲜重mrfw:mainrootfreshweight;主根干重mrdw:mainrootdryweight.
2.2.2不同居群三七遗传多样性
在物种水平上,26对引物在三七10个栽培群体248个单株中共检测到218个等位基因,等位基因的数目从2到23,平均为8.4;有效等位基因数(ae)范围从1.2227到11.5357,平均值为3.13;shannon信息指数(i)范围从0.4214到2.6436,平均值为1.21;观测杂合度(ho)范围从0.0936到0.8024,平均值为0.51;期望杂合度(he)范围从0.1825到0.9152,平均值为0.6;nei’s期望杂合度(h)范围从0.1821到0.9133,平均值为0.6;以上数据表明,三七物种水平遗传多样性丰富,见表7。
每个标记的相关信息可以根据他的pic值进行评估,根据nei’s(1973),每个ssr的pic值反映了基因的多样性,pic≥0.5为高度多态性,pic≤0.3为低度多态性,0.3≤pic≤0.5为中度多态性。根据表7可知,所有位点的平均pic为0.54(>0.5),为高度多态性,遗传变异大,最高pic值为位点ssr2-46(0.91)和ssr2-41(0.85),表明标记间的遗传多样性丰富,可作为关联分析的分子标记(表7)。遗传分化数值介于0.0064和0.2674之间,平均值为0.0446;这表明三七有4.46%的遗传变异存在于居群间,有95.54%存在于种群内。位点之间的基因流(nm)数值介于0.6849和38.93之间,平均值为10.3,这一结果表明三七种群间基因交流较大(表7)。
在种群水平上,所选材料观测等位基因数(a)范围从3.88到5.23,平均值为4.7;有效等位基因数(ae)范围从2.52到2.979,平均值为2.74;shannon信息指数(i)范围从0.9982到1.119,平均值为1.07;观测杂合度(ho)范围从0.4899到0.5507,平均值为0.5129;期望杂合度(he)范围从0.5688到0.6,平均值为0.5806;nei’s期望杂合度(h)范围从0.5456到0.5816,平均值为0.5675。从各遗传参数来看,三七群体间存在较大的遗传变异,见表8。
表726个ssr位点遗传多样性系数
表8三七不同栽培群体遗传多样性
三七中主要的有效成分包括三七皂苷r1、人参皂苷rg1、人参皂苷re、人参皂苷rb1和人参皂苷rd。通过对十个三七群体进行测定五种皂苷含量。从结果分析来看,三七不同栽培群体的5种单体皂苷含量、r1+rg1+rb1含量以及总皂苷含量之间存在显著差异。三七皂苷r1含量在zy(2.6%)群体中最高,zhy(1.2%)、lj(1.4%)群体中最低;人参皂苷rg1含量在zy(7.8%)、qb(7.6%)、dqyh(7.5%)群体中相对较高,且差异不显著;人参皂苷re含量在zy(1.4%)群体中最高,lj(0.4%)群体中最低,其他群体之间差异不显著;人参皂苷rb1(8.5%)在zy群体中含量最高,lj(2.3%)群体中最低;人参皂苷rd含量在zy(1.8%)和qb1(1.6%)两个群体中最高,lj(0.6%)群体中最低。r1+rg1+rb1三种皂苷总和在zy(18.8%)和qb1(16.9%)两个群体中最高,lj(7.3%)群体中最低。总皂苷含量在zy(22.0%)和qb1(19.6%)两个群体中最高,lj(8.3%)群体中最低。比较5种单体皂苷含量和总皂苷含量发现,他们在dqyh、mxyh群体,qb1、qb2群体间差异不显。
表9十个群体三七总皂苷含量
注:同列数值后不同小写字母表示差异显著(p﹤0.05)
2.2.3群体结构分析
通过structure2.2软件,对10份三七材料进行群体结构分析,以消除群体结构在关联分析中的假阳性。从图2可以看出,随着k值的增大,后验概率对数(lnp(d))也是逐渐增加的,若根据lnp(d)值无法确定合适的k值。根据evannog,2005.计算δ算的方法,很方便的计算出不同k取值范围下的δ值值,从而将10份材料划分为2个亚群。图3反应的是不同k取值下lnp(d)和δn的变化。图4是当k=2时的群体结构图,图5为三七群体hplc色图谱。
图4是利用structure软件对248份试验材料进行的群体结构分析,从图中可以看出,根据最佳k值(k=2),248份供试材料被分为2个亚群,图中横坐标代表个体编号,纵坐标表示个体被划分到不同亚群的概率。亚群1主要包括152份材料,分别是镇沅28份,矮杆28份,23份u6,滇七1号29份,丽江12份,抗七1号26份;亚群2主要包括95份材料,qb群体55份,中和营群体28份,苗乡1号12份。
2.2.4三七皂苷含量和表型性状相关分析
利用spass20.0的pearson相关分析,我们得到了三年七不同栽培居群的皂苷含量和表型性状的相关系数表10。综合各项指标相关结果,得出这样的相关性结论:在0.01水平显著正相关的指标:r1和rg1、rd、总皂苷含量;rg1和re、rb1、rd、总皂苷含量、茎数;re和rb1、rd、总皂苷含量;rb1和rd、总皂苷含量、茎数、复叶数;rd和总皂苷含量、茎数;总皂苷含量-茎数;总皂苷含量和复叶数、主根干重;小叶数和复叶数、茎粗、株高、复叶柄长、主根粗、主根干重;复叶数和茎粗、株高、复叶柄长、主根粗、主根鲜重、主根干重;茎粗和株高、中叶长、中叶宽、中叶长宽比、复叶柄长、主根粗、主根鲜重、主根干重;株高和复叶柄长、主根粗、主根干重;中叶长和中叶宽、中叶长宽比、主根粗、主根干重;中叶宽和复叶柄长、主根粗、主根干重;复叶柄长和主根粗、主根干重;主根粗和主根鲜重、主根干重;主根鲜重-主根干重。
在0.01水平显著负相关的指标:rg1-主根长;re-中叶长;rb1和中叶长宽比、主根长;rd-主根长;总皂苷含量和中叶长宽比、主根长;小叶数-主根长;复叶数-主根长;茎粗-主根长;中叶宽-主根长;复叶柄长-主根长;主根长-主根粗。
2.2.5三七皂苷含量和ssr标记的关联分析
本文采用tassel软件的混合线性模型(mlm)方法,对三七皂苷含量和13个表型性状进行关联分析。表11反映的是p<0.05,p<0.01条件下三七皂苷含量以及表型性状显著关联的标记,在26个标记中发现8个标记和三七表型、皂苷含量相关联,表型变异解释率范围为0.3%-9.6%,平均解释率为1.7%,其中一些ssr标记与2个或多个表型性状关联。
从表11可以看出,在p<0.05显著条件下,与表型性状相关的标记有7个,分别是1个与茎数相关,表型变异解释率为0.5%;1个与复叶数相关,表型变异解释率为0.8%;3个与主根长相关,表型变异解释率平均值为0.5%;3个与主根鲜重相关,表型变异解释率平均值为0.67%;2个与主根干重相关,表型变异解释率为0.6;同时这7个标记与至少两个以上性状关联。
在p<0.01显著条件下,有7个标记与表型和皂苷含量相关,分别是1个与小叶数相关,表型变异解释率为0.8%;2个与复叶数相相关,表型变异解释率为0.9%;2个与茎粗相关,表型变异解释率为1,4%;1个与rd含量相关,表型变异解释率为5.0%;1个与rg1含量相关,表型变异解释率为0.4%。
表10三七不同栽培群体皂苷含量和表型性状相关分析
注:*和**分别表示在0.05水平和0.01水平显著相关。note:*and**representsignificanceat0.05and0.01level,respectively总皂苷含量ts:totalsaponinscontent.
表11三七不同栽培群体皂苷,表型性状和ssr位点关联分析结果
p-marker:标记与表型性状的显著性;r2:标记对表型变异的解释率;*和**;在0.05好0.01水平的显著相关性。
为进一步研究与表型相关联的ssr标记的可靠性,用26个ssr标记对文山地区8个不同三七居群的208份材料进行全基因组扫描,在0.05和0.01水平条件下,检测到与表型和皂苷含量相关联的ssr标记8个,表型变异解释率为0.1%-17.06%,平均值为2.3%,见表12。
在p<0.05显著条件下,8个标记均与表型和皂苷含量相关联,其中ssr35与茎数,小叶数,re,rb1相关联;ssr2-6和复叶数,主根干重,主根鲜重和re相关联;ssr2-57与复叶柄长和中叶长宽比相关联;ssr43与主根干重,r1含量,株高,中叶长,主根鲜重相关联;ssr89和主根干重相关联,表型变异解释率为2.2%;ssr42与rd相关,表型变异解释率为2.2%;ssr9和主根粗关联,表型变异解释率为1.0%;ssr92与主根鲜重,中叶长宽比相关联,见表12。
在p<0.01显著条件下,有7个标记与表型性状和皂苷含量关联,其中ssr89和茎数,复叶柄长,中叶宽,中叶长宽比,主根长和主根粗相关联;ssr2-6和茎数和主根粗相关联;ssr43和小叶数,复叶数,复叶柄长和中叶宽相关联;ssr92和小叶数,复叶柄长以及主根干重关联;ssr35和复叶数,总皂苷,r1,rd,中叶长宽比以及主根长相关联;ssr2-57和复叶数,中叶长以及中叶宽相关联;ssr9和rd关联,表型变异解释率为3.8%,见表12。
表12文山地区三七不同栽培群体皂苷,表型性状和ssr位点关联分析结果
在两个不同地区间都检测到的位点有8个,同一性状与多个位点关联,分别是3个与小叶数相关,5个与复叶数相关,4个与茎粗相关,4个与株高相关,4个与中叶长相关,4个与中叶宽相关,4个与复叶柄长相关,4个与主根长相关,7个与主根粗相关,2个与总皂苷相关,3个与re相关,4个与rd相关,1个与rg1相关,4个与r1相关,2个与rb1相关;其中与主根鲜重和主根干重相关的7个位点相同,见表13。
表13不同地区都检测到的位点
以上内容是结合具体的优选实施方式对本发明所作的进一步详细说明,不能认定本发明的具体实施只局限于这些说明。对于本发明所属技术领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干简单推演或替换,都应当视为属于本发明的保护范围。
序列表
<110>云南农业大学
<120>三七ssr多态性引物、多态性检测方法及ssr标记在总皂苷含量确定上的应用
<130>2018
<160>32
<170>siposequencelisting1.0
<210>1
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>1
ctacattgtatttgccttttggc23
<210>2
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>2
taactcaaaactagccagaccca23
<210>3
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>3
cttcgtctccgtcttcttcacta23
<210>4
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>4
ctcgtccacgatccagatattta23
<210>5
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>5
gatcgccggttatgtatttgtat23
<210>6
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>6
tcctagtagtcgttgcacgtaga23
<210>7
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>7
ggtccttatatgttgtctgtcgc23
<210>8
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>8
caaagcaaaacttccggatacta23
<210>9
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>9
gcaatgccaataaccacaactat23
<210>10
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>10
agtcaaagccagaacaaacttca23
<210>11
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>11
gtcaaaacattgacatgcctaca23
<210>12
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>12
tattatgtccgttatgactgggg23
<210>13
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>13
atacaaccttggtaacatcctcg23
<210>14
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>14
gaagtgctttagttttgcagcat23
<210>15
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>15
tcctctgtcattgtcttatgtgc23
<210>16
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>16
gatcaacaatatcccaaaatcca23
<210>17
<211>22
<212>dna
<213>notoginsengradixetrhizoma
<400>17
agttttcatggttattgagtgg22
<210>18
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>18
attcaacaattttgcctcagaga23
<210>19
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>19
ggttgggtttattcctcactgat23
<210>20
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>20
tctccatcacacttattgcattg23
<210>21
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>21
tgctacctcgtgctaccatagat23
<210>22
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>22
atccgtttagaggatctttgacc23
<210>23
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>23
cggagataaggcttttccttgta23
<210>24
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>24
atcaatcctccattaatcttcca23
<210>25
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>25
catttctcttgttgaaaattggc23
<210>26
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>26
aggaaaaggggaaagagaagaag23
<210>27
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>27
ctccctctccatctcctacctat23
<210>28
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>28
tggaccaagtaccatatgacaga23
<210>29
<211>24
<212>dna
<213>notoginsengradixetrhizoma
<400>29
ccctaggatggtttatatccattg24
<210>30
<211>18
<212>dna
<213>notoginsengradixetrhizoma
<400>30
tcgcgacgacatgcaata18
<210>31
<211>25
<212>dna
<213>notoginsengradixetrhizoma
<400>31
caccaaaattattgatctcttttga25
<210>32
<211>23
<212>dna
<213>notoginsengradixetrhizoma
<400>32
tgatgtccatggtttctcctagt23
- 还没有人留言评论。精彩留言会获得点赞!