一种公猪有效精子数相关的分子遗传标记的制作方法
本公开涉及猪遗传基因技术领域,特别涉及一种公猪有效精子数相关的分子遗传标记。
背景技术:
近年来,随着养殖业的规模化、集约化发展,越来越多的猪场使用鲜精进行人工授精,人工授精已成为一种强有力的猪生产高度集约化工具。种公猪高品质精液不仅可以提供优良的遗传资源,而且直接影响母猪的繁殖力以及人工授精的成功率。因此输精之前对精液进行检测是必不可少的。通过对精液质量评定还能监控公猪健康状况和繁殖潜力,从而可优化个体遗传潜力,发挥最大繁殖能力。
有效精子数是评价精液品质的一项重要指标。在人工授精的过程中,由于精子形态和活力不佳等而导致有效精子数量降低,从而导致怀孕率下降。因此,在人工授精之前,必须对精子的形态以及活力进行分析检测。精子畸形是影响每次采精有效精子数的重要因素,精子畸形率是指畸形精子占总精子的百分率。公猪的畸形精子率一般不能超过18%,否则应弃去。畸形精子指巨型精子、短小精子、断尾、断头、顶体脱落、原生质、头大、双头、双尾、折尾等精子,一般不能直线运动,受精能力较差(猪精液的检测及测定标准,综合养猪)。现今常用吉姆萨染色法测定畸形率,此种方法计数准确但费时费力(质量监督检验检疫总局.gb23238—2009种猪常温精液[s].北京:中国标准出版社,2009)。畸形率较高,经过数月正常天气和良好的饲喂调整,再对种公猪精子进行形态观察,仍有较高的畸形率,则可认为畸形率高是由于种公猪遗传因素造成的,应予以淘汰(成华初,崔霞,吴云华.种公猪精子畸形率增高的原因及处理对策[j].中国畜牧兽医文摘,2017,33(09):70)。除畸形率外,每次采精的精液体积、密度、活力也影响有效精子数。
挖掘和利用新的提高有效精子数的基因对于种公猪的遗传育种有着重大意义。基于覆盖全基因组的高密度snp数据和大群体的性状表型记录,可通过全基因组关联分析技术(gwas)(hirschhorn,j.n.&daly,m.j.genome-wideassociationstudiesforcommondiseasesandcomplextraits.nat.rev.genet.6,95–108(2005))准确定位控制性状的候选基因。尽管该技术仍然存在一些缺陷(de,r.,bush,w.s.&moore,j.h.bioinformaticschallengesingenome-wideassociationstudies(gwas).methodsmol.biol.1168,63–81(2014)),其已被广泛应用于人类复杂疾病候选基因挖掘和畜禽重要经济性状关键基因的定位。经典的gwas一般基于plink(purcell,s.etal.plink:atoolsetforwhole-genomeassociationandpopulation-basedlinkageanalyses.am.j.hum.genet.813,559–575(2007))等软件对所有标记逐个进行单标记回归分析,继而设定一个显著阈值来筛选显著位点。这类方法往往面临计算强度大、过高估计标记效应、显著性阈值设定不合理等问题。为了进一步提高gwas的效率,新方法和软件不断被提出。其中,一步法全基因组关联分析(wssgwas(wang,h.,misztal,i.,aguilar,i.legarra,a.&muir,w.m.genome-wideassociationmappingincludingphenotypesfromrelativeswithoutgenotypes.genetres94,73–83(2012));(wang,h.etal.genome-wideassociationmappingincludingphenotypesfromrelativeswithoutgenotypesinasingle-step(ssgwas)for6-weekbodyweightinbroilerchickens.front.genet.5,1–10(2014)))同时利用系谱、历史个体表型记录和基因型数据进行关联分析,适用于大量个体拥有表型记录而只有少量个体拥有基因型数据的情况,尤其适用于畜禽重要经济性状的全基因组关联分析。基于gblupf90软件(misztal,i.etal.blupf90andrelatedprograms(bgf90).inproc.7thworldcongr.genet.appl.livest.prod.21–22(2002).doi:9782738010520),可轻易实现wssgwas。在不同的研究群体中,前人已经定位到了部分影响公猪精液性状的候选基因或者基因组区域,见参考文献(marques,d.b.d.d.etal.weightedsingle-stepgwasandgenenetworkanalysisrevealnewcandidategenesforsementraitsinpigs.genet.sel.evol.50,1–14(2018));(kaewmala,k.etal.associationstudyandexpressionanalysisofcd9ascandidategeneforboarspermqualityandfertilitytraits.anim.reprod.sci.125,170–179(2011));(kaewmala,k.etal.investigationintoassociationandexpressionofplczandcox-2ascandidategenesforboarspermqualityandfertility.reprod.domest.anim.47,213–223(2012));(gunawan,a.etal.investigationonassociationandexpressionofesr2asacandidategeneforboarspermqualityandfertility.reprod.domest.anim.47,782–790(2012));(diniz,d.b.etal.agenome-wideassociationstudyrevealsanovelcandidategeneforspermmotilityinpigs.anim.reprod.sci.151,201–207(2014)))。
技术实现要素:
针对上述技术问题,本公开提供一种公猪有效精子数相关的分子遗传标记,通过公猪有效精子数与全基因组分子遗传标记的关联分析,成功筛选影响公猪每次采精有效精子数的大效应分子遗传标记,将本公开应用于种公猪精液产量的选育,选留每次采精有效精子数量大的纯合公猪,可有效提高有效精子数量。
通过鉴定了一个影响公猪精子有效精子数的分子标记alga0123896,该标记不同基因型公猪单次采精有效精子数有极显著差异,筛选出影响公猪精子有效精子数的分子遗传标记,alga0123896属于nc_010444基因内含子序列,该位置为一个t>c突变,t>c即t为大频率的等位基因,c为小频率的等位基因,符号>为等位基因频率大小,标记是影响公猪精子有效精子数的等位基因,其中涉及的alga0123896遗传标记即snp号为alga0123896的突变位点,见ncbi中猪基因组数据库(sscrofall.1)。
本公开涉及的snp分子遗传标记(alga0123896),参阅ensembl数据库(http://asia.ensembl.org/sus_scrofa/search/new?db=core),得到登录号为alga0123896的nc_010444基因片段(rs号为rs81324094),alga0123896标记位于猪2号染色体147528447bp位置,该位置为一个t>c突变(突变位点),t>c即t为大频率的等位基因,c为小频率的等位基因,符号>为等位基因频率大小,且该突变属于nc_010444基因内含子序列。该snp分子遗传标记的突变位点的上下游100bp的核苷酸序列如下:
5’-atcaactatgactttcttttcagaaaaaaagcagctgcaattctacaccaggaatgcaggtaatcacacttaacaagtaatatagtgtctgctgcagctar(t/c)taaatctcgtatttttcaacctcacacaatgaatactcctcatcaattaaaagaaaaacaaaaacaactgcagttcccattgcggctcagtggtaacgaa-3’;r为突变位点,上述序列101位核苷酸处的r是t或c时,即r(t/c)时,导致上述序列多态性;当上述核苷酸序列的第101位核苷酸为c时,猪显著提高有效精子数,5’-和-3’分别表示核苷酸序列的5’端和3’端。
上述alga0123896标记基因型cc与tt公猪个体间单次采精有效精子数相差44.53亿,c等位基因显著提高有效精子数,选留每次采精有效精子数含量高的cc基因型纯合猪,通过持续选育,可有效提高有效精子数量,加快优质公猪的选育进度,(由于猪的dna是反向螺旋双链结构,两条链的突变位点核苷酸均为c时为cc基因型纯合猪,其中,每一条链有一个核苷酸序列,c表示一个突变位点为c,cc基因型是双链的突变位点都是c的纯合猪,同理,tt基因型为双链的突变位点都是t的纯合猪;ct基因型为一条链的突变位点是t另一条的突变位点是c的猪)。
筛选一种公猪有效精子数相关的分子遗传标记的方法具体包括以下步骤:
1、分子标记的获得的流程步骤
1.1、采集公猪的耳组织样品或者血样,提取总dna,并对dna进行质量检测。采用ggp50ksnp(geneseek,us)芯片进行基因分型,获得覆盖全基因组的snp标记基因型。
1.2、根据最新版的猪参考基因组(sscrofall.1),采用ncbi基因组比对程序(https://www.ncbi.nlm.nih.gov/)对所有snp标记的物理位置进行更新。基因组位置未知的snp不用于关联分析。
1.3对于所有常染色体上的snp标记,利用plink软件进行质量控制,标准为:个体检出率≥90%;snp检出率≥90%;小等位基因频率≥0.01;哈迪-温伯格平衡p值≥10-6。对于缺失基因型,采用beagle软件(version4.1)进行填充。
2、分子标记的验证的流程步骤
2.1、整理种猪系谱,主要包括公猪个体号、父亲、母亲和初生日期等信息。采用ultimatetmcasa(hamiltonthorneinc.,beverly,ma,usa)系统对新鲜精液进行分析获得精子有效精子数性状表型数据,用于表型-基因型关联分析。
2.2、采用加权的一步法全基因组关联分析法(weightedsinglestepgenome-wideassociationstudy,wssgwas)进行全基因组关联分析。该方法首先基于混合模型方程组来估计个体育种值,继而基于育种值模型与标记效应模型的等价关系将育种值转换为标记效应。本发明采用的全基因组关联分析模型如下:
y=xb+za+wp+age+intv+e
其中,y为有效精子数观测值向量;x,z和w为设计矩阵;b为固定效应向量(总体均值和年-季效应);
其中,a为基于系谱的亲缘关系矩阵;a22为a中有基因型个体对应的分块矩阵;gω=0.9g+0.1a22,
对于上述混合模型,采用ai-reml(averageinformationrestrictedmaximumlikelihood)法估计方差组分,并通过求解混合模型方程组获得育种值。通过迭代的方式获得标记权重,主要步骤如下:
第1步:初始化(t=1),d(t)=i,g(t)=λzd(t)z′,
第2步:通过ssgblup计算个体育种值;
第3步:通过公式
第4步:利用公式
第5步:利用公式
第6步:利用公式g(t+1)=λzd(t+1)z′计算亲缘关系矩阵用于下一轮迭代;
第7步:令t=t+1,并从第2步开始下一轮迭代。
上述步骤迭代三次,最终获得snp标记效应。将第三轮迭代输出的标记效应作为最终的结果。计算过程主要通过在r统计分析平台编程调用blupf90软件来实现,其中airemlf90程序用于方差组分估计,blupf90程序用于计算育种值,postgsf90用于计算标记效应。
3、不同基因型公猪单次采精有效精子数基因筛选
对于所有标记的效应值,取其绝对值画曼哈顿图,展示和筛选大效应的snp标记。并采用方差分析和多重比较(r统计分析平台),分析不同基因型群体公猪单次采精有效精子数差异情况。
本公开的有益效果为:本公开提供了一种公猪有效精子数相关的分子遗传标记,通过检测该分子标记,可以应用于种公猪精液产量的选育,选留每次采精有效精子数含量高的纯合公猪,通过持续选育,可有效提高有效精子数量,加快优质公猪的选育进度。
附图说明
通过对结合附图所示出的实施方式进行详细说明,本公开的上述以及其他特征将更加明显,本公开附图中相同的参考标号表示相同或相似的元素,显而易见地,下面描述中的附图仅仅是本公开的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图,在附图中:
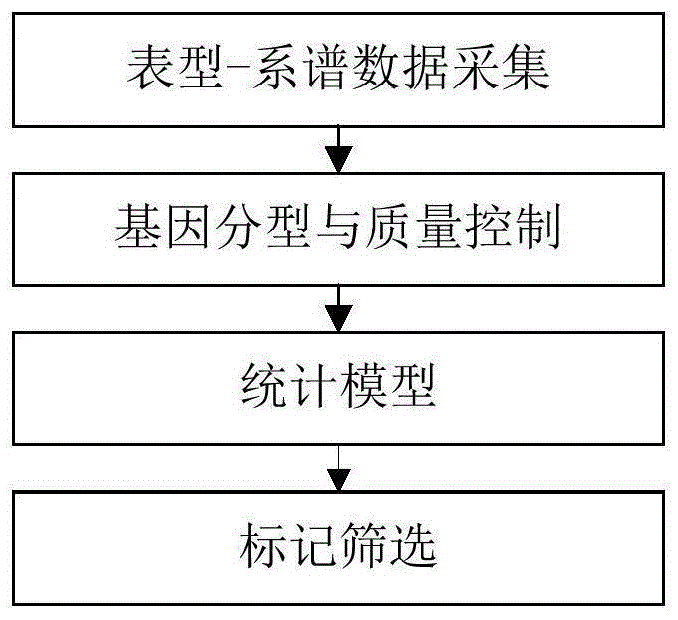
图1所示为本公开的筛选一种公猪有效精子数相关的分子遗传标记的方法工作流程图;
图2所示为alga0123896标记基因组位置及单次采精有效精子数全基因组snp效应分布。
具体实施方式
以下将结合实施例和附图对本公开的构思、具体结构及产生的技术效果进行清楚、完整的描述,以充分地理解本公开的目的、方案和效果。需要说明的是,在不冲突的情况下,本申请中的实施例及实施例中的特征可以相互组合。
如图1所示为根据本公开的筛选一种公猪有效精子数相关的分子遗传标记的方法工作流程图,下面结合图1来阐述根据本公开的筛选一种公猪有效精子数相关的分子遗传标记的方法。
本公开筛选一种公猪有效精子数相关的分子遗传标记的方法,具体包括以下步骤:
(1)表型-系谱数据采集
本专利的基础研究群体为杜洛克公猪,全部来自广西某规模化公猪站。完整系谱中包含12个世代5284头种猪,其中2015-2018年间记录了2693头公猪每次采精的精液体积、精子密度、精子活力、精子畸形率4个性状表型数据。精子畸形率通过ultimatetmcasa(hamiltonthorneinc.,beverly,ma,usa)系统对新鲜精液进行分析获得。总共获得143114条精液性状观测值(平均每头公猪53条数据),用于表型-基因型关联分析。通过上述4个性状计算单次采精有效精子数(亿),单次采精有效精子数的计算公式为:
有效精子数=精液体积×精子密度×精子活力×(1-精子畸形率)。
(2)基因分型与质量控制
采集1733头公猪的耳组织样品或者血样,提取总dna,并采用ggp50ksnp(geneseek,us)芯片进行基因分型,获得覆盖全基因组的50705个snp标记。根据最新版的猪参考基因组(sscrofall.1),采用ncbi基因组比对程序(https://www.ncbi.nlm.nih.gov/)对所有snp标记的物理位置进行更新。基因组位置未知的snp不用于关联分析。对于所有常染色体上的snp标记,利用plink软件进行质量控制,标准为:个体检出率≥90%;snp检出率≥90%;小等位基因频率≥0.01;哈迪-温伯格平衡p值≥10-6。对于缺失基因型,采用beagle软件(version4.1)进行填充。基于以上质量控制标准,剩余1623头公猪和28289个snp标记用于关联分析,其中1231头公猪既有单次采精有效精子数表型数据,也有基因型数据。
(3)统计模型
为了充分利用所有表型数据和基因型数据,本发明专利采用加权的一步法全基因组关联分析法(weightedsinglestepgenome-wideassociationstudy,wssgwas)进行全基因组关联分析。该方法首先基于混合模型方程组来估计个体育种值,继而基于育种值模型与标记效应模型的等价关系将育种值转换为标记效应。本发明采用的全基因组关联分析模型如下:
y=xb+za+wp+age+intv+e,
其中,y为有效精子数观测值向量;x,z和w为设计矩阵;b为固定效应向量(总体均值和年-季效应);
其中,a为基于系谱的亲缘关系矩阵;a22为a中有基因型个体对应的分块矩阵;gω=0.9g+0.1a22,
对应上述混合模型,采用ai-reml(averageinformationrestrictedmaximumlikelihood)法估计方差组分,并通过求解混合模型方程组获得育种值。通过迭代的方式获得标记权重,主要步骤如下:
第1步:初始化(t=1),d(t)=i,g(t)=λzd(t)z′,
第2步:通过ssgblup计算个体育种值;
第3步:通过公式
第4步:利用公式
第5步:利用公式
第6步:利用公式g(t+1)=λzd(t+1)z′计算亲缘关系矩阵用于下一轮迭代;
第7步:令t=t+1,并从第2步开始下一轮迭代。
上述步骤迭代三次,最终获得snp标记效应。将第三轮迭代输出的标记效应作为最终的结果。计算过程主要通过在r统计分析平台编程调用blupf90软件来实现,其中airemlf90程序用于方差组分估计,blupf90程序用于计算育种值,postgsf90用于计算标记效应。
(4)标记筛选
对于所有标记的效应值,取其绝对值画曼哈顿图,展示和筛选大效应的snp标记。并采用方差分析和多重比较(r统计分析平台),分析alga0123896标记不同基因型群体公猪单次采精有效精子数差异情况。
分析不同基因型公猪单次采精有效精子数:
对于所有标记的效应值,取其绝对值画曼哈顿图,展示和筛选大效应的snp标记(如图2所示),图2所示为alga0123896标记基因组位置及单次采精有效精子数全基因组snp效应分布。并采用方差分析和多重比较(r统计分析平台),分析不同基因型群体公猪单次采精有效精子数差异情况(表1)。
一种公猪有效精子数相关的分子遗传标记在影响公猪精子有效精子数辅助选择中的应用:
本公开鉴定了一个影响公猪精子有效精子数的分子标记alga0123896,由表1可知,该标记不同基因型公猪单次采精有效精子数有极显著差异(表1为alga0123896标记不同基因型公猪单次采精有效精子数);
表1alga0123896标记不同基因型公猪单次采精有效精子数
alga0123896标记基因型cc与tt公猪个体间单次采精有效精子数相差44.53亿,c等位基因显著提高有效精子数。
通过检测alga0123896标记基因型辅助种公猪选育,可通过选留cc纯合公猪进入公猪站,提高有效精子数,提高种公猪利用效率。
本公开的snp分子遗传标记(alga0123896标记)位于猪2号染色体147528447bp位置,该位置为一个t>c突变(突变位点),且该突变位于nc_010444基因内含子上。该snp分子遗传标记的突变位点的上下游100bp的核苷酸序列如下:
5’-atcaactatgactttcttttcagaaaaaaagcagctgcaattctacaccaggaatgcaggtaatcacacttaacaagtaatatagtgtctgctgcagctar(t/c)taaatctcgtatttttcaacctcacacaatgaatactcctcatcaattaaaagaaaaacaaaaacaactgcagttcccattgcggctcagtggtaacgaa-3’;r为突变位点,上述序列101位核苷酸处的r是t或c时,即r(t/c)时,导致上述序列多态性;当上述核苷酸序列的第101位核苷酸为c时,猪显著提高有效精子数,5’-和-3’分别表示核苷酸序列的5’端和3’端。
(上述序列在突变点位为c时的序列如序列表seqidno.1所示的核苷酸序列),序列表seqidno.1是本发明筛选得alga0123896遗传标记(即rs号为rs81324094)的突变位点上下游100bp的核苷酸序列。
主要参考文献:
1.质量监督检验检疫总局.gb23238—2009种猪常温精液[s].北京:中国标准出版社,2009;
2.成华初,崔霞,吴云华.种公猪精子畸形率增高的原因及处理对策[j].中国畜牧兽医文摘,2017,33(09):70;
3.hirschhorn,j.n.&daly,m.j.genome-wideassociationstudiesforcommondiseasesandcomplextraits.nat.rev.genet.6,95–108(2005);
4.de,r.,bush,w.s.&moore,j.h.bioinformaticschallengesingenome-wideassociationstudies(gwas).methodsmol.biol.1168,63–81(2014);
5.purcell,s.etal.plink:atoolsetforwhole-genomeassociationandpopulation-basedlinkageanalyses.am.j.hum.genet.813,559–575(2007);
6.wang,h.,misztal,i.,aguilar,i.legarra,a.&muir,w.m.genome-wideassociationmappingincludingphenotypesfromrelativeswithoutgenotypes.genetres94,73–83(2012);
7.wang,h.etal.genome-wideassociationmappingincludingphenotypesfromrelativeswithoutgenotypesinasingle-step(ssgwas)for6-weekbodyweightinbroilerchickens.front.genet.5,1–10(2014);
8.misztal,i.etal.blupf90andrelatedprograms(bgf90).inproc.7thworldcongr.genet.appl.livest.prod.21–22(2002).doi:9782738010520;
9.marques,d.b.d.d.etal.weightedsingle-stepgwasandgenenetworkanalysisrevealnewcandidategenesforsementraitsinpigs.genet.sel.evol.50,1–14(2018);
10.kaewmala,k.etal.associationstudyandexpressionanalysisofcd9ascandidategeneforboarspermqualityandfertilitytraits.anim.reprod.sci.125,170–179(2011);
11.kaewmala,k.etal.investigationintoassociationandexpressionofplczandcox-2ascandidategenesforboarspermqualityandfertility.reprod.domest.anim.47,213–223(2012);
12.gunawan,a.etal.investigationonassociationandexpressionofesr2asacandidategeneforboarspermqualityandfertility.reprod.domest.anim.47,782–790(2012);
13.diniz,d.b.etal.agenome-wideassociationstudyrevealsanovelcandidategeneforspermmotilityinpigs.anim.reprod.sci.151,201–207(2014)。
序列表
<110>佛山科学技术学院
<120>一种公猪有效精子数相关的分子遗传标记
<141>2019-06-05
<160>1
<170>siposequencelisting1.0
<210>1
<211>201
<212>dna
<213>sscrofa10.2
<220>
<221>gene
<222>(1)..(201)
<220>
<221>mutation
<222>(101)..(101)
<400>1
atcaactatgactttcttttcagaaaaaaagcagctgcaattctacaccaggaatgcagg60
taatcacacttaacaagtaatatagtgtctgctgcagctactaaatctcgtatttttcaa120
cctcacacaatgaatactcctcatcaattaaaagaaaaacaaaaacaactgcagttccca180
ttgcggctcagtggtaacgaa201
- 还没有人留言评论。精彩留言会获得点赞!