碱性蛋白酶PA3及其编码基因和应用的制作方法
本发明属于农业生物技术领域,具体涉及碱性蛋白酶pa3及其编码基因和应用。
背景技术:
蛋白酶指通过切割肽键实现蛋白质水解的一类酶,其来源十分广泛,大量分布于动物、植物、微生物中。相比于动植物来源的蛋白酶,微生物源蛋白酶具有培养方便、操作简单和产酶量高等便于工业化的批量生产的特点,因此,微生物源蛋白酶成为了目前的蛋白酶的重要来源。蛋白酶在细胞代谢过程中发挥着至关重要的作用,大量生理活动依靠蛋白酶执行,此外蛋白酶还具有工业应用特性,作为最主要的工业酶制剂之一,被广泛应用于各个领域,市场需求大,经济价值高。
以最适ph为标准,蛋白酶分为酸性蛋白酶、碱性蛋白酶及中性蛋白酶。其中碱性蛋白酶在蛋白酶制剂总销量中占比最大。碱性蛋白酶的最适ph在ph7.5-11.0之间均有分布,且通常在偏碱环境中保持稳定,少数碱性蛋白酶在酸性条件下也有一定的耐受性。碱性蛋白酶的蛋白分子量通常在18-34kda。工业用碱性蛋白酶主要来源于杆菌属,例如枯草芽孢杆菌、解淀粉芽孢杆菌等。目前已发现的碱性蛋白酶基本为丝氨酸蛋白酶,由于活性中心存在一个丝氨酸,当存在苯甲基磺酰氟(pmsf)、二异丙基磷酰氟(dfp)等抑制剂时,其活性被抑制。该类酶通常对位于芳香族氨基酸羧基端的肽键优先作用。常见的碱性蛋白酶有胰蛋白酶、枯草杆菌类蛋白酶等。
直接利用野生菌株发酵生产碱性蛋白酶存在产酶水平低、性质不稳定、分离纯化麻烦等缺点。随着基因工程技术的发展,许多优质的表达系统被建立以实现目标蛋白的高效表达。目前已成功构建蛋白表达系统有大肠杆菌表达系统、酵母表达系统、枯草芽孢杆菌表达系统、昆虫表达系统和哺乳动物表达系统等。其中毕赤酵母表达系统以其表达局限性小、产量高、可控性强、产品投入少、产物易分离及纯化效果好等特点成为备受青睐的表达系统,尤其是对于真菌来源的目的蛋白,其具有完善的翻译后修饰功能。利用毕赤酵母表达真菌来源的碱性蛋白酶有利于其工业化大规模生产,便于其更好地发挥应用价值。利用毕赤酵母表达外源基因时,对基因按照毕赤酵母偏好性进行密码子优化,目前,虽然现有技术根据不同的方法计算了毕赤酵母中密码子偏好性并且提出了偏好密码子和稀有密码子,但是对于部分氨基酸的偏好密码子的结果存在差异,因此在选择时,需结合外源基因的具体情况和宿主细胞类型,进行具体分析。在密码子优化过程中,除将稀有密码子替换成偏好密码子外,还需注意调整基因局部区域大at含量,避免使转录提前终止;并且还需提高翻译终止效率,同时,去除不利于mrna稳定的序列及二级结构,以提高整体的翻译效率,促进表达。因此,利用毕赤酵母表达外源基因时,仅进行偏好密码子替换并不能获得预期结果,需综合考虑基因、宿主细胞等因素。
技术实现要素:
本发明的目的在于提供一种碱性蛋白酶pa3。
本发明的再一目的在于提供上述碱性蛋白酶pa3的编码基因。
本发明的再一目的在于提供含有上述基因的重组表达载体。
本发明的再一目的在于提供含有上述基因重组菌株。
本发明的再一目的在于提供上述碱性蛋白酶pa3的制备方法。
本发明的再一目的在于提供上述碱性蛋白酶pa3的应用。
根据本发明具体实施方式的碱性蛋白酶pa3,其氨基酸序列如seqidno.1所示:
其中,该酶全长387个氨基酸,其中1-15位氨基酸构成信号肽,即“mrlakflallplaag”,16-106位氨基酸构成前导肽,即“apslarreepaplleargaqaipgkfivklregsplaalqqamsllggkadhvfqnvfsgfaasmnpavielmrnhpdveyieqdgkvnin”。
根据本发明具体实施方式的碱性蛋白酶pa3,其成熟蛋白的氨基酸序列如seqidno.2所示:
其中,该酶成熟蛋白全长281个氨基酸,理论分子量为28.2kda,等电点为8.74。
根据本发明具体实施方式的碱性蛋白酶基因pa3,编码上述碱性蛋白酶pa3。
根据本发明具体实施方式的碱性蛋白酶基因pa3,上述碱性蛋白酶除信号肽以外的372个氨基酸对应的核酸序列如seqidno.3所示:
根据本发明具体实施方式的碱性蛋白酶基因pa3,上述碱性蛋白酶包含信号肽在内的387个氨基酸对应的核酸序列如seqidno.4所示:
根据本发明具体实施方式的碱性蛋白酶pa3,其所对应的编码基因未经秘密子优化情况下的核酸序列如seqidno.5所示:
根据本发明具体实施方式的碱性蛋白酶基因的重组表达载体,优选为ppic9r-pa3。将本发明的碱性蛋白酶基因插入到表达载体合适的限制性酶切位点之间,使其核苷酸序列可操作的与表达调控序列相连接。作为本发明的一个最优选的实施方案,优选为将蛋白酶基因插入到质粒ppic9r上的ecori和noti限制性酶切位点之间,使该核苷酸序列位于aoxl启动子的下游并受其调控,得到重组酵母表达质粒ppic9r-pa3。
根据本发明具体实施方式的碱性蛋白酶基因的重组菌株,优选为重组菌株gs115/pa3。其中,优选所述宿主细胞为毕赤酵母(pichiapastoris)细胞,优选将重组酵母表达质粒转化毕赤酵母细胞(pichicpastoris)gs115,得到重组菌株gs115/pa3。
根据本发明具体实施方式的制备碱性蛋白酶pa3的方法,所述方法包括以下步骤:
(1)用包含碱性蛋白酶基因pa3的重组表达载体转化宿主细胞,得到重组菌株;
(2)培养重组菌株,诱导表达碱性蛋白酶pa3;
(3)分离并纯化碱性蛋白酶pa3。
本发明还提供了上述蛋白酶的应用,运用基因工程手段来产业化生产蛋白酶。
本发明的有益效果:
本发明提供了真菌(torrubiellahemipterigena)来源的碱性蛋白酶pa3及其基因,可应用于食品、饲料、制革、洗涤、医药等多个领域。根据本发明的技术方案就可以实现利用基因工程手段生产性质优良适合工业应用的碱性蛋白酶。
附图说明
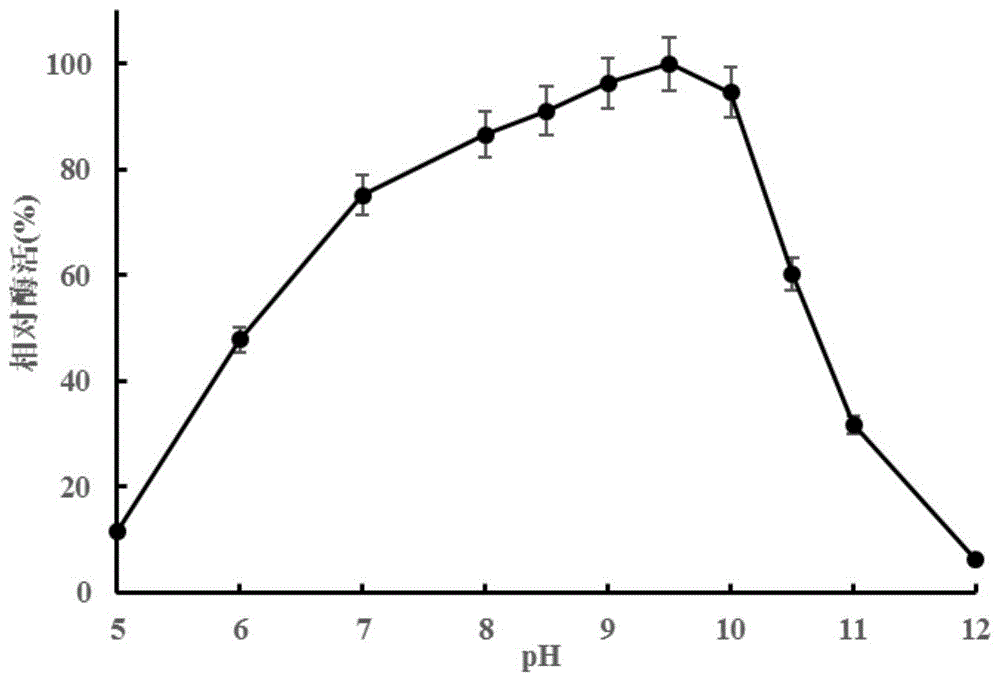
图1显示本发明重组蛋白酶pa3的最适ph值;
图2显示本发明重组蛋白酶pa3的ph稳定性;
图3显示本发明重组蛋白酶pa3最适反应温度;
图4显示本发明重组蛋白酶pa3热稳定性。
具体实施方式
试验材料和试剂
1、菌株及载体:表达宿主pichiapastorisgs115,表达质粒载体ppic9r。
2、生化试剂:限制性内切酶ecori、noti、bglii、fastpfudna聚合酶、assemblykit试剂盒、酪蛋白。
3、培养基:
lb培养基:0.5%酵母提取物,1%蛋白胨,1%nacl,ph7.0
ypd培养基:1%酵母提取物,2%蛋白胨,2%葡萄糖
md固体培养基:2%葡萄糖,1.5%琼脂糖,1.34%ynb,0.00004%biotin
牛奶双层筛选培养基:
上层:1%酵母提取物,2%蛋白胨,1.34%ynb,0.00004%biotin,0.5%甲醇(v/v),1.5%琼脂糖;
下层:4%奶粉,1.5%琼脂糖。
bmgy培养基:1%酵母提取物,2%蛋白胨,1%甘油(v/v),1.34%ynb,0.00004%biotin。
bmmy培养基:1%酵母提取物,2%蛋白胨,1.34%ynb,0.00004%biotin,0.5%甲醇(v/v)。
说明:本实施例中未做详细具体说明的分子生物学实验方法,均参照《分子克隆实验指南》(第三版)j.萨姆布鲁克一书中所列的具体方法进行,或者按照试剂盒和产品说明书进行。
实施例1获取碱性蛋白酶基因pa3及构建表达载体
根据genbank中公布的蛋白序列(序列号为:cej93023.1),对其进行密码子优化,化学合成获得优化后的基因,编号为pa3。
将基因pa3连接在puc57载体上,本发明通过同源重组的方法实现目的基因pa3与表达载体ppic9r的连接,完成表达载体的构建。
首先,通过pcr的方法,借助特异性引物向目的基因中引入同源区段。
表1pa3特异性引物
注:下划线标注为与载体ppic9r同源的重叠序列
以重组质粒puc57-pa3作为模板,通过特异性引物进行pcr扩增反应;同时,利用限制性内切酶ecori、noti对表达载体ppic9进行酶切,随后1%的琼脂糖凝胶电泳并回收pcr产物和酶切产物。回收后的表达载体ppic9r和携带同源区段的目的片段通过assemblykit试剂盒进行连接。
在50℃条件下连接15min,反应结束后置于冰上冷却数秒,后可重组产物直接用于转化或保存于-20℃。
连接产物转化入e.colitrans-t1感受态中,转化菌液涂布在lb平板(含100μg/ml氨苄青霉素)上37℃培养箱过夜培养,挑取克隆子用菌落pcr方法鉴定并测序,测序正确的重组质粒命名为ppic9r-pa3。
实施例2碱性蛋白酶pa3工程菌株的构建
大量提取重组ppic9-pa3质粒,利用限制性内切酶bglii对其进行线性化处理,线性化体系。
37℃酶切2h后,取10μl酶切产物进行琼脂糖凝胶电泳,确认是否酶切完全,后利用omegagelextractionkit试剂盒对酶切产物回收和纯化。将纯化回收的线性化产物电转入毕赤酵母感受态中(电转化仪参数:fungus,pic)。电转后的菌液涂布在md平板上,30℃培养48h。待md平板上长出转化子后,用灭菌后的牙签从md板上挑取36个单克隆于牛奶双层筛选平板上,30℃条件下培养72h。根据转化子周围是否出现水解圈及出圈先后和大小,判断转化子是否表达蛋白酶活性,并衡量其活性高低,从中筛选出高蛋白酶活性的转化子。
实施例3制备重组碱性蛋白酶pa3
(1)摇瓶水平表达重组碱性蛋白酶pa3
将挑选出的酶活较高的转化子接种于30mlypd液体培养基中,于30℃,200rpm摇床中培养48h,获得种子液。随后将种子液按照1%的接种量转接到300mlbmgy培养基中,于30℃,200rpm摇床中培养48h,之后将菌体转移到200mlbmmy培养基中,于30℃,200rpm摇床中培养48h,期间每隔24h,补加0.5%(v/v)甲醇。
(2)重组碱性蛋白酶pa3的纯化
发酵过程结束后,将菌液12000rpm离心10min,去除菌体和其它不溶物,收集上清,既为粗酶液。用5kda大小的膜包(vivascience,hannover,germany)对粗酶液进行浓缩,并通过透析(截留分子量:3kda)对粗酶液进行脱盐处理,将其置换到buffera(ph6.5,10mm磷酸氢二钠-柠檬酸缓冲液)中。脱盐后的酶液通过离子交换柱(阳离子交换柱hitrapsphp5ml)进行纯化,利用bufferb(在buffera基础上添加1mnacl,并调节ph保证其与buffera相同)进行梯度(0-1.0mnacl)洗脱,对于洗脱下来的具有酶活力的组份进行收集,利用sds-page验证其蛋白的纯度及分子量大小,并进行液相色谱-电喷雾离子化-质谱联用(liquidchromatography-electrosprayionization-tandemmassspectrometry,lc-esi-ms)鉴定。
实施例4重组碱性蛋白酶pa3基本酶学性质分析
采用福林酚试剂显色法对本发明的碱性蛋白酶进行活性分析。具体方法如下:500μl适当的稀释酶液和500μl底物(1%w/v酪蛋白),在ph9.5,60℃条件下反应20min,后加入1ml三氯乙酸(0.4mol/l)终止反应。将终止后的反应体系12000rpm离心3min后,取上清进行显色。显色反应体系如下:2.5ml0.4mol/l碳酸钠溶液,500μl上清和500μl福林酚试剂,40℃下反应20分钟,待其冷却至室温后读取其波长680nm的吸光值。所有反应均设有三个平行和空白对照。
蛋白酶的酶活(u)定义为:在给定条件下,每分钟释放1μg酪氨酸所需的酶量为一个酶活单位。
酶学性质研究所用酶液均达到电泳纯。本发明所用缓冲体系如下:乳酸-乳酸钠缓冲体系(50mmol/l,ph3.0-5.0),磷酸盐缓冲体系(200mmol/l,ph5.0-8.0),硼砂-氢氧化钠缓冲体系(100mmol/l,ph8.0-12.0)。
(1)重组碱性蛋白酶pa3的最适ph及ph稳定性
利用ph5.0-12.0的缓冲液将纯化后的酶液稀释适当倍数,在最适温度下,与处于相应ph值的底物反应20min,测定其活性,以确定重组酶的最适ph。
结果如图1所示,纯化后的pa3的最适ph为9.5,ph8.0-10.0均保持80%以上的相对酶活。当反应条件超过ph10.0后,pa3的酶活出现明显下降,在ph11.0条件下,相对酶活约为30%,ph12条件下,相对酶活不足10%。从整体趋势来看,pa3属于碱性蛋白酶,但在偏酸性的条件下,pa3依然保持一定活性,ph6.0时,pa3可以发挥约50%的活性。
将纯化后的酶液在37℃,ph3.0-12.0条件下预处理1h,对处理后的酶液进行适当稀释,在最适温度及ph下测定剩余酶活,以确定重组酶的ph稳定性。
结果如图2所示,pa3在ph稳定性方面也表现较好,在ph4.0-11.0条件下处理1h后,pa3的剩余酶活可以保持90%以上,这表明pa3可保持稳定的ph范围较为宽泛。
(2)重组碱性蛋白酶pa3的最适温度及热稳定性
将纯化后的酶液用最适ph对应的缓冲体系稀释适当倍数,测定在20℃-80℃的不同温度下的酶活,以确定重组酶最适温度。
结果如图3所示,pa3的最适温度为60℃,温度50℃-65℃之间,相对酶活保持在60%以上,50℃时还有55%以上的相对酶活,40℃时相对酶活为约为30%。65℃到70℃区段,酶活下降明显,70℃时,相对酶活约为14%。
将纯化后的酶液用最适ph对应的缓冲体系稀释到50μg/ml,分别在50℃、55℃、60℃下进行热处理,处理时间分别为2min、5min、10min、20min、30min和60min,处理后的样品置于冰上以终止酶的热损失,冷却后稀释适当倍数,在最适反应条件下测定剩余酶活,以确定其热稳定性,以未处理酶液所测的活性为100%。
结果如图4所示,pa3的热稳定性在中低温段表现良好,在50℃条件下处理1h几乎没有酶活损失;但当处理温度达到55℃时,酶活出现下降,处理1h后,损失50%以上活性;60℃时处理20min后几乎检测不到活性。
序列表
<110>中国农业科学院饲料研究所
<120>碱性蛋白酶pa3及其编码基因和应用
<160>5
<170>siposequencelisting1.0
<210>1
<211>387
<212>prt
<213>真菌(torrubiellahemipterigena)
<400>1
metargleualalyspheleualaleuleuproleualaalaglyala
151015
proserleualaargargglugluproalaproleuleuglualaarg
202530
glyalaglnalaileproglylyspheilevallysleuargglugly
354045
serproleualaalaleuglnglnalametserleuleuglyglylys
505560
alaasphisvalpheglnasnvalpheserglyphealaalasermet
65707580
asnproalavalilegluleumetargasnhisproaspvalglutyr
859095
ilegluglnaspglylysvalasnileasnalatyrthrthrglnthr
100105110
glyalaprotrpglyleuglyargileserhisargalalysglyser
115120125
thrsertyrthrtyraspthrseralaglygluglythrcysvaltyr
130135140
valileaspthrglyvalgluaspthrhisprogluphegluglyarg
145150155160
alalysleuilelysthrtyrtyrglyasnargaspglyhisglyhis
165170175
glythrhiscysserglythrileglyserlysthrtyrglyvalala
180185190
lyslysthrlysiletyrglyvallysvalleuaspaspasnglyser
195200205
glythrpheserasnileilealaglyvalaspphevalalaasnasp
210215220
tyrlysthrargglycysprolysglyalavalalasermetserleu
225230235240
glyglyglylysthrglnalavalasnaspalavalalaargleugln
245250255
argalaglyvalphevalalavalalaalaglyasnaspasnthrasp
260265270
alaalaasnthrserproalasergluproservalcysthrvalgly
275280285
alaserasplysaspaspvalargserthrpheserasntyrglyser
290295300
valvalaspilephealaproglythralaileleuserthrtrpile
305310315320
glyglyargthrasnthrileserglythrsermetalathrprohis
325330335
ilealaglyleualaalatyrleumetglylysaspglyalavalala
340345350
alaglyleucysalalysilealaglnthralathrargasnvalleu
355360365
argasnileproalaglythrileasnalaleualapheasnglyasn
370375380
prosergly
385
<210>2
<211>281
<212>prt
<213>真菌(torrubiellahemipterigena)
<400>2
alatyrthrthrglnthrglyalaprotrpglyleuglyargileser
151015
hisargalalysglyserthrsertyrthrtyraspthrseralagly
202530
gluglythrcysvaltyrvalileaspthrglyvalgluaspthrhis
354045
progluphegluglyargalalysleuilelysthrtyrtyrglyasn
505560
argaspglyhisglyhisglythrhiscysserglythrileglyser
65707580
lysthrtyrglyvalalalyslysthrlysiletyrglyvallysval
859095
leuaspaspasnglyserglythrpheserasnileilealaglyval
100105110
aspphevalalaasnasptyrlysthrargglycysprolysglyala
115120125
valalasermetserleuglyglyglylysthrglnalavalasnasp
130135140
alavalalaargleuglnargalaglyvalphevalalavalalaala
145150155160
glyasnaspasnthraspalaalaasnthrserproalaserglupro
165170175
servalcysthrvalglyalaserasplysaspaspvalargserthr
180185190
pheserasntyrglyservalvalaspilephealaproglythrala
195200205
ileleuserthrtrpileglyglyargthrasnthrileserglythr
210215220
sermetalathrprohisilealaglyleualaalatyrleumetgly
225230235240
lysaspglyalavalalaalaglyleucysalalysilealaglnthr
245250255
alathrargasnvalleuargasnileproalaglythrileasnala
260265270
leualapheasnglyasnprosergly
275280
<210>3
<211>1119
<212>dna
<213>真菌(torrubiellahemipterigena)
<400>3
gctccatccttggctagaagagaagaaccagctcctttgttggaagctagaggtgctcaa60
gctatcccaggtaagttcatcgtcaagttgagagagggttctccattggctgcattgcaa120
caagctatgtccttgcttggtggtaaggctgaccacgttttccagaacgttttctctggt180
ttcgccgcctctatgaacccagctgttattgagttgatgagaaaccatccagacgtcgag240
tacattgagcaggacggtaaggttaacatcaacgcctacactactcagactggtgctcca300
tggggtttgggtagaatttctcatagagctaagggttccacctcctacacttacgatact360
tccgctggtgagggtacttgtgtttacgttatcgacactggtgtcgaggacactcaccca420
gaatttgagggtagagccaagctgatcaagacctactacggtaacagagatggtcacggt480
catggtactcactgttccggtactattggttccaagacttacggtgtcgccaaaaagacc540
aaaatctacggtgtcaaggtcctggacgataacggttctggtactttctccaacattatc600
gccggtgttgacttcgttgccaacgactacaagactagaggttgtccaaagggtgctgtt660
gcctctatgtctcttggtggtggaaagactcaagctgttaacgacgctgttgctagattg720
caacgtgccggtgtttttgttgctgttgctgctggtaacgacaacactgatgctgctaat780
acttctccagcttctgagccatccgtctgtactgttggtgcttctgataaggacgacgtc840
agatccaccttctctaactacggttccgttgttgacatcttcgctccaggtactgctatc900
ttgtccacttggattggtggtaggactaacaccatctccggtacttctatggctactcca960
cacattgctggtttggctgcttacctgatgggtaaagatggtgcagttgctgcaggtttg1020
tgtgctaagattgctcagactgccaccagaaacgtcctgagaaatattccagctggtact1080
atcaacgccctggcctttaacggtaatccatctggttaa1119
<210>4
<211>1164
<212>dna
<213>真菌(torrubiellahemipterigena)
<400>4
atgcgtctggctaagttccttgctctactccctcttgccgctggcgctccatccttggct60
agaagagaagaaccagctcctttgttggaagctagaggtgctcaagctatcccaggtaag120
ttcatcgtcaagttgagagagggttctccattggctgcattgcaacaagctatgtccttg180
cttggtggtaaggctgaccacgttttccagaacgttttctctggtttcgccgcctctatg240
aacccagctgttattgagttgatgagaaaccatccagacgtcgagtacattgagcaggac300
ggtaaggttaacatcaacgcctacactactcagactggtgctccatggggtttgggtaga360
atttctcatagagctaagggttccacctcctacacttacgatacttccgctggtgagggt420
acttgtgtttacgttatcgacactggtgtcgaggacactcacccagaatttgagggtaga480
gccaagctgatcaagacctactacggtaacagagatggtcacggtcatggtactcactgt540
tccggtactattggttccaagacttacggtgtcgccaaaaagaccaaaatctacggtgtc600
aaggtcctggacgataacggttctggtactttctccaacattatcgccggtgttgacttc660
gttgccaacgactacaagactagaggttgtccaaagggtgctgttgcctctatgtctctt720
ggtggtggaaagactcaagctgttaacgacgctgttgctagattgcaacgtgccggtgtt780
tttgttgctgttgctgctggtaacgacaacactgatgctgctaatacttctccagcttct840
gagccatccgtctgtactgttggtgcttctgataaggacgacgtcagatccaccttctct900
aactacggttccgttgttgacatcttcgctccaggtactgctatcttgtccacttggatt960
ggtggtaggactaacaccatctccggtacttctatggctactccacacattgctggtttg1020
gctgcttacctgatgggtaaagatggtgcagttgctgcaggtttgtgtgctaagattgct1080
cagactgccaccagaaacgtcctgagaaatattccagctggtactatcaacgccctggcc1140
tttaacggtaatccatctggttaa1164
<210>5
<211>1164
<212>dna
<213>真菌(torrubiellahemipterigena)
<400>5
atgcgtctggctaagttccttgctctactccctcttgccgctggcgctcctagcctcgct60
agacgagaggagcctgctccccttttggaggctcgcggtgctcaggcaattcctggaaaa120
ttcattgtcaagctgcgagaaggcagtccacttgctgctctacagcaggccatgtcgctc180
ttgggcggcaaagcagaccacgtcttccagaacgtcttttctggtttcgctgcatccatg240
aatccagccgtcattgagttgatgcgcaaccatcctgatgtcgagtatatcgaacaggat300
ggcaaggtgaacatcaacgcttacaccacgcagaccggagctccatggggacttggtcga360
atttctcacagggcaaagggtagcacttcctacacctatgacacttcagctggcgaggga420
acctgtgtctatgtcatcgatactggtgtggaggacacccatcctgaatttgaaggccgt480
gccaagctcatcaagacctattacggcaacagggatggccatggccatggcactcactgc540
tctggaaccattggctccaagacctatggcgttgccaaaaagaccaagatttacggtgtc600
aaggtgttggacgataatggatcgggaaccttctccaacatcattgctggcgtggacttt660
gttgccaacgactacaagacccgcggctgccccaagggtgctgtagcatccatgtctctt720
ggtggtggtaagacacaggcagtcaacgatgctgttgcacgtctccagcgagctggtgtt780
tttgttgctgtcgccgctggaaacgacaacaccgatgccgccaacacgtctccagcctcg840
gagccatctgtctgcaccgttggtgctagcgacaaggacgatgtgcgatctaccttttcc900
aactatggatctgttgtagacatcttcgcgccaggcactgctatcctgtctacttggatc960
ggcggccgcactaacaccatctccggtacatccatggccactccccatattgctggtctg1020
gctgcctatctcatgggcaaggacggagctgttgccgctggtttgtgtgccaagattgcg1080
caaactgccactagaaatgttctccgcaacatccccgcaggcaccatcaatgcccttgca1140
ttcaatggtaaccctagcgggtaa1164
- 还没有人留言评论。精彩留言会获得点赞!
- 腈水解酶突变体及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白AsTMK1及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白EtSnRK2.2及其编码基因和应用的制造方法与工艺
- OsSAPK7蛋白及其编码基因在提高水稻白叶枯病抗性中的应用的制造方法与工艺
- 一种抗除草剂抗虫融合基因、编码蛋白及应用的制造方法与工艺
- 毛果杨PtrZFP103基因及其编码蛋白和应用的制造方法与工艺
- 大豆bHLH转录因子基因GmFER及其编码蛋白和应用的制造方法与工艺
- 一种花狭口蛙的基因及其编码的多肽和该多肽的用途的制造方法与工艺
- 一种泽陆蛙基因及其编码的多肽和该多肽的用途的制造方法与工艺
- 一种水稻MIT1基因、其编码蛋白及应用的制造方法与工艺