一种NTRK融合突变检测的探针组、试剂盒及方法与流程
本发明属于基因检测
技术领域:
,特别涉及一种ntrk融合突变检测的探针组、试剂盒及方法。
背景技术:
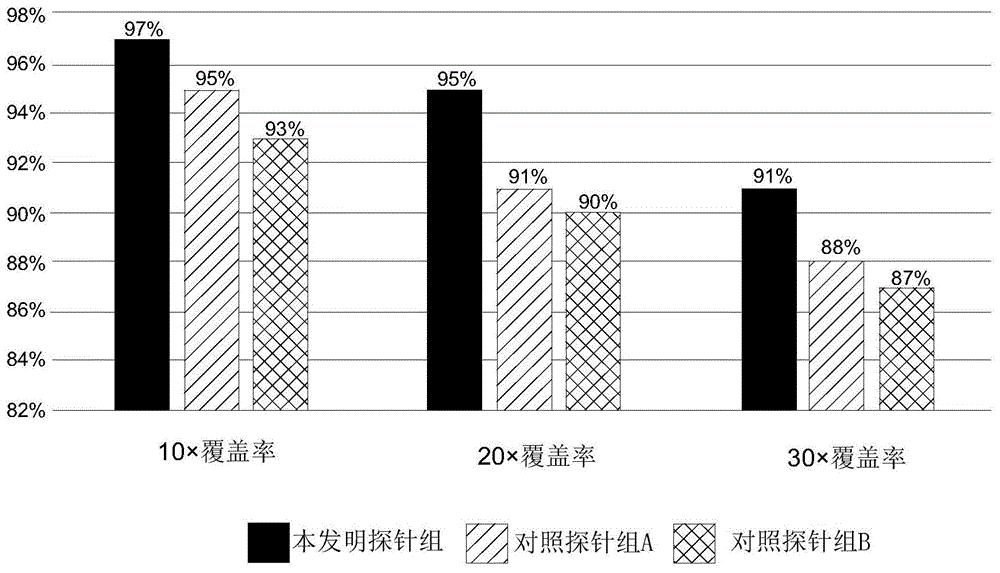
:ntrk(neurotrophinreceptorkinase)是神经营养因子受体络氨酸激酶。原肌球蛋白受体激酶(trk)家族包括trka、trkb和trkc三种蛋白,它们分别由ntrk1、ntrk2和ntrk3基因编码,这些蛋白通常在神经组织中表达。ntrk基因的融合突变促进了肿瘤的形成。trk途径的失调可导致神经发育障碍和各种中枢神经系统(cns)疾病,但当ntrk基因与不相关的基因融合时也会诱导癌症发生。据世界卫生组织估计,有0.2%的nsclc患者存在ntrk融合。trk融合蛋白将处于持续活跃状态,引发永久性的信号级联反应,驱动trk融合肿瘤的扩散和生长。因此,ntrk融合蛋白充当致癌驱动因子,促进癌细胞生长和存活。这一发现导致了ntrk基因融合作为癌症治疗的新靶点而出现。我们在检测ntrk融合的时候,还需要检测其耐药的位点突变。拉罗替尼(larotrectinib)是一款不分年龄和癌种类型针对ntrk融合的广谱trk抑制剂,主要抑制酪氨酸激酶的活性。trk家族蛋白是酪氨酸激酶,如果能抑制激酶活性,就能抑制癌症生长。还有针对其耐药位点的第二代trk靶向药物loxo-195已经出炉,专门来对抗耐药的新突变。目前,有多种方法可用于检测ntrk基因融合突变,包括fish、rt-pcr和ngs检测。其中,rt-pcr法只可检测已知突变,且由于ntrk融合断点具有多变性,极大可能发生漏检。再者,对于fish法来说,一组fish分离探针通常只用于一种基因的融合突变检测,较为单一,缺乏全面性,难以大范围的开展。基于此,有必要提供一种覆盖面广、准确性高、敏感性高且便于开展的ntrk突变检测方案。技术实现要素:为解决上述技术问题,本发明的目的在于提供一种ntrk融合突变检测的探针组、试剂盒及方法;利用该探针组的检测方法覆盖面广,准确性高,敏感性高且便于开展。为实现上述技术目的,达到上述技术效果,本发明通过以下技术方案实现:一种ntrk融合突变检测的探针组,包括核苷酸序列如seqidno.1~146所示的一个或多个探针。进一步的,上述探针组包括核苷酸序列如seqidno.1~146所示的全部探针。进一步的,该探针组中的探针序列的5’端具有标记物。本发明还提供了一种ntrk融合突变检测的试剂盒,该试剂盒包括上述探针组。此外,本发明还提供了一种ntrk融合突变检测的方法,包括以下步骤:(1)提取待检测样本的dna;(2)利用步骤(1)提取的dna构建待检测样本的dna文库;(3)获得检测ntrk融合突变的探针组;(4)使步骤(3)获得的探针组与步骤(2)构建的dna文库进行杂交,以捕获目标区域的dna序列;(5)对步骤(4)获得的杂交产物进行洗脱、分离,对捕获的目标区域的dna序列进行pcr扩增,获得扩增产物,对扩增产物进行纯化,得到测序文库,然后对测序文库进行质检;(6)利用高通量测序方法对步骤(5)获得的测序文库进行检测。进一步的,上述步骤(5)获得的测序文库的dna片段长度为350-450bp。更进一步的,步骤(2)中,首先将提取的dna片段化,然后利用该片段化的dna构建dna文库。上述步骤(5)中,利用探针上的标记物对杂交产物进行分离。进一步的,探针上的标记物为生物素,具体的,步骤(5)是利用链霉亲和素和生物素的亲和作用对杂交产物进行分离。本发明的有益效果是:(1)利用本发明设计的探针组进行ntrk融合突变检测的方法缩短了杂交时间,可以更加快速地检测非小细胞肺癌患者ntrk基因是否发生融合突变,与检测金标准fish法相比,具有检测灵敏度高、覆盖面广、通量高等优点,有利于大规模临床开展。(2)本发明通过ngs技术对ntrk基因融合突变进行全面检测,可以一次性检测患者存在的所有类型的突变,通过对基因进行靶向测序,可以发现何段未知基因序列和其他基因发生融合突变,且可以检测到耐药突变,从而可以在分子水平得到非常确凿的证据,与传统的fish杂交相比,证据更明确与直接,不需要人工的主观经验判断;ntrk基因融合突变是决定患者是否采用拉罗替尼进行治疗的有效指标;而且临床标本检测中是否存在ntrk基因融合突变,可为临床医生选择药物提供参考,做到进一步降低治疗风险,提高患者的生存质量。附图说明图1为本发明实施例的探针组与对比探针组在检测时的覆盖率对比结果。图2为本发明实施例中待测样本1的测序文库的质检结果图。图3为本发明实施例中待测样本2的测序文库的质检结果图。图4为本发明实施例中待测样本3的测序文库的质检结果图。图5为本发明实施例中待测样本4的测序文库的质检结果图。图6为本发明实施例中待测样本5的测序文库的质检结果图。图7为本发明实施例中待测样本6的测序文库的质检结果图。具体实施方式下面结合附图对本发明的较佳实施例进行详细阐述,以使本发明的优点和特征能更易于被本领域技术人员理解,从而对本发明的保护范围做出更为清楚明确的界定。实施例该实施例的样本来源为6例主诉为非小细胞肺癌患者的血液样品(患者均已签署知情同意书),以下为6名患者的基因检测过程。一、dna的提取全血样品使用qiaamptmdnaminikit(qiagen.cat.no.51304)试剂盒提取患者的核酸样品。具体的提取过程为:1.吸取20μlqiagenprotease(蛋白酶k100mg/ml)加入至无菌2ml离心管底部;2.将200μl全血样品加入到上述离心管内,然后加入4μlrnasea(核糖核酸酶a100mg/ml),吹打混匀,室温孵育5分钟,再加入200μl的bufferal(裂解缓冲液),涡旋混匀15秒;3.将离心管置于56℃水浴锅中孵育10分钟;4.瞬时离心,加入200μl乙醇(96-100%),涡旋混匀15秒,得到样品溶液;5.转移样品溶液到qiaampminispincolumn(分离柱)中,室温放置1分钟,8000rpm离心1分钟;6.将qiaampminispincolumn放在一个干净的2ml收集管中,丢弃装有滤液的管子;7.加入500μlbufferaw1(洗涤缓冲液1),8000rpm离心1分钟;8.弃滤液,向qiaampminispincolumn中加入500μlbufferaw2(洗涤缓冲液2),20000rpm离心3分钟;9.弃滤液,向qiaampminispincolumn的滤膜中心加入60μl56℃水浴预热的bufferae(溶解缓冲液),室温孵育5min,8000rpm离心1分钟;10.收集洗脱液,在装有洗脱液的1.5ml离心管上做好标记。11.使用thermofisher公司的nanodropone(超微量分光光度计)检测提取的dna:od260/od280在1.8-2.0范围内,浓度不低于5ng/μl;二、dna文库的构建1.使用片段化的酶或者机械打断的方法,将提取的dna片段化成200bp左右。2.使用kapahyperprepkit文库构建试剂盒进行文库构建:2-1.dna末端修补和末端加“a”2-1-1.取出试剂盒中的endrepair&a-tailingbuffer和endrepair&a-tailingenzyme置于冰上自然融解,混合均匀,瞬时离心备用;2-1-2.按照下表,在置于冰上的0.2mlpcr管中进行反应体系配制:已片段化的dna50μlendrepair&a-tailingbuffer7μlendrepair&a-tailingenzyme3μl总体积60μl将上述反应体系混合均匀,瞬时离心使全部反应液置于pcr管底部;在pcr仪上启动如下反应程序,热盖85℃,等温度稳定至20℃时将反应管放进pcr仪:20℃30min65℃30min4℃hold该过程利用endrepairenzyme对dna的末端补平,将末端修复,通过kapaa-tailingenzyme在经过末端修复的dna片段的3’末端添加碱基a。2-2.dna两端加接头2-2-1取出试剂盒中的ligationbuffer和dnaligase置于冰上自然融解,混合均匀,瞬时离心备用。2-2-2.从pcr仪上取出pcr反应管,置于冰上,按照下表进行接头连接反应体系配制:上一步反应产物60μladapterstock(15μm)5μlpcr-gradewater5μlligationbuffer30μldnaligase10μl总体积110μl将上述反应体系混合均匀,瞬时离心使全部反应液置于pcr管底部,即为连接体系pcr管;在pcr仪上启动如下反应程序,热盖85℃,等温度稳定至20℃时将反应管放进pcr仪:20℃15min4℃hold该反应过程实现在dna的双末端连接上接头。2-3.磁珠纯化2-3-1.提前将beads(磁珠)取出涡旋混匀,室温平衡30min后使用;2-3-2.按照要求向上述连接体系pcr管中加入beads,进行片段筛选,经纯化过程,具体为:离心后,将pcr管置于磁力架上,弃上清,然后,使用80%的乙醇清洗两遍,然后加入22μl去离子水,孵育5min,把该pcr管置于磁力架上,吸取上清,即为纯化后的产物,并将该纯化后的产物保存在新的pcr管中。2-4.纯化后的产物扩增2-4-1.取出试剂盒中的2*hifipcrmastermix和amplificationprimermix置于冰上自然融解,混合均匀,瞬时离心备用;2-4-2.按照下表在置于冰上的pcr管中进行反应体系配制:2*hifipcrmastermix25μlamplificationprimermix5μl纯化后产物20μl总体积50μl2-4-2.将上述pcr管放入pcr仪中启动以下程序:2-4-3.向pcr管加入beads,对扩增后产物进行纯化,纯化过程具体为:离心后,将pcr管置于磁力架上,弃上清,然后使用80%的乙醇清洗两遍,再加入22μl去离子水,孵育5min,将pcr管置于磁力架上,吸取上清到新的pcr管中,即为所得的dna文库。三、获得检测ntrk融合突变的探针组为了实现更高的覆盖率,通过对探针的类型、长度、序列、杂交条件等进行大量实验摸索,并针对ntrk融合基因自身的特征,创造性的设计了核苷酸序列如seqidno.1~146所示的探针组。在该实施例中,该探针组包含了核苷酸序列如seqidno.1~146所示的全部探针。其中,探针组中的探针序列的5’端具有标记物,在该实施例中,该标记物为生物素。本发明的探针组中的探针是基于杂交捕获的富集方法进行设计,为序列特异性的捕获探针,其与基因组上特定目标区域互补。探针是溶液形式的、经生物素标记的寡核苷酸序列,能够杂交并捕获预设的区域。本发明的探针组中的探针能够容忍结合位置存在多个错配,不会干扰与目标区域的杂交。可以克服常见的等位基因丢失问题。本发明的探针通常只结合目标区域,因此其测序范围也较为集中,测序深度更深,可有效避免探针分离到非目标的邻近区域,做到靶点区域与非靶点区域的测序数据的有效平衡,这也就提升了目标区域的总体覆盖度。另外,在ntrk发生融合区域中被证明难以富集的区域,通过适当增加相应探针的密度的方法,可以将捕获的特异性提高。本发明实施例的探针组包含了核苷酸序列如seqidno.1~146所示的全部探针,适当增大了探针密度,提高了捕获的特异性。四、杂交捕获1.在离心管中加入5μlhumancotdna,2μlblocking(试剂购自idt公司),再向离心管中加入500ng总量的dna文库;2.使用eppendorf公司的concentratorplus(真空浓缩机)真空离心抽干离心管中液体;3.将清洗与杂交试剂盒(试剂购自idt公司)放于室温融解,并平衡至室温。4.按下表,向浓缩干燥的离心管中加入相应试剂,得到杂交反应液:2*hybridizationbuffer8.5μlhybridizationbufferenhancer2.7μlprobemix4μlnuclease-freewater1.8μl注:本发明的探针组,即上述加入的probemix,是按照等量均匀混合而成的,使用前需涡旋混合均匀,瞬时离心后使用移液枪把probemix缓慢加入到杂交混合液(包括杂交缓冲液、杂交缓冲增强剂和无核酸酶水)中。5.将离心管中的反应液吹打混匀,室温孵育10min;然后涡旋混匀,瞬时离心。6.将离心管中全部的17μl杂交反应液转移到0.2ml低吸附pcr管中,将pcr管放入pcr仪中启动以下程序:95℃30sec65℃4h65℃hold利用上述程序杂交反应4小时,得到杂交产物。五、杂交产物洗脱、分离首先,取50μl链霉亲和素标记磁珠于室温下静置30min;按下表,在低吸附1.5ml离心管中配置链霉亲和素标记磁珠重悬液2*hybridizationbuffer8.5μlhybridizationbufferenhancer2.7μlnuclease-freewater5.8μl链霉亲和素标记磁珠50μl杂交结束后,向含有杂交文库的pcr管中加入链霉亲和素标记磁珠重悬液,进行洗脱分离,链霉亲和素标记磁珠通过探针吸附目标片段,pcr管涡旋后,放入磁力架,待液体澄清,弃上清,然后使用80%的乙醇清洗两遍,再加入22μl去离子水,孵育5min,将pcr管置于磁力架上,吸取上清到新的pcr管中,得到经洗脱分离后的目标片段。六、目标片段扩增1.按照下表向含有20μl的经洗脱分离后的目标片段的pcr管中加入下列试剂,配置扩增体系:2*kapahifihotstartreadymix25μllibraryamplificationprimer1.25μlnuclease-freewater3.75μl将pcr管置于pcr仪中,按下表程序进行扩增:2.对扩增后的产物进行纯化,得到测序文库。七、文库质检和ngs测序采用labchipgxtouch24nucleicacidanalyzer对纯化后的扩增产物(测序文库)进行质检,如图2-7显示的是6个待测样本的测序文库的质检结果,片段大小都在350-450bp之间,均满足要求;最后通过高通量测序方法,并使用2000/2500,systems等二代测序平台对6个测序文库进行检测,使用相对应的软件对检测结果进行全面解读,测序结果如下表所示:患者突变类型1etv6-ntrk32tpm3-ntrk13lmna-ntrk14etv6-ntrk35etv6-ntrk36未见突变结果表明,本发明检测了6例样品,其中5例样品中检测到了非小细胞肺癌的ntrk融合突变,1例样品未得到阳性结果。八、不同方法的结果比对采用fish杂交方法对5例阳性样品和1例阴性样品进行试验。fish杂交结果与上面测序的结果一致。九、不同探针组覆盖率的对比使用相同的待测样品,分别使用本发明探针组、对比探针组a(anti-pantrk探针)、对比探针组b(anbipin的ntrk基因断裂探针),进行杂交捕获。使用2000/2500,systems等二代测序平台对3个文库进行检测,使用相对应的软件对检测结果进行全面解读。从图1中可以看出,在10×/20×/30×覆盖率上,本发明都优于其他探针组,所以本发明的探针组覆盖均一度更高。以上所述仅为本发明的实施例,并非因此限制本发明的专利范围,凡是利用本发明说明书及附图内容所作的等效结构或等效流程变换,或直接或间接运用在其他相关的
技术领域:
,均同理包括在本发明的专利保护范围内。序列表<110>苏州璞瑞卓越生物科技有限公司<120>一种ntrk融合突变检测的探针组、试剂盒及方法<130>2019<160>146<170>siposequencelisting1.0<210>1<211>24<212>dna<213>人工序列(artificialsequence)<400>1gctcaggtcaaccggcaggaaacg24<210>2<211>25<212>dna<213>人工序列(artificialsequence)<400>2gggctgggaagtaagcgagtggagg25<210>3<211>27<212>dna<213>人工序列(artificialsequence)<400>3gatgttggaacttcttggctgggctga27<210>4<211>27<212>dna<213>人工序列(artificialsequence)<400>4tggatgttggaacttcttggctgggct27<210>5<211>27<212>dna<213>人工序列(artificialsequence)<400>5tgttgggaaagagcggcaatggtagtg27<210>6<211>27<212>dna<213>人工序列(artificialsequence)<400>6cttgttgggaaagagcggcaatggtag27<210>7<211>26<212>dna<213>人工序列(artificialsequence)<400>7acttgttgggaaagagcggcaatggt26<210>8<211>27<212>dna<213>人工序列(artificialsequence)<400>8cgacacggtgagtagaacgatttgggc27<210>9<211>26<212>dna<213>人工序列(artificialsequence)<400>9cggtgggaaagaacgcaaggaggaag26<210>10<211>24<212>dna<213>人工序列(artificialsequence)<400>10cggtgggaaagaacgcaaggagga24<210>11<211>25<212>dna<213>人工序列(artificialsequence)<400>11ggacggtgggaaagaacgcaaggag25<210>12<211>24<212>dna<213>人工序列(artificialsequence)<400>12gggacggtgggaaagaacgcaagg24<210>13<211>23<212>dna<213>人工序列(artificialsequence)<400>13gccagcgacgagcccgtagacac23<210>14<211>23<212>dna<213>人工序列(artificialsequence)<400>14ggtgggagcagccagcgacgagc23<210>15<211>25<212>dna<213>人工序列(artificialsequence)<400>15gggtagtgagggactggacgagggg25<210>16<211>27<212>dna<213>人工序列(artificialsequence)<400>16cgtcaagggcaaacaaacatcatcgca27<210>17<211>26<212>dna<213>人工序列(artificialsequence)<400>17gcgtcaagggcaaacaaacatcatcg26<210>18<211>25<212>dna<213>人工序列(artificialsequence)<400>18gctgggaagtaagcgagtggaggcg25<210>19<211>26<212>dna<213>人工序列(artificialsequence)<400>19aggagcccagggaggtaggttgtcgt26<210>20<211>26<212>dna<213>人工序列(artificialsequence)<400>20cgaggagcccagggaggtaggttgtc26<210>21<211>25<212>dna<213>人工序列(artificialsequence)<400>21gcgaggagcccagggaggtaggttg25<210>22<211>24<212>dna<213>人工序列(artificialsequence)<400>22ggcgaggagcccagggaggtaggt24<210>23<211>23<212>dna<213>人工序列(artificialsequence)<400>23cggcgaggagcccagggaggtag23<210>24<211>23<212>dna<213>人工序列(artificialsequence)<400>24cgtggacccagggaagcggagga23<210>25<211>27<212>dna<213>人工序列(artificialsequence)<400>25cgtagctgtccttgccgagtctgacct27<210>26<211>23<212>dna<213>人工序列(artificialsequence)<400>26cgagtggaggcggagtcggctgt23<210>27<211>25<212>dna<213>人工序列(artificialsequence)<400>27gggaagtaagcgagtggaggcggag25<210>28<211>23<212>dna<213>人工序列(artificialsequence)<400>28tggacccagggaagcggaggagc23<210>29<211>26<212>dna<213>人工序列(artificialsequence)<400>29ggaggaaggagtggaaggttgagggg26<210>30<211>27<212>dna<213>人工序列(artificialsequence)<400>30gaacgcaaggaggaaggagtggaaggt27<210>31<211>27<212>dna<213>人工序列(artificialsequence)<400>31ggaaagaacgcaaggaggaaggagtgg27<210>32<211>27<212>dna<213>人工序列(artificialsequence)<400>32tgggaaagaacgcaaggaggaaggagt27<210>33<211>23<212>dna<213>人工序列(artificialsequence)<400>33gcggacggagtcgggcggagcgg23<210>34<211>23<212>dna<213>人工序列(artificialsequence)<400>34cgcctgcgaccagctccacagca23<210>35<211>26<212>dna<213>人工序列(artificialsequence)<400>35caatacccgtgtcggtccattccctc26<210>36<211>23<212>dna<213>人工序列(artificialsequence)<400>36tgcggcgaggagcccagggaggt23<210>37<211>25<212>dna<213>人工序列(artificialsequence)<400>37gcatttccgccagtcagcttacccg25<210>38<211>24<212>dna<213>人工序列(artificialsequence)<400>38agacgatcccagtctcggttgcgg24<210>39<211>25<212>dna<213>人工序列(artificialsequence)<400>39ggtcaaccggcaggaaacgactcaa25<210>40<211>25<212>dna<213>人工序列(artificialsequence)<400>40gccagcgacgagcccgtagacactc25<210>41<211>23<212>dna<213>人工序列(artificialsequence)<400>41tgggcttgcgccgttgaaggtac23<210>42<211>23<212>dna<213>人工序列(artificialsequence)<400>42cttgggcttgcgccgttgaaggt23<210>43<211>23<212>dna<213>人工序列(artificialsequence)<400>43gaggcttgggcttgcgccgttga23<210>44<211>25<212>dna<213>人工序列(artificialsequence)<400>44ctcaggtcaaccggcaggaaacgac25<210>45<211>24<212>dna<213>人工序列(artificialsequence)<400>45cgccggtcctactggcctcactcc24<210>46<211>27<212>dna<213>人工序列(artificialsequence)<400>46catccatgagtaagtgcgtcaagggca27<210>47<211>25<212>dna<213>人工序列(artificialsequence)<400>47ggtccatgccttggcgtgaggataa25<210>48<211>26<212>dna<213>人工序列(artificialsequence)<400>48cagcgattgaatcctgggctttctgg26<210>49<211>27<212>dna<213>人工序列(artificialsequence)<400>49gccggtcctactggcctcactccctac27<210>50<211>27<212>dna<213>人工序列(artificialsequence)<400>50tgggaaagagcggcaatggtagtgttc27<210>51<211>24<212>dna<213>人工序列(artificialsequence)<400>51ccggcattcgggcctaagcactct24<210>52<211>27<212>dna<213>人工序列(artificialsequence)<400>52cggtcctactggcctcactccctacct27<210>53<211>25<212>dna<213>人工序列(artificialsequence)<400>53gccggtcctactggcctcactccct25<210>54<211>23<212>dna<213>人工序列(artificialsequence)<400>54gcgccggtcctactggcctcact23<210>55<211>24<212>dna<213>人工序列(artificialsequence)<400>55tggtccatgccttggcgtgaggat24<210>56<211>27<212>dna<213>人工序列(artificialsequence)<400>56cgccattctggcaattccagtcaaacg27<210>57<211>24<212>dna<213>人工序列(artificialsequence)<400>57tctcctgactaggcgaccggcaca24<210>58<211>23<212>dna<213>人工序列(artificialsequence)<400>58atccgccgtggacccagggaagc23<210>59<211>27<212>dna<213>人工序列(artificialsequence)<400>59cggcggaggaggaggagcagagggagg27<210>60<211>25<212>dna<213>人工序列(artificialsequence)<400>60cggcggaggaggaggagcagaggga25<210>61<211>23<212>dna<213>人工序列(artificialsequence)<400>61cggcggaggaggaggagcagagg23<210>62<211>27<212>dna<213>人工序列(artificialsequence)<400>62tccatgagtaagtgcgtcaagggcaaa27<210>63<211>27<212>dna<213>人工序列(artificialsequence)<400>63gtggccttatttgttgtcgcgggttta27<210>64<211>26<212>dna<213>人工序列(artificialsequence)<400>64ggtggccttatttgttgtcgcgggtt26<210>65<211>24<212>dna<213>人工序列(artificialsequence)<400>65tttccgccagtcagcttacccgct24<210>66<211>27<212>dna<213>人工序列(artificialsequence)<400>66cggtacaatacccgtgtcggtccattc27<210>67<211>26<212>dna<213>人工序列(artificialsequence)<400>67gcggtacaatacccgtgtcggtccat26<210>68<211>23<212>dna<213>人工序列(artificialsequence)<400>68cgggtcccaggttggcaactcaa23<210>69<211>23<212>dna<213>人工序列(artificialsequence)<400>69atggtccatgccttggcgtgagg23<210>70<211>23<212>dna<213>人工序列(artificialsequence)<400>70cccgctcggcccgtcccttcctc23<210>71<211>23<212>dna<213>人工序列(artificialsequence)<400>71gacccgctcggcccgtcccttcc23<210>72<211>23<212>dna<213>人工序列(artificialsequence)<400>72ccgacccgctcggcccgtccctt23<210>73<211>25<212>dna<213>人工序列(artificialsequence)<400>73tcaaccggcaggaaacgactcaacg25<210>74<211>25<212>dna<213>人工序列(artificialsequence)<400>74cgatcttcctcgggtcccaggttgg25<210>75<211>23<212>dna<213>人工序列(artificialsequence)<400>75cgccgccgctcccgccgccattt23<210>76<211>25<212>dna<213>人工序列(artificialsequence)<400>76gcaattccagtcaaacggcaagccc25<210>77<211>23<212>dna<213>人工序列(artificialsequence)<400>77acaccgcctgcgaccagctccac23<210>78<211>23<212>dna<213>人工序列(artificialsequence)<400>78cgccgccaccgccgccactctgc23<210>79<211>23<212>dna<213>人工序列(artificialsequence)<400>79aggcagtcctcgatccgctcgcc23<210>80<211>23<212>dna<213>人工序列(artificialsequence)<400>80tcccaggttggcaactcaacgcc23<210>81<211>23<212>dna<213>人工序列(artificialsequence)<400>81gccgtggacccagggaagcggag23<210>82<211>23<212>dna<213>人工序列(artificialsequence)<400>82gccgccgccaccgccgccactct23<210>83<211>23<212>dna<213>人工序列(artificialsequence)<400>83gctgcggcgaggagcccagggag23<210>84<211>27<212>dna<213>人工序列(artificialsequence)<400>84ggccgaaggtggtggttggtgggagca27<210>85<211>25<212>dna<213>人工序列(artificialsequence)<400>85ggccgaaggtggtggttggtgggag25<210>86<211>23<212>dna<213>人工序列(artificialsequence)<400>86ggccgaaggtggtggttggtggg23<210>87<211>23<212>dna<213>人工序列(artificialsequence)<400>87ccggcttgacagggcctgagggg23<210>88<211>24<212>dna<213>人工序列(artificialsequence)<400>88aggctcaggtcaaccggcaggaaa24<210>89<211>25<212>dna<213>人工序列(artificialsequence)<400>89gccgcgttgaacggtatgtgggaac25<210>90<211>23<212>dna<213>人工序列(artificialsequence)<400>90gccgcgttgaacggtatgtggga23<210>91<211>23<212>dna<213>人工序列(artificialsequence)<400>91ccctgccgcgttgaacggtatgt23<210>92<211>27<212>dna<213>人工序列(artificialsequence)<400>92ctgggctttctgggagccactgtcatc27<210>93<211>23<212>dna<213>人工序列(artificialsequence)<400>93ctgccgccgccaccgccgccact23<210>94<211>28<212>dna<213>人工序列(artificialsequence)<400>94gagtaaaccagtgtcctgtgccgtgaaa28<210>95<211>27<212>dna<213>人工序列(artificialsequence)<400>95ggtcagaggacagaaaggtcagcgagg27<210>96<211>28<212>dna<213>人工序列(artificialsequence)<400>96tgaggtcagaggacagaaaggtcagcga28<210>97<211>25<212>dna<213>人工序列(artificialsequence)<400>97ccagaaccgctccttcctcatccca25<210>98<211>25<212>dna<213>人工序列(artificialsequence)<400>98tcccagaaccgctccttcctcatcc25<210>99<211>27<212>dna<213>人工序列(artificialsequence)<400>99tgtatcccagaaccgctccttcctcat27<210>100<211>27<212>dna<213>人工序列(artificialsequence)<400>100tcagaggacagaaaggtcagcgagggt27<210>101<211>25<212>dna<213>人工序列(artificialsequence)<400>101ggtcggtcttcactcagcccatcgt25<210>102<211>29<212>dna<213>人工序列(artificialsequence)<400>102gctcagccctactccaacaaactgtcaaa29<210>103<211>25<212>dna<213>人工序列(artificialsequence)<400>103tgacaacctggtgagggcagaaagc25<210>104<211>25<212>dna<213>人工序列(artificialsequence)<400>104gggattgtggagccgaatgcctttt25<210>105<211>26<212>dna<213>人工序列(artificialsequence)<400>105gctttcccgagtatgctgctgacctg26<210>106<211>25<212>dna<213>人工序列(artificialsequence)<400>106gagggacaagggcaagggttgaaaa25<210>107<211>28<212>dna<213>人工序列(artificialsequence)<400>107atgggcttggagggagatgatggacact28<210>108<211>27<212>dna<213>人工序列(artificialsequence)<400>108atgggcttggagggagatgatggacac27<210>109<211>25<212>dna<213>人工序列(artificialsequence)<400>109cctctgcctggaccctttgggaatg25<210>110<211>28<212>dna<213>人工序列(artificialsequence)<400>110gaaggcagaagttgggttacaaggcaga28<210>111<211>27<212>dna<213>人工序列(artificialsequence)<400>111aagggctatcagttggcagaggcaggg27<210>112<211>26<212>dna<213>人工序列(artificialsequence)<400>112caagggctatcagttggcagaggcag26<210>113<211>25<212>dna<213>人工序列(artificialsequence)<400>113aggctactcctctggttggttgcgg25<210>114<211>25<212>dna<213>人工序列(artificialsequence)<400>114ctgacctcaggttatccacccgcct25<210>115<211>27<212>dna<213>人工序列(artificialsequence)<400>115aaagccgtatcactggcataaggaaag27<210>116<211>25<212>dna<213>人工序列(artificialsequence)<400>116tgcggcactctgggaaggaactctg25<210>117<211>25<212>dna<213>人工序列(artificialsequence)<400>117gttatccacccgcctctgcctccca25<210>118<211>25<212>dna<213>人工序列(artificialsequence)<400>118cccggctggttcctgctaggactac25<210>119<211>25<212>dna<213>人工序列(artificialsequence)<400>119gggtgagtgcccaaagtgctgaaaa25<210>120<211>25<212>dna<213>人工序列(artificialsequence)<400>120attctgcggcactctgggaaggaac25<210>121<211>25<212>dna<213>人工序列(artificialsequence)<400>121tatccacccgcctctgcctcccaaa25<210>122<211>25<212>dna<213>人工序列(artificialsequence)<400>122cctcaggttatccacccgcctctgc25<210>123<211>25<212>dna<213>人工序列(artificialsequence)<400>123tcactctgttgcccaggctaaatgc25<210>124<211>25<212>dna<213>人工序列(artificialsequence)<400>124aaacattctgcggcactctgggaag25<210>125<211>27<212>dna<213>人工序列(artificialsequence)<400>125gctggcacatgactccctcgtgtatgg27<210>126<211>25<212>dna<213>人工序列(artificialsequence)<400>126gctggcacatgactccctcgtgtat25<210>127<211>29<212>dna<213>人工序列(artificialsequence)<400>127aactgacatattgttgtgacattcagggg29<210>128<211>25<212>dna<213>人工序列(artificialsequence)<400>128aggttatccacccgcctctgcctcc25<210>129<211>25<212>dna<213>人工序列(artificialsequence)<400>129gacctcaggttatccacccgcctct25<210>130<211>25<212>dna<213>人工序列(artificialsequence)<400>130ccaaacattctgcggcactctggga25<210>131<211>25<212>dna<213>人工序列(artificialsequence)<400>131ggacctcctcccttctttcttcccc25<210>132<211>25<212>dna<213>人工序列(artificialsequence)<400>132gagatgaggatcttcgctcttgccc25<210>133<211>28<212>dna<213>人工序列(artificialsequence)<400>133gaatttcaacactttgtatggtctcccg28<210>134<211>28<212>dna<213>人工序列(artificialsequence)<400>134gagtctcactctgttgcccaggctaaat28<210>135<211>28<212>dna<213>人工序列(artificialsequence)<400>135cttgctggatagagttacctgtagccca28<210>136<211>29<212>dna<213>人工序列(artificialsequence)<400>136cccggctggttcctgctaggactacaatt29<210>137<211>29<212>dna<213>人工序列(artificialsequence)<400>137ttgctggatagagttacctgtagcccact29<210>138<211>25<212>dna<213>人工序列(artificialsequence)<400>138tatggctggcacatgactccctcgt25<210>139<211>25<212>dna<213>人工序列(artificialsequence)<400>139tctatggctggcacatgactccctc25<210>140<211>25<212>dna<213>人工序列(artificialsequence)<400>140catctatggctggcacatgactccc25<210>141<211>29<212>dna<213>人工序列(artificialsequence)<400>141cccaggcaaccactaatctactttctgtc29<210>142<211>27<212>dna<213>人工序列(artificialsequence)<400>142gcctggcctcattctggtctgtagttg27<210>143<211>26<212>dna<213>人工序列(artificialsequence)<400>143ggcctggcctcattctggtctgtagt26<210>144<211>25<212>dna<213>人工序列(artificialsequence)<400>144tggcctggcctcattctggtctgta25<210>145<211>29<212>dna<213>人工序列(artificialsequence)<400>145agaaggaaatggtctgtttaatacgccac29<210>146<211>25<212>dna<213>人工序列(artificialsequence)<400>146ggctcactgcaacctccatctcccg25当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1