一种一日龄鉴别鸡受精蛋性别的方法与流程
本发明涉及生物技术领域,特别涉及一种一日龄鉴别鸡受精蛋性别的方法。
背景技术:
家鸡受精蛋早期胚胎性别鉴定一直是行业难点。主要原因在于禽类胚胎发育有其区别于哺乳动物的特点:禽类胚胎发育分为体内和体外两个阶段,精卵结合后且仅在体内停留20~26小时左右即以蛋的形式娩出,并暂停发育,该阶段胚胎处于囊胚阶段,尚未有技术能够检测母鸡体内胚胎性别;受精蛋娩出后,适宜的温度下开始继续发育,此时受精卵外部有厚厚的蛋白,蛋白外部为结实的蛋壳,传统孵化技术通过“照蛋”只能分辨出是否为受精蛋,胚胎是否死亡,无法分辨性别。
目前常用的雌雄鉴别技术主要分为两类:一类是出雏后,通过一些伴性性状如“金银羽”、“快慢羽”和“横斑”等性状可在雏鸡出壳后24小时内实现快速鉴别雌雄,准确率超过95%。尽管该技术一定程度上满足了蛋鸡商品代养殖的需求,但是和母鸡等量的公鸡则被立即屠杀,造成很大了浪费和人道主义困扰。该技术的实施需要培育专门化品系(如慢羽系等),需要通过特殊杂交配套组合才能实现,无法直接通过本品种选育实现雌雄自别。另一类技术是直接对处于孵化阶段的受精蛋进行操作,判断性别。典型方法如光谱扫描,利用不同性别胚胎生理生化指标如:血红蛋白浓度等成分浓度不同,实现胚胎早期性别鉴定,该方法需要胚胎至少发育到7天以后(即胚胎性别分化到足够程度)才能实现。利用穿刺技术,提取胚胎内部分组织液,通过pcr(聚合酶链式反应),可以鉴别胚胎染色体组成,从而判断性别。即便在胚胎孵化7天后判断出受精蛋性别,已经入孵一周多的种蛋也无法另做他用,造成资源浪费。同时该类技术均需破坏蛋壳,无法实现工业化生产。
因此,需要一种能够在胚胎发育早期(如入孵后一天内)即可准确判断受精蛋性别的技术,既能保证母雏的正常生产,也不会对雄性种蛋造成浪费的技术,以克服现有技术存在的缺陷。
技术实现要素:
本发明的一个目的是解决至少上述问题,并提供至少后面将说明的优点。
本发明还有一个目的是提供一种一日龄鉴别鸡受精蛋性别的方法,能够在受精蛋入孵后一天内即可准确判断受精蛋性别,既能保证母雏的正常生产,也不会对雄性种蛋造成浪费的技术,该方法方便快捷、价格低廉,适于推广与应用。
为了实现本发明的这些目的和其它优点,提供了一种一日龄鉴别鸡受精蛋性别的方法,包括以下步骤:
在鸡z染色体上定点插入标签蛋白基因,并构建基因型为携带标签蛋白基因插入片段纯合子的纯系,再通过纯系与野生型个体进行配套组合,所生的种蛋入孵第一天通过光学设备照射就可判断种蛋的性别。
优选的是,所述标签蛋白基因为z染色体上定点位置插入可用于早期鉴别且可被检测到的标记基因。
优选的是,所述标签蛋白基因为绿色荧光蛋白基因。
优选的是,构建基因型为携带绿色荧光蛋白基因插入片段纯合子的纯系,具体包括以下的方法步骤:
步骤1:根据禽类z染色体基因组序列,设计引导rna序列;
步骤2:通过分子克隆,将引导rna序列插入到含有xcas9蛋白的aav-crispr质粒的多克隆位点上,构建crispr/cas基因切割质粒;
步骤3:根据z染色体上插入位点获取靶序列,再根据靶序列上下游1kb的基因序列分别扩增上游同源臂序列如seqidno:1所示和下游同源臂序列如seqidno:2所示,将上游同源臂序列和下游同源臂序列通过分子克隆与绿色荧光蛋白基因连接,形成供体基因序列,并将供体基因序列克隆到aav质粒中,构建crispr/cas基因供体质粒;
步骤4:将构建好的crispr/cas基因切割质粒和crispr/cas基因供体质粒分别进行avv病毒包装,得到两种病毒原液,再分别经纯化、浓缩后得到两种工作液病毒;
步骤5:将得到的两种工作液病毒共同注射到早期的鸡受精卵的胚盘中,并进行孵化;、
步骤6:孵化后得到的仔鸡f0代,通过表型和pcr扩增检测进行阳性鉴定;将阳性的f0代鸡与野生型鸡配种,并通过阳性鉴定方法,得到具有外源基因遗传能力的杂合子f1代鸡;;
步骤7:将步骤6中的f1代阳性杂合子公鸡和阳性母鸡纯繁产生的雏鸡,通过阳性鉴定方法,得到公鸡和母鸡均为基因修饰的纯合个体。
优选的是,步骤1中所述引导rna序列具体设计方式为:通过在位于z染色体上75842691-75842693bp上找到合适的pam位点,并获得该位点附近20-24bp的靶序列,然后将靶序列和tracerrna序列融合而成。
优选的是,步骤4中所述工作液病毒的滴度不小于1012vg/ml。
优选的是,步骤5中注射的位置为:在待注射种蛋平置并标记长轴和短轴的交点,酒精消毒后在交点处开窗,并找到胚盘上方的明区。
优选的是,步骤5中所述工作液病毒的注射体积为0.5-2μl。
优选的是,步骤6和步骤7中阳性鉴定时,pcr扩增检测的条件为:95℃预变性5min;94℃变性30s-60℃退火30s-72℃延伸30s,循环25次;72℃延伸5min;4℃保存。
优选的是,所述纯系与野生型个体进行配套组合,具体配套组合形式为:不携带绿色荧光蛋白的野生型公鸡和步骤7中筛选出的携带绿色荧光蛋白的母鸡交配,所产生的种蛋依据有无绿色荧光判断雌雄,能检测到绿色荧光反应的为雄性种蛋,不能检测到绿色荧光反应的雌性种蛋。
优选的是,所述光学设备是能以波长395nm的紫外光或450-490nm的蓝光作为激发光照射种蛋的设备。
本发明至少包括以下有益效果:
本发明利用基因编辑技术,将绿色荧光蛋白基因插入鸡性染色体z,并构建基因型均为携带绿色荧光蛋白插入片段纯合子的纯系。通过该纯系与野生型个体进行配套组合,其种蛋入孵第一天即可准确判断种蛋性别:雌性种蛋不携带绿色荧光蛋白基因可用于大规模蛋鸡生产,携带插入基因的雄性种蛋可用作生物反应器等用途。相较于其他胚胎性别鉴定方法,本发明胚胎雌性鉴定在种蛋入孵第一天完成,准确性高,该方法不破坏蛋壳,对种蛋应激小,并且早期性别鉴定后的胚胎可及时处理,避免浪费孵化资源。
本发明的其它优点、目标和特征将部分通过下面的说明体现,部分还将通过对本发明的研究和实践而为本领域的技术人员所理解。
附图说明
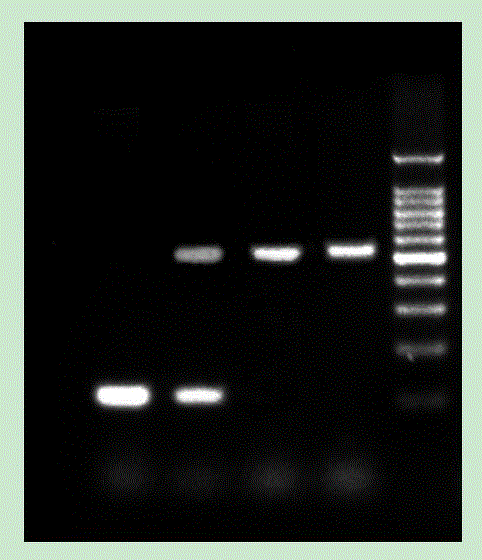
图1为本发明所述的f0代基因修饰动物基因型鉴定结果,泳道从左到右分别为wt(未经修饰的野生型基因)、雄性杂合、雄性纯合、雌性阳性,marker最小为200bp,marker最亮条带为1000bp;
图2为所述的构建得到的纯系和野生型个体配套组合形式。
具体实施方式
下面对本发明做进一步的详细说明,以令本领域技术人员参照说明书文字能够据以实施。
应当理解,本文所使用的诸如“具有”、“包含”以及“包括”术语并不排除一个或多个其它元件或其组合的存在或添加。
本发明提供一种一日龄鉴别鸡受精蛋性别的方法,包括以下步骤:
在鸡z染色体上定点插入标签蛋白基因,并构建基因型为携带标签蛋白基因插入片段纯合子的纯系,再通过纯系与野生型个体进行配套组合,所生的种蛋入孵第一天通过光学设备照射就可判断种蛋的性别。
在上述方案中,鸡z染色体上两个基因间找到合适的pam位点,通过克隆的方式将外源报告基因插入,本申请是通过插入标签蛋白基因,通过插入的标签蛋白的表达,可通过表型和基因型共同鉴定筛选出后代中携带标签蛋白插入片段纯合子的纯系,通过纯系和野生型个体的配套组合,所生的种蛋中携带一份标签蛋白等位基因的即为雄性种蛋,而不携带插入标签蛋白插入片段的基因,表现为野生型,即为雌性种蛋。
一个优选方案中,所述标签蛋白基因为z染色体上定点位置插入可用于早期鉴别且可被检测到的标记基因。
一个优选方案中,所述标签蛋白基因为绿色荧光蛋白基因。
上述方案中,采用绿色荧光蛋白基因便于早期鉴别,通过光学设备照射根据是否有绿色或蓝色荧光就能方便的检测种蛋的性别。
一个优选方案中,构建基因型为携带绿色荧光蛋白基因插入片段纯合子的纯系,具体包括以下的方法步骤:
步骤1:根据禽类z染色体基因组序列,设计引导rna序列;
步骤2:通过分子克隆,将引导rna序列插入到含有xcas9蛋白的aav-crispr质粒的多克隆位点上,构建crispr/cas基因切割质粒;
步骤3:根据z染色体上插入位点获取靶序列,再根据靶序列上下游1kb的基因序列分别扩增上游同源臂序列如seqidno:1所示和下游同源臂序列如seqidno:2所示,将上游同源臂序列和下游同源臂序列通过分子克隆与绿色荧光蛋白基因连接,形成供体基因序列,并将供体基因序列克隆到aav质粒中,构建crispr/cas基因供体质粒;
步骤4:将构建好的crispr/cas基因切割质粒和crispr/cas基因供体质粒分别进行avv病毒包装,得到两种病毒原液,再分别经纯化、浓缩后得到两种工作液病毒;
步骤5:将得到的两种工作液病毒共同注射到早期的鸡受精卵的胚盘中,并进行孵化;、
步骤6:孵化后得到的仔鸡f0代,通过表型和pcr扩增检测进行阳性鉴定;将阳性的f0代鸡与野生鸡配种,并通过阳性鉴定方法,得到具有外源基因遗传能力的杂合子f1代鸡;;
步骤7:将步骤6中的f1代中阳性杂合子公鸡和阳性母鸡纯繁产生的雏鸡,通过阳性鉴定方法,得到公鸡和母鸡均为基因修饰的纯合个体。
一个优选方案中,步骤1中所述引导rna序列具体设计方式为:通过在位于z染色体上75842691-75842693bp上找到合适的pam位点,并获得该位点附近20-24bp的靶序列,然后将靶序列和tracerrna序列融合而成。
一个优选方案中,步骤4中所述工作也病毒的滴度不小于1012vg/ml。
一个优选方案中,步骤5中注射的位置为:在待注射种蛋平置并标记长轴和短轴的交点,酒精消毒后在交点处开窗,并找到胚盘上方的明区;
所述工作液病毒的注射体积为0.5-2μl。
一个优选方案中,步骤6和步骤7中阳性鉴定时,pcr扩增检测的条件为:95℃预变性5min;94℃变性30s-60℃退火30s-72℃延伸30s,循环25次;72℃延伸5min;4℃保存。
一个优选方案中,所述纯系与野生型个体进行配套组合,具体配套组合形式为:不携带绿色荧光蛋白的野生型公鸡和步骤7中筛选出的携带绿色荧光蛋白的母鸡交配,所产生的种蛋依据有无绿色荧光判断雌雄,能检测到绿色荧光反应的为雄性种蛋,不能检测到绿色荧光反应的雌性种蛋。
一个优选方案中,所述光学设备是能以波长395nm的紫外光或450-490nm的蓝光作为激发光照射种蛋的设备。
另外,本申请主要采用的技术手段是在鸡z染色体上定点插入可用于早期鉴别的,可被检测到的标签蛋白基因,故不仅限于下面具体方法中所使用的具体基因编辑方法、所使用的质粒、所使用的标签蛋白基因。
为了达到在性染色体上进行特异性基因编辑,发明人使用目前最先进的第三代基因组定点编辑技术:crispr/cas(成簇规律间隔短回文重复序列)技术。该技术基本原理为tracerrna与目标序列融合形成的引导rna(grna)和cas蛋白形成切割复合体;该复合体可以特异性的识别基因组上的前间隔序列临近基序(pam)附近的靶序列,并将靶序列切开,形成双链断裂(dsb)。机体内存在天然的dna断裂修复机制非同源末端链接(nhei)和同源重组(hr);当有供体dna存在的情况下,会发生同源重组,此时供体dna会插入到dna断裂位置,最终达到插入外源报告基因的目的。本申请根据此方法,设计了针对鸡z染色体非编码区绿色荧光蛋白基因的定点插入方法。
实施例1
一种一日龄鉴别鸡受精蛋性别的方法,选用某白壳蛋鸡品系。
1、构建基因修饰鸡纯系
(1)引导rna序列设计
根据禽类z染色体基因组序列,在位于z染色体上两个基因的间隔序列中寻找pam位点,并获得其附近20-24个碱基的靶序列,与已知的tracerrna序列融合,设计引导rna序列,其中,正向grna序列如seqidno:3所示;反向grna序列如seqidno:4所示,整反向序列分别合成后,95℃退火合成双链dna。
(2)基因切割质粒构建
通过加在grna序列正方向两侧的酶切位点序列,直接连接到含有bsmb1酶切位点的aav-crispr质粒(该质粒含有xcas9蛋白)的多克隆位点上,该位点位于真核启动子下游,从而构建crispr/cas基因切割质粒。
(3)供体质粒构建
发生同源重组的必要前提条件是供体基因序列上下游需要有和dna断裂位置上下游相匹配的同源序列。根据靶序列上下游各1kb的基因组序列,设计pcr扩增引物,分别扩增靶序列上游和下游约1kb的同源臂,通过分子克隆手段将上同源臂如seqidno:1所示和下游同源臂如seqidno:2所示连接到绿色荧光蛋白基因(egfp,如seqidno:5所示)的5’端和3’端,形成完整的供体基因序列;最后将该供体基因序列克隆到aav质粒中,构建crispr/cas供体质粒。
(4)aav病毒包装
将构建好的crispr/cas基因切割质粒及供体质粒,连同辅助质粒、包装质粒,按照25:15:13的比例,分别转染到hek293t细胞中。轻轻震荡混匀后,将细胞置于37℃,5%co2培养箱中培养,转染后24h观察转染效率,并在培养3天后,收集细胞培养上清,进行病毒纯化。得到的病毒原液经纯化、浓缩后得到可用于转基因操作的工作液病毒。为保证足够的病毒感染效率,工作液病毒的滴度不小于1012vg/ml。
(5)鸡胚基因修饰操作
将得到的两种工作液病毒共同注射到早期的鸡受精卵的胚盘中(1d-5d),操作如下:将待注射种蛋平置并标记长轴与短轴的交点,酒精消毒后在交点处开窗,大小以能看到胚盘为宜。在体视显微镜下找到胚盘上方清楚地卵黄膜表面(明区),用显微注射针吸取0.5-2ul的病毒,注射到明区位置,注射完毕后用石蜡膜将种蛋重新封闭,并进行孵化。
(6)f0代基因修饰动物鉴定及传代
孵化得到的仔鸡即为f0代,对f0动物进行阳性鉴定。
阳性动物标准:1、表型,即动物皮肤组织在488nm左右的蓝色激光发光下,显示为绿色;2、以鸡组织dna为模板的pcr扩增,可以检测到egfp基因;将阳性的f0代动物与野生型动物配种,得到阳性的f1代动物(鉴定方法与f0动物相同),此时的f1代动物即为具有外源基因(绿色荧光蛋白)遗传能力的杂合子动物。
pcr鉴定引物:f:caactacaccctatgcctg
r:ttccctcccctctagggag
扩增条件:95℃预变性5min;94℃变性30s-60℃退火30s-72℃延伸30s,循环25次;72℃延伸5min;4℃保存。
设计基因修饰动物pcr鉴定引物时,上下游引物分别位于z染色体基因组靶序列的两侧。阳性动物由于在靶序列位置插入了外源的荧光标记基因,扩增后会得到1028bp的产物,其中含有egfp基因;而非基因修饰动物,则只有230bp的产物,两种不同产物可以通过琼脂糖凝胶电泳进行分离判别,此方法同样可以鉴别杂合子和纯合子雄性个体。因为杂合子个体存在基因修饰的z染色体和未经修饰的z染色体,所以pcr产物的电泳条带有两条(阳性的大条带和阴性的短条带);而纯合子个体的z染色体均为基因修饰过的,所以产物只有一条大的条带,如图1所示。
(7)获得基因修饰纯合子动物:
上述步骤(6)得到的母鸡因只携带一条z染色体,阳性即为基因修饰纯合个体。阳性杂合子公鸡与阳性母鸡纯繁产生的雏鸡,重复步骤(6)中pcr检测方法,淘汰pcr扩增结果显示两条带的个体,所剩的公母鸡均为基因修饰纯合个体,该公母鸡即可进行闭锁继代纯繁,保持基因修饰纯合特性。
2、构建杂交配套组合
(1)采集野生型公鸡精液,用精液稀释液稀释5倍,按公母比1:45对母鸡进行人工授精,每6天输精一次。
(2)收集合格种蛋,送入蛋库。
上述的构建原理为:将1构建得到的纯系和野生型个体可通过如图2所示的配套组合形式,生产种蛋。
3、种蛋鉴别
(1)使用波长为395nm紫外光或450-490nm的蓝光作为激发光,照射种蛋;
(2)通过光学设备能够检测到绿色荧光反应的即为雄性种蛋,未检测到绿色荧光反应的即为雌性种蛋。
本申请还与现有技术中其他性别鉴定方法进行了效果对比,如表1
表1鸡不同性别鉴别方法的效果对比
从表1中可看出,翻肛人工劳动强度大,鉴别准确性低;依据伴性遗传性状如金银羽和快慢羽等,需在出雏后24小时内完成,尽管可以降低劳动强度增加鉴别准确性,但是大量公雏被扑杀,造成人道主义困扰的同时也是对生产资源的浪费;远红外扫描和穿刺检测需要在种蛋入孵7天后才能进行,前者鉴别准确性低,后者需要破坏蛋壳,均无法实现工业化应用,并且入孵超过7天的种蛋无法另作他用,也是对资源的浪费。本发明可在种蛋入孵1天内完成雌雄鉴别,雌性种蛋为完全野生型,对正常生产不产生任何影响,雄性种蛋刚入孵1天,完全不影响做其他用途。
尽管本发明的实施方案已公开如上,但其并不仅仅限于说明书和实施方式中所列运用,它完全可以被适用于各种适合本发明的领域,对于熟悉本领域的人员而言,可容易地实现另外的修改,因此在不背离权利要求及等同范围所限定的一般概念下,本发明并不限于特定的细节和这里示出与附图。
序列表
<110>南京农业大学首都医科大学附属北京同仁医院
<120>一种一日龄鉴别鸡受精蛋性别的方法
<160>5
<170>siposequencelisting1.0
<210>1
<211>1047
<212>dna
<213>人工序列(artificialsequence)
<400>1
ctgctagatcattttgattctgttctacgtgtcccaaaataggcccgacttcccaccttt60
ggcctcaactcatgtctctttcatatctcctttttctttctagtactccggcacaggcag120
gcagagcatctcatcatgacacaggtaaaacctcccaacagcaccaagaggatgcaaact180
aggaagagctgctttaaccatcctagattcagtagccagctagtaagcaagttccctaaa240
tctccttcccagctggtgtcatcctgacttgtcctaggcaggactttcatctgttcccaa300
actttccatcaatctctcccgattcttaaatccttcttgctgtaagcacagcgactctga360
tttattcttgtgcaaacccctccttccttggctgttaatcggtctagtgccatcctattc420
tggagcactgctttggataaaggggacacttcttcttggattgctcgtaatgcatccgtt480
gtgccattctctatgatttccagagttgcagagatattgactctagctttttctagctgg540
gcaactcctagagccgggataagggctctcggaaagcagtggtatcccttgttctatata600
gctaggggtttgtatgtacgcttcgtttgaatcccaagggtcctcaggagtccccgaggg660
atctcctcatgcaccatggtctgggctattaagtatcctgtagtgcaagtccctgtccaa720
tgtaaaggtactgctttccgtgcctgagcgtccccacataaccaccatgtggtatccatg780
aggtatctgacatggcctggccatcttttcctgaagagtgaacgatgctcccatccctac840
aattccgagtatgttcttctctgagagccagtgtgactcgttatcgattctaggatcaca900
ccccacactttggttccacccccaactacaccctatgcctgtcaagttctgtcagccaac960
ccccaccccacctttaccgtcagtttttctgtctacagagatagacctagtagagttagt1020
aactagtcaatgcaatgtaggtccctt1047
<210>2
<211>1053
<212>dna
<213>人工序列(artificialsequence)
<400>2
ttgtggcttgtcttctatcccaaacttacactttccgtgctctagccaacctggaagggt60
ccaatgtagtgccaaaatcccataaacgggcacctttccctcccctctagggagggcatt120
gcatatccaacaagatttgttgagagcctggcccattttctcagttcttcggaagtagct180
cttctcttgccaagctgtgactactacccccctcatactgaccattctgaaactcttatc240
cgtgctttcggatcctgattttcagcggtcctctctcttgtgctgtccactgtggtcctg300
gtgctttcttcaggcaggtatgatgtatccacggtgtaagcccctctaccttggccgtgg360
tgtgggtggtcaggagaagttggtagggtccttcccactttggttccagaggtacatctg420
agggagattgtacgtaaacacggtcgcctggcaatacatggtgcactggagcgtcagtgc480
tccagtcaatcagttcccagtcatgtgcttctttgttggaccacctttgcaaaaatgtgg540
gcctcgatctgctgagattgctccaggagccctgaaccttggaataaacatcatgtaaca600
gcatttttgttgcttcccgaaccttaggagtcctccagggaaatacctctggccatcccc660
aaaaggtatcagttagtgccaacacccagcgatcccccccttttcttgggggtttcagga720
agtcactttgtcagtgttgccctggaaggttccctttcccaataggcccacttgtagccc780
tttgctggtactaggatcgtgctttgtaacctctgtttacggtctggaattaaagccctc840
ttcaacgacctgctctaccccctgatccaccatcttctctccagtttccggtacagtgtt900
ctctttctgatgtccttgacaatggatggtacccacgcttaccttagtgaagatttctgt960
aacttctgggggtacaccactgtttgtcccagtttgtatgtaagccagacttaccatttt1020
cagtcagttcatcttgtttgacccttagctcca1053
<210>3
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>3
caccgtgcaatgtaggtcccttttgtgg28
<210>4
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>4
cacgttacatccagggaaacacccaaa27
<210>5
<211>798
<212>dna
<213>人工序列(artificialsequence)
<400>5
atggtgagcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctggac60
ggcgacgtaaacggccacaagttcagcgtgtccggcgagggcgagggcgatgccacctac120
ggcaagctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggcccacc180
ctcgtgaccaccctgacctacggcgtgcagtgcttcagccgctaccccgaccacatgaag240
cagcacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatcttc300
ttcaaggacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctg360
gtgaaccgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcac420
aagctggagtacaactacaacagccacaacgtctatatcatggccgacaagcagaagaac480
ggcatcaaggtgaacttcaagatccgccacaacatcgaggacggcagcgtgcagctcgcc540
gaccactaccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaaccac600
tacctgagcacccagtccgccctgagcaaagaccccaacgagaagcgcgatcacatggtc660
ctgctggagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaagtcc720
ggagctgcggccgctgccgctgcggcagcggccgaattccccgggctcgagaagcttgga780
tccaccggatctagataa798
- 还没有人留言评论。精彩留言会获得点赞!