一种重组多肽连接酶原及其制备、激活方法与应用与流程
本发明属于生物技术领域,特别涉及一种重组多肽连接酶原及其制备、激活方法与应用。
背景技术:
生物活性肽具有多种人体代谢和生理调节功能,易消化吸收,有促进免疫、激素调节、抗菌、抗病毒、降血压、降血脂等作用。与线性多肽相比,环肽不但具有较高的结构稳定性、热稳定性和生物利用度,而且能够抗水解,在化学变性剂条件下能保持完整生物活性,更为重要的是环化能够不同程度地增强多肽原有生理活性。因此,将线性多肽首尾环化成环状一直是国际生物化学与食品药品领域致力解决的关键问题。
受制备技术的限制,目前环肽主要用于价格昂贵的药物开发。目前来说,制备环肽的主要方法有:天然环肽分离精制、化学合成及微生物发酵合成。然而,天然产物中环肽含量非常低,分离提纯困难;化学合成需要繁琐的基团保护,降低了反应产率和产品纯度;发酵法则需要特殊菌株,能生产的环肽种类少。相对于上述环肽合成方式,近年来环肽的酶法合成表现出极高的应用前景,还可克服其它制备方法产量和纯度低、副产物产生、分离纯化复杂等缺点。
2014年,新加坡学者首次报道了从药用植物蝶豆(clitoriaternatea)豆荚中分离纯化出特异性天冬酰氨内肽酶butelase1,发现其能高效催化线性多肽分子内环化,具有底物范围广、识别基序简单、催化速度快等特点。但是,植物提取容易受原料限制,提取工艺耗时长、产率低,耗时3天从1kg豆荚中仅得到的5mg天然butelase1。因此,通过重组异源表达技术生产和纯化生产活性butelase1是研究热点。截止目前,在原核表达方面仅有澳大利亚学者amym.james通过大肠杆菌异源表达全长氨基酸序列的butelase1,但其活性远不及天然提取的butelase1。也有学者尝试利用酵母表达系统重组表达butelase1,但是,与大肠杆菌原核表达系统相比,真核的酵母表达体系表达量低,表达周期长,分离纯化成本高。
技术实现要素:
本发明的第一目的在于克服现有技术的缺点与不足,提供一种重组多肽连接酶原;所述的重组多肽连接酶原的体外自激活能力获得了显著的提高。
本发明的第二目的在于提供一种重组多肽连接酶原的制备方法,实现了所述的重组多肽连接酶原的高效可溶表达。
本发明的第三目的在于提供一种重组多肽连接酶原的激活方法,所述激活方法获得的重组多肽连接酶原激活产物活性强,催化效率高。
本发明的第四目的在于提供所述的重组多肽连接酶原或其激活产物的应用。
本发明的目的通过下述技术方案实现:
一种重组多肽连接酶原,具有seqidno:1所示氨基酸序列。所述重组多肽连接酶原的氨基酸序列与野生型蝶豆源多肽连接酶原的氨基酸序列seqidno:2相比,具有以下氨基酸突变:338位赖氨酸突变为天冬氨酸;358位脯氨酸突变为天冬酰胺;359位谷氨酸突变为天冬氨酸,将1~20位信号肽截短,以及n末端融合有mbp标签。
编码所述的重组多肽连接酶原的dna分子,具有seqidno:3所示的核苷酸序列。所述的核苷酸序列基于大肠杆菌表达系统对密码子进行了优化,能显著提高异源基因在宿主菌中的表达效率。
一种重组表达载体,含有编码所述的重组多肽连接酶原的核苷酸序列。
优选的,所述的重组表达载体含有如seqidno:3所示的核苷酸序列。
一种重组多肽连接酶原的制备方法,包括如下步骤:
将编码所述的重组多肽连接酶原的核苷酸序列克隆入表达载体中构建重组表达载体;再将所述的重组表达载体转化原核表达系统中进行诱导表达,固液分离收集菌体沉淀,纯化后得到所述的重组多肽连接酶原。
所述的原核表达系统优选为大肠杆菌表达系统;更优选为大肠杆菌rosetta(de3)。
所述的重组表达载体的优选为通过t7启动子进行表达。
所述的表达载体优选为含有mbp标签的表达载体,更优选为pmal-c5x或pmal-c5x-his,可以极大地增加目的蛋白表达的可溶性。
所述的诱导表达的具体操作步骤优选为:将阳性转化体接种于lb培养液进行培养,当菌液od600达到0.5~0.9时,加入终浓度为0.1~0.6mmol/l的iptg,15~30℃诱导12~28h;所述的lb培养液中含有卡那霉素30~50μg/ml和氨苄青霉素50~100μg/ml。
所述的将阳性转化体接种于lb培养液进行培养的具体操作为:
(1)将所述的阳性转化体在lb培养液中于25~40℃振荡培养过夜得到种子液;
(2)将种子液接种到lb培养液中,于25~40℃,150~250r/min条件下培养。
步骤(2)中所述的接种量优选按与lb培养液的体积比为1:50~1:150进行接种。
所述的纯化的步骤优选为:裂解细胞后收集上清液,将上清液过亲和层析柱,洗脱得到纯化后的重组多肽连接酶原。
所述的裂解细胞的方法优选为:用超声破碎法冰浴裂解细胞。
所述的超声破碎法中,优选将菌体于缓冲液a中重悬后进行;所述的缓冲液a的配方为20mmnah2po4-na2hpo4、200mmnacl、1mmedta、1mmdtt,ph=7。
所述的缓冲液a的用量优选为每克菌体加入10ml缓冲液a。
所述的超声破碎法的条件优选为:功率为50~200w,超声1~5s,间隔1~5s,持续2~30min。
所述的裂解细胞后收集上清液的操作优选为:裂解后的菌体进行冷冻离心后弃沉淀,过膜,得到所述的上清液。
所述的冷冻离心的条件优选在4℃条件下12000r/min离心40min。
所述的过膜优选为用0.22μm微孔滤膜过滤。
所述的亲和层析柱优选为mbp标签亲和层析柱。
所述的洗脱优选为先用缓冲液a平衡,再用缓冲液b洗脱;所述的缓冲液b的配方为:20mmnah2po4-na2hpo4、100mmnacl、1mmedta、10mm麦芽糖,ph=7。
一种重组多肽连接酶原的激活方法,包括如下步骤:将重组多肽连接酶原先在ph为3.5~6的环境下进行第一次透析,再于ph为4.5~7的环境下进行第二次透析,固液分离去除沉淀,得到所述的重组多肽连接酶原激活产物。
所述的激活方法优选在25~45℃下进行。
所述的第一次透析优选在包含如下组分的缓冲液ⅰ中进行:20~50mm乙酸-乙酸钠、50~200mmnacl、1~5mmedta、2~5mmdtt。
所述的第一次透析的时间优选为8~24h。
所述的第二次透析优选在包含如下组分的缓冲液ⅱ中进行:20~50mmna2hpo4-nah2po4或na2hpo4-柠檬酸、50~200mmnacl、1~5mmedta、0.5~1mmdtt。
所述的第二次透析的时间优选为2~8h。
所述的透析优选在透析袋中进行,所述的透析袋优选为截留分子量为3~10kda的透析袋。
一种重组多肽连接酶原激活产物,通过所述的重组多肽连接酶原激活方法得到。
所述的重组多肽连接酶原或所述的重组多肽连接酶原激活产物的应用,包括用于多肽或蛋白质的环化、多肽与多肽或蛋白质之间的连接、蛋白质与蛋白质之间的连接、蛋白质或多肽与其它化合物的连接中。
当用于多肽或蛋白质的环化时,所述的多肽或蛋白质的c端含有天冬酰胺-组氨酸-缬氨酸(nhv)序列,n端第一位氨基酸是除了脯氨酸以外的19种常见氨基酸,n端第二位氨基酸残基必须是异亮氨酸、亮氨酸、缬氨酸或者半胱氨酸(i、l、v或c);所述的多肽或蛋白质优选为线性多肽或蛋白质。
所述的环化的形式为,多肽或蛋白质的c端的组氨酸-缬氨酸(hv)序列断裂,然后n端和c端首尾以酰胺键相连即得到环肽。
当用于多肽与多肽或蛋白质之间的连接、蛋白质与蛋白质之间的连接时,至少有一个多肽或蛋白质的c端含有天冬酰胺-组氨酸-缬氨酸(nhv)序列,另一个多肽或蛋白质满足n端第一位氨基酸是除了脯氨酸以外的19种常见氨基酸,n端第二位氨基酸残基必须是异亮氨酸、亮氨酸、缬氨酸或者半胱氨酸(i、l、v或c)。
所述的连接的形式为,一条多肽或蛋白质的c端的组氨酸-缬氨酸(hv)序列断裂,然后和另一条多肽或蛋白质的n端形成酰胺键即得到连接产物。
所述的应用的条件优选为:温度为25~60℃,ph为4.5~7。
所述的应用优选在含有如下成分的缓冲液中进行:20~50mmna2hpo4-nah2po4、na2hpo4-柠檬酸或2-吗啉乙磺酸,50~200mmnacl,1~5mmedta,0.5~1mmdtt。
所述的缓冲液优选还含有0.05%~0.3%的tritonx-100。
本发明相对于现有技术具有如下的优点及效果:
1、本发明采用大肠杆菌原核表达的方法,制备了可溶性的且能被高效激活的重组多肽连接酶原,目的蛋白含量占总蛋白的60~70%,表达量高,且分离纯化只需一次亲和层析,简单高效,易于大量表达,适合工业化生产。
2、本发明针对原核表达体系进行了密码子优化,同时定点突变了野生型butelase1的三个氨基酸,可以增强重组多肽连接酶原的体外自激活能力,同时提高重组多肽连接酶原激活产物的催化效率。
3、本发明利用带有mbp标签的表达载体(例如pmal-c5x-his载体)来进行目的蛋白的表达,使得表达的蛋白在n端具有一个mbp促溶标签。不但增加了目的蛋白的表达量和可溶性,而且,经mbp标签亲和层析,可以简化重组多肽连接酶原的分离纯化。
4、本发明采用低ph激活的方法,获得的激活产物具有和天然提取的多肽连接酶butelase1相同的功能作用,可以进行多肽或蛋白质的环化、多肽与多肽或蛋白质之间的连接、蛋白质与蛋白质之间的连接、蛋白质或多肽与其它化合物的连接;而且,与现有技术中其它的大肠杆菌原核表达得到的多肽连接酶butelase1相比,具有更高的催化效率和更专一的环化活性。
5、相对于现有技术中通过天然提取方法获得butelase1,本发明的方法无需以蝶豆作为原料,只需要简单的培养基作为原料,制备多肽连接酶的过程不受原料限制,在生物化工、医药和食品等领域具有广阔的前景;相对于现有技术中通过酵母表达系统重组表达获得butelase1,本发明的方法表达周期更短,表达量更高,分离纯化更简单。
附图说明
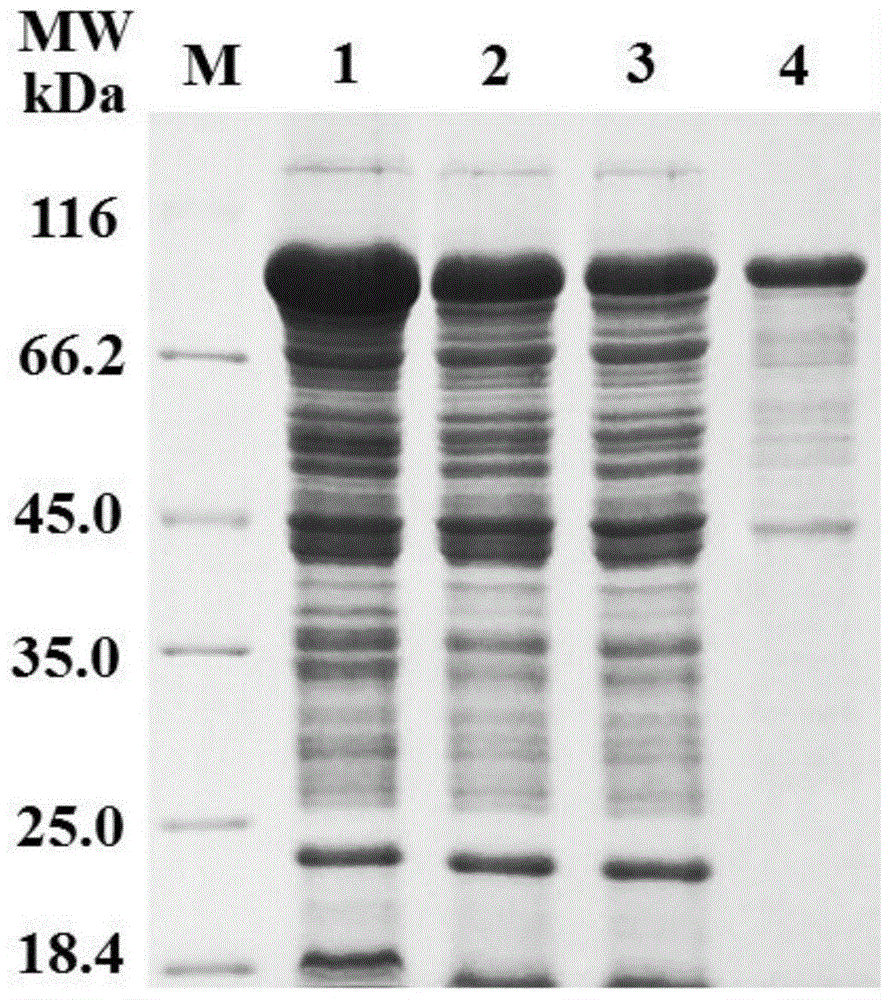
图1为mbp标签亲和层析纯化重组多肽连接酶原的sds-page图;其中,泳道1~4依次为总蛋白、上清蛋白、穿透蛋白和洗脱蛋白。
图2为重组多肽连接酶原激活的sds-page图;其中,泳道1和2分别是重组多肽连接酶原和重组多肽连接酶原激活产物。
图3为重组多肽连接酶原激活前后的相对酶活图。
图4为重组多肽连接酶原激活产物环化多肽kb1-nhv的液相色谱图。
图5为重组多肽连接酶原激活产物环化多肽kb1-nhv的产物峰的maldi-tof图。
图6为重组多肽连接酶原激活产物环化抗菌肽sod6-nhv的maldi-tof图。
图7为重组多肽连接酶原激活产物环化抗菌肽p113-nhv的maldi-tof图。
图8为重组多肽连接酶原激活产物进行多肽分子间连接的maldi-tof图。
图9为重组多肽连接酶原激活产物环化向日葵胰蛋白酶抑制剂的maldi-tof图。
图10是实施例11中不同菌株表达得到的重组多肽连接酶原激活产物的相对酶活结果分析图。
图11是实施例12中野生型重组多肽连接酶原激活产物和突变型重组多肽连接酶原激活产物的相对酶活结果分析图。
具体实施方式
下面结合实施例及附图对本发明作进一步详细的描述,但本发明的实施方式不限于此。
除无特别说明,本发明中用到的各种试剂、原料均为可以从市场上购买的商品或者可以通过公知的方法制得的产品。
实施例1构建含编码重组多肽连接酶原的核苷酸序列的重组载体
构建含编码重组多肽连接酶原的核苷酸序列的重组载体,包括如下步骤:
(1)根据所述重组多肽连接酶原的氨基酸序列,进行大肠杆菌表达系统的密码子优化后,获得能在大肠杆菌中进行高效表达的dna分子,利用拼接pcr的方法,人工合成编码所述重组多肽连接酶原的dna分子,具体如seqidno:3所示。
(2)pmal-c5x-his载体的构建
通过ndei:catatg和bamhi:ggatcc两个酶切位点,将基因片段“gtcgacaagcttgcggccgcactcgagcaccaccaccaccaccactgagatccggctgctaacggatcc”接入pmal-c5x载体(由广州英赞生物科技有限公司提供),得到的pmal-c5x-his载体能够表达带有his标签的目的蛋白。
(3)将编码重组多肽连接酶原的dna分子与表达载体pmal-c5x-his进行重组
合成引物序列如下,ndei-fp:acgccatatgattcgtgatgattttctgcg;sali-rp:acgcgtcgactcacacgctaaaccccg。利用pcr技术对重组多肽连接酶原的dna分子进行扩增,以人工合成编码所述重组多肽连接酶原的dna分子为模板,加入25μl2×pfumaxhifipcrpromix(由广州英赞生物科技有限公司生产,enzyvalley,货号p217)、上下游引物ndei-fp和sali-rp各1μl以及适量的灭菌水,进行pcr扩增。扩增条件为:98℃30s;98℃10s,58℃30s,72℃30s,共30个循环;72℃5min。琼脂糖凝胶电泳回收大小约1.4kb的目的基因片段,以ndei和sali(购于neb)双酶切目的基因片段和载体pmal-c5x-his,并用t4dna酶(购于天根生物科技有限公司)进行连接反应,连接产物转化至dh5a感受态细胞(购于天根生物科技有限公司)中。
(4)挑取单菌落进行菌落pcr鉴定,并将阳性单克隆送往测序公司测序验证,证实编码重组多肽连接酶原的dna分子的正确性,培养验证正确的感受态细胞,并提取质粒,所获得的质粒即为含有编码重组多肽连接酶原的dna分子的重组载体。
实施例2重组多肽连接酶原在重组大肠杆菌中的表达和鉴定
将实施例1获得的重组载体转化到宿主细胞大肠杆菌rosetta(de3)(购于上海唯地生物技术有限公司)中,挑选大肠杆菌阳性转化体,接种于含有少量卡那霉素30μg/ml和氨苄青霉素50μg/ml的lb培养液中,在摇床中于25℃振荡培养过夜作为种子液,取种子液按体积比1:150的比例接种至含有卡那霉素30μg/ml和氨苄青霉素50μg/ml的lb培养液中,于25℃,150r/min条件下培养至菌液od600达到0.5,向每瓶菌液加入iptg至终浓度为0.6mmol/l,于15℃诱导表达28h后,离心收集菌体沉淀。
取iptg诱导前后菌液各1ml,12000r/min离心1min后取沉淀,加入150μl缓冲液a(20mmnah2po4-na2hpo4、200mmnacl、1mmedta、1mmdtt,ph7.0),震荡均匀,冰浴超声破碎,(超声条件为:功率50w,超声1s,间隔1s,持续2min),离心取上清液,沉淀物加入150μl缓冲液a,震荡重悬。取上清及沉淀组分悬液进行sds-page分析,观察蛋白表达情况。sds-page电泳检测发现,诱导后的大肠杆菌菌体中表达有可溶性的重组多肽连接酶原。
实施例3重组多肽连接酶原纯化
将实施例1获得的重组载体转化到宿主细胞大肠杆菌rosetta(de3)中,挑选大肠杆菌阳性转化体,接种于含有少量卡那霉素40μg/ml和氨苄青霉素75μg/ml的lb培养液中,在摇床中于40℃振荡培养过夜作为种子液,取种子液按体积比1:50的比例接种至含有卡那霉素40μg/ml和氨苄青霉素75μg/ml的lb培养液中,于40℃,250r/min条件下培养至菌液od600达到0.9,向每瓶菌液加入iptg至终浓度为0.3mmol/l,于28℃诱导表达15h后,离心收集菌体沉淀,-20℃冻存备用。
取1l发酵液发酵得到的冻存菌体,按每克菌体加入10ml缓冲液a(20mmnah2po4-na2hpo4、200mmnacl、1mmedta、1mmdtt,ph7.0)重悬菌体,用超声波细胞破碎仪冰浴裂解菌体,超声条件为:功率200w,超声5s,间隔5s,持续30min。将裂解后菌体放入高速冷冻离心机,4℃下12000r/min离心30min,取上清液用0.22μm微孔滤膜过滤,备用。
微滤膜滤过液用mbptraptmhp5ml层析柱进行分离。将层析柱接入快速蛋白质纯化仪柱位阀中,用超纯水清洗系统和柱子,再用缓冲液a平衡柱子,然后用样品泵将滤过液上样,用缓冲液a清洗柱子,将没有结合上柱子的蛋白或者是杂质冲洗干净;再用缓冲液b(20mmnah2po4-na2hpo4、100mmnacl、1mmedta、10mm麦芽糖,ph7.0)进行一步洗脱,蛋白变性电泳检测洗脱峰,收集含有目的蛋白重组多肽连接酶原的洗脱峰,收集得到的体积为8ml,浓度为4.0mg/ml,即1l发酵液纯化得到的32mg重组多肽连接酶原。洗脱液的蛋白浓度采用紫外分光光度法,测量波长为280nm,摩尔吸光系数133620m-1cm-1,分子量为94239da。取样20μl,用sds-page蛋白电泳检测。结果如图1所示,泳道1~4依次为总蛋白、上清蛋白、穿透蛋白和洗脱蛋白,洗脱蛋白中分子量为94.3kda位置是目的蛋白重组多肽连接酶原的条带;出乎意料地发现,本发明只需要通过一次mbp标签亲和层析,就得到了电泳纯的重组多肽连接酶原,无需再进行其他纯化步骤(如ni离子亲和层析等);同时,通过sds-page灰度分析法,确定本发明中目的蛋白含量占总蛋白的60~70%,表达量高。
实施例4重组多肽连接酶原的激活
将实施例1获得的重组载体转化到宿主细胞大肠杆菌rosetta(de3)中,挑选大肠杆菌阳性转化体,接种于含有少量卡那霉素50μg/ml和氨苄青霉素100μg/ml的lb培养液中,在摇床中于37℃振荡培养过夜作为种子液,取种子液按体积比1:100的比例接种至含有卡那霉素50μg/ml和氨苄青霉素100μg/ml的lb培养液中,于37℃,200r/min条件下培养至菌液od600达到0.8,向每瓶菌液加入iptg至终浓度为0.1mmol/l,于18℃诱导表达18h后,离心收集菌体沉淀。按实施例3所述步骤分离纯化制备重组多肽连接酶原,4℃冷藏备用。
将重组多肽连接酶原在37℃条件下,用10kda的透析袋在缓冲液ⅰ(20mm乙酸-乙酸钠、100mmnacl、1mmedta、5mmdtt,ph4.5)中透析12h激活后,将装有蛋白液的透析袋转移至缓冲液ⅱ(20mmna2hpo4-nah2po4、100mmnacl、1mmedta、0.5mmdtt,ph6.0)中透析6h,离心去除沉淀,即得到具有环化活性的重组多肽连接酶原激活产物。取样20μl,用sds-page蛋白电泳检测。结果如图2所示,泳道1和2分别是重组多肽连接酶原和重组多肽连接酶原激活产物,激活产物在约40kda左右出现蛋白条带,说明在低ph条件下,重组多肽连接酶原能够很好地自激活。
进一步对激活前的重组多肽连接酶原与激活产物的相对酶活进行测试。在50μl的反应缓冲液(20mmnah2po4-na2hpo4、50mmnacl、1mmedta、0.5mmdtt、0.05%tritonx-100,ph6.0)中,加入0.5μm激活前的重组多肽连接酶原或激活产物和50μmkalatab1-nhv多肽(氨基酸序列为glpvcgetcvggtcntpgctcswpvctrnhv)。37℃温育30min后,加入终浓度为100mm的hcl来终止反应。反应液用0.22μm微孔滤膜过滤,然后取20μl通过hplc进行分析。酶溶液的摩尔浓度采用紫外分光光度法,测量波长为280nm,摩尔吸光系数133620m-1cm-1。
通过液相峰面积计算环化产物的得率,将重组多肽连接酶原激活产物环化kb1-nhv多肽产物的得率定为酶活100%,从而来计算相对酶活。结果如图3所示,未进行激活的重组多肽连接酶原没有活性,激活后得到的激活产物活性显著;进一步说明了所述方法可以成功激活所述的重组多肽连接酶原。
实施例5重组多肽连接酶原激活产物的活性测试
将实施例3获得的重组多肽连接酶原在45℃条件下,用3kda的透析袋在缓冲液ⅰ(50mm乙酸-乙酸钠、200mmnacl、5mmedta、2mmdtt,ph6.0)中透析24h激活后,将装有蛋白液的透析袋转移至缓冲液ⅱ(50mmna2hpo4-nah2po4、200mmnacl、5mmedta、1mmdtt,ph7.0)中透析8h,离心去除沉淀,即得到具有环化活性的重组多肽连接酶原激活产物。
在50μl的反应缓冲液(20mmnah2po4-na2hpo4、50mmnacl、1mmedta、0.5mmdtt、0.05%tritonx-100,ph6.0)中,加入0.5μm重组多肽连接酶原激活产物和50μmkalatab1-nhv多肽(氨基酸序列为glpvcgetcvggtcntpgctcswpvctrnhv)。37℃温育20min后,加入终浓度为100mm的hcl来终止反应。反应液用0.22μm微孔滤膜过滤,然后取20μl通过hplc进行分析鉴定。结果如图4所示,kalatab1-nhv多肽的环化产物在线性多肽约1min后出峰,约有70%的产率;为了鉴定产物峰的身份,接取产物峰液相的流出液,取1μl点板通过maldi-tof进行分析鉴定,结果如图5所示,产物的分子量为2898.33da,和理论的环化产物分子量吻合,即该产物峰就是环化产物峰。所以本发明利用大肠杆菌原核表达系统生产的重组多肽连接酶原激活产物具有良好的环化多肽的活性。
环化判定:线性多肽kbi-nhv分子量为3152.65da,反应之后得到环化产物cyclic-kb1的分子量为2898.33da。
实施例6重组多肽连接酶原激活产物合成环状抗菌肽的应用
将实施例3获得的重组多肽连接酶原在25℃条件下,用5kda的透析袋在缓冲液ⅰ(30mm乙酸-乙酸钠、50mmnacl、3mmedta、4mmdtt,ph3.5)中透析8h激活后,将装有蛋白液的透析袋转移至缓冲液ⅱ(30mmna2hpo4-柠檬酸、50mmnacl、2mmedta、0.75mmdtt,ph4.5)中透析2h,离心去除沉淀,即得到重组多肽连接酶原激活产物。
在50μl的反应缓冲液(50mmnah2po4-na2hpo4、200mmnacl、5mmedta、1mmdtt、0.3%tritonx-100,ph7.0)中,加入0.5μm重组多肽连接酶原激活产物和50μm菠菜叶细胞壁抗菌肽sod6-nhv多肽(氨基酸序列为gifsnmyartpagyfrgpagyaanhv)或人唾液组胺素5的片段p113-nhv多肽(氨基酸序列为glakrhhgykrkfhnhv)。60℃温育2h后,加入终浓度为100mm的hcl来终止反应。反应液用0.22μm微孔滤膜过滤,经ziptip脱盐柱脱盐后,取1μl点板通过maldi-tof进行分析鉴定。结果如图6和7,所述重组多肽连接酶原激活产物能够很好地将线性的sod6-nhv或p113-nhv抗菌肽进行首尾连接的环化,并且都是表现出专一性的环化作用,达到了合成环状抗菌肽的目的,根据madli-tof质谱峰强计算环化产物cyclic-sod6的得率为95%,环化产物cyclic-p113的得率为95%。
环化判定:线性多肽sod6-nhv分子量为2789.12da,反应之后得到环化产物cyclicsod6的分子量为2534.83da。
环化判定:线性多肽p113-nhv分子量为2085.38da,反应之后得到环化产物cyclicp113的分子量为1831.12da。
实施例7重组多肽连接酶原激活产物对多肽进行分子间连接的应用
将实施例3获得的重组多肽连接酶原在37℃条件下,用10kda的透析袋在缓冲液ⅰ(20mm乙酸-乙酸钠、100mmnacl、2mmedta、5mmdtt,ph5.0)中透析16h激活后,转移至缓冲液ⅱ(20mmna2hpo4-nah2po4、100mmnacl、1mmedta、0.5mmdtt,ph6.5)中透析4h,离心去除沉淀,即得到重组多肽连接酶原激活产物。
在50μl的反应缓冲液(30mmnah2po4-柠檬酸、100mmnacl、2mmedta、0.75mmdtt、0.1%tritonx-100,ph4.5)中,加入0.5μm重组多肽连接酶原激活产物、50μm八肽kalvinhv(简称k)和1mm六肽giggir(简称g)。25℃温育1h后,加入终浓度为100mm的hcl来终止反应。反应液用0.22μm微孔滤膜过滤,经ziptip脱盐柱脱盐后,取1μl点板通过maldi-tof进行分析鉴定。结果如图8,所述重组多肽连接酶原激活产物能够很好地将八肽kalvinhv和六肽giggir进行分子间连接,得到产物十二肽kalvingiggir(简称kg),根据madli-tof质谱峰强计算连接产物的得率为80%。
连接判定:线性八肽kalvinhv(简称k)分子量为893.03da,线性六肽giggir(简称g)分子量为571.66da,反应之后得到连接产物十二肽kalvingiggir(简称kg)的分子量为1210.47da。
实施例8重组多肽连接酶原激活产物环化向日葵胰蛋白酶抑制剂
在50μl的反应缓冲液(20mm2-吗啉乙磺酸、100mmnacl、1mmedta、0.5mmdtt,ph5.5)中,加入1mg/ml实施例4获得的重组多肽连接酶原激活产物、250μm多肽底物向日葵胰蛋白酶抑制剂sfti-nhv线形肽(氨基酸序列:grctksippicfpnhv)。37℃温育1h后,加入终浓度为100mm的hcl来终止反应。反应液用0.22μm微孔滤膜过滤,经ziptip脱盐柱脱盐后,取1μl点板通过madli-tof进行分析鉴定。结果如图9,所述的重组多肽连接酶原激活产物能够将多肽底物sfti-nhv进行环化。根据madli-tof质谱峰强计算环化产物的得率为75%,未检测到水解产物,说明该酶制剂的催化效率和环化专一性优良。
环化判定:线性多肽sfti-nhv分子量为1769.11da,反应之后得到环化产物cyclicsfti的分子量为1514.83da。
实施例9本发明中重组多肽连接酶原激活产物与现有butelase1的比较
2014年新加坡学者从1kg药用植物蝶豆(clitoriaternatea)豆荚中分离纯化得到的5mg天然butelase1,工艺耗时长且复杂,提取产率也较低,大量生产时会受到蝶豆原料来源的限制。本发明采用大肠杆菌原核表达的方法,获得了体外自激活能力显著提高的重组多肽连接酶原,表达量高,且分离纯化只需一次亲和层析,简单高效,易于大量表达,适合工业化生产。激活所述的重组多肽连接酶原后得到的重组多肽连接酶原激活产物的生物活性及催化效率出众。
本发明所得的重组多肽连接酶原激活产物与2019年澳大利亚学者amym.james异源表达的butelase1(简称butelase1(2019))相比,具有更高的催化效率和更专一的环化活性。如实施例8中所述,所述重组多肽连接酶原激活产物能催化向日葵胰蛋白酶抑制剂环化,酶反应1h环化产物的得率为75%,未检测到水解产物。在相同的酶反应条件下,butelase1(2019)催化向日葵胰蛋白酶抑制剂反应24h后,根据报道的madli-tof质谱峰强计算环化产物的得率仅约为10%,并且约1%的底物被水解。由此可见,所述重组多肽连接酶原激活产物比butelase1(2019)有更高的催化效率和更专一的环化活性。
本发明所得的重组多肽连接酶原与2019年国内学者nipi酵母表达系统重组表达获得的butelase1(简称rbtase)相比,具有表达周期更短,表达量更高,分离纯化更简单的优势。本发明的重组多肽连接酶原表达周期为15h,表达量为每升发酵液得到32mg目的蛋白,分离纯化只需一次mbp标签柱亲和层析。rbtase表达周期为96h,表达量为每升发酵液得到16mg目的蛋白,分离纯化需联用凝胶排阻层析和ni离子亲和层析才能达到纯度符合要求的目的蛋白。
实施例10表达载体的筛选
在本发明的研究过程中,曾尝试过用pet-28a、phue-ubiquitin、pet-28a-sumo和pmal-c5x-his四种载体(载体均由广州英赞生物科技有限公司提供)来表达重组多肽连接酶原。
构建的pet-28a重组载体,表达的重组蛋白为组氨酸标签+全长多肽连接酶原,氨基酸序列如seqidno:4所示,将pet-28a重组载体转入大肠杆菌rosetta(de3)中进行表达。结果为无目标蛋白的表达。
构建的phue-ubiquitin重组载体,表达的重组蛋白为组氨酸标签+泛素标签+全长多肽连接酶原,氨基酸序列如seqidno:5所示,将phue-ubiquitin重组载体转入大肠杆菌rosetta(de3)中进行表达,结果为少量目标蛋白为可溶表达,大部分均为包涵体表达。
构建的pet-28a-sumo重组载体,表达的重组蛋白为组氨酸标签+sumo标签+全长多肽连接酶原,氨基酸序列如seqidno:6所示,将phue-ubiquitin重组载体转入大肠杆菌rosetta(de3)中进行表达。结果为目标蛋白为可溶表达,但是目标蛋白未能和镍离子亲和层析柱有效结合,即不能纯化得到重组多肽连接酶原。
构建的pmal-c5x-his重组载体,表达的重组蛋白为mbp标签+全长多肽连接酶原,氨基酸序列如seqidno:1所示,将pmal-c5x-his重组载体转入大肠杆菌rosetta(de3)中进行表达。结果为目标蛋白为可溶表达,利用一次mbptraptmhp5ml层析柱纯化即能得到重组多肽连接酶原。
综上,经过多种载体的表达筛选研究,本发明优选采用含有mbp标签的表达载体,更优选的表达载体如pmal-c5x、pmal-c5x-his等载体。
实施例11表达菌株的筛选
在本发明研究过程中,曾尝试过用大肠杆菌bl21(de3)(购于天根生物科技有限公司)和大肠杆菌rosetta(de3)表达菌株,来表达重组多肽连接酶原。
将实施例1获得的重组载体转化到宿主细胞大肠杆菌bl21(de3)和大肠杆菌rosetta(de3)中,分别挑选大肠杆菌阳性转化体进行诱导表达,结果均为重组多肽连接酶原可溶表达。利用一次mbptraptmhp5ml层析柱纯化均能得到重组多肽连接酶原,并在实施例4所描述的激活条件和酶活测试条件下进行激活和活性测试,结果如图10所示,大肠杆菌bl21(de3)表达的重组蛋白激活得到产物没有环化活性;大肠杆菌rosetta(de3)表达的重组蛋白激活得到的产物则具有良好的环化活性。
综上,经过表达菌株的筛选研究,得到优选的表达菌株为大肠杆菌rosetta(de3)。
实施例12突变位点的优化研究
在本发明的研究过程中,曾尝试过两种突变方案。
突变方案一的重组多肽连接酶原的氨基酸序列如seqidno:1所示,与野生型蝶豆源多肽连接酶原的氨基酸序列如seqidno:2相比,具有以下氨基酸突变:338位赖氨酸突变为天冬氨酸;358位脯氨酸突变为天冬酰胺;359位谷氨酸突变为天冬氨酸。
突变方案二的重组多肽连接酶原的氨基酸序列如seqidno:7所示,与野生型蝶豆源多肽连接酶原的氨基酸序列如seqidno:2相比,具有以下氨基酸突变:368位赖氨酸突变为谷氨酸;369位赖氨酸突变为谷氨酸。
野生型重组多肽连接酶原、所述突变方案一重组多肽连接酶原和所述突变方案二重组多肽连接酶原,在实施例2所记载的表达条件下进行表达,在实施例3所记载的纯化条件下进行纯化,在实施例4所记载的激活条件下进行激活。得到相应的产物1、产物2和产物3。在实施例4所记载的酶活测试条件下进行活性测试。结果如图11所示,突变方案一所获得的产物2相对酶活比产物1好,说明其催化效率更高,为正向突变;突变方案二所获得的产物3和产物1活性无明显差别。
综上,突变方案一可以有效地提高重组多肽连接酶原激活产物的催化效率,效果显著,可以作为优选方案。
上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。
序列表
<110>华南理工大学
<120>一种重组多肽连接酶原及其制备、激活方法与应用
<160>17
<170>siposequencelisting1.0
<210>1
<211>853
<212>prt
<213>人工序列(artificialsequence)
<400>1
metlysileglugluglylysleuvaliletrpileasnglyasplys
151015
glytyrasnglyleualagluvalglylyslyspheglulysaspthr
202530
glyilelysvalthrvalgluhisproasplysleugluglulysphe
354045
proglnvalalaalathrglyaspglyproaspileilephetrpala
505560
hisaspargpheglyglytyralaglnserglyleuleualagluile
65707580
thrproasplysalapheglnasplysleutyrprophethrtrpasp
859095
alavalargtyrasnglylysleuilealatyrproilealavalglu
100105110
alaleuserleuiletyrasnlysaspleuleuproasnproprolys
115120125
thrtrpglugluileproalaleuasplysgluleulysalalysgly
130135140
lysseralaleumetpheasnleuglngluprotyrphethrtrppro
145150155160
leuilealaalaaspglyglytyralaphelystyrgluasnglylys
165170175
tyraspilelysaspvalglyvalaspasnalaglyalalysalagly
180185190
leuthrpheleuvalaspleuilelysasnlyshismetasnalaasp
195200205
thrasptyrserilealaglualaalapheasnlysglygluthrala
210215220
metthrileasnglyprotrpalatrpserasnileaspthrserlys
225230235240
valasntyrglyvalthrvalleuprothrphelysglyglnproser
245250255
lysprophevalglyvalleuseralaglyileasnalaalaserpro
260265270
asnlysgluleualalysglupheleugluasntyrleuleuthrasp
275280285
gluglyleuglualavalasnlysasplysproleuglyalavalala
290295300
leulyssertyrgluglugluleuvallysaspproargilealaala
305310315320
thrmetgluasnalaglnlysglygluilemetproasnileprogln
325330335
metseralaphetrptyralavalargthralavalileasnalaala
340345350
serglyargglnthrvalaspglualaleulysaspalaglnthrasn
355360365
serserserasnasnasnasnasnasnasnasnasnasnleuglyile
370375380
gluglyargileserhismetileargaspasppheleuargleupro
385390395400
serglnalaserlysphepheglnalaaspaspasnvalgluglythr
405410415
argtrpalavalleuvalalaglyserlysglytyrvalasntyrarg
420425430
hisglnalaaspvalcyshisalatyrglnileleulyslysglygly
435440445
leulysaspgluasnileilevalphemettyraspaspilealatyr
450455460
asngluserasnprohisproglyvalileileasnhisprotyrgly
465470475480
seraspvaltyrlysglyvalprolysasptyrvalglygluaspile
485490495
asnproproasnphetyralavalleuleualaasnlysseralaleu
500505510
thrglythrglyserglylysvalleuaspserglyproasnasphis
515520525
valpheiletyrtyrthrasphisglyglyalaglyvalleuglymet
530535540
proserlysprotyrilealaalaseraspleuasnaspvalleulys
545550555560
lyslyshisalaserglythrtyrlysserilevalphetyrvalglu
565570575
sercysgluserglysermetpheaspglyleuleuprogluasphis
580585590
asniletyrvalmetglyalaseraspthrglyglusersertrpval
595600605
thrtyrcysproleuglnhisproserproproproglutyraspval
610615620
cysvalglyaspleupheservalalatrpleugluaspcysaspval
625630635640
hisasnleuglnthrgluthrpheglnglnglntyrgluvalvallys
645650655
asnlysthrilevalalaleuilegluaspglythrhisvalvalgln
660665670
tyrglyaspvalglyleuserlysglnthrleuphevaltyrmetgly
675680685
thraspproalaasnaspasnasnthrphethrasplysasnserleu
690695700
glythrproargaspalavalserglnargaspalaaspleuilehis
705710715720
tyrtrpglulystyrargargalaasnaspglyserserarglysala
725730735
glualalyslysglnleuarggluvalmetalahisargmethisile
740745750
aspasnservallyshisileglylysleuleupheglyileglulys
755760765
glyhislysmetleuasnasnvalargproalaglyleuprovalval
770775780
aspasptrpaspcysphelysthrleuileargthrphegluthrhis
785790795800
cysglyserleuserglutyrglymetlyshismetargserpheala
805810815
asnleucysasnalaglyilearglysgluglnmetalaglualaser
820825830
alaglnalacysvalserileproaspasnprotrpserserleuhis
835840845
alaglypheserval
850
<210>2
<211>482
<212>prt
<213>蝶豆(clitoriaternatea)
<400>2
metlysasnproleualaileleupheleuilealathrvalvalala
151015
valvalserglyileargaspasppheleuargleuproserglnala
202530
serlysphepheglnalaaspaspasnvalgluglythrargtrpala
354045
valleuvalalaglyserlysglytyrvalasntyrarghisglnala
505560
aspvalcyshisalatyrglnileleulyslysglyglyleulysasp
65707580
gluasnileilevalphemettyraspaspilealatyrasngluser
859095
asnprohisproglyvalileileasnhisprotyrglyseraspval
100105110
tyrlysglyvalprolysasptyrvalglygluaspileasnpropro
115120125
asnphetyralavalleuleualaasnlysseralaleuthrglythr
130135140
glyserglylysvalleuaspserglyproasnasphisvalpheile
145150155160
tyrtyrthrasphisglyglyalaglyvalleuglymetproserlys
165170175
protyrilealaalaseraspleuasnaspvalleulyslyslyshis
180185190
alaserglythrtyrlysserilevalphetyrvalglusercysglu
195200205
serglysermetpheaspglyleuleuprogluasphisasniletyr
210215220
valmetglyalaseraspthrglyglusersertrpvalthrtyrcys
225230235240
proleuglnhisproserproproproglutyraspvalcysvalgly
245250255
aspleupheservalalatrpleugluaspcysaspvalhisasnleu
260265270
glnthrgluthrpheglnglnglntyrgluvalvallysasnlysthr
275280285
ilevalalaleuilegluaspglythrhisvalvalglntyrglyasp
290295300
valglyleuserlysglnthrleuphevaltyrmetglythrasppro
305310315320
alaasnaspasnasnthrphethrasplysasnserleuglythrpro
325330335
arglysalavalserglnargaspalaaspleuilehistyrtrpglu
340345350
lystyrargargalaprogluglyserserarglysalaglualalys
355360365
lysglnleuarggluvalmetalahisargmethisileaspasnser
370375380
vallyshisileglylysleuleupheglyileglulysglyhislys
385390395400
metleuasnasnvalargproalaglyleuprovalvalaspasptrp
405410415
aspcysphelysthrleuileargthrphegluthrhiscysglyser
420425430
leuserglutyrglymetlyshismetargserphealaasnleucys
435440445
asnalaglyilearglysgluglnmetalaglualaseralaglnala
450455460
cysvalserileproaspasnprotrpserserleuhisalaglyphe
465470475480
serval
<210>3
<211>2562
<212>dna
<213>人工序列(artificialsequence)
<400>3
atgaaaatcgaagaaggtaaactggtaatctggattaacggcgataaaggctataacggt60
ctcgctgaagtcggtaagaaattcgagaaagataccggaattaaagtcaccgttgagcat120
ccggataaactggaagagaaattcccacaggttgcggcaactggcgatggccctgacatt180
atcttctgggcacacgaccgctttggtggctacgctcaatctggcctgttggctgaaatc240
accccggacaaagcgttccaggacaagctgtatccgtttacctgggatgccgtacgttac300
aacggcaagctgattgcttacccgatcgctgttgaagcgttatcgctgatttataacaaa360
gatctgctgccgaacccgccaaaaacctgggaagagatcccggcgctggataaagaactg420
aaagcgaaaggtaagagcgcgctgatgttcaacctgcaagaaccgtacttcacctggccg480
ctgattgctgctgacgggggttatgcgttcaagtatgaaaacggcaagtacgacattaaa540
gacgtgggcgtggataacgctggcgcgaaagcgggtctgaccttcctggttgacctgatt600
aaaaacaaacacatgaatgcagacaccgattactccatcgcagaagctgcctttaataaa660
ggcgaaacagcgatgaccatcaacggcccgtgggcatggtccaacatcgacaccagcaaa720
gtgaattatggtgtaacggtactgccgaccttcaagggtcaaccatccaaaccgttcgtt780
ggcgtgctgagcgcaggtattaacgccgccagtccgaacaaagagctggcaaaagagttc840
ctcgaaaactatctgctgactgatgaaggtctggaagcggttaataaagacaaaccgctg900
ggtgccgtagcgctgaagtcttacgaggaagagttggtgaaagatccgcgtattgccgcc960
actatggaaaacgcccagaaaggtgaaatcatgccgaacatcccgcagatgtccgctttc1020
tggtatgccgtgcgtactgcggtgatcaacgccgccagcggtcgtcagactgtcgatgaa1080
gccctgaaagacgcgcagactaattcgagctcgaacaacaacaacaataacaataacaac1140
aacctcgggatcgagggaaggatttcacatatgattcgtgatgattttctgcgtctgccg1200
agccaggcgagcaagtttttccaggcggatgataatgtggaaggcacccgttgggccgtt1260
ctggttgcgggctctaaaggctatgttaattatcgccatcaggcggatgtttgtcatgcc1320
tatcagattctgaaaaaaggcggtctgaaagatgaaaatatcattgtgtttatgtatgat1380
gatattgcctataatgagagtaatccacatccgggcgtgatcattaatcatccgtatggt1440
agcgatgtgtacaagggcgttcctaaagattatgttggtgaggatattaatcctccgaat1500
ttttatgcagtgctgctggccaataagtctgcactgaccggcaccggctcaggcaaagtt1560
ctggatagcggtccgaatgatcatgtttttatctattatacggatcatggcggcgccggc1620
gtgttagggatgccgagcaaaccgtatatcgccgcgtctgatctgaatgatgtgctgaag1680
aaaaaacacgcaagcggtacatataagtcaatcgtgttttatgtggaatcttgcgaaagt1740
ggctcaatgtttgatggcttactgccagaagatcataatatctatgtgatgggtgcgtca1800
gatacaggtgaatcttcttgggtgacgtattgtccactgcaacatccgagtcctcctcca1860
gaatatgatgtttgcgttggcgatctgttttcagttgcttggttagaagattgcgatgtg1920
cataacctccagacggaaacctttcagcagcagtatgaagtggttaaaaacaaaaccatc1980
gttgccctgattgaagatggcacacatgtggttcagtatggggacgtgggcctgtctaaa2040
cagaccttatttgtgtatatgggtactgatccagccaatgataacaacacctttacggat2100
aaaaatagcttagggacgccacgcgatgccgtgtctcagcgcgatgccgacttaattcat2160
tattgggaaaaatatcgtcgcgcaaatgatggtagtagtcgcaaagccgaagccaaaaaa2220
cagttacgcgaagttatggcacatcgcatgcatattgataatagcgtgaagcacatcggc2280
aagctgctgtttggcattgaaaaaggccataaaatgctgaataatgtgcgccctgcggga2340
ctgccggtggtggatgattgggattgctttaaaaccctgattcgtacctttgaaacccat2400
tgcgggagcctgagcgaatatggcatgaaacacatgcgcagctttgccaatctgtgcaat2460
gcgggcattcgtaaagaacagatggccgaagcgagcgcgcaggcgtgcgtgagcattccg2520
gacaatccgtggagcagcctgcatgcggggtttagcgtgtga2562
<210>4
<211>483
<212>prt
<213>人工序列(artificialsequence)
<400>4
metglyserserhishishishishishisserserglyleuvalpro
151015
argglyserhismetileargaspasppheleuargleuprosergln
202530
alaserlysphepheglnalaaspaspasnvalgluglythrargtrp
354045
alavalleuvalalaglyserlysglytyrvalasntyrarghisgln
505560
alaaspvalcyshisalatyrglnileleulyslysglyglyleulys
65707580
aspgluasnileilevalphemettyraspaspilealatyrasnglu
859095
serasnprohisproglyvalileileasnhisprotyrglyserasp
100105110
valtyrlysglyvalprolysasptyrvalglygluaspileasnpro
115120125
proasnphetyralavalleuleualaasnlysseralaleuthrgly
130135140
thrglyserglylysvalleuaspserglyproasnasphisvalphe
145150155160
iletyrtyrthrasphisglyglyalaglyvalleuglymetproser
165170175
lysprotyrilealaalaseraspleuasnaspvalleulyslyslys
180185190
hisalaserglythrtyrlysserilevalphetyrvalglusercys
195200205
gluserglysermetpheaspglyleuleuprogluasphisasnile
210215220
tyrvalmetglyalaseraspthrglyglusersertrpvalthrtyr
225230235240
cysproleuglnhisproserproproproglutyraspvalcysval
245250255
glyaspleupheservalalatrpleugluaspcysaspvalhisasn
260265270
leuglnthrgluthrpheglnglnglntyrgluvalvallysasnlys
275280285
thrilevalalaleuilegluaspglythrhisvalvalglntyrgly
290295300
aspvalglyleuserlysglnthrleuphevaltyrmetglythrasp
305310315320
proalaasnaspasnasnthrphethrasplysasnserleuglythr
325330335
proarglysalavalserglnargaspalaaspleuilehistyrtrp
340345350
glulystyrargargalaprogluglyserserarglysalagluala
355360365
lyslysglnleuarggluvalmetalahisargmethisileaspasn
370375380
servallyshisileglylysleuleupheglyileglulysglyhis
385390395400
lysmetleuasnasnvalargproalaglyleuprovalvalaspasp
405410415
trpaspcysphelysthrleuileargthrphegluthrhiscysgly
420425430
serleuserglutyrglymetlyshismetargserphealaasnleu
435440445
cysasnalaglyilearglysgluglnmetalaglualaseralagln
450455460
alacysvalserileproaspasnprotrpserserleuhisalagly
465470475480
pheserval
<210>5
<211>558
<212>prt
<213>人工序列(artificialsequence)
<400>5
metglyserserhishishishishishisserserglyleuvalpro
151015
argglyserhismetglnilephevallysthrleuthrglylysthr
202530
ilethrleugluvalgluproseraspthrilegluasnvallysala
354045
lysileglnasplysgluglyileproproaspglnglnargleuile
505560
phealaglylysglnleugluaspglyargthrleuserasptyrasn
65707580
ileglnlysgluserthrleuhisleuvalleuargleuargglygly
859095
ileargaspasppheleuargleuproserglnalaserlysphephe
100105110
glnalaaspaspasnvalgluglythrargtrpalavalleuvalala
115120125
glyserlysglytyrvalasntyrarghisglnalaaspvalcyshis
130135140
alatyrglnileleulyslysglyglyleulysaspgluasnileile
145150155160
valphemettyraspaspilealatyrasngluserasnprohispro
165170175
glyvalileileasnhisprotyrglyseraspvaltyrlysglyval
180185190
prolysasptyrvalglygluaspileasnproproasnphetyrala
195200205
valleuleualaasnlysseralaleuthrglythrglyserglylys
210215220
valleuaspserglyproasnasphisvalpheiletyrtyrthrasp
225230235240
hisglyglyalaglyvalleuglymetproserlysprotyrileala
245250255
alaseraspleuasnaspvalleulyslyslyshisalaserglythr
260265270
tyrlysserilevalphetyrvalglusercysgluserglysermet
275280285
pheaspglyleuleuprogluasphisasniletyrvalmetglyala
290295300
seraspthrglyglusersertrpvalthrtyrcysproleuglnhis
305310315320
proserproproproglutyraspvalcysvalglyaspleupheser
325330335
valalatrpleugluaspcysaspvalhisasnleuglnthrgluthr
340345350
pheglnglnglntyrgluvalvallysasnlysthrilevalalaleu
355360365
ilegluaspglythrhisvalvalglntyrglyaspvalglyleuser
370375380
lysglnthrleuphevaltyrmetglythraspproalaasnaspasn
385390395400
asnthrphethrasplysasnserleuglythrproarglysalaval
405410415
serglnargaspalaaspleuilehistyrtrpglulystyrargarg
420425430
alaprogluglyserserarglysalaglualalyslysglnleuarg
435440445
gluvalmetalahisargmethisileaspasnservallyshisile
450455460
glylysleuleupheglyileglulysglyhislysmetleuasnasn
465470475480
valargproalaglyleuprovalvalaspasptrpaspcysphelys
485490495
thrleuileargthrphegluthrhiscysglyserleuserglutyr
500505510
glymetlyshismetargserphealaasnleucysasnalaglyile
515520525
arglysgluglnmetalaglualaseralaglnalacysvalserile
530535540
proaspasnprotrpserserleuhisalaglypheserval
545550555
<210>6
<211>566
<212>prt
<213>人工序列(artificialsequence)
<400>6
methishishishishishismetseraspsergluvalasnglnglu
151015
alalysprogluvallysprogluvallysprogluthrhisileasn
202530
leulysvalseraspglysersergluilephephelysilelyslys
354045
thrthrproleuargargleumetglualaphealalysargglngly
505560
lysglumetaspserleuargpheleutyraspglyileargilegln
65707580
alaaspglnthrprogluaspleuaspmetgluaspasnaspileile
859095
glualahisarggluglnileglyileargaspasppheleuargleu
100105110
proserglnalaserlysphepheglnalaaspaspasnvalglugly
115120125
thrargtrpalavalleuvalalaglyserlysglytyrvalasntyr
130135140
arghisglnalaaspvalcyshisalatyrglnileleulyslysgly
145150155160
glyleulysaspgluasnileilevalphemettyraspaspileala
165170175
tyrasngluserasnprohisproglyvalileileasnhisprotyr
180185190
glyseraspvaltyrlysglyvalprolysasptyrvalglygluasp
195200205
ileasnproproasnphetyralavalleuleualaasnlysserala
210215220
leuthrglythrglyserglylysvalleuaspserglyproasnasp
225230235240
hisvalpheiletyrtyrthrasphisglyglyalaglyvalleugly
245250255
metproserlysprotyrilealaalaseraspleuasnaspvalleu
260265270
lyslyslyshisalaserglythrtyrlysserilevalphetyrval
275280285
glusercysgluserglysermetpheaspglyleuleuprogluasp
290295300
hisasniletyrvalmetglyalaseraspthrglyglusersertrp
305310315320
valthrtyrcysproleuglnhisproserproproproglutyrasp
325330335
valcysvalglyaspleupheservalalatrpleugluaspcysasp
340345350
valhisasnleuglnthrgluthrpheglnglnglntyrgluvalval
355360365
lysasnlysthrilevalalaleuilegluaspglythrhisvalval
370375380
glntyrglyaspvalglyleuserlysglnthrleuphevaltyrmet
385390395400
glythraspproalaasnaspasnasnthrphethrasplysasnser
405410415
leuglythrproarglysalavalserglnargaspalaaspleuile
420425430
histyrtrpglulystyrargargalaprogluglyserserarglys
435440445
alaglualalyslysglnleuarggluvalmetalahisargmethis
450455460
ileaspasnservallyshisileglylysleuleupheglyileglu
465470475480
lysglyhislysmetleuasnasnvalargproalaglyleuproval
485490495
valaspasptrpaspcysphelysthrleuileargthrphegluthr
500505510
hiscysglyserleuserglutyrglymetlyshismetargserphe
515520525
alaasnleucysasnalaglyilearglysgluglnmetalagluala
530535540
seralaglnalacysvalserileproaspasnprotrpserserleu
545550555560
hisalaglypheserval
565
<210>7
<211>853
<212>prt
<213>人工序列(artificialsequence)
<400>7
metlysileglugluglylysleuvaliletrpileasnglyasplys
151015
glytyrasnglyleualagluvalglylyslyspheglulysaspthr
202530
glyilelysvalthrvalgluhisproasplysleugluglulysphe
354045
proglnvalalaalathrglyaspglyproaspileilephetrpala
505560
hisaspargpheglyglytyralaglnserglyleuleualagluile
65707580
thrproasplysalapheglnasplysleutyrprophethrtrpasp
859095
alavalargtyrasnglylysleuilealatyrproilealavalglu
100105110
alaleuserleuiletyrasnlysaspleuleuproasnproprolys
115120125
thrtrpglugluileproalaleuasplysgluleulysalalysgly
130135140
lysseralaleumetpheasnleuglngluprotyrphethrtrppro
145150155160
leuilealaalaaspglyglytyralaphelystyrgluasnglylys
165170175
tyraspilelysaspvalglyvalaspasnalaglyalalysalagly
180185190
leuthrpheleuvalaspleuilelysasnlyshismetasnalaasp
195200205
thrasptyrserilealaglualaalapheasnlysglygluthrala
210215220
metthrileasnglyprotrpalatrpserasnileaspthrserlys
225230235240
valasntyrglyvalthrvalleuprothrphelysglyglnproser
245250255
lysprophevalglyvalleuseralaglyileasnalaalaserpro
260265270
asnlysgluleualalysglupheleugluasntyrleuleuthrasp
275280285
gluglyleuglualavalasnlysasplysproleuglyalavalala
290295300
leulyssertyrgluglugluleuvallysaspproargilealaala
305310315320
thrmetgluasnalaglnlysglygluilemetproasnileprogln
325330335
metseralaphetrptyralavalargthralavalileasnalaala
340345350
serglyargglnthrvalaspglualaleulysaspalaglnthrasn
355360365
serserserasnasnasnasnasnasnasnasnasnasnleuglyile
370375380
gluglyargileserhismetileargaspasppheleuargleupro
385390395400
serglnalaserlysphepheglnalaaspaspasnvalgluglythr
405410415
argtrpalavalleuvalalaglyserlysglytyrvalasntyrarg
420425430
hisglnalaaspvalcyshisalatyrglnileleulyslysglygly
435440445
leulysaspgluasnileilevalphemettyraspaspilealatyr
450455460
asngluserasnprohisproglyvalileileasnhisprotyrgly
465470475480
seraspvaltyrlysglyvalprolysasptyrvalglygluaspile
485490495
asnproproasnphetyralavalleuleualaasnlysseralaleu
500505510
thrglythrglyserglylysvalleuaspserglyproasnasphis
515520525
valpheiletyrtyrthrasphisglyglyalaglyvalleuglymet
530535540
proserlysprotyrilealaalaseraspleuasnaspvalleulys
545550555560
lyslyshisalaserglythrtyrlysserilevalphetyrvalglu
565570575
sercysgluserglysermetpheaspglyleuleuprogluasphis
580585590
asniletyrvalmetglyalaseraspthrglyglusersertrpval
595600605
thrtyrcysproleuglnhisproserproproproglutyraspval
610615620
cysvalglyaspleupheservalalatrpleugluaspcysaspval
625630635640
hisasnleuglnthrgluthrpheglnglnglntyrgluvalvallys
645650655
asnlysthrilevalalaleuilegluaspglythrhisvalvalgln
660665670
tyrglyaspvalglyleuserlysglnthrleuphevaltyrmetgly
675680685
thraspproalaasnaspasnasnthrphethrasplysasnserleu
690695700
glythrproarglysalavalserglnargaspalaaspleuilehis
705710715720
tyrtrpglulystyrargargalaprogluglyserserarglysala
725730735
glualaglugluglnleuarggluvalmetalahisargmethisile
740745750
aspasnservallyshisileglylysleuleupheglyileglulys
755760765
glyhislysmetleuasnasnvalargproalaglyleuprovalval
770775780
aspasptrpaspcysphelysthrleuileargthrphegluthrhis
785790795800
cysglyserleuserglutyrglymetlyshismetargserpheala
805810815
asnleucysasnalaglyilearglysgluglnmetalaglualaser
820825830
alaglnalacysvalserileproaspasnprotrpserserleuhis
835840845
alaglypheserval
850
<210>8
<211>69
<212>dna
<213>人工序列(artificialsequence)
<400>8
gtcgacaagcttgcggccgcactcgagcaccaccaccaccaccactgagatccggctgct60
aacggatcc69
<210>9
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>9
acgccatatgattcgtgatgattttctgcg30
<210>10
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>10
acgcgtcgactcacacgctaaaccccg27
<210>11
<211>31
<212>prt
<213>人工序列(artificialsequence)
<400>11
glyleuprovalcysglygluthrcysvalglyglythrcysasnthr
151015
proglycysthrcyssertrpprovalcysthrargasnhisval
202530
<210>12
<211>26
<212>prt
<213>人工序列(artificialsequence)
<400>12
glyilepheserasnmettyralaargthrproalaglytyrphearg
151015
glyproalaglytyralaalaasnhisval
2025
<210>13
<211>17
<212>prt
<213>人工序列(artificialsequence)
<400>13
glyleualalysarghishisglytyrlysarglysphehisasnhis
151015
val
<210>14
<211>8
<212>prt
<213>人工序列(artificialsequence)
<400>14
lysalaleuvalileasnhisval
15
<210>15
<211>6
<212>prt
<213>人工序列(artificialsequence)
<400>15
glyileglyglyilearg
15
<210>16
<211>12
<212>prt
<213>人工序列(artificialsequence)
<400>16
lysalaleuvalileasnglyileglyglyilearg
1510
<210>17
<211>16
<212>prt
<213>人工序列(artificialsequence)
<400>17
glyargcysthrlysserileproproilecyspheproasnhisval
151015
- 还没有人留言评论。精彩留言会获得点赞!
- 一种dna文库及构建转录激活子样效应因子核酸酶质粒的方法
- 人转录共激活因子pc4在制备诊断、治疗或预防肿瘤的制剂中的应用的制作方法
- 一种转录激活子样效应因子核酸酶的内源活性检测方法
- 一种“转录激活子样效应因子-功能基团-雌激素受体”功能蛋白及其应用的制作方法
- 一种多模块dna文库及转录激活子样效应因子核酸酶质粒的构建方法
- 用VP16替代Nanog转录激活域CD2的Nanog基因突变体、携带其的载体
- 一种新的多肽——人转录激活辅因子37.07和编码这种多肽的多核苷酸的制作方法
- 一对转录激活子样效应因子核酸酶l3和r1及其编码基因与应用的制作方法
- 一对转录激活子样效应因子核酸酶及其编码基因与应用的制作方法
- 一对转录激活子样效应因子核酸酶及其编码基因与应用的制作方法