一种利用古菌分子标记OTU300快速检测城市绿地土壤全氮含量的方法与流程
本发明属于生物
技术领域:
,涉及一种利用古菌分子标记otu300快速检测城市绿地土壤全氮含量的方法。
背景技术:
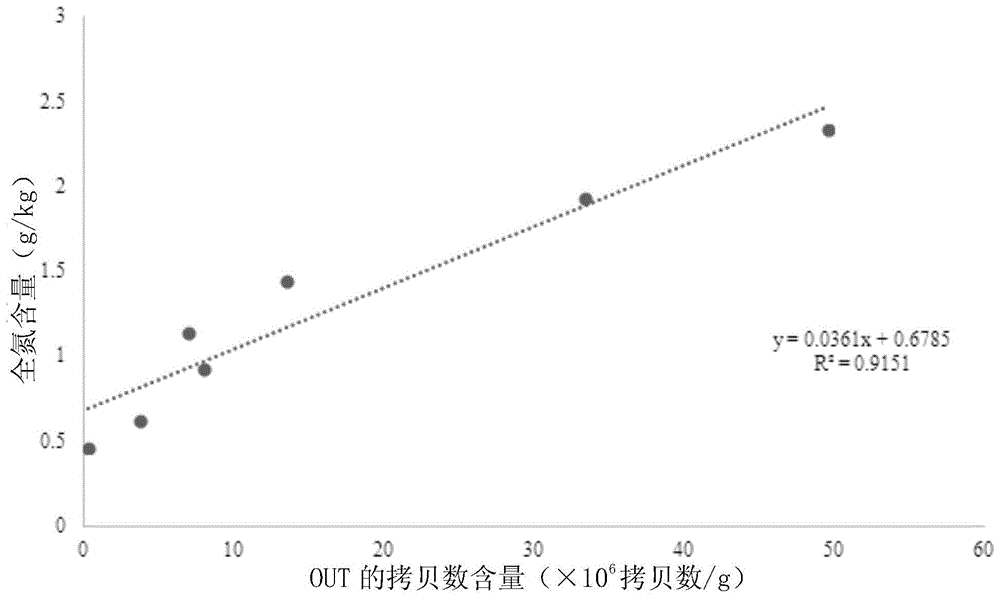
:城市园林绿化建设是生态文明建设和美丽中国建设的重要组成部分,对优化城市环境,提高人民生活质量,促进城市可持续发展起着重要作用。城市绿地土壤作为城市生态环境的物质基础,其质量直接影响着植物的健康生长以及生态效益、景观功能的发挥,在一定程度上决定着城市绿地质量。因此,对城市绿地土壤质量进行科学客观的监测和评价,是城市绿地管理的重要参考和依据。目前对城市绿地土壤质量指标的检测主要是沿用传统的理化检测方法,样品需求量大,样品前处理过程复杂、周期长,尤其所需要的人力资源成本较高,难以实现对大批量样品的快速检测。土壤微生物对土壤环境的变化具有综合性、敏感性和功能性的特点,探索通过检测特定微生物类群的丰度来实现对绿地土壤质量指标的快速和自动化检测技术具有重要的现实意义。土壤全氮是土壤肥力质量的重要指标,土壤全氮包括所有形式的有机态和无机态氮,是标志土壤氮素总量和供应植物有效氮素的源和库,能够综合反映土壤氮素状况、评估土壤供氮能力。土壤全氮受成土母质、地形、气候、生物等自然因素以及绿化养护等人为因素共同影响,其在土壤中的分布表现出显著的空间异质性。土壤全氮已成为世界各国土壤质量分析和实验室测定的例行项目。技术实现要素:本发明的目的是提供一种利用古菌分子标记otu300快速检测城市绿地土壤全氮含量的方法。本发明提供了引物探针组,由特异引物对和特异探针组成;所述特异引物对由序列表的序列4所示的引物524f-10-ext和序列表的序列5所示的引物arch958r-mod组成;所述特异探针的核苷酸序列如序列表的序列1所示。所述特异探针为taqman探针。所述taqman探针的5’末端具有荧光基团,3’末端具有荧光淬灭基团。所述荧光基团具体可为fam。所述荧光淬灭基团具体可为tamra。本发明还保护所述引物探针组在检测或辅助检测土壤全氮含量中的应用。本发明还保护所述引物探针组在比较不同地块的土壤全氮含量中的应用。本发明还保护一种试剂盒,包括所述引物探针组。所述试剂盒的功能为如下(a)或(b):(a)检测或辅助检测土壤全氮含量;(b)比较不同地块的土壤全氮含量所述试剂盒还包括记载有如下所述方法甲或方法乙的载体。本发明还提供了一种dna分子(探针),如序列表的序列1所示。本发明具体提供了一种taqman探针,其核苷酸序列如序列表的序列1所示。所述taqman探针的5’末端具有荧光基团,3’末端具有荧光淬灭基团。所述荧光基团具体可为fam。所述荧光淬灭基团具体可为tamra。本发明还保护所述dna分子或所述taqman探针在检测或辅助检测土壤全氮含量中的应用。本发明还保护所述dna分子或所述taqman探针在比较不同地块的土壤全氮含量中的应用。本发明还保护一种检测土壤全氮含量的方法(方法甲),包括如下步骤:以土壤样品的总dna为模板,进行实时荧光定量pcr;实时荧光定量pcr的扩增引物对由序列表的序列4所示的引物524f-10-ext和序列表的序列5所示的引物arch958r-mod组成;实时荧光定量pcr的探针的核苷酸序列如序列表的序列1所示;实时荧光定量pcr获得ct值;根据ct值计算拷贝数,通过土壤样品中的拷贝数含量计算得到土壤样品中的全氮含量。实时荧光定量pcr的探针为taqman探针。所述taqman探针的5’末端具有荧光基团,3’末端具有荧光淬灭基团。所述荧光基团具体可为fam。所述荧光淬灭基团具体可为tamra。所述拷贝数为所述探针的靶标片段的拷贝数。通过土壤样品中的拷贝数含量计算得到土壤样品中的全氮含量的实现方法为:将土壤样品的拷贝数含量代入线性方程,得到土壤样品的全氮含量。线性方程为:y=0.0361x+0.6785;r2=0.9151;y代表全氮含量,单位为g/kg;x代表目标out的拷贝数含量,单位为×106拷贝数/g。根据ct值计算拷贝数的方法为:将ct值代入标准曲线方程,得到拷贝数。所述标准曲线方程的制备方法为:将序列表的序列2所示的dna分子与pmd18-t载体连接,得到otu300标准品质粒;采用otu300标准品质粒制作以拷贝数的对数为自变量且以ct值为因变量的标准曲线方程。拷贝数的对数为拷贝数以10为底的对数。土壤样品的总dna的制备方法:采用mobio强力dna提取试剂盒(mobiolaboratories,carlsbad,inc.,ca,usa)提取土壤样品的总dna。实时荧光定量pcr具体采用96实时荧光定量pcr仪。实时荧光定量pcr的反应体系(20μl):10μlpremixextaq(takara,dalian,china)、0.4μl引物524f-10-ext、0.4μl引物arch958r-mod、0.2μl探针、2μl模板溶液和7μl无菌水。反应体系中,引物524f-10-ext的浓度为0.2μm,引物arch958r-mod的浓度为0.2μm,探针的浓度为0.1μm。反应体系中,模板dna的含量为7ng。实时荧光定量pcr的反应程序:95℃预变性120s;95℃变性10s、60℃退火延伸45s,45个循环。本发明还保护一种比较不同地块的土壤全氮含量的方法(方法乙),包括如下步骤:将两个以上地块的土壤样品分别按照方法甲进行检测;根据检测结果比较各个地块的土壤全氮含量。以上任一所述土壤为绿地土壤。以上任一所述地块为绿地地块。以上任一所述土壤为城市绿地土壤。以上任一所述地块为城市绿地地块。以上任一所述土壤为长江三角洲的绿地土壤。以上任一所述地块为长江三角洲的绿地地块。以上任一所述土壤为长江三角洲的城市绿地土壤。以上任一所述地块为长江三角洲的城市绿地地块。以上任一所述土壤为公园绿地土壤。以上任一所述地块为公园绿地地块。以上任一所述土壤为城市公园绿地土壤。以上任一所述地块为城市公园绿地地块。以上任一所述土壤为长江三角洲的公园绿地土壤。以上任一所述地块为长江三角洲的公园绿地地块。以上任一所述土壤为长江三角洲的城市公园绿地土壤。以上任一所述地块为长江三角洲的城市公园绿地地块。长江三角洲为上海市、江苏省和浙江省。以上任一所述土壤样品取自0-20cm表层土壤。采用本发明提供的方法检测土壤全氮含量或比较不同地块的土壤全氮含量,具有如下优点:样品需求量小,无需前处理,所需时间短,人力成本低,可以实现对大批量样品的快速自动化检测。本发明对于土壤样品的测评具有重要应用推广价值。附图说明图1为目标otu的拷贝数含量与土壤全氮含量的线性关系。具体实施方式以下的实施例便于更好地理解本发明,但并不限定本发明。下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的试验材料,如无特殊说明,均为自常规生化试剂商店购买得到的。以下实施例中的定量试验,均设置三次重复实验,结果取平均值。土壤质量指标的测定依据相关国家标准、行业标准和地方标准进行,详见表1。表1土壤质量指标测定方法检测指标检测方法phly/t1239-1999森林土壤ph值的测定电导率ly/t1251-1999森林土壤水溶性盐分分析(电导法)有机质ny/t1121.6-2006土壤有机质的测定全氮ly/t1228-2015森林土壤氮的测定(凯氏定氮法)有效氮ly/t1228-2015森林土壤氮的测定(碱解-扩散法)全磷ly/t1232-2015森林土壤磷的测定(碱熔钼锑抗比色法)全钾ly/t1234-2015森林土壤钾的测定(碱熔法)有效磷db31/t661-2012附录fab-dtpa浸提/电感耦合等离子体质谱仪有效钾db31/t661-2012附录fab-dtpa浸提/电感耦合等离子体发射光谱仪有效硫db31/t661-2012附录fab-dtpa浸提/电感耦合等离子体质谱仪有效氯参考db31/t661-2012中附录e(水饱和浸提)交换性钠参考db31/t661-2012中附录e(水饱和浸提)全砷gb/t22105.2-2008土壤质量总汞、总砷、总铅的测定原子荧光法全铜全消解电感耦合等离子体质谱仪方法全锌全消解电感耦合等离子体质谱仪方法全铅全消解电感耦合等离子体质谱仪方法全铬全消解电感耦合等离子体质谱仪方法全镍同上全消解电感耦合等离子体质谱仪方法有效钙参照db31/t661-2012附录fab-dtpa浸提/电感耦合等离子体发射光谱仪有效锰db31/t661-2012附录fab-dtpa浸提/电感耦合等离子体发射光谱仪有效锌db31/t661-2012附录fab-dtpa浸提/电感耦合等离子体发射光谱仪全汞usepa7473-2007热分解齐化原子吸收光度法注:与所参照的方法相比,有效钙检测的区别点仅在于检测目标物为有效钙。实施例1、发现与土壤全氮含量相关的out一、土壤样品的采集2017年11月,在上海市16个行政区的代表性公园绿地设置研究样地。采样方法:土壤样品采集遵循多点混合的采样原则,每块样地选择8个取样点,利用直径2.5cm土钻分别采集0-20cm表层土壤,之后混合成1个土壤样品。共采集土壤样品76个。土壤样品混合均匀,过2mm筛,去除植物根系、碎石等杂物。然后将每个土壤样品分为两份。一份样品自然风干,然后取样用于步骤二中测定土壤化学性质;另一份样品于-80℃保存,然后取样用于步骤三中土壤总dna提取。二、土壤质量指标的分析测定步骤一中自然风干的土壤样品,进行土壤质量指标的分析测定。土壤质量指标的测定结果见表2。表2土壤质量指标检测结果检测指标最小值最大值平均值ph5.328.797.92电导率(μs/cm)60.70656.52142.57有机质(g/kg)7.1046.6227.59全氮(g/kg)0.472.341.13有效氮(mg/kg)25.85152.6484.68全磷(g/kg)0.421.000.68全钾(g/kg)15.8025.4319.08有效磷(mg/kg)0.8548.208.66有效钾(mg/kg)28.20397.89188.90有效硫(mg/kg)13.0696.6552.36有效氯(mg/l)4.161900.0041.46交换性钠(mg/l)3.30993.0021.60全砷(mg/kg)4.8113.508.73全铜(mg/kg)16.3899.8436.05全锌(mg/kg)87.38223.87125.52全铅(mg/kg)18.2452.1829.01全铬(mg/kg)55.40101.0072.90全镍(mg/kg)27.4044.5936.62有效钙(mg/kg)198.36366.95282.89有效锰(mg/kg)8.5529.4616.76有效锌(mg/kg)1.8129.589.05全汞(mg/kg)0.050.570.21三、土壤古菌种群多样性分析1、步骤一中-80℃保存的土壤样品,提取总dna。提取土壤样品的总dna,采用mobio强力dna提取试剂盒(mobiolaboratories,carlsbad,inc.,ca,usa)。每个土壤样品重复提取两次,将两次提取的总dna混合,得到dna样本。76个土壤样品,得到相应的76个dna样本。所有dna样本均-80℃保存。利用nanodrop2000超微量分光光度计和0.8%琼脂糖凝胶电泳(5vcm-1,45min)检测dna质量。76个dna样本od260/od280均在1.8-2.0范围之间,od260/od280最大值为1.98,od260/od280最小值为1.81。2、古菌16srrna基因扩增及高通量测序以dna样本为模板,采用引物524f-10-ext和引物arch958r-mod组成的引物对进行pcr扩增。完成pcr扩增后,将pcr产物进行2%琼脂糖凝胶电泳,然后利用genejet凝胶回收试剂盒(thermoscientific)对目的条带进行切胶纯化,然后构建测序文库,采用illuminamiseq测序平台(illumina,sandiego,ca,usa)进行测序。引物524f-10-ext和引物arch958r-mod为古菌通用引物,靶序列位于古菌16srrna基因v4-v5可变区。524f-10-ext(序列表的序列4):5'-tgycagccgccgcggtaa-3';arch958r-mod(序列表的序列5):5'-yccggcgttgavtccaatt-3';y代表c或t;v代表g、a或c。pcr扩增的反应体系为30μl。有效成分为15μlhigh-fidelitypcrmastermix(newenglandbiolabs)、引物和模板dna。反应体系中,引物524f-10-ext和引物arch958r-mod的浓度均为0.2μm。反应体系中,模板dna的含量为10ng。pcr扩增的反应程序:95℃预变性3min;95℃变性30s、55℃退火30s、72℃延伸45s,33个循环;72℃延伸5min。3、高通量数据分析及结果高通量测序结果生物信息学分析具体步骤如下:1)根据样品特异标签从原始数据中对同一样品序列进行抽提,形成单独的文件,并去除标签及引物序列;2)利用flash(v1.2.7)软件进行序列拼接;3)利用qiime(v1.7.0)软件对测序完成后的原始序列进行质量过滤;4)利用uchime软件检测嵌合体,并将之删除;5)利用uparse(v7.0.1001)软件在95%的相似性水平上划分操作分类单元(otus);6)由于单拷贝序列的可靠性受到质疑,因此在后续分析中去除单拷贝序列;7)为了去除样品间不同测序深度的影响,样品otu表格均一化到相同的测序深度;8)基于rdp数据库对otus进行序列比对,并确定其分类地位。每个样本选取27765条古菌16srrna基因序列用于后续分析。在95%的序列相似度下对古菌16srrna基因序列进行归类,共获得580个otus。四、土壤古菌类群与土壤质量指标的相关性分析将步骤三得到的580个otus分别与步骤二得到的各个土壤化学指标数据进行pearson相关性分析。结果表明:580个古菌otus中,土壤中古菌otu300丰度与土壤全氮含量的相关性最强,土壤中古菌otu69丰度与土壤全氮含量的相关性排第二;土壤中古菌otu300丰度与土壤全氮含量呈显著正相关,相关系数为r为0.486;土壤中古菌otu69丰度与土壤全氮含量的相关系数为-0.293。可利用检测土壤中古菌otu300或古菌otu69的丰度来反映土壤全氮的含量。实施例2、建立otu与土壤化学特征间的线性关系一、古菌marker基因探针设计根据测序结果,设计用于检测otu300的探针。probe300-1探针:5'-aggatactacagagctagga-3'。probe300-2探针(序列表的序列1):5'-ttggcttcgcgctaacccagt-3'。probe300-1探针为taqman探针,5’末端具有荧光基团fam,3’末端具有荧光淬灭基团tamra。probe300-2探针为taqman探针,5’末端具有荧光基团fam,3’末端具有荧光淬灭基团tamra。根据测序结果,设计用于检测otu69的探针。probe69-1探针:5'-acccgctcaacggttgggct-3'。probe69-2探针:5'-tgatgggatggcctcgagct-3'。probe69-1探针为taqman探针,5’末端具有荧光基团fam,3’末端具有荧光淬灭基团tamra。probe69-2探针为taqman探针,5’末端具有荧光基团fam,3’末端具有荧光淬灭基团tamra。二、土壤中古菌marker基因拷贝数的测定根据实测值,从实施例1的步骤一的土壤样品中随机选择全氮含量呈梯度分布的土壤样品。1、取土壤样品,提取总dna。总dna的提取方法同实施例1的步骤三的1。2、取模板溶液(即步骤1得到的总dna溶液),利用96实时荧光定量pcr仪进行荧光定量pcr(探针法)。采用引物524f-10-ext和引物arch958r-mod组成的引物对。采用probe300-1探针或probe300-2探针或probe69-1探针或probe69-2探针。反应体系(20μl):10μlpremixextaq(takara,dalian,china)、0.4μl引物524f-10-ext、0.4μl引物arch958r-mod、0.2μl探针、2μl模板溶液和7μl无菌水。反应体系中,引物524f-10-ext的浓度为0.2μm,引物arch958r-mod的浓度为0.2μm,探针的浓度为0.1μm。反应体系中,模板dna的含量为7ng。反应程序:95℃预变性120s;95℃变性10s、60℃退火延伸45s,45个循环。通过溶解曲线确定扩增特异性。将ct值代入标准曲线方程,得到目标otu的拷贝数,再计算得到土壤样品中目标otu的拷贝数含量(单位为拷贝数/g,即每g干重的土壤样品中目标otu的拷贝数)。标准曲线方程的制作方法见步骤3。3、制作荧光定量pcr标准曲线将序列表的序列2所示的dna分子(从土壤总dna中扩增得到的)与pmd18-t载体连接,得到otu300标准品质粒。采用te缓冲液作为溶剂,制备含有不同浓度otu300标准品质粒的标准品质粒溶液(通过nanodrop2000超微量分光光度计测定标准品质粒溶液中的dna浓度,换算dna拷贝数,即为otu300拷贝数);将各个标准品质粒溶液分别作为模板溶液,按照步骤2的方法进行检测(采用probe300-1探针或probe300-2探针),获得以otu300拷贝数的对数(以10为底的对数)为自变量且以ct值为因变量的标准曲线方程。将序列表的序列3所示的dna分子(从土壤总dna中扩增得到的)与pmd18-t载体连接,得到otu69标准品质粒。采用te缓冲液作为溶剂,制备含有不同浓度otu69标准品质粒的标准品质粒溶液(通过nanodrop2000超微量分光光度计测定标准品质粒溶液中的dna浓度,换算dna拷贝数,即为otu69拷贝数);将各个标准品质粒溶液分别作为模板溶液,按照步骤2的方法进行检测(采用probe69-1探针或probe69-2探针),获得以otu69拷贝数的对数(以10为底的对数)为自变量且以ct值为因变量的标准曲线方程。荧光定量pcr扩增有效性在90%和110%之间。三、古菌marker基因拷贝数与土壤化学特征间线性关系的建立土壤样品的全氮实测值(单位为g/kg)、ct值和土壤样品目标otu的拷贝数含量(单位为拷贝数/g)的结果见表3。土壤样品的全氮实测值为实施例1的步骤二中获得的数据。ct值和土壤样品中目标otu的拷贝数含量为本实施例的步骤二获得的数据。利用probe300-2探针进行荧光定量pcr,土壤中目标otu相对丰度(体现为土壤中目标otu拷贝数)与土壤全氮含量具有良好的线性关系。表3注:土壤样品1,采集自奉贤区的公园绿地;土壤样品2,采集自黄浦区的公园绿地;土壤样品3,采集自虹口区的公园绿地;土壤样品4,采集自松江区的公园绿地;土壤样品5,采集自青浦区的公园绿地;土壤样品6,采集自徐汇区的公园绿地;土壤样品7,采集自宝山区的公园绿地。利用probe300-2探针进行荧光定量pcr,土壤中目标otu的拷贝数含量与土壤全氮含量的线性关系见图1。线性方程为:y=0.0361x+0.6785;r2=0.9151;y代表全氮含量(g/kg),x代表目标out的拷贝数含量(×106拷贝数/g)。实施例3、未知城市绿地土壤样品全氮含量的分子检测在上海植物园、南京玄武湖公园、苏州吴江公园内分别随机选择3块绿地。采样方法:土壤样品采集遵循多点混合的采样原则,每块样地选择8个取样点,利用直径2.5cm土钻分别采集0-20cm表层土壤,之后混合成1个土壤样品。土壤样品混合均匀,过2mm筛,去除植物根系、碎石等杂物。然后将每个土壤样品分为两份。一份样品自然风干,然后取样检测全氮含量,获得全氮含量实测值(单位为g/kg);另一份样品于-80℃保存,然后取样用于土壤总dna提取。1、提取土壤样品的总dna。采用mobio强力dna提取试剂盒(mobiolaboratories,carlsbad,inc.,ca,usa)。每个土壤样品重复提取两次,将两次提取的总dna混合,得到dna样本。2、取模板溶液(即步骤1得到的dna样本),利用96实时荧光定量pcr仪进行荧光定量pcr(探针法)。反应体系(20μl):10μlpremixextaq(takara,dalian,china)、0.4μl引物524f-10-ext、0.4μl引物arch958r-mod、0.2μlprobe300-2探针、2μl模板溶液和7μl无菌水。反应体系中,引物524f-10-ext的浓度为0.2μm,引物arch958r-mod的浓度为0.2μm,probe300-2探针的浓度为0.1μm。反应体系中,模板dna的含量为7ng。反应程序:95℃预变性120s;95℃变性10s、60℃退火延伸45s,45个循环。将ct值代入标准曲线方程,得到otu300的拷贝数,再计算得到土壤样品中otu300的拷贝数含量(单位为拷贝数/g,即每g干重的土壤样品中otu300的拷贝数)。标准曲线方程的制作方法见实施例2的步骤二的3。3、将土壤样品中otu300的拷贝数含量代入线性方程,得到土壤样品的全氮含量的计算值(单位为g/kg)。线性方程为:y=0.0361x+0.6785;r2=0.9151;y代表全氮含量(g/kg),x代表otu300的拷贝数含量(×106拷贝数/g)。土壤样品中otu300的拷贝数含量(单位为拷贝数/g)、土壤样品的全氮含量的计算值(单位为g/kg)、土壤样品的全氮含量的实测值(单位为g/kg)的结果见表4。表4sequencelisting<110>上海市园林科学规划研究院<120>一种利用古菌分子标记otu300快速检测城市绿地土壤全氮含量的方法<130>gncyx200032<160>5<170>patentinversion3.5<210>1<211>21<212>dna<213>artificialsequence<400>1ttggcttcgcgctaacccagt21<210>2<211>466<212>dna<213>artificialsequence<400>2tgtcagccgccgcggtaataccagcaccccgagtggtcgggacgattattgggcctaaag60catccgtagccggttctgcaagtctcccgttaaatccaactgctcaacaggtgggctgcg120gaggatactacagagctaggaggcaggagaggcaagcggtactcagtgggtaggggtaaa180atccgttgatccattgaagaccaccagtggcgaaggcggcttgccagaatgcgctcgacg240gtgagggatgaaagctgggggagcaaaccggattagatacccgggtagtcccagccgtaa300acgatgcagactcggtgatgggttggcttcgcgctaacccagtgccgcagggaagccgtt360aagtctgccgcctgggtagtacggtcgcaagactgaaacttaaaggaattggcgggggag420caccacaaggggtgaagcctgcggttcaattggattcaacgccgga466<210>3<211>466<212>dna<213>artificialsequence<400>3tgtcagccgccgcggtaataccagcaccccgagtggtcgggacgattattgggcctaaag60catccgtagccggtcatgcaagtcttccgttaaatccacccgctcaacggttgggctgcg120gaggatactacgtggctaggaggcgggagaggcaagcggtactcagtgggtaggggtaaa180atcctttgatccattgaagaccaccagtggcgaaggcggcttgccagaacgcgctcgacg240gtgagggatgaaagctgggggagcaaaccggattagatacccgggtagtcccagctgtaa300acgatgcagactcggtgatgggatggcctcgagctatcccagtgccgcagggaagccgtt360aagtctgccgcctggggagtacggtcgcaagactgaaacttaaaggaattggcgggggag420caccacaaggggtgaagcctgcggttcaattggattcaacgccgga466<210>4<211>18<212>dna<213>artificialsequence<400>4tgycagccgccgcggtaa18<210>5<211>19<212>dna<213>artificialsequence<400>5yccggcgttgavtccaatt19当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1