与脊尾白虾生长相关的SNP标记所在基因、检测引物及应用的制作方法
本发明属于甲壳动物遗传育种技术领域,具体涉及一种与脊尾白虾生长相关的snp标记所在基因、分型引物及应用。
背景技术:
脊尾白虾(exopalaemoncarinicauda)隶属于十足目,长臂虾科,白虾属,主要分布于中国大陆沿岸和朝鲜半岛西岸的浅海低盐水域,以渤海和黄海产量最大。脊尾白虾具有环境适应力强、繁殖周期短、繁殖季节长、抗病力强等特点,目前全国脊尾白虾养殖面积超过40万亩,年产量约5万吨,年产值超过30亿元,已发展成我国海水养殖特色经济虾类之一。随着养殖规模的扩大,目前养殖主要依靠捕捞野生亲虾放入池塘自然繁殖,存在个体间差异大、生长速度缓慢等问题,影响养殖效益。因此,对脊尾白虾生长性状进行遗传改良是促进脊尾白虾养殖业发展的重要举措。
生长性状是水产动物优良品种培育过程中重要的测试指标,也是直接的育种目标。甲壳动物的生长发育主要依赖于蜕皮过程中肌肉的重塑和生长速率。肌肉的生长发育是一个受多种基因和信号分子调控的复杂过程,肌肉生长抑制素(myostatin,mstn)基因是哺乳动物肌肉生长发育及分化的重要负调控基因,其基因突变或蛋白功能的缺失会促进肌细胞增殖和肌纤维增大。mstn基因的多态性与水产动物的生长性状存在显著相关关系,研究表明,中华绒螯蟹mstn基因第一个外显子的一个snp位点对其体重和壳长性状具有显著的影响。
分子标记辅助育种(molecularmarker-assistedbreeding,mas)是借助与性状紧密相关的分子标记对具有性状优势的等位基因或基因型的个体进行直接选择育种,是分子生物学和基因组学的研究结果应用到水产养殖品种选育的技术。开展dna水平的标记辅助育种是科学发展的必然,其提高了选择的准确性,降低了育种成本,加快遗传进展。但甲壳动物的分子标记辅助育种尚处于初级阶段,需要筛选获得更多与经济性状相关的重要snp标记并应用于分子标记辅助育种中。
技术实现要素:
本发明要解决的技术问题在于提供一种脊尾白虾生长相关的snp标记所在基因、分型引物及应用,即找到对脊尾白虾生长起重要调控作用的mstn基因上与脊尾白虾体长、体重性状显著相关的snp标记,并用于选育快速生长的脊尾白虾。
本发明是通过如下技术方案来实现的:
本发明首先提供一种与脊尾白虾生长相关的snp标记所在基因,所述snp标记位于mstn基因的外显子区域,所述基因的基因组序列为seqidno.1,所述基因序列的5’端第567个位点,其碱基为c或t,即为snp标记m567。
所述snp标记m567的cc基因型个体的体长、体重性状显著的大于tt基因型和ct基因型的个体(p<0.05),且该位点未改变氨基酸的构成。
本发明还提供一种用于检测上述snp标记m567的引物,包括正向引物:5'-atcaggttcaatgctccaca-3',反向引物:5'-gagcctctaccactaaaccaag-3'。
本发明利用所述引物在脊尾白虾快速生长品种选育中的应用,利用所述引物筛选具有snp标记m567cc基因型的脊尾白虾育种材料,其具体的操作方法如下:
(1)提取脊尾白虾待检测样品的基因组dna;
(2)以步骤(1)获得的基因组dna为模板,利用检测snp标记m567的引物进行pcr扩增,获得pcr产物;
(3)对步骤(2)获得pcr扩增产物进行测序,根据测序结果确定待测样品在m567标记的基因型,确定待测个体是否具有快速生长的潜力,即筛选m567标记为cc基因型的个体。
本发明与现有技术相比的有益效果:
本发明提供的脊尾白虾快速生长相关的snp标记m567不仅能够快速获得稳定的生长性状突出的脊尾白虾,而且能够不受脊尾白虾生长阶段、性别等限制,可以用于脊尾白虾的早期选育,降低亲本筛选的盲目性,节约了养殖成本,缩短了育种周期。
附图说明
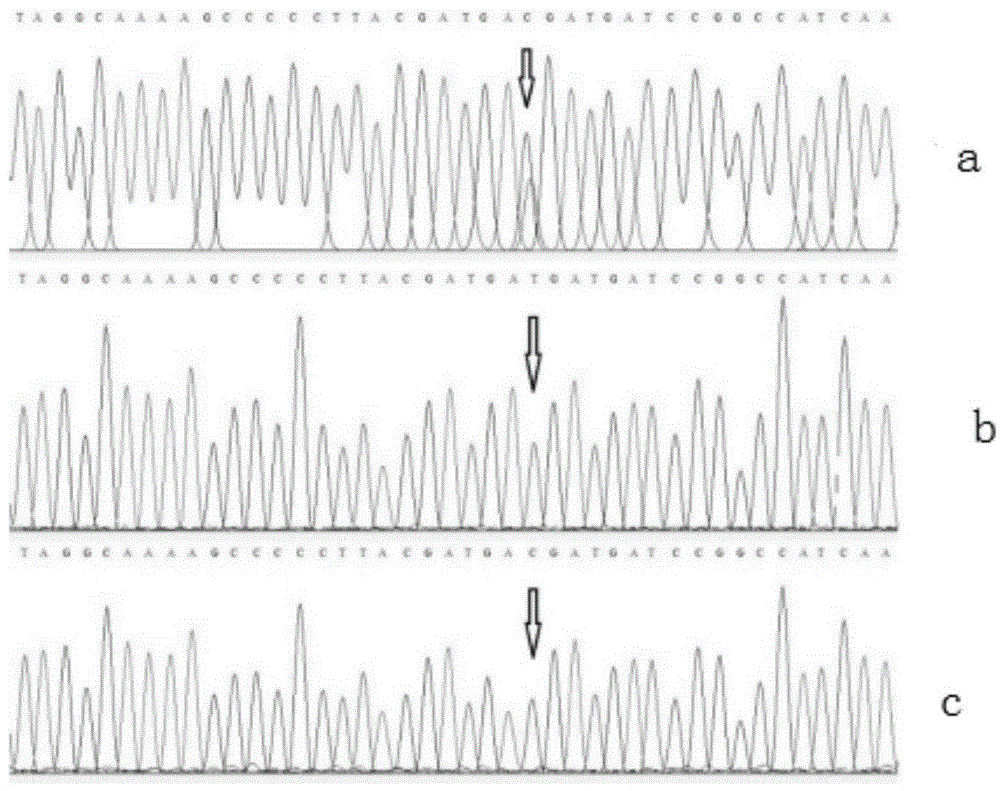
图1为m567标记扩增产物ct、tt、cc基因型在序列seqidno.1的567bp处的测序峰图:a为ct基因型,b为tt基因型,c为cc基因型。
具体实施方式
以下的实施例对本发明作进一步描述,是对本发明进行说明而非对其加以限定。下述实施例中的实验方法如无特殊说明,均为常规方法。下述实施例中所用的试验材料如无特殊说明,均为常规生化试剂商店可以购买得到的。
实施例1获得脊尾白虾mstn基因的基因组序列
具体步骤如下:
(1)脊尾白虾dna的提取。参照传统的酚氯仿的方法提取脊尾白虾肌肉组织的dna。取脊尾白虾肌肉组织0.1g放在2ml的离心管中,迅速加入裂解缓冲液,加入30μl蛋白酶k用于除去蛋白质,60℃温育1-2h,期间每隔20分钟上下颠倒混匀一次;待澄清后,冷却5分钟,加入等体积(0.8ml)的酚氯仿异戊醇(25:24:1),置于摇床上,低速混匀20分钟;室温12000g离心20分钟,吸取上清0.6ml,加入等体积的冷的异丙醇(-20℃),上下颠倒几次,出现白色絮状的沉淀;用95%的乙醇洗涤dna两次,待完全干燥后用1×te溶解。用1%琼脂糖胶检测dna的质量,测定浓度后,把dna稀释到50ng/μl,4℃或者-20℃保存备用。
(2)目的基因pcr扩增。根据脊尾白虾mstn基因cdna编码区的全长,在编码区的两端设计引物,获得mstn基因组dna全长,包括正向引物序列:5'-ttgttagcctttgccctcc-3',反向引物序列:5'-tggtcgtaatacagcatcttca-3'。
以脊尾白虾基因组dna为模板,设计引物进行pcr扩增。反应体系为:2μl基因组dna,正向引物和反向引物各1μl,2μlmg2+plus10×lapcrbufferii,1.6μl2.5mmdntpmixture,0.4μl5u/μltakaralataq和12μl无菌水。pcr反应程序如下:95℃30s;95℃40s,55℃,40s,72℃2min,35个循环;72℃10min。扩增产物使用1%琼脂糖凝胶电泳检测。
(3)目的片段的克隆及测序分析。
pcr产物使用天根琼脂糖凝胶dna回收试剂盒回收目的片段,与peasy-t1(北京全式金生物技术有限公司)克隆载体连接,转化至对应的trans1-t1感受态细胞中,经鉴定后挑选阳性克隆,送生工生物工程(上海)股份有限公司进行测序,获得mstn基因的基因组全长序列。经过与mstn基因的cdna序列进行比对,明确该基因包含3个外显子和2个内含子。
实施例2筛选snp标记及其遗传多样性分析
(1)snp标记的筛选
选择脊尾白虾大个体和小个体分别10尾,提取其基因组dna,等量混合。以混合dna为模板,正向引物:5'-ttgttagcctttgccctcc-3',反向引物:5'-tggtcgtaatacagcatcttca-3',进行pcr反应。挑选10个pcr产物送公司进行pcr产物直接测序,利用seqman软件进行序列的拼接和查找snp标记,初步筛查获得12个具有多态性的标记,其中m567标记位于mstn基因的第2个外显子区域,5’端的567bp处,见图1。
(2)m567标记的遗传样性分析
选择30个脊尾白虾野生群体,提取其基因组dna。以野生个体的基因组dna为模板,在m567标记附近设计引物检测其基因型,正向引物:5'-atcaggttcaatgctccaca-3',反向引物:5'-gagcctctaccactaaaccaag-3',进行pcr反应扩增。获得的pcr产物进行测序,识别seqidno.1序列中567bp处的基因型,并计算其遗传参数(表1)。利用popgene1.32计算观测杂合度和期望杂合度。多态信息含量(pic)根据botstein的方法计算。
实施例3脊尾白虾m567标记与生长性状的关联分析
所用脊尾白虾群体为日照海辰水产有限公司养殖场养殖的脊尾白虾家系,从中随机选出96个脊尾白虾个体,对每个个体的体长、体重性状进行测量,同时提取其基因组dna。利用正向引物:5'-atcaggttcaatgctccaca-3',反向引物:5'-gagcctctaccactaaaccaag-3'对每一个脊尾白虾个体进行pcr扩增和测序,获得每个个体在m567标记的基因型。利用spss22.0软件的广义线性模型glm进行m567标记与生长性状的相关性分析。结果表明,mstn基因中的m567标记与体长、体重性状存在显著相关,m567标记的cc基因型个体的体长、体重性状显著的大于tt基因型和ct基因型的个体(p<0.05)(表2)。
表1脊尾白虾mstn基因snp标记遗传多态性分析
表2mstn基因m567标记与生长性状的关联分析
序列表
<110>中国水产科学研究院黄海水产研究所
<120>与脊尾白虾生长相关的snp标记所在基因、检测引物及应用
<160>5
<170>siposequencelisting1.0
<210>1
<211>1851
<212>dna
<213>脊尾白虾(exopalaemoncarinicauda)
<400>1
atgctttggataaggtatatcttgttagcctttgccctcctgcagtcgctgacggaggca60
cagcgaagaaaaaccacggggaaacccggccgcaaaaggaaccaagtcgaaaggagacct120
cgctcagatggcccctcagaacacatggaggaaactgaagatattcctccacctcactgg180
gcaagtggaggcagtggtgggggggagagtggtgggggtggtggtgggggaggaatcggt240
ggaggaggaggaggaggaggaggtggtggtggaggtggaggaggaggaggaggaggtgga300
ggaggtggaggagagggaggagagcgtgcccatcggcatggccatcaggttcaatgctcc360
acatgtttcaacaatattttgcaaattcggagagagagacggatcgaagagattaaagat420
cgagtgcttacggccacgggcctcggaactccacccgatatgaccggggttgtgttgacg480
gaaaatccaaacatacaaaccattatacacgaaatggaaaagagtgcgccccctcattcc540
tttaggcaaaagcccccttacgatgacgatgatccggccatcaagagagaaaagatcttc600
tctccagtagaaccaggtaacatctacgcttcacgctcagctcctccgggtctccgaatt660
ccggcggacatgaacgccctctacttcaagctagacctcgggacgctcctcaccagggtc720
aagagagccatcctacacgtctggctcaaacccatgcggtccgaactcgacagacaagtt780
tcgatcactgtttacaaaatataccgcattaataacacggatttcctagataaaacggaa840
gtaacgacactacggaaggaattcgacgccttagaagggaactgggtcaagattcctgtg900
tacaagctcctgcaggagtggctcagtaaaccagatgaaaaccttggtttagtggtagag960
gctctagactcgcagggccgtcaagtggctgtgacagatcctacagagtctcctaaaaat1020
gtaagtggcctcaagaaaacatttggtctactatttagatgactaattatcaagtatttt1080
atattgggatctattatttttttttaattgaaataaatactatcccccaaatcaattatt1140
tttcattggactaaatactaaccatcttaatttttttcattgaaatatattttctattgg1200
attatatactatcttatcaattatttttcactgaaatctatttttcattggactctccat1260
cttataaattgggctatttttccttgttggagcccttgggtttatagaatctttcttttc1320
caactagggttgtagcttgactaataataataataatacttcccgcttcatagtcctcag1380
ccatgaagagttgggagacccagacctacatggccgaaatctatctaaatactaaaaatc1440
taaaaacccgtgccactcttccaggcccctctcctagaaatccacaccgaggaaaaccgc1500
cgggggcgcggccgtcgcaacagcggcagcgtcgactgcacgaacaacgacacggagaaa1560
tgctgtcgctacccgctctcggtgaacttcgtcgagatgggctgggacttcatcgtggcc1620
cccaaggtctacgacgccaacttctgcaacggcgagtgcccttacttgtacgcccaaaaa1680
tacgcccacagcgccctcgtccagaagatgaacagcagcagcgccaagcacggcccttgc1740
tgcggggcgaggaagctctctcccatgaagatgctgtattacgaccacgaccacaagatc1800
aaatttgacaccatccaagacatggtggtggatcgctgcggatgctcttaa1851
<210>2
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>2
atcaggttcaatgctccaca20
<210>3
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>3
gagcctctaccactaaaccaag22
<210>4
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>4
ttgttagcctttgccctcc19
<210>5
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>5
tggtcgtaatacagcatcttca22
- 还没有人留言评论。精彩留言会获得点赞!