陆地棉GhEBF1基因、蛋白及其应用
本发明涉及分子生物学技术领域,涉及一种陆地棉ghebf1基因、蛋白及其应用,特别涉及一种与陆地棉黄萎病抗性相关的ghebf1基因、蛋白及其应用。
背景技术:
棉花是重要的经济作物,在国民经济中占有重要地位,它不仅是纺织、化工、医药和国防工业的重要原料,而且是重要的出口创汇商品。其中,陆地棉(gossypium.hirsutum)是主要种植种类,占我国棉花面积的99%以上。黄萎病是棉花生产中最重要病害之一,1930年代传入我国,1950年代在我国生产棉省少数地区发生及为害,随着带菌棉种的调运,传播十分迅速,1990年代初我国主产棉区为害逐年加重。
引起黄萎病发生的主要病原物是大丽轮枝菌(verticilliumdahliae)。由于黄萎病是一类主要土传病害,其病原菌寄主范围广、易变异,没有专化寄生关系,在不良环境条件下,可以形成具有长期存活的休眠体微菌核等特点,防治极为困难,是我国棉花可持续发展的主要障碍之一。国内外的长期研究表明,综合防治是防治黄萎病最经济有效的措施,而选育抗病品种是最重要、最经济的、最生态的综合防治方法,但由于其抗病性是由多基因控制的数量性状,再加上其流行为害的复杂性,培育抗病品种的难度高,一直以来,我国缺乏抗病品种(品系),尤其是占我国种植面积99.9%的陆地棉的抗病品种更是奇缺,这也是导致该病时不时流行为害的重要原因。
病原相关的模式分子引发的免疫反应(pathogenassociatedmolecularpatterntriggeredimmunity,pti)在植物抵抗病原物的侵染过程中发挥着重要的作用,植物的识别受体(patternrecognitionreceptors,prrs)能识别病原物表面的相关模式分子(pathogenassociatedmolecularpatterns,pamps),促发pti。富含亮氨酸重复序列受体激(lrr-rks)类的prrs依赖lrr-rkbak1的调节来传递信号,bak1(bri1-associatedreceptorkinase1)还与lrr-rkbri1相互作用,bak1和bri1(brassinosteroidinsen-sitive1))是类固醇激素油菜素(brassinosteroid,br)的主要受体,能够促进br信号的传递,在植物的免疫机制中,外源病原体侵入的信号能被bak1接收,经过信号递级传递,br信号激活磷酸酶pp2a,使bzr1和bes1去磷酸化,与大量基因的启动子结合,从而调节这些基因的表达;br也是介导植物生长的正调节因子。
br是植物生活史中广泛的发育和生理过程所必需的激素,在植物生长发育及逆境应答中也发挥重要作用。在过去的大多数报道中,都是研究brs的生物合成或信号转导相关的基因,与植株矮化、延迟开花,衰老等一系列表型相关。有研究表明,bes1/bzr1是br信号转导途径的唯一转录因子。在病原物侵染植物的过程中,br也起着重要的作用,albrecht等研究发现,br信号可能通过调控富含亮氨酸重复序列的受体样激酶(lrr-rlk)bak1下游的免疫信号,在植物生长过程中发挥重要的免疫调节作用,是病原菌侵染过程中潜在的调控位点。生理研究还表明,brs促进细胞伸长,增强对环境胁迫的耐受性和对病原体侵染的抵抗力,从而提高作物产量。lozano-duranr等对拟南芥的研究发现,bzr1可诱导几种wrky转录因子(wrky15、wrky18、wrky11)和hbi1的表达,这些转录因子对早期免疫反应具有负面控制作用;此外,bzr1还与wrky40结合,介导br与免疫信号之间的拮抗作用,最终bzr1介导的转录改变将导致pti信号的抑制。
目前已公布的ebf基因,多来源于模式植物和草本植物,部分林木也克隆获得了ebf基因,如马尾松、杨树等。陆地棉作为我国主要的种植种,具有重要的经济价值,但目前尚无ebf同源基因的研究报道。
技术实现要素:
本发明的目的是提供一种陆地棉ghebf1基因、蛋白及其应用,以解决上述现有技术存在的问题,通过从陆地棉叶片中克隆获得与陆地棉抗病相关的ebf基因,通过vigs和在拟南芥中超表达证实了其在高抗黄萎病品种的叶片和根系中的协同性表达,依据转基因拟南芥抗黄萎病性评价,证实其提高了抗黄萎病性的能力;进一步通过将该基因转入优质高产感病棉花品种中,可以将感病品种改造成为高抗黄萎病优质高产棉花品种,甚至可以用于其他农作物的抗黄萎病性改造,这对于获取抗黄萎病的陆地棉或其他农作物新品种具有重要意义。
为实现上述目的,本发明提供了如下方案:
本发明提供一种基因,所述基因包括如下任一种所示的dna分子:
1)如seqidno:1所示的dna分子;
2)如seqidno:1所示序列自5’末端第270位核苷酸序列所示的dna分子;
3)与1)或2)限定的dna序列具有至少70%同源性且编码与植物黄萎病抗性相关蛋白的dna分子。
本发明还提供由所述的基因编码的蛋白。
优选的是,包括以下任一项所述蛋白质:
1)由如seqidno:2所示的氨基酸序列组成的蛋白质;
2)将如seqidno:2所示的氨基酸序列经过取代和/或缺失和/或添加一个或几个氨基酸残基且与植物黄萎病抗性相关的由seqidno:2衍生的蛋白质。
本发明还提供扩增所述基因全长或者任意片段的引物对,包括如seqidno:3和seqidno:4所示的核苷酸序列。
本发明还提供所述的基因或所述的蛋白的应用,包括1)或2):
1)促进植物叶片和根系协同表达;
2)提高植物抗黄萎病能力。
优选的是,所述植物为陆地棉或拟南芥。
本发明还提供一种培育转基因植物的方法,包括将所述的基因导入目的植物中获取转基因植物的步骤,所述转基因植物具有如下1)或2)中表型:
1)所述转基因植物的叶片和根系协同表达;
2)所述转基因植物黄萎病发病率和病情指数降低。
优选的是,所述植物为陆地棉或拟南芥。
本发明公开了以下技术效果:
本发明提供了一种陆地棉与黄萎病抗性相关基因ghebf1及其编码的蛋白,通过从棉花叶片中克隆获得与陆地棉抗黄萎病相关的ghebf1基因,通过rt-pcr证实了其在抗黄萎病棉花品种叶片和根系中的协同性表达,依据转基因拟南芥抗黄萎病性评价,证实其提高了抗黄萎病性的能力,沉默该基因则可以使高抗病棉花品种的抗黄萎病能力直线下降。因此,如果将本发明提供的基因转入陆地棉或其他农作物,则可以提高优质高产感病棉花或其他农作物品种的抗黄萎病性的遗传改良,这对于获取具有优良高抗黄萎病陆地棉或其他农作物新品种具有重要意义。
附图说明
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动性的前提下,还可以根据这些附图获得其他的附图。
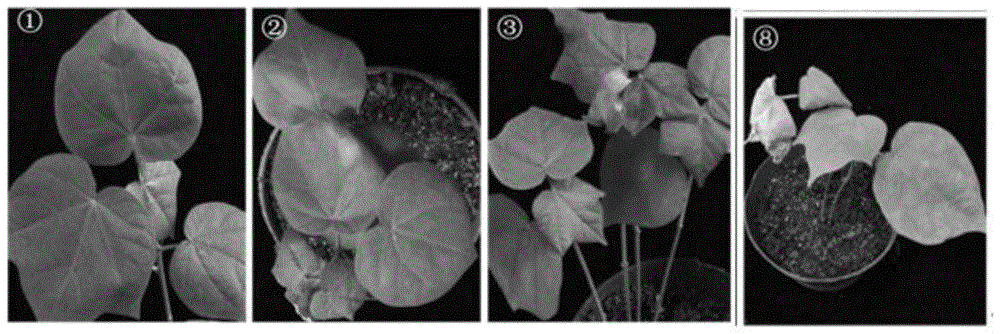
图1为实施例2通过vigs技术沉默抗黄萎病陆地棉品种kv-3的ghebf1基因,各沉默植株接种大丽轮枝菌v9913周后发病情况;其中,①:未接菌野生型中植棉kv-3;②:接菌野生型中植棉kv-3;③:转化空载体(clcrv-00)中植棉kv-3接菌v991;⑧:沉默抗病品种中植棉kv3的ghebf1基因接菌v991;
图2为ghebf1的全长序列(包含启动子和终止子以及seqidno:1所示序列)。
具体实施方式
现以实施例方式对本发明技术方案进行具体说明,但其不应该被认为是对本发明的限制,而应理解为是对本发明的某些方面、特性和实施方案的更详细的描述。
下述实施例中所使用的试验方法如无特殊说明,均为常规方法;所使用的材料、试剂等,如无特殊说明,为可从商业途径得到的试剂和材料。
实施例1陆地棉ghebf1基因的克隆
1、rna的提取
使用rnapreppure植物多酚多糖总rna提取试剂盒,分别提取棉花品种中植棉kv3叶片样品rna。
2、cdna的合成
2.1中间片段cdna的合成
中间片段cdna的合成使用fastquantcdna第一链合成试剂盒反转录cdna。
2.23’端的cdna的合成
3’端的cdna的合成体系如下:
表1
按上述体系混匀,离心后,置pcr仪上42℃60min,70℃15min,反应结束后冰上冷却,置于-20℃保存。
3.35’端的cdna的合成
5’race的cdna的合成体系如下:
第一步:
表2
混匀,短暂离心后置于冰上。
第二步:
表3
充分混匀后,将这11μl产物放置在pcr仪中,设置反应的程序:72℃3min→42℃2min。结束后,冷却1min后备用。
第三步:
表4
混匀,短暂离心。
第四步:
表5
用移液枪轻轻吸打混匀,短暂离心。置于pcr仪中,设置程序:42℃90min→70℃10min。用适量tricine-edtabuffer稀释反应所得cdna后,-20℃保存。
3.引物设计
race引物(表4-1)均由primer5.0设计而成,gsp、upm引物试剂盒自带,设计的引物送往生工合成。
表6race引物
4、ghebf1全长克隆
4.1目的基因中间片段克隆
根据已知的cdna片段,设计中间片段引物,按照下述体系混匀进行pcr扩增。
表7
pcr程序:94℃3min;94℃30s,58℃30s,72℃1min,35个循环;72℃10min;4℃保存。琼脂凝胶电泳分析扩增产物。
4.2目的基因3’末端克隆
3’race采用巢式pcr法扩增,
3’race的第一轮pcr扩增体系:
表8
按上述体系混匀,短暂离心后,进行pcr扩增。
pcr程序:94℃3min;94℃30s,55℃30s,72℃2min,20个循环;72℃10min;4℃保存。琼脂凝胶电泳分析扩增产物。之后把第一轮pcr扩增产物稀释50倍,进行第二轮pcr扩增。
3’race的第二轮pcr扩增体系:
表9
按上述体系混匀,短暂离心后,进行pcr扩增。
pcr程序:94℃3min;94℃30s,55℃30s,72℃1min,30个循环;72℃10min;4℃保存。琼脂凝胶电泳分析扩增产物,之后胶回收、连接和转化,挑取阳性克隆送生工测序。
4.3目的基因5’末端克隆
用适量tricine-edtabuffer稀释反应所得5’racecdna后,进性5’race的pcr扩增,pcr体系如下:
step1:
表10
轻轻混匀,短暂离心后,置于冰上。
step2:
表11
按照上述程序配好体系后,轻轻混匀,短暂离心,按照以下程序进行pcr的扩增。
pcr程序:
表12
反应结束后,进行凝胶琼脂糖电泳分析,观察条带情况,若出现弥散条带或者无条带,进行以下操作:
(1)本次模板为此前pcr扩增产物50倍稀释产物(tricine-edtabuffer)
(2)引物采用upms和5’ighx,各1μl,采用上述中的pcr体系,并设置反应程序为:94℃3s,65℃30s,72℃1min,20个循环;4℃保存。
反应结束后,进行凝胶琼脂糖电泳分析,之后胶回收、连接和转化,挑取阳性克隆送生工测序。
4.4ghebf1全长克隆
将中间片段、3’race片段和5’race片段通过dnaman拼接,设计ghebf1全长引物qghebf1-f、qghebf1-r(表6),进行全长pcr扩增。选取5’合成的cdna,用tricine-edtabuffer稀释5倍作为模板。
反应体系:
表13
pcr程序:94℃3min;94℃30s,55℃30s,72℃2min,30个循环;72℃10min;4℃保存。琼脂凝胶电泳分析扩增产物,进行胶回收,加a尾,体系如下:
表14
反应条件:72℃30min
将全长pcr反应液连接到t1simple载体上,转化大肠杆菌dh5α,挑取阳性克隆送生工测序。
经鉴定,基因ghebf1的cdna序列全长为2877bp(见图2),5’端utr(非翻译区)269bp,3’端utr区670bp,开放阅读框(orf)1938bp,共编码646aa,序列如seqidno:1所示。
实施例2大丽轮枝菌胁迫下沉默抗黄萎病陆地棉品种中植棉kv3对黄萎病抗病性影响
(1)材料
以野生型抗黄萎病陆地棉品种中植棉kv3和沉默ghebf1基因的抗黄萎病陆地棉品种中植棉kv3为实验材料。
(2)实验分组
对照组:中植棉kv3(中国农业科学院植物保护研究所提供);
实验组:沉默ghebf1基因的抗黄萎病陆地棉中植棉kv3,转化空载体(clcrv-00)中植棉kv3(获取方法参考张华崇硕士论文“vigs技术解析陆地棉抗黄萎病相关基因即ghb2功能初步鉴定”,中国农业科学院研究生院,2016;以及任玉红硕士论文“棉花黄萎病防治技术及两个抗病相关基因功能研究”,中国农业科学院研究生院,2018)。
(3)检测抗病性黄萎病菌系:强致病力落叶菌系v991(中国农业科学院植物保护研究所提供)。
(4)试验设计和性状测定
种子播种前用浓硫酸脱绒处理后,均挑选饱满大小一致的种子进行后续试验,用70%乙醇浸泡种子5min进行表面灭菌,而后用3%的过氧化氢浸泡种子2h,最后用无菌水冲洗干净。灭菌完的种子点种于花盆中(营养土:蛭石=2:1),放置于温度24℃,16h光照,8h黑暗,相对湿度为70%的温室生长,当棉株长出真叶时,移至烧杯中,采用营养栽培法培养棉株(具体营养栽培法参考本实验室张华崇硕士论文“vigs技术解析陆地棉抗黄萎病相关基因即ghb2功能初步鉴定”,中国农业科学院研究生院,2016;以及任玉红硕士论文“棉花黄萎病防治技术及两个抗病相关基因功能研究”,中国农业科学院研究生院,2018)。
v991菌悬液的制备:首先将保存的v991接种于pda培养基中,在26℃条件下培养一周,将活化的v991于查氏培养基中26℃,200rpm,5-6d;将达到所需浓度的菌液过滤至灭菌的烧杯中,用血球计数板测定孢子悬浮液的浓度,稀释孢子悬浮液的浓度至107个/ml。
(5)测定结果
在含营养土蛭石培养花盆中生长的野生型和转基因植株根系均能良好生长。在棉花苗生长一月后,用落叶型强致病力病菌v991孢子悬浮液(浓度107个/ml),采用蘸根法接种病菌,接种5、10、15、21天调查黄萎病发病情况。
结果表明,沉默ghebf1基因使抗病品种中植棉kv-3在接菌黄萎病菌强致病力菌系v991后,发病率大幅度提高,极显著降低植株抗病性(见图1),发病率和病情指数见表15。把抗病品种中植kv-3的ghebf1基因沉默,黄萎病病情指数达到56.1±2.3,极显著高于野生型的病情指数9.5±1.2。说明ghebf1基因在陆地棉抗黄萎病性中占有重要作用。表中大写字母代表1%水平的显著差异。
表15实施例2不同处理组黄萎病病情指数比较
实施例3大丽轮枝菌(verticilliumdahliae)胁迫下转超表达ghebf1拟南芥黄萎病抗性检测
(1)材料
以转ghebf1基因的第二代拟南芥和野生型拟南芥为实验材料(获取方法见本实验室张华崇硕士论文“vigs技术解析陆地棉抗黄萎病相关基因即ghb2功能初步鉴定”,中国农业科学院研究生院,2016;以及任玉红硕士论文“棉花黄萎病防治技术及两个抗病相关基因功能研究”,中国农业科学院研究生院,2018)。
(2)栽培基质
培养基:1/2ms培养基;1/2ms+50mg/l卡那霉素培养基;
栽培基质:蛭石+1/4ms营养液
(3)试验设计和抗病性测定
野生型拟南芥在1/2ms培养基中培养,转基因拟南芥在1/2ms+50mg/l卡那霉素培养基中培养,待幼苗长到一定大小后移栽至营养钵,移栽7天后进行大丽轮枝菌胁迫处理;超表达植株采用灌根法接种黄萎病菌v991。
共设置3个处理条件:1)对照(ck):野生型col-0型拟南芥和转化空载体分别接种v991后,分别作为阳性和阴性对照;2)超表达ghebf1植株(即第二代纯合后代)。采用灌根法接种黄萎病菌(大丽轮枝菌)v991(孢子悬浮液的浓度107个/ml)。接菌15天后进行拍照观察、发病率测定。
(4)测定结果
野生型col-0型拟南芥和转化空载体和接种v991发病率和病情指数差异不明显,但超表达ghebf1植株黄萎病发病率和病情指数极显著低于野生型col-0型拟南芥和转化空载体,见表16(表中大写字母代表1%水平的显著差异)。
大丽轮枝菌(verticilliumdahliae)胁迫下,转化ghebf1基因发病极显著低于野生型。过表达ghebf1的植株发病率和病情指数极显著低于野生型,这意味着ghebf1基因在植株响应大丽轮枝菌环境中起到了重要作用。说明ghebf1基因具有提高植物抗黄萎病的能力,在功能上证明了ghebf1在陆地棉抗黄萎病性能上起到重要作用。
表16实施例3不同处理组黄萎病病情指数比较
以上所述的实施例仅是对本发明的优选方式进行描述,并非对本发明的范围进行限定,在不脱离本发明设计精神的前提下,本领域普通技术人员对本发明的技术方案做出的各种变形和改进,均应落入本发明权利要求书确定的保护范围内。
序列表
<110>中国农业科学院植物保护研究所
<120>陆地棉ghebf1基因、蛋白及其应用
<160>4
<170>siposequencelisting1.0
<210>1
<211>1938
<212>dna
<213>人工序列(artificialsequence)
<400>1
atgcctgctcttgttaattacagtggtgatgatgatttctataatggagggtccttttat60
acaaattctactgacttgggtagattgtgttcaatcagtttccaagttgatgtgtactgc120
ccgcctcgtaaaagaacgcgtattagtgccccatttcactttggagaagctgaggttgag180
cagaataagcaaccatcgatcgatgtcctccctgatgaatgccttgttgagatctttaaa240
cgcatttctgggggtaaagaaaggagctcctgtgcttgtgtctctaagcactggcttatg300
cttctgaccagcattcgcaagggtgaatatgagtcatcaaaggtggtcaaggagaacgtt360
ggttcgaattcgggtgatgttgagatgatcttatccgaggaagatgatgggtatcttaca420
aggtgcttagaaggaaagaaagcaactgacatgaggcttgctgcggtggcggttggaaca480
agtggtcatggtggtttaggaaagctttctattaggggaagcagctcttcccgtggagta540
actaatttcgggctgtctgccgtagctcgtggttgtccttctctaaaggtactttctttg600
tggaatgttccgcgtgttggagatgaaggtctgtgcgagatagctaaagaatgccatttg660
ttggagaagcttgatctttgtcagtgcccccacctttcaaacaaggggctaattgccatc720
gccgccaactgccctaatctgactgcgttgagcatccaatcttgcccaaagattggtaat780
gagggcctccaagctatcggaaagctttgccccaagctgcagtccatctctatcaaggac840
tgcccacttgttggggatcatggagtttcaagcctgttgtcatcagcatcttccgtcctt900
tcaaaggttaagcttcagggtttgagcatcacagatttttctcttgctgtgatcgggcac960
tatggcaagtctgtgactaatctaatgctgagtggtctacaaaatgtgagcgagaaggga1020
ttttgggtgatgggtaatgctcagggtctgcaaaagttggtctcttttacaattgcttct1080
tgctggggagtaactgatgtgagtcttgaagccattggaaagggctgcgctaacctgaag1140
cagatgtgccttcgcaggtgttgctttgtttctggtgatggattggtggcttttgccaag1200
tctgccggttctcttgagtgcttgcagttggaggagtgcaacagggtcactcaatccggg1260
gttatcggtgttctctcaaactgtgggttgaaatctcttaccctagtaaagtgtatggga1320
attaaggatatatccttggaagctcctttgtcttcctcttgcaattcccttaaatccttg1380
tctataaggaactgcccaggattcggaacagccagcctggctatggtgggcagattgtgc1440
cctcagcttcagcatgtagacctgagcggactttgcggcattacagatgcaggtcttctg1500
cctcttctagagaattgcgaggcaggacttgtgaaagtgaatctgagcggctgcttaaac1560
ttgactgatgaggtcgttttgcacttgaccaagctacatggcgcaaccctcgagttgctc1620
aatctcaacggttgcagaaggattactgatgcaagtttggctgcagttgcagaaaattgt1680
gttttcctcagcgacttggatgtgtcaaggtgtgcaatcaccgatgtcggtattgcagcc1740
ttgtctcacgcagagcaactcaacttgcaagtcctttcgttttcaggttgttcaggagta1800
tcaaacaaaagcatgaccttcctaaaaaaattgggcaagaccctggtggggttgaatctc1860
cagcactgcaattcaatcagcacccaaaccattgagctgctcgtggaaagcttgtggaga1920
tgcgacatccttgtctaa1938
<210>2
<211>643
<212>prt
<213>人工序列(artificialsequence)
<400>2
metproalaleuvalasntyrserglyaspaspaspphetyrasngly
151015
glyserphetyrthrasnserthraspleuglyargleucysserile
202530
serpheglnvalaspvaltyrcysproproarglysargthrargile
354045
seralaprophehispheglyglualagluvalgluglnasnlysgln
505560
proserileaspvalleuproaspglucysleuvalgluilephelys
65707580
argileserglyglylysgluargsersercysalacysvalserlys
859095
histrpleuleuleuthrserilearglysglyglutyrgluserser
100105110
lysvalvallysgluasnvalglyserasnserglyaspvalglumet
115120125
ileleuserglugluaspaspglytyrleuthrargcysleuglugly
130135140
lyslysalathraspmetargleualaalavalalavalglythrser
145150155160
glyhisglyglyleuglylysleuserileargglyserserserser
165170175
argglyvalthrasnpheglyleuseralavalalaargglycyspro
180185190
serleulysvalleuserleutrpasnvalproargvalglyaspglu
195200205
glyleucysgluilealalysglucyshisleuleuglulysleuasp
210215220
leucysglncysprohisleuserasnlysglyleuilealaileala
225230235240
alaasncysproasnleuthralaleuserileglnsercysprolys
245250255
ileglyasngluglyleuglnalaileglylysleucysprolysleu
260265270
glnserileserilelysaspcysproleuvalglyasphisglyval
275280285
serserleuleuserseralaserservalserlysvallysleugln
290295300
glyleuserilethrasppheserleualavalileglyhistyrgly
305310315320
lysservalthrasnleumetleuserglyleuglnasnvalserglu
325330335
lysglyphetrpvalmetglyasnalaglnglyleuglnlysleuval
340345350
serphethrilealasercystrpglyvalthraspvalserleuglu
355360365
alaileglylysglycysalaasnleulysglnmetcysleuargarg
370375380
cyscysphevalserglyaspglyleuvalalaphealalysserala
385390395400
glyserleuglucysleuglnleugluglucysasnargvalthrgln
405410415
serglyvalileglyvalleuserasncysglyleulysserleuthr
420425430
leuvallyscysmetglyilelysaspileserleuglualaproleu
435440445
sersersercysasnserleulysserleuserileargasncyspro
450455460
glypheglythralaserleualametvalglyargleucysprogln
465470475480
leuglnhisvalaspleuserglyleucysglyilethraspalagly
485490495
leuleuproleuleugluasncysglualaglyleuvallysvalasn
500505510
leuserglycysleuasnleuthraspgluvalvalleuhisleuthr
515520525
lysleuhisglyalathrleugluleuleuasnleuasnglycysarg
530535540
argilethraspalaserleualaalavalalagluasncysvalphe
545550555560
leuseraspleuaspvalserargcysalailethraspvalglyile
565570575
alaalaleuserhisalagluglnleuasnleuglnvalleuserphe
580585590
serglycysserglyvalserasnlyssermetthrpheleulyslys
595600605
leuglylysthrleuvalglyleuasnleuglnhiscysasnserile
610615620
serthrglnthrilegluleuleuvalgluserleutrpargcysasp
625630635640
ileleuval
<210>3
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>3
atgcctgctcttgttaat18
<210>4
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>4
ttagacaaggatgtcgca18
- 还没有人留言评论。精彩留言会获得点赞!