一种利用RNA干扰机制诱导多位点切割基因组实现选择性杀死细胞的DNA系统的制作方法
一种利用rna干扰机制诱导多位点切割基因组实现选择性杀死细胞的dna系统
技术领域
1.本发明涉及分子生物学领域,具体而言涉及一种利用rna干扰机制诱导多位点切割基因组实现选择性杀死细胞的dna系统。
背景技术:
2.rna干扰机制
3.rna干扰(rna interference,rnai)是指由单链或双链rna(double-stranded rna,dsrna)诱发的同源mrna高效特异性降解的现象。rnai具有如下特征:1)rnai是转录后水平的基因沉默机制;2)rnai具有很高的特异性,只降解与之序列相应的单个内源基因的mrna;3)rnai抑制基因表达具有很高的效率,表型可以达到缺失突变体表型的程度,而且相对很少量的dsrna分子(数量远远少于内源mrna的数量)就能完全抑制相应基因的表达,是以催化放大的方式进行的;4)rnai抑制基因表达的效应可以穿过细胞界限,在不同细胞间长距离传递和维持信号甚至传播至整个有机体以及可遗传等特点;5)dsrna不得短于21个碱基,并且长链dsrna也在细胞内被dicer酶切割为21bp左右的sirna,并由sirna或者mirna来介导mrna切割。而且大于30bp的dsrna不能在哺乳动物中诱导特异的rna干扰,而是细胞非特异性和全面的基因表达受抑和凋亡;6)atp依赖性:在去除atp的样品中rna干扰现象降低或消失显示rna干扰是一个atp依赖的过程。可能是dicer和risc的酶切反应必须由atp提供能量。
4.小干扰rna(sirna)
5.病毒基因、人工转入基因、转座子等外源性基因随机整合到宿主细胞基因组内,并利用宿主细胞进行转录时,常产生一些dsrna。宿主细胞对这些dsrna迅即产生反应,其胞质中的核酸内切酶dicer将dsrna切割成多个具有特定长度和结构的小片段rna(大约21~23bp),即sirna(小干扰rna,small interfering rna)。sirna在细胞内rna解旋酶的作用下解链成正义链和反义链,继之由反义sirna再与体内一些酶(包括内切酶、外切酶、解旋酶等)结合形成rna诱导的沉默复合物(rna-induced silencing complex,risc)。risc与外源性基因表达的mrna的同源区进行特异性结合,risc具有核酸酶的功能,在结合部位切割mrna,切割位点即是与sirna中反义链互补结合的两端。被切割后的断裂mrna随即降解,从而诱发宿主细胞针对这些mrna的降解反应。sirna不仅能引导risc切割同源单链mrna,而且可作为引物与靶rna结合并在rna聚合酶(rna-dependent rna polymerase,rdrp)作用下合成更多新的dsrna,新合成的dsrna再由dicer切割产生大量的次级sirna,从而使rnai的作用进一步放大,最终将靶mrna完全降解。
6.sirna通常作为rnai的工具被人工合成,但是后来发现很多生物包括线虫还有人类一些特殊的细胞也会合成内源性sirna,以用于基因表达的调控。
7.微小rna(microrna,mirna)
8.mirna是一类长度约22个碱基的非编码小rna,广泛存在于动物、植物和病毒中。
mirna主要通过与ago2等蛋白形成rna沉默复合物(rna-induced silencing complex,risc)在转录后水平调控基因的表达。由mirna和ago蛋白形成的risc复合物通过碱基配对的方式识别目标序列,依赖于mirna与靶标序列碱基互补的程度可以直接切割靶标rna或者通过促进rna降解以及抑制翻译的方式调控基因表达。
9.mirna与靶标序列的识别主要依赖于种子序列(第2-8位碱基),但是其他部分的碱基也参与靶标序列的识别过程。当mirna与靶标序列完全互补配对或者接近完全互补(除种子序列以外只有极少的错配),可以诱导ago2蛋白直接切割靶标序列。如果mirna的种子序列可以完全匹配靶标序列,而其余部分的匹配程度不足以诱导ago2切割,则会利用复杂的机制促进rna降解以及抑制翻译过程。
10.目前人体细胞内发现的mirna种类已经超过2000余种。细胞中mirna的表达根据细胞类型和细胞状态的不同呈现高度的特异性,这种表达特异性已越来越广泛被用作细胞的特征标签,用于区别不同的细胞类型和细胞状态。以人体细胞为例,研究表明人体组织内不同类型的细胞有明显差异的mirna表达谱,许多细胞具有高度特异性的mirna表达,例如肝细胞中的mir-122,肌肉细胞中的mir-1,神经元细胞中的mir-124等。研究表明,细胞内mirna的表达组成在个体发育以及疾病发生和发展过程中均呈现高度的表达变化。
11.mirna在细胞的增殖分化、新陈代谢、细胞凋亡和个体生长发育、组织器官功能以及疾病的发生发展等多种生物学过程中发挥重要的调控作用。例如,在癌症研究方面,迄今已经发现多种可以促进癌细胞生长或转移的mirna,比如mir-17/92、mir-222/221、mir-21、mir-155等。
12.除了参与调控各项生命活动外,近年来mirna在临床应用方面也受到了越来越多的关注。在治疗疾病方面,一方面通过降低病变细胞中高表达的具有促进疾病作用的mirna的表达水平或者调控活性,可以起到抑制缓解疾病的目的。另一方面,通过向病变细胞靶向递送功能性mirna,可以降低疾病相关基因的表达水平或者抑制免疫逃逸相关的基因,促进免疫细胞的杀伤效果。虽然利用mirna在疾病治疗方面的应用已经取得了不少进展,但是这些研究大多处在初步研究阶段,对于其治疗效果和安全性等方面的研究还很不充分。目前对于mirna的应用仍然是基于mirna和靶基因之间的调控关系来实现的。但是由于每种mirna平均可以调控数百个靶基因的表达,而每种靶基因又往往处在多种mirna的调控之下,因而呈现出非常复杂的冗余性和关联性。因此通过降低或者递送单个或少量几个mirna来调控致病基因的表达水平,一方面可能没有明显调控效果,另一方面可能会带来严重的副作用。突破基于mirna和靶基因之间的调控关系的思路和方法有望避免以上缺陷,拓展mirna的应用,并有望推动mirna在临床治疗领域的实际应用。
技术实现要素:
13.本发明人利用mirna或sirna介导的sgrna释放策略创建了crispr-cas9平台,该平台可以被细胞内特定的mirna或sirna打开从而诱导crispr-cas9多位点切割基因组,从而靶向杀伤病变细胞。
14.据此,本发明提供了一种利用rna干扰机制诱导多位点切割基因组实现选择性杀死细胞的dna系统,所述细胞杀伤系统包括以下几个部分:
15.1.表达核酸酶crispr-cas9蛋白的质粒;
16.2.表达前体单链引导rna(pre-sgrna)的质粒,所述前体合成引导rna的含有与目标mirna或sirna完全互补的序列以及合成引导rna(sgrna)的序列;
17.所述细胞杀伤系统用于靶向杀伤表达特定mirna或sirna的细胞。
18.在本发明的一个具体实施方案中,所述核酸酶crispr-cas9蛋白为streptococcus pyogenes cas9(spycas9)。
19.在本发明的另一个具体实施方案中,所述前体合成引导rna在合成引导rna(sgrna)的序列两端均含有与目标mirna或sirna完全互补的序列。
20.在本发明的另一个具体实施方案中,所述前体合成引导rna在合成引导rna(sgrna)的序列两端含有的与目标mirna或sirna完全互补的序列是相同的序列。
21.在本发明的另一个具体实施方案中,所述合成引导rna的序列与细胞基因组中重复元件的部分序列互补,以使得所述sgrna可以将所述核酸酶crispr-cas9蛋白引导至重复元件上产生多次切割。
22.在本发明的另一个具体实施方案中,所述前体合成引导rna可以单独表达一种或者以串联形式表达多种。
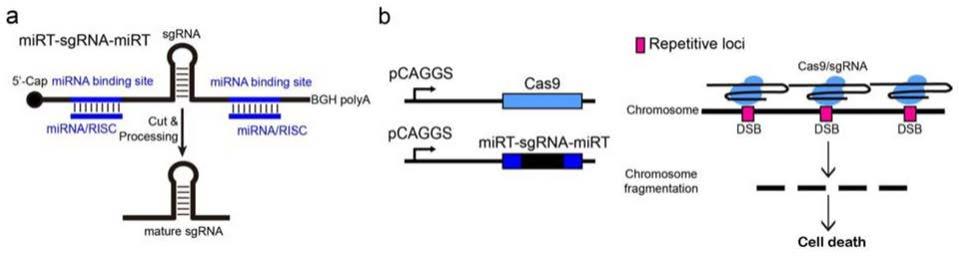
23.在本发明的另一个具体实施方案中,所述以串联形式表达的多种前体合成引导rna的序列两端含有的与目标mirna或sirna完全互补的序列是相同的序列。
24.在本发明的另一个具体实施方案中,所述以串联形式表达的前体合成引导rna与细胞基因组中重复元件序列互补的部分是不同序列。
25.在本发明的另一个具体实施方案中,所述合成引导rna的序列与细胞基因组中重复元件的部分序列互补,其中所述重复元件为人类细胞基因组中的高度重复元件alu元件(alu elements)。
26.本发明提供的dna靶向杀伤系统通过利用rna干扰机制和crispr-cas9系统相结合,将原本对基因表达起抑制作用的mirna或sirna转变为激活基因表达的信号,从而有效地实现了诱导crispr-cas9蛋白至重复元件上产生多次切割,破坏细胞基因组完整性,可以用于靶向杀伤表达目标mirna或sirna的细胞。
附图说明
27.参考随附的附图,本发明更多的目的、功能和优点将通过本发明实施方式的如下描述得以阐明,其中:
28.图1为micr-cut系统设计原理。
29.(a)pre-sgrna的设计原理。分别在由ii型启动子表达产生的sgrna的5’端和3’端插入与目标mirna完全互补配对的靶标序列。当细胞中有目标mirna表达时,可以通过risc复合物对靶标序列产生切割,从而去掉pre-sgrna两端的修饰,产生具有功能活性的sgrna。(b)micr-cut系统设计原理。sgrna设计成为靶向基因组上高度重复序列。(c)micr-cut系统由特定mirna诱导激活后,通过靶向切割基因组重复序列,造成过多dsb,从而实现特异性细胞杀伤。
30.图2为检验micr-cut系统对hela细胞的杀伤效果。
31.micr-cut系统对hela细胞的杀伤作用。向转染nc或mir-294的hela细胞中分别转染cas9、294t-alu1或者cas9、294t-alu2表达质粒。每组数据归一化到各自的nc组。图中所
示数据为平均值
±
标准差。2次独立重复实验。
32.图3为利用pre-sgrna串联形式的micr-cut系统特异性杀死hela细胞。
33.(a)串联形式的micr-cut pre-sgrna设计形式。mirna激活pre-sgrna后可以同时产生两种分别靶向不同基因组重复位点的sgrna,。(b)不同转染条件下hela细胞的存活情况。向转染nc或者mir-294的hela细胞中分别转染不同的pre-sgrna和cas9表达质粒。mock对照组为转染nc、不表达sgrna的空载体和cas9。3次独立重复实验。比例尺为200μm。(c)不同转染条件下hela细胞的相对数目。数据归一化到mock对照组。图中所示数据为平均值
±
标准差。3次独立重复实验。统计方法:two-tailed unpaired student’s t-test。
34.图4为利用pre-sgrna串联形式的micr-cut系统特异性杀死u2os细胞。
35.(a)不同转染条件下u2os细胞的存活情况。向转染nc或者mir-294的u2os细胞中分别转染不同的pre-sgrna和cas9表达质粒。mock对照组为转染nc、不表达sgrna的空载体和cas9。3次独立重复实验。比例尺为200μm。(b)不同转染条件下u2os细胞的相对数目。数据归一化到mock对照组。图中所示数据为平均值
±
标准差。3次独立重复实验。统计方法:two-tailed unpaired student’s t-test。
36.图5为利用micr-cut系统特异性杀死混合体系中的特定类型细胞。(a)不同转染条件下,混合体系中hela
mir-122/gfp
和hela
mir-302/dsred
细胞的存活情况。向hela
mir-122/gfp
和hela
mir-302/dsred
混合体系中分别转染cas9以及不同的pre-sgrna表达质粒。mock对照组转染不表达pre-sgrna的空载体和cas9。3次独立重复实验。比例尺为200μm。(b)不同转染条件下,混合体系中hela
mir-122/gfp
和hela
mir-302/dsred
细胞的比例。图中所示数据为平均值
±
标准差。3次独立重复实验。统计方法:two-tailed unpaired student’s t-test。
具体实施方式
37.crispr-cas9系统
38.crispr全称是clustered regularly interspaced short palindromic repeats,中文译作周期间隔短回文重复序列簇,是一类发现于细菌和古细菌中的基因组序列。cas9全称是crispr-associated protein 9,属于一类核酸酶,cas蛋白最初于2005-2006年左右被鉴定发现。此后几年(2007-2011)的一系列研究逐渐揭示crispr/cas系统作为细菌和古细菌免疫系统对抗病毒侵染的机制:研究发现crispr序列实际上来源于侵入细菌的质粒或者病毒dna,并且crispr序列可以被转录并加工产生短的rna,这些rna片段与cas蛋白结合起到对抗病毒的作用,因此这些rna被称为cas related rna,简称crrna。后来又发现细菌需要同时表达另外一种rna来激活cas蛋白的活性,即tracrrna(trans-activation cas related rna,反式激活crrna)。最终在2012年的研究中证明cas9可以和tracrrna、crrna结合,切割质粒dna;研究同时发现tracrrna和crrna可以被串联成一条rna,即sgrna。至此,crispr/cas9系统作为基因编辑工具的条件已经具备,并在2013年被成功应用在哺乳动物的基因编辑上,crispr-cas9时代正式开始。
39.crispr-cas9系统是目前应用最为广泛的基因编辑系统。如下图所示,这个系统主要由两部分构成:cas9和sgrna。cas9为核酸酶,可以切割dna,造成双链断裂(double strand break,dsb);sgrna全称为合成引导rna(synthetic guide rna),当cas9和sgrna结合以后,sgrna可以激活并引导cas9蛋白定位到基因组特定位点,由此启动cas9的基因编辑
或调控活性。
40.sgrna:合成引导rna(sgrna)通常为一段长度约110个核苷酸的序列。该序列主要由两部分构成:前端部分为长度约40个核苷酸的crrna,其5’端包含有人为自主设计的和目标基因的dna碱基互补配对的约20个核苷酸的序列,具有引导cas9:sgrna复合物定位到基因组目标区域的功能;后端部分为约70个核苷酸的tracrrna序列,该部分与crrna部分互补配对,形成特定结构,具有招募cas9蛋白,促进cas9:sgrna复合物与目标序列结合的功能。crrna与trarna之间一般包含4个核苷酸连接序列。本发明使用sgrna序列由ucsf的bo huang于2013年发表,源于酿脓链球菌(s.pyogenes)。
41.在通常的应用中,一般使用u6启动子来表达sgrna。这种启动子通过iii型rna聚合酶转录产生有活性的sgrna。而本发明中使用ii型rna聚合酶的启动子进行转录,那么转录出来的sgrna由于含有5’帽子和3’多聚核苷酸尾巴(polya),不能够发挥功能,因此被称为无活性sgrna)。需要通过设计去掉5’帽子和3’polya才能够将其转变为有活性的sgrna。
42.alu重复元件
43.alu元件是灵长类基因组中特有的、散在分布一类转座元件,也是人类基因组中丰度最高的重复元件,约占人类基因组序列的11%,拥有超过100万个拷贝。alu元件属于短散在序列(short interspersed elements,sines),长度约为300bp,因末端含有alu限制酶的切割位点而得名。研究表明,alu元件在人类演化以及多种疾病的产生中均发挥重要的作用。
44.micr-cut dna平台
45.本发明人建立了一种利用rna干扰机制诱导多位点切割基因组实现选择性杀死细胞的dna系统。本系统包含2个部分:由caggs启动子驱动表达的cas9核酸酶蛋白;由caggs启动子驱动表达的pre-sgrna。本研究将pre-sgrna的spacer序列设计成可以靶向基因组高度重复的元件;当mirna激活pre-sgrna后,cas9可以和sgrna组成功能性复合物对基因组进行多位点切割,造成大量dna双链断裂(dsb),从而引起细胞死亡。我们将这一系统命名为micr-cut dna系统。
46.在本发明的一个具体实施方案中,所选择重复元件为人类细胞基因组中的alu元件(alu elements)。因此可设计靶向该重复元件中部分序列的sgrna,并在该sgrna序列的两端连接与目标mirna或sirna序列完全互补的rna序列,这个就是所述的前体单链引导rna(pre-sgrna,在本发明中也表示为mirt-sgrna-mirt,mirt表示与目标mirna完全互补的序列),在pre-sgrna的两端分别有5’帽子和3’polya,使得该pre-sgrna处于无活性的状态。
47.将该平台导入靶细胞后,如果该靶细胞中没有目标mirna或sirna的表达,则整个系统处于未被激活的状态,cas9蛋白不会切割基因组dna。如果该靶细胞中表达目标mirna或sirna,则该目标mirna或sirna就会与pre-sgrna两端的与其完全互补的rna序列相结合,从而启动mirna或sirna介导的rna切割机制而导致该完全互补rna序列被切割。而该完全互补rna序列的切割则导致sgrna与其两端的5’帽子和3’polya相脱离从而具有引导活性。有活性的sgrna就会将dcas9蛋白引导至基因组中alu元件处,使得cas9核酸酶蛋白切割此处的dna,造成双链断裂。由于alu元件在人类细胞基因组中存在数十万计的重复,因此cas9可以多次切割dna以破坏细胞基因组的完整性,从而杀死细胞。以上过程就是本发明的主要原理。
48.在本发明的具体实施方案中,所述重复元件可以是alu序列,也可以是人体细胞基因组中其他高度重复的元件。
49.在本发明的具体实施方案中,与目标mirna或sirna完全互补的rna序列存在所述sgrna的两端。
50.在本发明的具体实施方案中,存在于所述sgrna两端的与目标mirna或sirna完全互补的rna序列是相同序列。
51.在本发明的具体实施方案中,所述前体合成引导rna可以单独表达一种或者以串联形式表达两种。
52.在本发明的具体实施方案中,所述以串联形式表达的多种前体合成引导rna的序列两端含有的与目标mirna或sirna完全互补的序列可以是相同或不同的序列,即不同的前体合成引导rna可以针对于相同或不同的mirna或sirna。
53.在本发明的具体实施方案中,所述以串联形式表达的前体合成引导rna与细胞基因组中重复元件序列互补的部分可以是不同或相同的序列。
54.本发明提供的dna靶向杀伤系统通过利用rna干扰机制和crispr-cas9系统相结合,将原本对基因表达起抑制作用的mirna或sirna转变为激活基因表达的信号,从而有效地诱导crispr/cas9系统进行多位点切割基因组以破坏细胞基因组的完整性,可以用于靶向杀伤表达目标mirna或sirna的细胞。因此具有重大的实际意义和广泛的应用前景。
55.术语和缩写
56.在本说明书中使用了一些术语和缩写,其含义如下所述,未特别说明的术语和缩写具有本领域技术人员公知的含义。
57.rnai:rna干扰(rna interference);
58.sirna:小干扰rna(small interfering rna);
59.mirna:微小rna(microrna)
60.mir:各种mirna的缩写,通常后面跟有数字及字母编号以表示其命名,各种mir的编号及其序列在本领域中都是公知的。
61.mirt:表示与目标mirna或sirna完全互补的序列
62.sgrna:合成引导rna(synthetic guide rna);
63.pre-sgrna:前体合成引导rna,含有与目标mirna或sirna完全互补的序列以及sgrna的序列,亦可表示为mirt-sgrna或者mirt-sgrna-mirt;
64.crispr:周期间隔短回文重复序列簇(clustered regularly interspaced short palindromic repeats);
65.cas9:周期间隔短回文重复序列簇相关蛋白9(crispr-associated protein9);
66.micr-cut dna:可被mirna或sirna诱导crispr/cas9系统进行多位点切割基因组以破坏细胞基因组的完整性,从而靶向杀伤细胞的实验平台系统(micr-cutting dna system);
67.通过下面的参考示范性实施例,本发明的目的和功能以及用于实现这些目的和功能的方法将得以阐明。然而,本发明并不受限于以下所公开的示范性实施例;可以通过不同形式来对其加以实现。说明书的实质仅仅是帮助相关领域技术人员综合理解本发明的具体细节。
68.在下文中,将参考附图描述本发明的实施例。在附图中,相同的附图标记代表相同或类似的部件,或者相同或类似的步骤。
69.实施例1
70.材料和方法
71.质粒和载体的构建
72.将cas9连接于含有由pgk启动子驱动的潮霉素抗性基因的piggybac载体中,所述cas9由caggs启动子驱动。将pre-sgrna(两端含有与目标mirna或sirna完全互补的rna序列的sgrna序列,简称为mirt-sgrna-mirt)连接于含有由pgk启动子驱动的博来霉素(zeocin)抗性基因的piggybac载体中,所述pre-sgrna也由caggs启动子驱动。为了制备对目标mirna或sirna敏感的mirt-sgrna-mirt构建体,使用fastpfu聚合酶(transgene公司),以含有sgrna的质粒为模板(所述sgrna的序列与alu元件的部分序列互补),用与mirt相对应的引物进行pcr。pcr产物与ecori-hf(20u,neb公司)和bamhi-hf(20u,neb公司)在37℃下孵育2h,用旋转柱(magen公司)纯化,并用t4连接酶(life technology公司)连接到经过ecori和bamhi消化的piggybac载体上。
73.细胞培养,质粒转染和小rna转染
74.1)细胞培养
75.hela和u2os细胞培养在高糖培养基中(dmem+10%fbs+p/s)。培养皿不需要特殊处理。在细胞生长到70%-80%汇合度时进行传代培养。传代时,将旧培养基吸除,用1
×
pbs清洗一遍后,加入0.1%trypsin,于37℃消化3-5分钟;加入适量新鲜培养基终止消化后,用1ml无菌枪头轻轻吹打10-15次后,按照1:3比例进行接种,轻轻摇匀后置于37℃,5%co2条件下进行培养。一般2天传代1次。
76.2)细胞转染和细胞系构建
77.本研究中,质粒转染使用试剂完成;mirna和sirna转染使用dharmafect完成。转染操作按照说明书进行。
78.hela
mir-122/gfp
和hela
mir-302/dsred
:在转染前16-20小时,将将2.0x105个hela接种到12孔板中;转染时,按照试剂使用说明,将1μgpb-caggs-pri-mir122/ef1a-gfp或者pb-caggs-pri-mir302/ef1a-dsred表达质粒和0.5μg pbase转染到细胞中;转染24小时后,将培养基换成含有200μg/ml hygromycin b的高糖型dmem培养基;筛选培养1周后利用流式细胞分选仪分选荧光强度最高的10%细胞群进行培养扩增,用于后续实验。
79.利用micr-cut dna系统杀伤hela或u2os细胞:在转染前16-20小时,将3.0x104个hela或者u2os细胞接种到48孔板中;使用dharmafect分别转染nc和mir-294mimics,终浓度为20nm;转染后6小时使用试剂进行质粒转染,每孔转染100ng cas9、100ng pre-sgrna和50ng pbase表达质粒;转染后48小时,将细胞按照1:1比例重新接种到24孔板中,培养基中含有100μg/ml zeocin;培养2天后,将细胞重新接种到不含zeocin的新的24孔板中继续培养2-3天,进行成像和细胞计数。
80.统计学分析
81.除非另有说明,数据以平均值
±
sd表示。我们进行双尾不成对student’st检验以确定统计学显著性。p值《0.05被认为是具有统计学显著性。
82.实施例2
83.检验micr-cut系统对hela细胞的杀伤效果。
84.本研究首先选择了hela细胞基因组中高重复序列的alu元件,并设计了两条靶向alu元件的sgrna,这两条sgrna在基因组上分别含有~100,000和~80,000个匹配的靶位点(使用cas-offinder软件预测)。本研究将这两条sgrna构建成为可以由mir-294激活的pre-sgrna(简称为294t-alu1,294t-alu2)。实验结果见图2,表明将具有核酸酶活性的cas9和pre-sgrna表达载体转染到hela细胞中,两种pre-sgrna在转染mir-294条件下均可以引起50%左右的hela细胞数目减少。该实验结果初步说明可以利用micr-cut dna系统通过靶向切割重复序列的方式特异性杀伤细胞。
85.实施例3
86.利用pre-sgrna串联形式的micr-cut系统特异性杀死hela细胞。
87.为了增强micr-cut系统的杀伤效果,本研究选择将两条靶向不同位点的pre-sgrna采用串联成为单个pre-sgrna的形式,这样当mirna激活pre-sgrna后可以同时产生两种不同的sgrna,分别靶向不同的基因组重复位点。本研究分别构建了mir-294和mir-21靶向激活的pre-sgrna(简称为21t-alu1-alu2;294t-alu1-alu2)。
88.实验结果见图3,可以看出:当转染cas9和21t-alu1-alu2表达质粒后,hela细胞数目显著减少;而转染cas9和294t-alu1-alu2对hela细胞生长没有显著影响,只有当外源转染mir-294后才可以显著减少hela细胞数目。该实验结果进一步证明micr-cut dna系统可以通过mirna诱导的方式特异性地杀伤细胞。
89.实施例4
90.利用pre-sgrna串联形式的micr-cut系统特异性杀死u2os细胞。
91.为了验证micr-cut系统可以用于杀死其他类型的肿瘤细胞,本研究选择骨肉瘤细胞系u2os细胞重复了细胞杀伤实验。实验结果见图4,表明当转染cas9和21t-alu1-alu2表达质粒后,u2os细胞数目显著减少;而转染cas9和294t-alu1-alu2对u2os细胞生长没有显著影响,只有当外源转染mir-294后才可以显著减少u2os细胞数目。该实验结果证明了micr-cut dna系统在杀伤肿瘤细胞中的通用性。
92.实施例5
93.利用micr-cut系统特异性杀死混合体系中的特定类型细胞。
94.接下来本研究探究了micr-cut dna系统是否可以在混合细胞中选择性杀死表达特异mirna的细胞。为了避免转染效率和细胞生长速度等因素的影响,本研究分别构建了过表达mir-122的绿色荧光标记的hela细胞(简称为hela
mir-122/gfp
)和过表达mir-302的红色荧光标记的hela细胞(简称为hela
mir-302/dsred
),mir-122和mir-302在野生型hela细胞中均不表达。
95.接下来本研究构建了mir-122和mir-302激活的串联形式的pre-sgrna(简称为122t-alu1-alu2;302t-alu1-alu2)。实验结果见图5,如图5所示,当转染cas9和122t-alu1-alu2表达质粒后,混合细胞中表达mir-122的绿色hela细胞比例显著降低,表达mir-302的红色hela细胞比例显著升高;而转染cas9和302t-alu1-alu2表达质粒后,混合细胞中表达mir-302的红色hela细胞比例显著降低,表达mir-122的绿色hela细胞比例显著升高。该实验结果初步证明micr-cut系统可以在混合细胞中选择性杀伤表达特异mirna的细胞。
96.通过公开的上述实施例,本发明的其他实施例对于本领域技术人员都是易于想到和理解的。说明书中的具体描述仅被认为是示例性的,本发明的真正范围和主旨均由本发明的权利要求书所限定。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1