RNA中m5C和m6A双重分析逻辑光电化学传感方法
rna中m5c和m6a双重分析逻辑光电化学传感方法
技术领域
1.本发明属于生物基因技术领域,尤其涉及一种rna中m5c和m6a双重分析逻辑光电化学传感方法。
背景技术:
2.目前,rna甲基化是rna转录后最主要的表观遗传修饰方式,rna甲基化包括n6
‑
甲基腺嘌呤m6a、n7
‑
甲基鸟嘌呤m7g、5
‑
甲基胞嘧啶m5c、n1
‑
甲基腺嘌呤m1a、假尿嘧啶um等五种类型,其中m6a和m5c是发生最高频的两类修饰,而m6a叠合m5c(即特定rna序列中腺嘌呤a和胞嘧啶c均同时发生甲基化修饰)占rna甲基化比率≧95%。mrna和lncrna等非编码小rna 通过甲基化修饰调控rna的可变剪切、转录本组装和蛋白质翻译等;在肿瘤发生中,直接参与调节抑癌基因失活和癌基因表达。大量研究表明,m6a和m5c 修饰水平的偏倚与肿瘤、代谢性疾病、神经缺陷、心血管疾病和异常分化等密切相关。rna甲基化分析已成为自身免疫性疾病、代谢性疾病等基因表观紊乱疾病临床诊断,和肿瘤早期预警与去甲基化治疗方案选择的新手段,rna甲基化临床适宜检测技术研究具有重大意义。
3.依据检测原理不同,迄今已研究建立的rna甲基化分析技术主要包括以下三类:
4.一是基于免疫共沉淀和全基因组测序建立的rna甲基化测序分析 (merip
‑
seq):merip
‑
seq分析是利用m6a和m5c特异性抗体筛选携带m6a 和m5c的mrna片段并将其沉淀,构建相应的cdna文库后进行高通量测序。该技术是目前唯一可实现甲基化定位和丰度分析以及甲基化碱基类别鉴定的 rna甲基化检测技术。但由于该方法的分辨率仅约100个bp,测序长度受限,且易受跨越残基的大峰影响;另一方面,测试结果受测序深度的影响,文库构建过程复杂;同时,由于检测后庞大的数据处理和对检测设备的特殊要求,导致其难以在临床实验室推广应用。
5.二是基于高效液相色谱和质谱技术为基础建立的液相色谱
‑
串联质谱技术:即lc
‑
ms/ms,该技术可测定基因组rna甲基化水平,能够对含痕量rna的生物样本(如血浆)进行全rna甲基化丰度的准确测定。但由于分析前需进行 rna提取和多重洗脱,并需采用核酸外切酶将rna水解为单核苷酸,因而存在检测过程繁冗、洗脱纯度干扰和酶解不完全等缺陷。
6.第三类是基于特定位点切割与夹板提取结合薄层色谱技术而构建的 scarlet分析。scarlet是一种整合了特定位点切割、放射性标记、夹板辅助提取以及薄层色谱等多种技术的rna测序法。该类技术分辨率高,但由于涉及放射性污染,以及同样存在酶解效率和测序后庞大的数据处理等缺陷,而限制了其在临床医学实验室的开展。
7.光电化学传感技术是以电化学生物传感技术为研究背景,利用光学和电学信号转换作为基础,通过光电活性材料发生电荷转移所产生光电流信号响应的变化来实现对待测物灵敏检测的一种新型检测技术,其结合了电化学技术的传统优点和光电化学技术的优势,使用光学信号作为激发光源,产生较低的背景信号,对检测干扰小。同时,它还具有仪器操作简便、检测灵敏度高和易于实时监测等优点,便于临床实验室推广使用,在生物化学、蛋白质组学和药物筛选等领域具有广阔的应用前景。
8.在疾病早期诊断中,一种位点甲基化指标并不具有代表性,往往需要通过多个指标联合诊断,这就需要建立灵敏、高效的多位点同时检测技术。目前国内外虽已有光电化学传感技术用于rna甲基化定量分析的报道,但rna中m5c 和m6a双重分析的光电化学传感技术未见报道。
9.因此,建立一种具有临床实验室推广应用前景的rna甲基化分析技术,已成为代谢性疾病、自身免疫性疾病和肿瘤等基因表观紊乱疾病临床检测的迫切需求。
10.通过上述分析,现有技术存在的问题及缺陷为:
11.(1)现有基于免疫共沉淀和全基因组测序建立的rna甲基化测序分析技术,由于分辨率仅约100个bp,测序长度受限,且易受跨越残基的大峰影响;测试结果受测序深度的影响,文库构建过程复杂;同时,由于检测后庞大的数据处理和对检测设备的特殊要求,导致其难以在临床实验室推广应用。
12.(2)现有基于高效液相色谱和质谱技术为基础建立的液相色谱
‑
串联质谱技术,由于分析前需进行rna提取和多重洗脱,并需采用核酸外切酶将rna水解为单核苷酸,因而存在检测过程繁冗、洗脱纯度干扰和酶解不完全等缺陷。
13.(3)现有基于特定位点切割与夹板提取结合薄层色谱技术而构建的 scarlet分析技术,由于涉及放射性污染,以及同样存在酶解效率和测序后庞大的数据处理等缺陷,而限制了其在临床医学实验室的开展。
14.(4)目前国内外虽已有光电化学传感技术用于rna甲基化定量分析的报道,但rna中m5c和m6a双重分析的光电化学传感技术未见报道。
15.解决以上问题及缺陷的难度为:建立一种同时满足高灵敏、操作简便、干扰小、设备要求低、易于临床推广使用的分析rna甲基化的技术是最大的难度。
16.解决以上问题及缺陷的意义为:m6a和m5c修饰水平的偏倚与肿瘤、代谢性疾病、神经缺陷、心血管疾病和异常分化等密切相关。rna甲基化分析已成为自身免疫性疾病、代谢性疾病等基因表观紊乱疾病临床诊断,和肿瘤早期预警与去甲基化治疗方案选择的新手段,因此构建rna甲基化临床适宜检测技术研究具有重大意义。
技术实现要素:
17.针对现有技术存在的问题,本发明提供了一种用于肿瘤早期预警检测的双重分析的逻辑光电化学传感器,还涉及一种rna中m5c和m6a双重分析的逻辑光电化学传感方法。
18.本发明是这样实现的,一种用于肿瘤早期预警检测的双重分析的逻辑光电化学传感器,包括:
19.不同浓度的m5c靶序列与10
‑7m m6a靶序列共同孵育后的dna步行器1,用于在同一根电极表面检测光电信号;
20.当不同浓度的m6a靶序列与10
‑7m m5c靶序列共同孵育后的dna步行器2,用于在同一根电极表面检测光电信号。
21.所述dna步行器1,用于在同一根电极表面检测的465nm波长下的光电信号;
22.所述dna步行器2,用于在同一根电极表面检测的625nm波长下的光电信号。
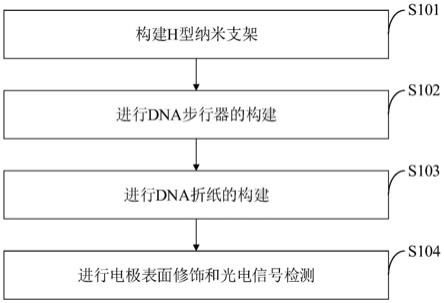
23.本发明另一目的在于提供一种rna中m5c和m6a双重分析的逻辑光电化学传感方法,所述rna中m5c和m6a双重分析的逻辑光电化学传感方法包括以下步骤:
24.步骤一,构建h型纳米支架;
25.步骤二,进行dna步行器的构建;
26.步骤三,进行dna折纸的构建;
27.步骤四,进行电极表面修饰和光电信号检测。
28.进一步,步骤一中,所述构建h型纳米支架,包括:
29.用四条寡核苷酸序列构建h型纳米支架;
30.配置tm杂交缓冲液,用该缓冲液稀释a、b、a1、b1,等比杂交使其终浓度为2μm,快速退火得到h型结构,并用15%的聚丙烯酰胺凝胶电泳在150v 的电压下验证其是否正确形成;其中,所述tm杂交缓冲液由10mm tris
‑
盐酸、1mm edta以及12.5mm氯化镁组成。
31.进一步,所述退火,包括:
32.于90℃10min,迅速退火到4℃持续0.5h以上,取出放入4℃储存备用。
33.进一步,所述a的核苷酸序列如seq id no:1所示,所述b的核苷酸序列如seq id no:2所示,所述a1的核苷酸序列如seq id no:3所示,所述b1的核苷酸序列如seq id no:4所示。
34.进一步,步骤二中,所述dna步行器的构建,包括:
35.用链霉亲和素标记的fe3o4磁珠作为支撑基体构建dna步行器,生物素标记l1和s1链,将s1和outputa在37℃杂交2h形成双链结构;
36.用结合缓冲液清洗磁珠三次,将磁珠重悬于结合缓冲液中,将l1和 s1/outputa按照一定比例加入一定量的磁珠中,37℃振荡混合40min,使磁珠与 l1和s1充分反应结合,并用tm缓冲液清洗备用;l2、s2和磁珠的连接体的制备方法同理。
37.进一步,所述l1的核苷酸序列如seq id no:5所示,所述s1的核苷酸序列如seq id no:6所示,所述outputa的核苷酸序列如seq id no:7所示,所述l2的核苷酸序列如seq id no:8所示,所述s2的核苷酸序列如seq idno:9所示。
38.进一步,步骤三中,所述dna折纸的构建,包括:
39.用xa,xb,x2,x3,x4五种寡核苷酸链制备x型结构作为dna折纸的支架,制备程序与h型结构一样,杂交终浓度也为2μm,同样用15%的聚丙烯酰胺凝胶电泳验证结果;
40.用x型结构进行滚环扩增,在滚环扩增出的链上用staple1,staple2,staple3 构建dna折纸结构,将ptc
‑
nh2和mb分别嵌入dna折纸结构中作为光电信号介质;
41.将ptc
‑
nh2嵌入dna折纸a中,mb嵌入dna折纸b中,产生的信号分别对应m6a和m5c靶序列,分别在465nm和625nm的波长下测量光电流。
42.进一步,所述xa的核苷酸序列如seq id no:10所示,所述xb的核苷酸序列如seq id no:11所示,所述x2的核苷酸序列如seq id no:12所示,所述 x3的核苷酸序列如seq id no:13所示,所述x4的核苷酸序列如seq id no: 14所示,所述staple1的核苷酸序列如seq id no:15所示,所述staple2的核苷酸序列如seq id no:16所示,所述staple3的核苷酸序列如seq id no:17所示。
43.进一步,步骤四中,所述电极表面修饰和光电信号检测,包括:
44.将巯基修饰的捕获探针通过金
‑
硫键固定在电沉积后的玻碳电极表面;
45.将m6a和m5c靶序列与h型结构孵育;在dna步行器1中加入移动链w1,当 m6a靶序列存在时将a链置换下来,a和w1与l1共同组成步行器的足,f1链是步行器的引发剂,在f1加入
其中后,步行器开始运行,将磁珠上的outputa置换下来,通过少量的a即可获得大量的outputa达到信号放大的作用。
46.在dna步行器2中加入移动链w2,当m5c靶序列存在时将b链置换下来,b 和w2与l2共同组成步行器的足,f2链是步行器的引发剂,在f2加入其中后,步行器开始运行,将磁珠上的outputb置换下来,通过少量的b即可获得大量的 outputb达到信号放大的作用。
47.电极表面的捕获探针与dna折纸支架的xa通过outputa连接起来,即可把含有光电信号介质的dna折纸捕获到电极表面在465nm波长下产生与m6a 靶序列浓度相关的光电信号;同理,当m5c靶序列存在时,在625nm波长下产生与m5c靶序列浓度相关的光电信号;当两者均存在时即产生两种信号,两者均不存在时即无信号产生。
48.进一步,所述w1的核苷酸序列如seq id no:18所示,所述f1的核苷酸序列如seq id no:19所示,所述w2的核苷酸序列如seq id no:20所示,所述f2的核苷酸序列如seq id no:21所示,所述outputb的核苷酸序列如 seq id no:22所示,phosphorylated linear dna的核苷酸序列如seq id no: 23所示,ligation template dna的核苷酸序列如seq id no:24所示。
49.结合上述的所有技术方案,本发明所具备的优点及积极效果为:本发明提供的rna中m5c和m6a双重分析的逻辑光电化学传感方法,检测灵敏度高、高效,具有临床实验室推广应用前景。实验结果表明,当不同浓度的m5c靶序列与10
‑7m m6a靶序列共同孵育后在同一根电极表面检测的465nm波长下的光电信号,从结果可看出,不同浓度的m5c靶序列不影响m6a靶序列的光电信号;当不同浓度的m6a靶序列与10
‑7m m5c靶序列共同孵育后在同一根电极表面检测的625nm波长下的光电信号,从结果可看出,不同浓度的m6a靶序列不影响 m5c靶序列的光电信号;故说明此体系具有能同时检测两种靶序列的能力。
附图说明
50.为了更清楚地说明本发明实施例的技术方案,下面将对本发明实施例中所需要使用的附图做简单的介绍,显而易见地,下面所描述的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下还可以根据这些附图获得其他的附图。
51.图1是本发明实施例提供的rna中m5c和m6a双重分析的逻辑光电化学传感方法流程图。
52.图2是本发明实施例提供的rna中m5c和m6a双重分析的逻辑光电化学传感方法原理图。
53.图3是本发明实施例提供的验证h型纳米支架是否正确形成的结果示意图;
54.图中:泳道1
‑
10分别为:a,b,a1,b1,a1+b,a1+b1,a1+b+a,a1+b1+b, a1+b1+a+b,marker。
55.图4是本发明实施例提供的x型结构和rca扩增产物的电泳图;
56.图中:泳道1
‑
3均是x型结构,泳道4是环状模板,泳道5
‑
7是rca扩增产物,泳道8是marker。
57.图5是本发明实施例提供的当不同浓度的m5c靶序列与10
‑
7m m6a靶序列共同孵育后在同一根电极表面检测的465nm波长下的光电信号示意图。
58.图6是本发明实施例提供的当不同浓度的m6a靶序列与10
‑
7m m5c靶序列共同孵育后在同一根电极表面检测的625nm波长下的光电信号示意图。
59.图7是本发明实施例提供的光电信号变化曲线图。
具体实施方式
60.为了使本发明的目的、技术方案及优点更加清楚明白,以下结合实施例,对本发明进行进一步详细说明。应当理解,此处所描述的具体实施例仅仅用以解释本发明,并不用于限定本发明。
61.针对现有技术存在的问题,本发明提供了一种rna中m5c和m6a双重分析的逻辑光电化学传感方法,下面结合附图对本发明作详细的描述。
62.本发明提供一种用于肿瘤早期预警检测的双重分析的逻辑光电化学传感器,包括:
63.不同浓度的m5c靶序列(10
‑6m,10
‑7m,10
‑8m,10
‑9m)与10
‑7m m6a靶序列共同孵育后的dna步行器1,用于在同一根电极表面检测光电信号;
64.当不同浓度的m6a靶序列(10
‑6m,10
‑7m,10
‑8m,10
‑9m)与10
‑7m m5c靶序列共同孵育后的dna步行器2,用于在同一根电极表面检测光电信号。
65.所述dna步行器1,用于在同一根电极表面检测的465nm波长下的光电信号;
66.所述dna步行器2,用于在同一根电极表面检测的625nm波长下的光电信号。
67.如图1所示,本发明实施例提供的rna中m5c和m6a双重分析的逻辑光电化学传感方法包括以下步骤:
68.s101,构建h型纳米支架;
69.s102,进行dna步行器的构建;
70.s103,进行dna折纸的构建;
71.s104,进行电极表面修饰和光电信号检测。
72.下面结合实施例对本发明的技术方案作进一步描述。
73.如图2所示,本发明实施例提供的rna中m5c和m6a双重分析的逻辑光电化学传感方法包括以下步骤:
74.1、构建h型纳米支架
75.用四条寡核苷酸序列构建h型纳米支架。具体过程如下,配置tm杂交缓冲液(10mm tris
‑
盐酸、1mm edta、12.5mm氯化镁),用该缓冲液稀释a、b、a1、b1,等比杂交使其终浓度为2μm,然后快速退火(90℃10分钟,迅速退火到4℃持续半小时以上,取出放入4℃储存备用)得到h型结构。用15%的聚丙烯酰胺凝胶电泳在150v的电压下验证其是否正确形成。结果如图3所示。
76.2、dna步行器(dna walker)的构建
77.用链霉亲和素标记的fe3o4磁珠作为支撑基体构建dna步行器。生物素标记l1和s1链,将s1和outputa在37℃杂交2小时形成双链结构。接下来,先用结合缓冲液清洗磁珠三次,然后将磁珠重悬于结合缓冲液中,将l1和s1/outputa按照一定比例加入一定量的磁珠中,37℃振荡混合40分钟,使磁珠与l1和s1充分反应结合,然后用tm缓冲液清洗备用。同理l2、s2和磁珠的连接体也是上述方法制备。
78.4、dna折纸的构建
79.用xa,xb,x2,x3,x4五种寡核苷酸链制备x型结构作为dna折纸的支架,制备程序与h型结构一样,杂交终浓度也为2μm,同样用15%的聚丙烯酰胺凝胶电泳验证结果。然后用x型结构进行滚环扩增,在滚环扩增出的链上用staple1,staple2,staple3构建dna折纸结构,然后将ptc
‑
nh2和mb分别嵌入dna折纸结构中作为光电信号介质。ptc
‑
nh2嵌入dna折纸a中,mb嵌入dna折纸b中,产生的信号分别对应m6a和m5c靶序列。分别在465nm和625nm的波长下测量光电流。
80.图4是x型结构和rca扩增产物的电泳图,泳道1
‑
3均是x型结构,泳道4是环状模板,泳道5
‑
7是rca扩增产物,泳道8是marker。
81.5、电极表面修饰和光电信号检测
82.将巯基修饰的捕获探针通过金
‑
硫键固定在电沉积后的玻碳电极表面。
83.将m6a和m5c靶序列与h型结构孵育。在dna步行器1中加入移动链w1,当m6a靶序列存在时将a链置换下来,a和w1与l1共同组成步行器的足,f1链是步行器的引发剂,在f1加入其中后,步行器开始运行,将磁珠上的outputa置换下来,通过少量的a即可获得大量的outputa达到了信号放大的作用。
84.在dna步行器2中加入移动链w2,当m5c靶序列存在时将b链置换下来,b和w2与l2共同组成步行器的足,f2链是步行器的引发剂,在f2加入其中后,步行器开始运行,将磁珠上的outputb置换下来,通过少量的b即可获得大量的outputb达到了信号放大的作用。
85.电极表面的捕获探针与dna折纸支架的xa通过outputa连接起来,即可把含有光电信号介质的dna折纸捕获到电极表面在465nm波长下产生与m6a靶序列浓度相关的光电信号。同理,当m5c靶序列存在时,在625nm波长下产生与m5c靶序列浓度相关的光电信号。当两者均存在时即产生两种信号,两者均不存在时即无信号产生。
86.图5是当不同浓度的m5c靶序列与10
‑7mm6a靶序列共同孵育后在同一根电极表面检测的465nm波长下的光电信号,从结果可看出,不同浓度的m5c靶序列不影响m6a靶序列的光电信号。
87.图6是当不同浓度的m6a靶序列与10
‑7mm5c靶序列共同孵育后在同一根电极表面检测的625nm波长下的光电信号,从结果可看出,不同浓度的m6a靶序列不影响m5c靶序列的光电信号。
88.故说明此体系具有能同时检测两种靶序列的能力。
89.本体系所用的所有序列如表1所示。
90.表1本体系所用的所有序列
91.s1gacatgtcgcatataggactagggccgtaagttagtgagaseqidno:1
92.outputataacttacggccctagtcctatatgcgacseqidno:2
93.f1tctcactaacttacggccctagtcctatatgcgacseqidno:3
94.l1t25
‑
tcaacatcagtseqidno:4
95.w1ctgataagcta
‑
t30
‑
gtcctatatgcgacatgtcseqidno:5
96.atagcttatcagactgatgttgaseqidno:6
97.bgtagcctatgcagcattgccagseqidno:7
98.a1gaactctggcaatgctggaatagtctgactacaacttaagctaseqidno:8
99.b1atcagtcaacatcagtctgttgtagtcagactattcaggctacseqidno:9
100.s2tcaacatcagtctgataagctagtcagtgaaacagtattgseqidno:10
101.f2caatactgtttcactgactagcttatcagactgatseqidno:11
102.l2t25
‑
ctggcaatgctseqidno:12
103.w2gcataggctac
‑
t30
‑
cttatcagactgatgttgaseqidno:13
104.outputbtgtttcactgactagcttatcagactgatseqidno:14
105.phosphorylatedlineardna:
106.atgcccagccctgtaagatgaagatagcgcacaatggtcggattctcaactcgtattctcaactcgtattctcaactcgtctctgccctgacttcseqidno:15
107.ligationtemplatednacagggctgggcatagaagtcagggcagagseqidno:16
108.staple1cagccctgtaagatgaagatagcgtctatgccseqidno:17
109.staple2ccctgactcacaatggtcggattccgtctctgseqidno:18
110.staple3tctcaacttcaactcgtattctcaactcgtatseqidno:19
111.xacctgccgtgctcaccgaatgctaggggtcgcatataggactseqidno:20
112.xbcctgccgtgctcaccgaatgctagggatcagtctgataagcseqidno:21
113.x2ggcaagctaatggtgagcacggcaggaaaaaaaaaacccccccccccagcctaagagttgagcaseqidno:22
114.x3ctcatgccatagtccattagcttgccaaaaaaaaaacccccccccccagcctaagagttgagcaseqidno:23
115.x4ccctagcattcggactatggcatgagaaaaaaaaaacccccagcctaagagttgagcaseqidno:24.
116.下面结合具体实验数据对本发明的技术效果作进一步描述。如图7所示。
117.左边曲线是无滚环扩增且未形成折纸结构时的光电信号图,由于仅存在x型引物结构,负载的光电信号介质过少,因此电流值仅为29na(465nm)和17na(625nm)。中间曲线为有滚环扩增但无折纸结构时的光电信号图,由于滚环扩增产物未形成折纸结构所以负载的光电信号介质较少,电流值仅为43na(465nm)和26na(625nm),而当折纸结构形成时,由于负载了大量光电介质电流强度增加如最右边曲线所示,电流值分别为69na(465nm)和34na(625nm)。结果表明,本实验所提出的信号放大策略是可行的。
118.以上所述,仅为本发明的具体实施方式,但本发明的保护范围并不局限于此,任何熟悉本技术领域的技术人员在本发明揭露的技术范围内,凡在本发明的精神和原则之内所作的任何修改、等同替换和改进等,都应涵盖在本发明的保护范围之内。
119.<110>中国人民解放军陆军军医大学
120.<120>一种rna中m5c和m6a双重分析的逻辑光电化学传感方法
121.<160>24
122.<210>1
123.<211>40
124.<212>dna
125.<213>人工序列(artificialsequence)
126.<400>1
127.gacatgtcgcatataggactagggccgtaagttagtgaga
128.<210>2
129.<211>29
130.<212>dna
131.<213>人工序列(artificial sequence)
132.<400>2
133.taacttacggccctagtcctatatgcgac
134.<210>3
135.<211>35
136.<212>dna
137.<213>人工序列(artificial sequence)
138.<400>3
139.tctcactaacttacggccctagtcctatatgcgac
140.<210>4
141.<211>13
142.<212>dna
143.<213>人工序列(artificial sequence)
144.<400>4
145.t25
‑
tcaacatcagt
146.<210>5
147.<211>33
148.<212>dna
149.<213>人工序列(artificial sequence)
150.<400>5
151.ctga taag cta
‑
t30
‑
gtcc tata tgcg acat gtc
152.<210>6
153.<211>22
154.<212>dna
155.<213>人工序列(artificial sequence)
156.<400>6
157.tagcttatcagactgatgttga
158.<210>7
159.<211>22
160.<212>dna
161.<213>人工序列(artificial sequence)
162.<400>7
163.gtagcctatgcagcattgccag
164.<210>8
165.<211>43
166.<212>dna
167.<213>人工序列(artificial sequence)
168.<400>8
169.gaactctggcaatgctggaatagtctgactacaacttaagcta
170.<210>9
171.<211>43
172.<212>dna
173.<213>人工序列(artificial sequence)
174.<400>9
175.atcagtcaacatcagtctgttgtagtcagactattcaggctac
176.<210>10
177.<211>40
178.<212>dna
179.<213>人工序列(artificial sequence)
180.<400>10
181.tcaacatcagtctgataagctagtcagtgaaacagtattg
182.<210>11
183.<211>35
184.<212>dna
185.<213>人工序列(artificial sequence)
186.<400>11
187.caatactgtttcactgactagcttatcagactgat
188.<210>12
189.<211>13
190.<212>dna
191.<213>人工序列(artificial sequence)
192.<400>12
193.t25
‑
ctggcaatgct
194.<210>13
195.<211>33
196.<212>dna
197.<213>人工序列(artificial sequence)
198.<400>13
199.gcataggctac
‑
t30
‑
cttatcagactgatgttga
200.<210>14
201.<211>29
202.<212>dna
203.<213>人工序列(artificial sequence)
204.<400>14
205.tgtttcactgactagcttatcagactgat
206.<210>15
207.<211>95
208.<212>dna
209.<213>人工序列(artificial sequence)
210.<400>15
211.atgcccagccctgtaagatgaagatagcgcacaatggtcggattctcaactcgtattctcaactcgtattctcaactc gtctctgccctgacttc
212.<210>16
213.<211>29
214.<212>dna
215.<213>人工序列(artificial sequence)
216.<400>16
217.caggg ctggg catag aagtc agggc agag
218.<210>17
219.<211>32
220.<212>dna
221.<213>人工序列(artificial sequence)
222.<400>17
223.cagccctgtaagatgaagatagcgtctatgcc
224.<210>18
225.<211>32
226.<212>dna
227.<213>人工序列(artificial sequence)
228.<400>18
229.ccctgactcacaatggtcggattccgtctctg
230.<210>19
231.<211>32
232.<212>dna
233.<213>人工序列(artificial sequence)
234.<400>19
235.tctcaacttcaactcgtattctcaactcgtat
236.<210>20
237.<211>41
238.<212>dna
239.<213>人工序列(artificial sequence)
240.<400>20
241.cctgccgtgctcaccgaatgctaggggtcgcatataggact
242.<210>21
243.<211>41
244.<212>dna
245.<213>人工序列(artificial sequence)
246.<400>21
247.cctgccgtgctcaccgaatgctagggatcagtctgataagc
248.<210>22
249.<211>64
250.<212>dna
251.<213>人工序列(artificial sequence)
252.<400>22
253.ggcaagctaatggtgagcacggcaggaaaaaaaaaacccccccccccagcctaagagttgagca <210>23
254.<211>64
255.<212>dna
256.<213>人工序列(artificial sequence)
257.<400>23
258.ctcatgccatagtccattagcttgccaaaaaaaaaacccccccccccagcctaagagttgagca
259.<210>24
260.<211>64
261.<212>dna
262.<213>人工序列(artificial sequence)
263.<400>24
264.ccctagcattcggactatggcatgagaaaaaaaaaacccccccccccagcctaagagttgagca
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1