一种滤除DNase高通量测序数据中DNA碱基倾向性偏差的方法与流程
本发明属于分子生物信息检测与分析领域,具体涉及一种有效提高DNase高通量测序数据的检测信息准确性的滤除DNase高通量测序数据中DNA碱基倾向性偏差的方法。
背景技术:
目前,DNA蛋白结合位点的检测主要采用染色质免疫共沉淀技术(Chromatin Immunoprecipitation,ChIP)。而将ChIP实验结果与高通量测序技术相结合的ChIP-Seq技术,则能有效地在全基因组范围内检测目的功能蛋白在DNA上的结合位点。ChIP-Seq的原理是:首先通过染色质免疫共沉淀技术(ChIP)利用与目的蛋白特异性结合的酶来富集结合有目的蛋白的DNA片段,并对其进行纯化与文库构建。然后对富集得到的DNA片段进行高通量测序,再将测序获得的数百万条读数序列精确定位到基因组上,从而获得全基因组范围内结合有目的蛋白的DNA区段信息,进而通过各种分析算法得到目的蛋白DNA结合位点。
然而,ChIP-Seq技术也有诸多不足之处,首先是富集目的蛋白的结合酶具有特异性,从而导致某些蛋白因找不到合适的特异结合酶而无法进行检测;其次,一次实验只能检测一种蛋白,耗时耗力,成本高,无法大规模使用;第三,更为重要的是,由于实验获取的与目的蛋白结合的DNA片段较长,测序时只能对其两端进行部分测序,由于测序区域并不是结合位点本身,因此,ChIP-Seq技术对DNA蛋白结合位点的检测分辨率无法达到单碱基。
针对上述问题,近几年产生了一种新的DNA蛋白结合位点检测技术--基于DNase高通测序信息的DNA蛋白结合位点检测技术,即DNase-Seq技术。DNase-Seq的原理是:首先利用DNase核酸剪切酶对DNA进行酶切处理。则没有DNA蛋白结合的DNA区域将被DNase核酸剪切酶随机地切断,而有DNA蛋白结合的DNA区域由于受到结合蛋白的阻碍特异性不被切断。随后,对酶切处理过的DNA片段进行纯化与文库构建,再进行测序,从而获得全基因组范围内DNase核酸剪切酶的酶切信息。在酶切信息中,蛋白结合位点处的酶切信息将特异性减弱,就像在DNA上留下一个个足迹一样,从而可以精确鉴定DNA结合蛋白在DNA分子上的结合位点。
与ChIP-Seq技术相比,DNase-Seq技术的优点非常突出。首先,由于不具有特异性,DNase-Seq可一次性在全基因组范围内同时检测多种DNA蛋白的结合位点;其次,由于一次性检测多种DNA蛋白的结合位点,DNase-Seq大幅提高了检测效率并降低了检测成本,使大规模进行DNA蛋白结合位点检测成为可能;第三,更为重要的是,由于测序起始位置就是酶切位置,DNase-Seq对DNA蛋白结合位点的检测分辨率可达单碱基。
然而,近期发现DNase核酸剪切酶在切割DNA时存在一定的DNA碱基倾向性,这将对 DNA蛋白结合位点的识别产生不利的影响。如何去除该倾向性已成为基于DNase-Seq的DNA蛋白结合位点识别的一个关键问题。
技术实现要素:
本发明的目的在于提供一种滤除DNase高通量测序数据中DNA碱基倾向性偏差的方法。
本发明的目的是这样实现的:
(1)DNase-Seq实验数据酶切位点区域DNA碱基获取
依据DNase-Seq实验数据在基因组中的位置,提取每一个实验数据对应酶切位点附近区域的DNA碱基。本发明选用酶切位点附近6个位点的碱基,即以酶切位点为中心,左右各取3个碱基。
(2)DNase-Seq实验数据DNA碱基倾向性获取
本发明选用酶切位点附近6个位点的碱基,每个碱基有A、C、G、T等4种取值,则6个位点碱基共有4096种碱基组合。通过统计整个DNase-Seq实验数据酶切位点处这4096种碱基组合出现的频次,即可获得DNase-Seq实验数据的DNA碱基倾向性。
(3)DNA碱基倾向性去除
设有m个蛋白结合位点,每个结合位点包含n个碱基,则:第i个结合位点的DNase检测信号为:[Si1,Si2,…,Sin]。其值和为:
考虑DNase的DNA碱基倾向性,则第i个结合位点第j列的DNase检测信号为:Sij=[(1-w)Pij+wBij]Ri。其中,Pij为第i个结合位点第j列处与DNA结合蛋白的蛋白结构相对应的DNase的固有切割概率,Bij为第i个结合位点第j列处与该处DNA碱基倾向性相对应的DNase的切割概率。Pij是稳定的,可用于DNA蛋白结合位点识别,而Bij是不稳定的,应予以滤除。
具体滤除方法如下:
其中,Sij,Ri可从实验数据中直接得到。Bij则根据前一步骤获取的DNase-Seq实验数据的DNA碱基倾向性得到。w为权值,取值范围为[0,1]之间,需要进一步确定。
对于m个蛋白结合位点,当权值w取不同值时,会得到不同的[Pi1,Pi2,…,Pin],1≤i≤m。设则当m个[Pi1,Pi2,…,Pin]与[P1,P2,...,Pn]之间的m个相关性值的中位值最大时, 此时的w值为最优值。
本发明的有益效果在于:通过所发明的方法可以精确地滤除DNase高通量测序数据中含有的DNA碱基倾向性偏差,以生成更加准确的DNase-Seq测序结果,从而为后续更高层次的应用分析提供数据保障。
附图说明
图1为DNase-Seq实验数据DNA碱基倾向性直方图。
图2为w权值的评价值变化曲线。
图3为本发明流程图。
具体实施方式
下面结合附图对本发明做进一步描述。
作为DNA蛋白结合位点检测的新技术,DNase-Seq技术具有众多突出的优点。由于不具有特异性,DNase-Seq可一次性在全基因组范围内同时检测多种DNA蛋白的结合位点;由于一次性检测多种DNA蛋白的结合位点,DNase-Seq大幅提高了检测效率并降低了检测成本,使大规模进行DNA蛋白结合位点检测成为可能;由于测序起始位置就是酶切位置,DNase-Seq对DNA蛋白结合位点的检测分辨率可达单碱基。
然而,近期发现DNase核酸剪切酶在切割DNA时存在一定的DNA碱基倾向性,这将对DNA蛋白结合位点的识别产生不利的影响。本发明即是针对该问题提出的一种滤除DNase高通量测序数据中DNA碱基倾向性偏差的方法。
1、DNase-Seq实验数据酶切位点区域DNA碱基获取
依据DNase-Seq实验数据在基因组中的位置,提取每一个实验数据对应酶切位点附近区域的DNA碱基。本发明选用酶切位点附近6个位点的碱基,即以酶切位点为中心,左右各取3个碱基。
2、DNase-Seq实验数据DNA碱基倾向性获取
本发明选用酶切位点附近6个位点的碱基,每个碱基有A、C、G、T等4种取值,则6个位点碱基共有4096种碱基组合。通过统计整个DNase-Seq实验数据酶切位点处这4096种碱基组合出现的频次,即可获得DNase-Seq实验数据的DNA碱基倾向性。
3、DNA碱基倾向性去除
设有m个蛋白结合位点,每个结合位点包含n个碱基,则:第i个结合位点的DNase检测信号为:[Si1,Si2,…,Sin]。其值和为:
考虑DNase的DNA碱基倾向性,则第i个结合位点第j列的DNase检测信号为: Sij=[(1-w)Pij+wBij]Ri。其中,Pij为第i个结合位点第j列处与DNA结合蛋白的蛋白结构相对应的DNase的固有切割概率,Bij为第i个结合位点第j列处与该处DNA碱基倾向性相对应的DNase的切割概率。Pij是稳定的,可用于DNA蛋白结合位点识别,而Bij是不稳定的,应予以滤除。
具体滤除方法如下:
其中,Sij,Ri可从实验数据中直接得到。Bij则根据前一步骤获取的DNase-Seq实验数据的DNA碱基倾向性得到。w为权值,取值范围为[0,1]之间,通过下述方法确定:
对于m个蛋白结合位点,当权值w取不同值时,会得到不同的[Pi1,Pi2,…,Pin],1≤i≤m。设则当m个[Pi1,Pi2,…,Pin]与[P1,P2,...,Pn]之间的m个相关性值的中位值最大时,此时的w值为最优值。
4、实验验证
从UCSC国际生物信息网站下载人类基因组碱基序列数据,以及国际ENCODE计划UW大学测得的人类K562细胞系DNase-Seq测序数据和NFYA转录因子ChIP-Seq测序数据。
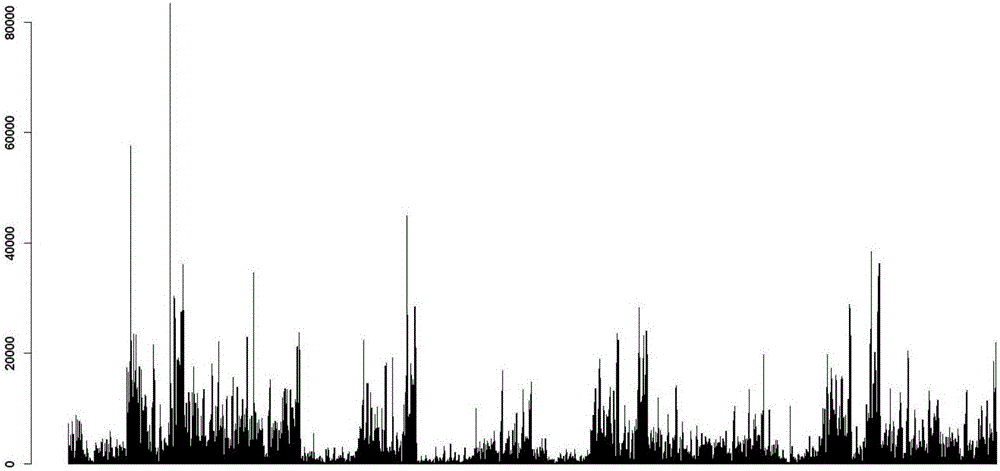
根据每个DNase-Seq测序数据酶切位点在人类基因组中的位置,提取附近6个位点的碱基,即以酶切位点为中心,左右各取3个碱基。统计酶切位点处4096种碱基组合出现的频次,获得DNase-Seq实验数据的DNA碱基倾向性。该倾向性的直方图如图1所示(横轴为碱基组合,纵轴为频次)。由图1可见,DNase-Seq实验数据存在明显的DNA碱基倾向性。
根据NFYA转录因子的ChIP-Seq测序数据,识别出953个NFYA蛋白结合位点。每个结合位点包含201个碱基。
利用本发明方法对DNase-Seq实验数据进行DNA碱基倾向性滤除。当w取某一权值时,每个结合位点滤除DNA碱基倾向性的DNase检测信号为[Pi1,Pi2,…,Pin],1≤i≤953。计算每个结合位点[Pi1,Pi2,…,Pin]与[P1,P2,...,Pn]之间的Pearson相关值,这里n取值为201。选取953个相关值的中位值作为该w值是否优异的评价值。让w值由0到1变化,获得如图2所示的w值的评价值变化曲线(横轴为w值,纵轴评价值)。由图2可见,当w值为0.15时,评价值达到最大并不再增加,此时的w值应为最优值,并进而得到与之对应的滤除DNA碱基倾向性的DNase-Seq检测信息。
作为DNA蛋白结合位点检测的新技术,DNase-Seq技术具有突出优点。由于不具有特异性,DNase-Seq可一次性在全基因组范围内同时检测多种DNA蛋白的结合位点;由于一次性检测多种DNA蛋白的结合位点,DNase-Seq大幅提高了检测效率并降低了检测成本,使大规模进行DNA蛋白结合位点检测成为可能;由于测序起始位置就是酶切位置,DNase-Seq对DNA蛋白结合位点的检测分辨率可达单碱基。然而,DNase核酸剪切酶在切割DNA时存在一定的DNA碱基倾向性,这将对DNA蛋白结合位点的识别产生不利的影响。本发明即是针对该问题提出的一种滤除DNase高通量测序数据中DNA碱基倾向性偏差的方法。
- 还没有人留言评论。精彩留言会获得点赞!
- 一种构建Hi‑C高通量测序文库的方法与制造工艺
- 桑葚病原菌高通量鉴定及种属分类方法及其应用与制造工艺
- 基于高通量测序技术检测乳腺癌/卵巢癌易感基因的多重PCR特异性引物、试剂盒及方法与制造工艺
- 基于高通量测序检测人EGFR基因变异的质控方法及试剂盒与制造工艺
- 基于高通量测序技术的小麦病原微生物检测方法及其应用与制造工艺
- 一种基于二代高通量测序技术的地中海贫血症的基因检测试剂盒的制造方法与工艺
- 一种基于集群的高通量数据分析方法与制造工艺
- 高通量双折射干涉成像光谱仪装置及其成像方法与制造工艺
- 高通量测序检测人循环肿瘤DNA EGFR基因的捕获探针及试剂盒的制造方法与工艺
- 一种高通量测序检测无创亲子鉴定的方法与制造工艺