一种知识图谱三元组置信度评价方法与流程
[0001]
本发明涉及一种知识图谱三元组置信度评价方法,更具体的说,尤其涉及一种包含评估阶段、融合阶段和校验阶段的知识图谱三元组置信度评价方法。
背景技术:
[0002]
不同靶点、药物作为实体,靶点、药物之间的相互作用作为关系,将相关知识以属性的形式存储于实体和关系中,相互交织形成一个巨大的图谱,并支持查询、推理、智能分析等功能,该图谱就被称为“药物-靶点知识图谱(drug-target knowledge graph,dt kg)”。dt kg在有效揭示药物-靶点之间复杂的物理、生物化学作用规律,发现药物-靶点之间尚未发现的隐含作用关系,进而发现新型药物或者开发现有药物的新用途是生物医药领域知识图谱研究的重要方向。
[0003]
知识图谱在构建过程中难免出现错误。为了发现知识图谱中的错误,提高知识图谱的质量,进而提升知识驱动的学习任务的性能,学界引入知识图谱三元组置信度的概念。知识图谱三元组置信度(kg triple trustworthiness),用于衡量三元组所表达知识的真实程度。知识图谱三元组置信度的取值范围为[0,1],值越接近0表示该三元组是错误的概率越大,反之,值越接近1则表示该三元组是真实的概率越大。
[0004]
现有的知识图谱三元组置信度评价方法可以概括为3类,分类原则是按照知识图谱三元组置信度评价方法的适用阶段来划分的,如图1中1、2和3所示。第一类置信度评价方法用于“从文本数据中抽取三元组”的过程,典型的案例有:德国马克思普朗克信息研究中心的knowlife知识库。第二类置信度评价方法用于embedding过程,embedding旨在将所有实体和关系编码成连续的向量空间。在embedding过程中进行置信度评价并剔除数据噪声是近年科研人员研究的热点,典型的方法有:scef(a novel support-confidence-aware kg embedding framework)、ckrl(a novel confidence-aware knowledge representation learning framework)、transt(a novel translating embedding learning approach with triple trustiness)等。第三类置信度评价方法直接对三元组进行评估,可以衡量知识推理得到的三元组的可靠性,同时也适用于动态知识库的置信度评价。典型的方法有:kgttm(a knowledge graph triple trustworthiness measurement model)、ctranse(knowledge graph embedding on uncertain knowledge graphs by using adapting confidence-margin-based loss function for translation-based models)等。
[0005]
现有的知识图谱三元组置信度评价方法如表1所示,、列举了7种方法:
[0006]
表1
[0007]
方法名称适用阶段年份knowlife从文本中提取实体及关系2015scefembedding2019kgttm三元组2019
transtembedding2019ckrlembedding2018confgcn节点属性预测2019ctranseembedding2019
[0008]
(1)knowlife实现了一种通用且可扩展的自动构建生物医学知识库的方法,它从科学出版物、健康门户网站和在线社区资源中自动提取信息,并在自动信息提取过程中引入置信度评价规则,用于定量衡量抽取得到的实体及关系数据的可靠性,从而提高生物医学知识库的质量。
[0009]
(2)scef是一种支持置信度感知的知识图嵌入框架,该框架在传统的基于翻译模型的基础上,结合置信度构建能量函数,通过具有三重置信度(文本、知识图和三元组)的知识表示学习来实现知识图的完善和矫正。
[0010]
(3)kgttm是一个知识图谱三元组置信度的度量模型,它从实体层面、关系层面和知识图谱全局层面量化三元组的语义正确性和所表达事实的真实程度。
[0011]
(4)transt是一种基于实体类型、实体描述等信息计算三元组置信度的模型,它通过基于交叉熵的损失函数来优化模型,进而提高知识嵌入学习的性能。
[0012]
(5)ckrl是一种基于置信度的知识表示学习框架,它引入了基于结构信息的置信度的概念,通过使用三元组的实体、关系和实体间路径的向量信息构建能量方程,提升了知识表示学习和知识图谱噪声探测的效果。
[0013]
(6)confgcn模型用于“预测节点属性”任务的可靠性,可以用于评估图中节点标签的得分及其置信度。
[0014]
(7)ctranse是一种基于翻译的模型,它用于处理知识图在自动更新时引入的错误,该模型采用基于置信度的损失函数来完成对动态知识图的嵌入表示学习。
[0015]
但现有的知识图谱三元组置信度评价方法存在如下缺点:
[0016]
1、考虑因素不全面,置信度得分不可靠。现有置信度评价方法考虑了知识图谱全局层面、实体层面和关系层面的置信度影响因素,但是未将科研文献、数据来源两个因素考虑在内,这导致最终得到的置信度得分不可靠。
[0017]
2、计算复杂度高,可解释性差。现有方法通过机器学习模型评价三元组置信度(例如:kgttm基于rnn进行知识图谱全局层面的置信度评价,sematyp通过构建逻辑回归模型进行置信度评价),模型计算复杂度高,且可解释性差。
[0018]
3、置信度评价局限于embedding过程。现有的绝大多数置信度评价方法适用于embedding过程中,这些方法无法直接评价通过知识推理和自动化方法构建的三元组的质量。
技术实现要素:
[0019]
本发明为了克服上述技术问题的缺点,提供了一种知识图谱三元组置信度评价方法。
[0020]
本发明的知识图谱三元组置信度评价方法,包括评估阶段、融合阶段和校验阶段,其特征在于:所述评估阶段通过以下步骤来实现:
[0021]
a).实体层面评估;
[0022]
a-1).数据源角度对实体的评价,待评估的实体包括化合物、疾病、蛋白质、基因、通路、细胞系、药品、产品、靶点、酶、蛋白质-化合物共计11种,对于每种实体的数据源置信度n
r
参考关联开放数据云the linked open data cloud中的lod打分,对于没有进行lod打分的pubchem、rcsb pdb、drugbank和dto本体数据源分别给出5星、5星、5星和4星的打分;实体的数据源置信度n
r
的取值等于lod打分的星数,如果同一实体在2个或2个以上的数据源总出现,则其数据源置信度n
r
取最高打分值;
[0023]
a-2).文献共现角度对实体的评价,在文献库中查询与实体相关的文献,实体的文献共现角度置信度lca通过公式(1)进行求取:
[0024][0025]
其中,lca表示实体的文献共现角度置信度,n表示与实体相关的文献数目,f表示文献的影响因子,l为文献引用量,t为不同文献类别对应的打分值,i表示第i篇文献,α、β、θ表示权值;
[0026]
a-3).外链规模角度对实体的评价,实体的外链规模置信度n
l
用生物医药知识图谱中实体外部链接的数量表示,实体外链规模越大,实体数据的可靠性越高,通过实体外链个数衡量实体的可信性,实体的外链规模置信度n
l
等于实体的外链数目;
[0027]
a-4).文本描述角度对实体的评价,实体文本描述是对实体概念、类别、功能信息的描述,有文本描述的实体,它的数据可靠性更高;如果步骤的实体,如果a-1)中的数据源中存在相应实体的文字描述,则该实体的文本描述置信值d的取值为1,不存在则文本描述置信值d取值为0;
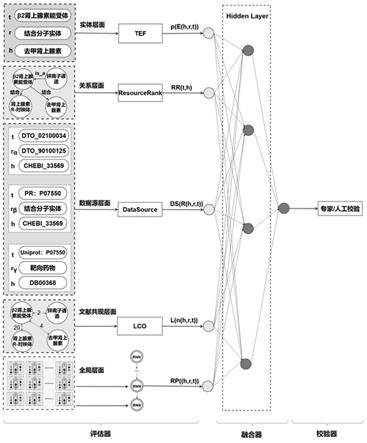
[0028]
a-5).实体重要性角度对实体的评价,在生物医药知识图谱中实体节点被链接的数量和质量直接决定了该节点在整个图谱中的重要性;采用pagerank算法来衡量某个实体在知识图谱中的重要性,来表征实体重要性置信度,pagerank算法如公式(2)所示:
[0029][0030]
其中,p1、p2、
…
、p
i
、
…
、p
n
表示知识图谱中的节点,表示待研究节点p
j
的入度,表示待研究节点p
j
的出度,n表示知识图谱中的节点数,表示节点p
j
的pagerank值,所有节点的pagerank值构成知识图的pagerank向量,q表示知识图中节点继续扩展的概率,其取值为0.5;
[0031]
a-6).实体的度的角度对实体的评价,实体节点的入度和出度反映了知识图谱中实体信息的富集程度和实体与其它实体间的关联强度;实体的度的角度的置信度n
s
通过公式(3)进行求取:
[0032]
n
s
=n
in
+n
out
ꢀꢀ
(3)
[0033]
其中,n
s
表示实体的度的角度的置信度,n
in
表示实体节点的入度,n
out
表示实体节点的出度;
[0034]
b).关系层面评估;
[0035]
b-1).数据源角度对关系层面的评价,对于生物医药知识图谱中实体间的关系,通
常用三元组(h,r,t)来表示,其中,h为头实体,t为尾实体,,r为实体间关系;如果三元组数据来自高质量的数据源,则表明两个实体间的关联性很强,三元组信息的置信度很高;关系层面的数据源置信度n
′
in
参考关联开放数据云the linked open data cloud中的lod打分,对于没有进行lod打分的pubchem、rcsb pdb、drugbank和dto本体数据源分别给出5星、5星、5星和4星的打分;关系层面的数据源置信度n
′
in
的取值等于lod打分的星数,如果同一实体在2个或2个以上的数据源总出现,则其关系层面的数据源置信度n
′
in
取最高打分值;
[0036]
b-2).文献共现角度对关系层面的评价,在文献库中查询与实体对(h,t)相关的文献,实体对(h,t)的文献共现角度置信度lca
′
通过公式(4)进行求取:
[0037][0038]
其中,lca
′
表示实体对(h,t)的文献共现角度置信度,n
′
表示与实体对(h,t)相关的文献数目,f表示文献的影响因子,l为文献引用量,t为不同文献类别对应的打分值,i表示第i篇文献,α、β、θ表示权值;
[0039]
b-3).对实体间已知关系层面的评价,生物医药知识图谱构建过程中创建的实体关系,称为已知关系,采用resourcerank算法衡量已知关系的置信度,得到已知关系的置信度resourcerank;
[0040]
b-4).对实体间未知关系层面的评价,现有知识图谱中不存在的、需要通过推理得到的实体关系,称为未知关系;采用ksp算法衡量未知关系的置信度,通过图谱中两个实体间的前k条最短路径数目来评价关系强度,得到未知关系的置信度ksp;
[0041]
c).知识图谱全局层面评估;
[0042]
通过n
total
/m对知识图谱全局层面进行评估,以衡量知识图谱全局层面的信息密度,进而评估整个知识图谱所含数据的可信度;其中n
total
为知识图谱所有实体节点的总度数,总度数为所有实体节点入度和出度的和,m为知识图谱中实体节点的总数。
[0043]
本发明的知识图谱三元组置信度评价方法,所述融合阶段通过以下步骤来实现:结合生物医药知识图谱数据质量情况、药物-靶点关系预测任务因素,生物医药知识图谱三元组置信值通过公式5求取:
[0044][0045]
其中,confidence表示三元组置信值,其为正数,置信值越大,置信度越高;confidence置信值由实体层面、关系层面和知识图谱全局三个层面的11个置信度评估器加权得到,最终将置信值归一化到[0,1]区间;在指定的知识图谱中,若置信值小于阈值0.6,则表明该三元组的数据是不可靠的。
[0046]
本发明的知识图谱三元组置信度评价方法,所述校验阶段用于评估知识图谱三元组最终的置信值是否合理,进而优化评估器及融合器的设计;校验器包含专家抽样校验和自动化校验两种方法;专家抽样校验:专家抽样校验方法借助医药领域专家进行人工校验,专家校验的范围是:置信值得分处于[0.9,1]范围内、且三元组中包含现有药物或热门靶点的数据;专家校验的方法是:对三元组所涉及的药物、靶点进行研究,根据专业知识和经验
核定高置信值的三元组数据是否可靠;
[0047]
自动化校验:自动化校验方法是借助分子对接技术对三元组的置信值进行校验,自动化校验的范围是:置信值的范围是[0.6,0.9],随机采样其中10%的三元组;自动化校验的方法是:采用discovery studio 2018client中的libdock和gold打分函数对三元组涉及的药物-靶点数据进行分子对接计算,根据最终的打分值判断置信值是否可靠;
[0048]
校验阶段的结果会反馈给评估阶段和融合阶段,对于校验结果和置信值严重负相关的数据,深入调研其原因,进而对融合阶段各方法的权重进行调整,从而完善整套知识图谱三元组置信度评价方法。
[0049]
本发明的知识图谱三元组置信度评价方法,步骤a-2)和步骤b-2)中的文献库包括cas、patent、pubmed、wikipedia和doi,所述的取值α、β、θ的取值分别为0.7、0.2和0.1;不同文献类别对应的打分值t如表1所示:
[0050]
表1
[0051]
文献类别打分值cas1.0patent0.8pubmed1.0wikipedia0.5doi1.0。
[0052]
本发明的知识图谱三元组置信度评价方法,步骤b-3)中的已知关系对关系层面的评价过程中,采用resourcerank算法衡量已知关系的置信度;resourcerank算法用于刻画两个实体间的关联强度,该算法的思路是:如果实体对(h,t)之间的关联性很强,那么会有非常多的资源从头部实体h,通过所有关联路径传递到尾部实体t;具体通过以下步骤来实现:
[0053]
b-3-1).构建一个以头实体h为中心的有向图;
[0054]
b-3-2).利用公式(6)迭代计算图中的资源,直到其收敛,并计算尾实体t的资源保留值;
[0055][0056]
其中,m
t
是所有通向尾结点t的集合,od(e
i
)是节点e
i
的出度,bw
eit
是节点e
i
到尾结点t的带宽,带宽即路径数目;对于m
t
中每个节点e
i
,从节点e
i
到尾结点t转移的资源量为设每个节点的资源流都有相同的η概率可以直接跳转到随机节点,并且随机流向尾节点t的这部分资源是1/n,n是节点总个数;
[0057]
b-3-3).利用步骤b-3-2)中的r(t|h)、头节点h的入度id(h)、头结点h的出度od(h)、尾节点t的入度id(t)、尾节点t的出度od(t)、头结点到尾节点的深度dep,总计6个特征构造特征向量v,并通过激活函数将v转换成概率值rr(h,t),rr(h,t)即为置信度resourcerank,用于衡量头结点h和尾节点t之间存在一个或多个关系的可能性,其通过公
式(7)进行求取:
[0058][0059]
其中,φ是非线性激活函数,w
i
和b
i
是训练时可以调节的参数矩阵,rr(h,t)值的范围在[0,1]之间,其值越接近1表明h和t之间越有可能存在关系。
[0060]
本发明的有益效果是:本发明的知识图谱三元组置信度评价方法,首先,评估阶段从实体、关系和知识图谱全局三个层面,数据源、文献共现、外链规模、文本描述、实体重要性、实体的度多个角度对三元组的置信度进行评价,得到11个置信度,然后,校验阶段,将11个置信度评估器加权融合得到最终的置信值,校验阶段对最终的置信值的合理性进行校验,并将校验的结果反馈给评估阶段和融合阶段,用于优化评估阶段的设计,或者调整融合阶段的权重。可见,本本发明的知识图谱三元组置信度评价方法,可高效、快速、大规模的发掘知识图谱数据中的错误,进而提升整个知识图谱系统的数据质量;可以对链接预测、关系推理等机器学习任务的结果进行数据可靠性校验。
附图说明
[0061]
图1为三类置信度评价方法的适用阶段的示意图;
[0062]
图2为本发明的知识图谱三元组置信度评价方法原理架构图;
[0063]
图3为本发明中resourcerank算法原理示意图;
[0064]
图4为评估阶段计算置信度的典型案例示意图。
具体实施方式
[0065]
下面结合附图与实施例对本发明作进一步说明。
[0066]
如图2所示,给出了本发明的知识图谱三元组置信度评价方法原理架构图,本发明的知识图谱三元组置信度评价方法用于评估生物医药知识图谱中三元组的可靠程度,知识图谱三元组置信度评价方法包含:评估器、融合器和校验器三个阶段,知识图谱三元组数据经过评估器会产生多个置信值打分,融合器按照一定权重将多个打分融合,生成最终的置信值。校验器会对最终的置信值的合理性进行校验,并将校验的结果反馈给评估器和融合器,用于优化评估器的设计,或者调整融合器的权重。
[0067]
评估器从实体、关系和知识图谱全局等三个层面,数据源、文献共现、外链规模、文本描述、实体重要性、实体的度多个角度对三元组的置信度进行评价,具体方法如表2所示:
[0068]
表2
[0069]
[0070][0071]
本发明的知识图谱三元组置信度评价方法,包括评估阶段、融合阶段和校验阶段,其特征在于:所述评估阶段通过以下步骤来实现:
[0072]
a).实体层面评估;
[0073]
a-1).数据源角度对实体的评价,待评估的实体包括化合物、疾病、蛋白质、基因、通路、细胞系、药品、产品、靶点、酶、蛋白质-化合物共计11种,对于每种实体的数据源置信度n
r
参考关联开放数据云the linked open data cloud中的lod打分,对于没有进行lod打分的pubchem、rcsb pdb、drugbank和dto本体数据源分别给出5星、5星、5星和4星的打分;实体的数据源置信度n
r
的取值等于lod打分的星数,如果同一实体在2个或2个以上的数据源总出现,则其数据源置信度n
r
取最高打分值;
[0074]
如表3所示,给出了lod数据源质量评价表:
[0075]
[0076][0077]
a-2).文献共现角度对实体的评价,在文献库中查询与实体相关的文献,实体的文献共现角度置信度lca通过公式(1)进行求取:
[0078][0079]
其中,lca表示实体的文献共现角度置信度,n表示与实体相关的文献数目,f表示文献的影响因子,l为文献引用量,t为不同文献类别对应的打分值,i表示第i篇文献,α、β、θ表示权值;
[0080]
该步骤中,文献库包括cas、patent、pubmed、wikipedia和doi,所述的取值α、β、θ的取值分别为0.7、0.2和0.1;不同文献类别对应的打分值t如表1所示:
[0081]
表1
[0082][0083][0084]
a-3).外链规模角度对实体的评价,实体的外链规模置信度n
l
用生物医药知识图谱中实体外部链接的数量表示,实体外链规模越大,实体数据的可靠性越高,通过实体外链个数衡量实体的可信性,实体的外链规模置信度n
l
等于实体的外链数目;
[0085]
a-4).文本描述角度对实体的评价,实体文本描述是对实体概念、类别、功能信息的描述,有文本描述的实体,它的数据可靠性更高;如果步骤的实体,如果a-1)中的数据源中存在相应实体的文字描述,则该实体的文本描述置信值d的取值为1,不存在则文本描述置信值d取值为0;
[0086]
a-5).实体重要性角度对实体的评价,在生物医药知识图谱中实体节点被链接的数量和质量直接决定了该节点在整个图谱中的重要性;采用pagerank算法来衡量某个实体在知识图谱中的重要性,来表征实体重要性置信度,pagerank算法如公式(2)所示:
[0087][0088]
其中,p1、p2、
…
、p
i
、
…
、p
n
表示知识图谱中的节点,表示待研究节点p
j
的入度,表示待研究节点p
j
的出度,n表示知识图谱中的节点数,表示节点p
j
的pagerank值,所有节点的pagerank值构成知识图的pagerank向量,q表示知识图中节点继续扩展的概率,其取值为0.5;
[0089]
a-6).实体的度的角度对实体的评价,实体节点的入度和出度反映了知识图谱中实体信息的富集程度和实体与其它实体间的关联强度;实体的度的角度的置信度n
s
通过公式(3)进行求取:
[0090]
n
s
=n
in
+n
out
ꢀꢀ
(3)
[0091]
其中,n
s
表示实体的度的角度的置信度,n
in
表示实体节点的入度,n
out
表示实体节点的出度;
[0092]
b).关系层面评估;
[0093]
b-1).数据源角度对关系层面的评价,对于生物医药知识图谱中实体间的关系,通常用三元组(h,r,t)来表示,其中,h为头实体,t为尾实体,r为实体间关系;如果三元组数据来自高质量的数据源,则表明两个实体间的关联性很强,三元组信息的置信度很高;关系层面的数据源置信度n
′
in
参考关联开放数据云the linked open data cloud中的lod打分,对于没有进行lod打分的pubchem、rcsb pdb、drugbank和dto本体数据源分别给出5星、5星、5星和4星的打分;关系层面的数据源置信度n
′
in
的取值等于lod打分的星数,如果同一实体在2个或2个以上的数据源总出现,则其关系层面的数据源置信度n
′
in
取最高打分值;
[0094]
b-2).文献共现角度对关系层面的评价,在文献库中查询与实体对(h,t)相关的文献,实体对(h,t)的文献共现角度置信度lca
′
通过公式(4)进行求取:
[0095][0096]
其中,lca
′
表示实体对(h,t)的文献共现角度置信度,n
′
表示与实体对(h,t)相关的文献数目,f表示文献的影响因子,l为文献引用量,t为不同文献类别对应的打分值,i表示第i篇文献,α、β、θ表示权值;
[0097]
b-3).对实体间已知关系层面的评价,生物医药知识图谱构建过程中创建的实体关系,称为已知关系,采用resourcerank算法衡量已知关系的置信度,得到已知关系的置信度resourcerank;
[0098]
如图3所示,给出了本发明中resourcerank算法原理示意图,从节点(实体)a到节点e的边(关系)非常密集,这说明(a,e)两个实体间存在较高的关联强度,实体a和e之间存在关系。但是,节点g和节点f之间没有直接关联的边,这说明实体g和f之间不存在关系。
[0099]
该步骤中,采用resourcerank算法衡量已知关系的置信度;resourcerank算法用于刻画两个实体间的关联强度,该算法的思路是:如果实体对(h,t)之间的关联性很强,那么会有非常多的资源从头部实体h,通过所有关联路径传递到尾部实体t;具体通过以下步骤来实现:
[0100]
b-3-1).构建一个以头实体h为中心的有向图;
[0101]
b-3-2).利用公式(6)迭代计算图中的资源,直到其收敛,并计算尾实体t的资源保留值;
[0102][0103]
其中,m
t
是所有通向尾结点t的集合,od(e
i
)是节点e
i
的出度,是节点e
i
到尾结点t的带宽,带宽即路径数目;对于m
t
中每个节点e
i
,从节点e
i
到尾结点t转移的资源量为设每个节点的资源流都有相同的η概率可以直接跳转到随机节点,并且随机流向尾节点t的这部分资源是1/n,n是节点总个数;
[0104]
b-3-3).利用步骤b-3-2)中的r(t|h)、头节点h的入度id(h)、头结点h的出度od(h)、尾节点t的入度id(t)、尾节点t的出度od(t)、头结点到尾节点的深度dep,总计6个特征构造特征向量v,并通过激活函数将v转换成概率值rr(h,t),rr(h,t)即为置信度resourcerank,用于衡量头结点h和尾节点t之间存在一个或多个关系的可能性,其通过公式(7)进行求取:
[0105][0106]
其中,φ是非线性激活函数,w
i
和b
i
是训练时可以调节的参数矩阵,rr(h,t)值的范围在[0,1]之间,其值越接近1表明h和t之间越有可能存在关系。
[0107]
b-4).对实体间未知关系层面的评价,现有知识图谱中不存在的、需要通过推理得到的实体关系,称为未知关系;采用ksp算法衡量未知关系的置信度,通过图谱中两个实体间的前k条最短路径数目来评价关系强度,得到未知关系的置信度ksp;
[0108]
c).知识图谱全局层面评估;
[0109]
通过n
total
/m对知识图谱全局层面进行评估,以衡量知识图谱全局层面的信息密度,进而评估整个知识图谱所含数据的可信度;其中n
total
为知识图谱所有实体节点的总度数,总度数为所有实体节点入度和出度的和,m为知识图谱中实体节点的总数。
[0110]
融合阶段通过以下步骤来实现:结合生物医药知识图谱数据质量情况、药物-靶点关系预测任务因素,生物医药知识图谱三元组置信值通过公式5求取:
[0111][0112]
其中,confidence表示三元组置信值,其为正数,置信值越大,置信度越高;confidence置信值由实体层面、关系层面和知识图谱全局三个层面的11个置信度评估器加权得到,最终将置信值归一化到[0,1]区间;在指定的知识图谱中,若置信值小于阈值0.6,则表明该三元组的数据是不可靠的。
[0113]
校验阶段用于评估知识图谱三元组最终的置信值是否合理,进而优化评估器及融合器的设计;校验器包含专家抽样校验和自动化校验两种方法;专家抽样校验:专家抽样校验方法借助医药领域专家进行人工校验,专家校验的范围是:置信值得分处于[0.9,1]范围内、且三元组中包含现有药物或热门靶点的数据;专家校验的方法是:对三元组所涉及的药物、靶点进行研究,根据专业知识和经验核定高置信值的三元组数据是否可靠;
[0114]
自动化校验:自动化校验方法是借助分子对接技术对三元组的置信值进行校验,自动化校验的范围是:置信值的范围是[0.6,0.9],随机采样其中10%的三元组;自动化校验的方法是:采用discovery studio 2018client中的libdock和gold打分函数对三元组涉及的药物-靶点数据进行分子对接计算,根据最终的打分值判断置信值是否可靠;
[0115]
校验阶段的结果会反馈给评估阶段和融合阶段,对于校验结果和置信值严重负相关的数据,深入调研其原因,进而对融合阶段的权重进行调整,从而完善整套知识图谱三元组置信度评价方法。
[0116]
如图4所示,给出了评估阶段计算置信度的典型案例示意图,以(去甲肾上腺素,结合分子实体,β2肾上腺素能受体)三元组为例,简述评估器计算置信度的过程:实体层面,用基于平移的能量函数算法(tef)计算去甲肾上腺素和β2肾上腺素能受体存在结合关系的可能性。首先计算(去甲肾上腺素,结合分子实体,β2肾上腺素能受体)三元组的能量函数,以实现实体和关系的低维分布式表示。然后利用sigmoid函数将能量函数转换成实体对(去甲肾上腺素,β2肾上腺素能受体)构成结合分子实体关系的概率,通过得到的概率值来衡量两个实体存在结合关系的可能性。关系层面,用resourcerank算法计算药物和靶点的关系类型和关联强度。resourcerank算法以去甲肾上腺素和β2肾上腺素能受体为中心节点建立一个深度为2的子图,然后基于生成的子图计算由头实体(去甲肾上腺素)流入尾实体(β2肾上腺素能受体)的资源数量,如果实体对(去甲肾上腺素,β2肾上腺素能受体)之间的关联性很强,那么会有非常多的资源从头实体(去甲肾上腺素),通过所有关联路径传递到尾实体(β2肾上腺素能受体)。数据源层面,datasource算法,对三元组所在的药物靶点本体(drug target ontology)、蛋白质本体(protein ontology)、uniprot的数据源进行综合的质量评价。首先,在药物靶点本体、蛋白质本体和uniprot数据源中均包含(去甲肾上腺素,结合分子实体,β2肾上腺素能受体)三元组的数据。其次,这一数据在三个数据源中的质量是有差异的,datasource算法参照关联开放数据云(lod,the linked open data cloud)中对不同数据源质量的打分制定了《lod数据源质量评价表》,根据既定的规则实现数据源层面的置信度评价。文献共现层面,文献共现算法(lco)用文献共现次数定量标识实体对的关联强度。首先,算法筛选出包含(去甲肾上腺素,结合分子实体,β2肾上腺素能受体)三元组的文
献。然后,以文献数目为主,并参照文献的影响因子、引用量、期刊类别等信息按照一定权重进行加权计算,最终得到用于标识实体对关联强度的置信值。知识图结构层面,可达路径推理算法(rp)用于评价有向图中头尾实体之间存在的语义相关性和三元组之间蕴含的复杂推理模式。首先,考虑路径与目标三元组的语义相关性因素,基于语义距离的路径选择算法进行可达路径的选择。接着,将选定的可达路径映射到一个低维向量,利用递归神经网络(rnn)得到最终的输出向量,它可以表示每条路径的语义信息。最后,将向量进行非线性处理得到值rp((h,r,t)),用来表示知识图谱中图结构层面的置信度。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1