一种减轻松枯梢病病原菌危害寄主植物的真菌群落结构调控方法与流程
本发明涉及防治松枯梢病的方法,尤其涉及一种基于内生真菌群落结构调控减轻松枯梢病危害寄主植物的方法,属于松枯梢病的生物防治领域。
背景技术:
松枯梢病是世界范围内针叶树上最常见和分布最广的重要病害之一,病症主要表现为顶芽枯死、枯针、枯梢或丛枝等,在某些寄主上还能导致树干溃疡、流脂、坏死以及根茎腐烂和木材蓝变等,幼树和大树均可受害。松枯梢病已经使世界许多国家的松树人工林遭受了严重危害和重大损失,引起世界各国林业工作者的普遍关注。
松枯梢病(sphaeropsissapinea)引起的危害具有发病广、危害严重、病期久等特征,分布在全世界多个国家,主要发生国为南非、美国、新西兰、中国、澳大利亚、荷兰、智利等。病情严重者枝梢枯死,引起相当大的损失。据研究,因病砍伐未成熟病树造成的木材损失达28%,潜在的木材损失达55%。该病已在我国中南、西南、华南、华东、西北等10余个省份的松林中发生,对松树人工林造成了严重的危害,损失巨大,已将该病害列入中国重要的森林病害。
植物叶片内生存着大量不同性质的微生物,如真菌、细菌、酵母菌等。它们与植物病原菌具有相同的生态位,与植物长期共同生存,组成了独特的微生物群落,形成植物微生物生态体系,在植物—微生物复杂的微生态关系中发挥着重要作用。微生物种间关系主要包括:共生,抗生,竞争,寄生关系。
迄今为止,尚未有通过调控寄主植物内生微生物群落结构实现减轻松枯梢病病原菌对寄主植物的危害的生物防治报道。
技术实现要素:
本发明的主要目的是提供一种减轻松枯梢病病原菌危害寄主植物的真菌群落结构调控方法,通过调节寄主植物内生真菌群落结构,使真菌群落向着不利于松杉球壳孢(s.sapinea)生长的结构转变,使松枯梢病原菌难于增殖、扩散,有效降低其对寄主植物的危害。
为实现上述目的,本发明所采取的技术方案是:
本发明提供了一种减轻松枯梢病病原菌危害寄主植物的真菌群落结构调控方法,该方法包括:调节寄主植物内生真菌群落结构,其中,所述的调节寄主植物内生真菌群落结构的方式包括提高与枯梢病病原菌负相关的内生真菌相对丰度或降低与枯梢病病原菌正相关的内生真菌相对丰度;或者在提高与枯梢病病原菌负相关的内生真菌相对丰度的同时降低与枯梢病病原菌正相关的内生真菌相对丰度。
作为本发明一种优选的实施方式,本发明中所述的枯梢病病原菌是松杉球壳孢(s.sapinea);所述的寄主植物是松树,优选为赤松。
本发明中所述与枯梢病病原菌负相关的内生真菌包括但不限于:jattaeataediosa、出芽短梗霉(aureobasidiumpullulans);所述与枯梢病病原菌正相关的内生真菌包括但不限于:直孢圆盘菌(orbiliarectispora),phaeothecoideasp.,devriesiasp.,cryptococcussp.,orbiliasp.。
作为本发明一种优选的实施方式,所述提高与枯梢病病原菌负相关的内生真菌的相对丰度是将jattaeataediosa的相对丰度提升至0.1%~0.6%,将出芽短梗霉(aureobasidiumpullulans)的相对丰度提升至3%以上;优选的,将出芽短梗霉(aureobasidiumpullulans)的相对丰度提升至5-10%。
所述提高与枯梢病病原菌负相关的内生真菌的相对丰度的方式包括直接在寄主植物上喷施枯梢病病原菌负相关的内生真菌的发酵液或直接将枯梢病病原菌负相关的内生真菌的菌种接种到寄主植物上。
作为本发明一种优选的实施方式,所述降低与枯梢病病原菌正相关的内生真菌的相对丰度是将与枯梢病病原菌正相关的内生真菌的相对丰度降低到0.5%以下;所述降低与枯梢病病原菌正相关的内生真菌的相对丰度的方式包括喷洒拮抗菌剂。
作为本发明一种优选的实施方式,所述降低与枯梢病病原菌正相关的内生真菌相对丰度的方式还包括清理或修剪已感染松枯梢病的树枝;其中,所述清理或修剪已感染松枯梢病的树枝的时间为每年的8-9月。
本发明整体技术方案详述
本发明在昆嵛山林区设置的9块永久样地内进行松枯梢病病情调查,对样地内赤松的枯梢病发病情况进行统计。然后对赤松针叶进行采样,分别选取无病斑针叶及染病针叶,表面消毒后进行dna提取并测序,分析内生真菌群落结构。
本发明通过比较无病斑赤松与染病赤松的内生真菌群落结构,发现无病斑赤松内生真菌多样性指数高于染病赤松,其中与枯梢病病原菌松杉球壳孢呈负相关性(p<0.05)的真菌包括:jattaeataediosa,aureobasidiumpullulans等。当内生真菌j.taediosa相对丰度为0.1%~0.6%或a.pullulans相对丰度为5%~10%时,赤松针叶内枯梢病菌得到抑制,赤松呈未发病的状态。此外,与松杉球壳孢(s.sapinea)呈正相关性的真菌包括:orbiliarectispora,phaeothecoideasp.,devriesiasp.,cryptococcussp.,orbiliasp.等,当上述菌相对丰度低于0.5%时,不利于松杉球壳孢(s.sapinea)的生长传播。
本发明减轻松枯梢病病原菌危害松属植物的调控方法还包括:对寄主植物染病枯梢的清理;对寄主植物进行修枝。
其中所述清理修剪包括:每年8月至9月,在感染枯梢病严重的样地内,清理枯梢枯枝,避免病原菌二次传播;对多余枝条进行修剪,减少多余的蒸腾作用,改善树体的水分及养分条件,增强树势。
本发明技术方案与现有技术相比,具有以下有益效果是:
本发明通过对植物内生真菌群落结构的调控,使得内生真菌群落结构向着不利于松杉球壳孢生长、传播的方向转变,使之难于向周围扩散转移,绿色、安全、无污染、可持续。
本发明所涉及到的术语定义
除非另外定义,否则本文所用的所有技术及科学术语都具有与本发明所属领域的普通技术人员通常所了解相同的含义。
术语“内生真菌”:是指其生活史的一定阶段或全部阶段生活于植物的各种组织或器官内部的真菌,并与植物建立和谐联合关系,其中的某些类群可以产生各种化学物质,并且能通过竞争或其他作用来抑制杀死某些致病菌。
术语“群落结构”:群落是指生态学中,在一个群落生境内相互之间具有直接或间接关系的所有生物,或生物的总和。而描述组成群落的生物种群在群落中所处的位置和存在的状态为群落结构。
术语“相关性”:用以观察微生物属或种间(或otu)的相互作用,通过计算斯皮尔曼(spearman)关联系数等方法,找寻微生物在不同环境下的可能的相互“协作”或“竞争”关系。
附图说明
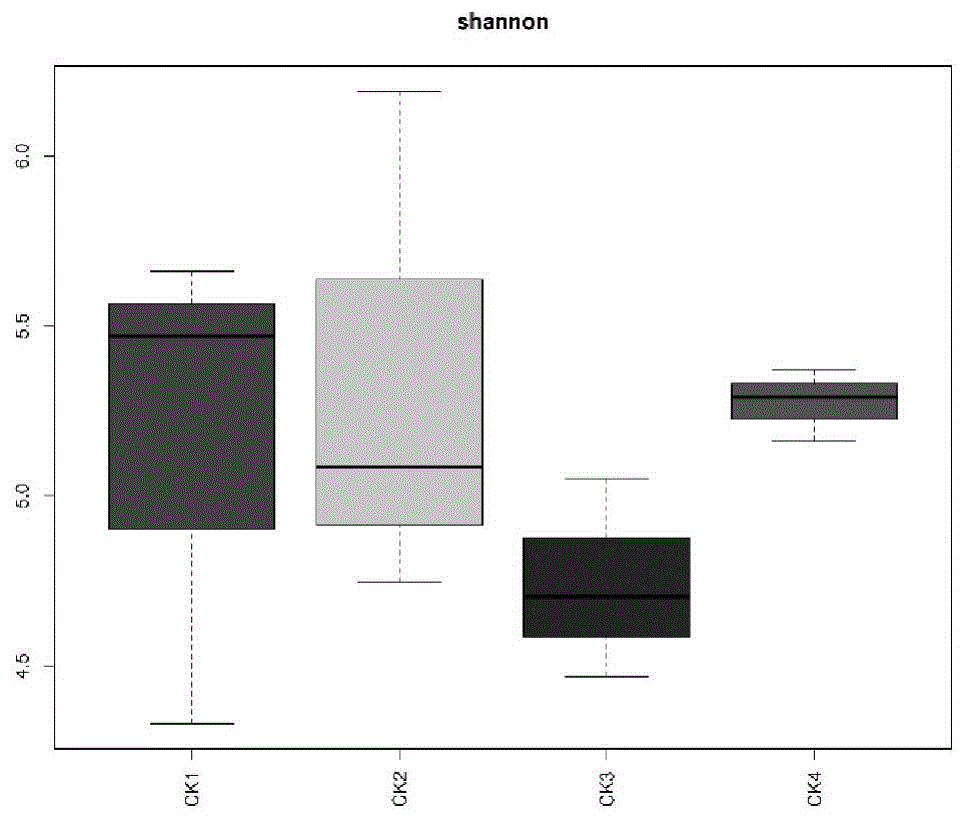
图1为不同枯梢病侵染情况的针叶内生真菌多样性指数柱状图。
图2为sphaeropsissapinea与负相关真菌的相对丰度变化趋势。
图3为sphaeropsissapinea与正相关真菌的相对丰度变化趋势。
具体实施方式
下面结合具体实施例来进一步描述本发明,本发明的优点和特点将会随着描述而更为清楚。但是应理解所述实施例仅是范例性的,不对本发明的范围构成任何限制。本领域技术人员应该理解的是,在不偏离本发明的精神和范围下可以对本发明技术方案的细节和形式进行修改或替换,但这些修改或替换均落入本发明的保护范围。
实施例1基于内生真菌群落结构调控松枯梢病
(1)寄主植物接种
对昆嵛山林间样地的赤松接种jattaeataediosa、aureobasidiumpullulans菌或喷施j.taediosa、a.pullulans发酵液,可直接将发酵液喷施到梢头及枝干,提高相对丰度,以抑制松杉球壳孢s.sapinea的生长繁殖。
(2)对病枝的清理及枝条修剪
将感染枯梢病的病枝及时清除,防止病原菌进一步扩散蔓延。对生长旺盛的林分,及时采取“留优去劣”的修枝或间伐,以减缓无效蒸腾作用,增强树势。
实施例2基于内生真菌群落结构调控松枯梢病
具体方法同实施例1,区别在于,对昆嵛山林间样地的赤松喷施orbiliarectispora,phaeothecoideasp.,devriesiasp.,cryptococcussp.,orbiliasp.等菌的拮抗菌剂,上述菌类多为常见病害的致病菌,通过拮抗上述病原菌的手段,同时抑制松杉球壳孢(s.sapinea)的生长。
实施例3基于内生真菌群落结构调控松枯梢病
具体方法同实施例1,区别在于,对昆嵛山林间样地的赤松接种jattaeataediosa、aureobasidiumpullulans菌或喷施j.taediosa、a.pullulans发酵液,并喷施拮抗orbiliarectispora,phaeothecoideasp.,devriesiasp.,cryptococcussp.,orbiliasp.等菌的拮抗菌剂,提高j.taediosa、a.pullulans的相对丰度,并抑制多种病原菌的生长。
实验例1松枯梢病与内生真菌群落结构分析
1实验方法
1.1研究区概况及样地设置
昆嵛山(121°41′34″e—121°48′04″e,37°11′50″n—37°17′22″n),属暖温带落叶阔叶林区,是胶东半岛上赤松分布最集中的区域,亦是以天然赤松为主要保护对象的森林生态类型保护区,赤松在该区域从山麓至海拔800m均有分布,并与落叶阔叶林共同组成了地带性天然次生林植被。该区域受暖温带季风气候影响,气候温和,无霜期达200~220d,年降水量为800~1200mm。该区域土壤以棕壤以及酸性粗骨土为主,海拔高处有少量山顶草甸土,土壤质地多为砂质壤类型,层次分明,结构较为疏松,有机质含量低。
根据林区生态系统结构、功能以及立地环境特点,在林区范围内设置9块赤松纯林永久样地,每块样地规格为30m×30m,样地保护带宽度为30m,实测并记录样地的海拔、坡向、坡位、坡度等指标,确保各样地立地条件一致。
1.2内生真菌测定
1.2.1样品采集
选取9个标准样地,在样地中分别选取5株赤松平均木,分别采集无病斑赤松的针叶ck1,染病赤松上的无病斑针叶ck2,病斑长度小于针叶长度二分之一的针叶ck3,病斑长度大于针叶长度二分之一的针叶ck4。采集后使用液氮速冻,干冰运输,保存于-80℃冰箱中。
采用ctab方法提取针叶样本基因组dna,利用琼脂糖凝胶电泳、od280检测dna的浓度和纯度。用无菌水在离心管中稀释样品质量浓度至1ng·μl-1,以稀释后样品基因组dna为模板,依据测序区域的选择,使用带有barcode的特异引物(newenglandbiolabs公司的
1.2.2物种注释与丰度计算
采用uparse软件聚类分析所有样品effectivetags序列,将序列聚类成为otus(默认97%的一致性),构建otus时选取代表性序列(otus中出现频数最高的序列),用greengene和rdpclassifier数据库将代表性序列集合注释分析样品物种,结合物种组成信息得到样品中真菌物种丰度。
1.2.3真菌多样性及spearman相关性分析
利用qiime(version1.7.0)软件依据样本文库的otus丰度信息计算样品的真菌多样性:shannon指数。
选取属水平总丰度前50的物种,计算物种之间的spearman相关系数,构建相关优势菌与病原菌的关联表格(相关系数阈值>0.3,且p<0.01)。
1.2.4数据分析
应用软件mothur中的summary.single分析alpha多样性(shannon多样性指数)。采用mothur中的commandsummarysingle分析beta多样性。
2实验结果
2.1无病斑针叶及染病针叶内生真菌多样性
图1表明,无病斑针叶(ck1与ck2)的内生真菌多样性指数高于染病较轻针叶ck3,在染病较重的ck4针叶中内生真菌多样性指数再次升高。在无病斑针叶中,内生真菌多样性较高,松杉球壳孢受其它微生物的影响,未表现出病斑;随着寄主植物生长势减弱,内生真菌多样性降低,与松杉球壳孢呈负相关性的内生真菌丰度减少,松杉球壳孢种群数量增加,针叶上出现病斑。
2.2spearman分析种间相关性
将测序获得的内生真菌相对丰度进行spearman相关性分析,表2为与松杉球壳孢(s.sapinea)相关性显著的内生真菌分析结果,结果表明:jattaeataediosa,aureobasidiumpullulans与松杉球壳孢(s.sapinea)呈负相关性,orbiliarectispora,phaeothecoideasp.,devriesiasp.,cryptococcussp.,orbiliasp.等真菌与松杉球壳孢(s.sapinea)呈正相关性。
研究表明,通过微生物的相互作用可以合理推测处于互利共生关系的微生物呈正相关关系,而处于竞争关系的微生物呈负相关关系,通过调整内生真菌的群落结构能够达到抑制松杉球壳孢(s.sapinea)的效果。
实验例2松枯梢病原菌与相关真菌的相对丰度显著分析
1实验方法
1.1筛选样地
选择实验例1的9块30m×30m的样地,对样地的内生真菌相对丰度进行比较。
1.2sphaeropsissapinea丰度变化
负相关性真菌与病原菌相对丰度变化:通过spss软件对9个样地内针叶的aureobasidiumpullulans和s.sapinea的相对丰度进行duncan显著性检验(置信度为95%),绘制病原菌变化折线图。
正相关性真菌与病原菌相对丰度变化:通过spss软件对9个样地内针叶的orbiliarectispora和s.sapinea的相对丰度进行duncan显著性检验(置信度为95%),绘制病原菌变化折线图。
2实验结果
由图2可知,病原菌s.sapinea相对丰度随着负相关性真菌a.pullulans的相对丰度的增高而呈显著的下降趋势。当a.pullulans相对丰度较低(低于3.00%)时,病原菌相对丰度最高可达2.00%,针叶有病斑;当a.pullulans相对丰度高于3.00%时,病原菌相对丰度降低,并维持在一个较低且平稳的水平,且针叶无病斑。
图3显示,当o.rectispora的相对丰度低于1.00%时,病原菌s.sapinea相对丰度维持在稳定的低水平(0.40%-0.55%),对应的针叶无病斑;随着o.rectispora的相对丰度高于1.00%,病原菌相对丰度呈现出显著的上升趋势,并逐渐趋于稳定状态,针叶表现为有病斑。
序列表
<110>中国林业科学研究院森林生态环境与保护研究所
<120>一种减轻松枯梢病病原菌危害寄主植物的真菌群落结构调控方法
<130>bj-2003-190912a
<160>2
<170>siposequencelisting1.0
<210>1
<211>22
<212>dna
<213>artificalsequence
<400>1
cttggtcatttagaggaagtaa22
<210>2
<211>18
<212>dna
<213>artificalsequence
<400>2
tgcgttcttcatcgatgc18
- 还没有人留言评论。精彩留言会获得点赞!