敲除yncD‑aroC双基因的甲型副伤寒沙门菌减毒疫苗的制作方法与工艺
敲除yncD-aroC双基因的甲型副伤寒沙门菌减毒疫苗技术领域本发明涉及一种敲除yncD基因和aroC基因的甲型副伤寒沙门菌减毒疫苗。
背景技术:
伤寒仍然是全球性的公共卫生问题。伤寒是由伤寒沙门菌、甲型、乙型和丙型副伤寒沙门菌引起的人类肠道传染病据世界卫生组织估计,全球每年的伤寒患者有1600-3300万感染者,死亡病例在50-60万左右[1]。伤寒病例长期位居我国法定甲乙类传染病的前10位,各地每年仍有大量散发病例,还时有局部的爆发流行,严重威胁着人们的身体健康。而近两年的统计数据显示,甲型副伤寒沙门菌在所有伤寒病原中的分离比率和感染绝对数量逐年上升,所占比例从18%到47%不等。这一变化在我国表现尤为突出,在我国主要流行区,甲型副伤寒沙门菌已逐渐取代伤寒沙门菌,成为伤寒病的主要病原。例如最新的数据显示在广西,甲型副寒和伤寒发病率分别为180/1O万和30/10万,甲型副伤寒占所有病例的85.8%。随着流行区人群和旅行者中甲型副伤寒感染病例的增加,一些地区开始出现多重耐药的甲型副伤寒沙门菌。过去伤寒病的预防一直以针对伤寒沙门菌感染为主,正在使用的两种伤寒疫苗(Vi疫苗和伤寒沙门菌Ty21a减毒活疫苗)对甲型副伤寒均缺乏有效的预防作用。而目前市场上尚没有可以应用的甲型副伤寒疫苗。因此相关的疫苗研制工作刻不容缓。接种疫苗的基本目的是诱导机体的免疫反应,进而对病原体的感染起到免疫保护的作用。现代分子生物学技术的发展使得我们可以通过基因敲除技术构建遗传背景清楚的减毒疫苗株。这种方式往往需要对影响细菌生长的某些重要基因或者与细菌致病性有关的毒力基因进行敲除操作。到目前为止,采取这种方式,已经针对沙门菌构建了很多的减毒株,涉及的基因包括aro,pur,htrA,ompR,ompF,ompC,galE,cya,crpandphoP等。申请人拥有的公开号为CN102172399B的发明专利,是通过将沙门菌yncD基因敲除得到的沙门菌减毒疫苗,虽然该技术也可以用于甲型副伤寒疫苗株的构建,但是和双基因敲除的减毒疫苗相比较,却存在一个关键问题,即由于该减毒疫苗只敲除了一个基因,因此在实际使用过程中存在回复突变的可能性,也就是细菌的毒力恢复到基因敲除前的水平,对接种者的健康和安全造成威胁。而双基因的敲除则可以在保持疫苗原有的免疫原型和免疫保护性的前提下,最大程度的规避潜在的毒力回复突变风险,以保证接种者的健康和安全。当然,在实际的疫苗制备过程中,如果涉及两个以上的毒力基因敲除,减毒株的构建就必须把握一个平衡,即首先要将病原体的毒力减弱,使其接种宿主后不会导致疾病发生;但同时还需保证其能有效诱导机体的免疫反应,不至于减毒过低而导致免疫保护效果不佳,从而丧失疫苗株的作用,因此,对于所敲除基因的选择就成了问题的关键所在。
技术实现要素:
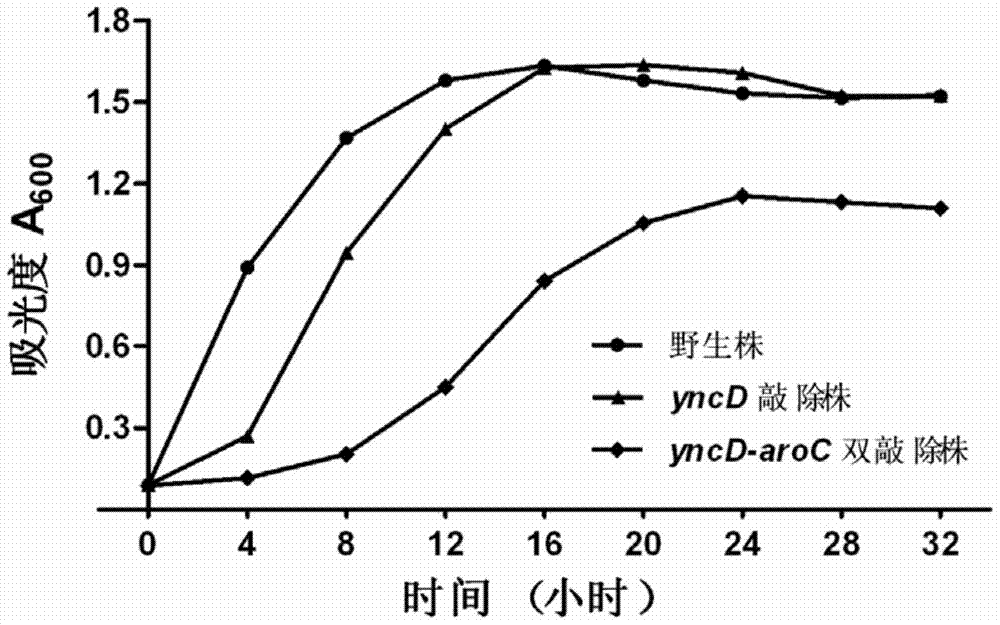
本发明的目的是提供一种敲除yncD-aroC双基因的甲型副伤寒沙门菌减毒疫苗,所述疫苗为将yncD-aroC双基因敲除而实现减毒的甲型副伤寒沙门菌减毒疫苗,其不仅相对于甲型副伤寒沙门菌野生株毒力明显减弱,与yncD单基因敲除株比较,其毒力也进一步降低。表现在其生长速率显著减低,在单核巨噬细胞中的胞内生存能力也较yncD单敲株显著减弱。但同时双基因敲除株仍然具有良好的免疫原性,对接种过疫苗株的小鼠具有显著的保护效果,并且疫苗株是两个毒力基因的敲除,最大程度避免了毒力回复的可能性。本发明所述的甲型副伤寒沙门菌减毒疫苗,包含甲型副伤寒沙门菌yncD-aroC敲除株。所述敲除株是通过甲型副伤寒沙门菌yncD基因敲除载体敲除yncD基因和通过甲型副伤寒沙门菌aroC基因敲除载体敲除aroC基因。所述甲型副伤寒沙门菌yncD基因敲除载体包含pYG4质粒和中间去掉yncD基因序列同时保证阅读框不发生变化的yncD基因上下游序列SEQIDNO:14;所述甲型副伤寒沙门菌aroC基因敲除载体包含pYG4质粒和中间去掉aroC基因序列同时保证阅读框不发生变化的aroC基因上下游序列SEQIDNO:15-16。所述沙门菌yncD基因敲除载体采用引物为SEQIDNO:1-4所示序列的PCR方法制得;所述沙门菌aroC基因敲除载体采用引物为SEQIDNO:7-10所示序列的PCR方法制得。本发明的另一个目的是提供一种甲型副伤寒沙门菌yncD双基因的敲除载体以及甲型副伤寒沙门菌aroC该敲除载体能够准确敲除甲型副伤寒沙门菌yncD-aroC。甲型副伤寒沙门菌yncD基因敲除载体,包含pYG4质粒序列和中间去掉yncD基因序列同时保证阅读框不发生变化的yncD基因上下游序列,所述中间去掉yncD基因序列同时保证阅读框不发生变化的yncD基因上下游序列如SEQIDNO:17-18所示;所述甲型副伤寒沙门菌yncD基因敲除载体采用引物为SEQIDNO:1-4所示序列的PCR方法制得。甲型副伤寒沙门菌aroC基因敲除载体,包含pYG4质粒序列和中间去掉aroC基因序列同时保证阅读框不发生变化的aroC基因上下游序列,所述中间去掉aroC基因序列同时保证阅读框不发生变化的aroC基因上下游序列如SEQIDNO:15-16所示;所述甲型副伤寒沙门菌aroC基因敲除载体采用引物为SEQIDNO:7-10所示序列的PCR方法制得。yncD敲除株的构建:首先设计两对引物,其序列分别为SEQIDNO:1-4,分别扩增yncD基因上游和下游各1000bp片段,由于两对引物中的内侧引物(SEQIDNO:2和SEQIDNO:3)有21个碱基的重叠,故可以采用2个外侧引物(SEQIDNO:1和SEQIDNO:4),两端设计了NdeⅠ酶切位点)对前述PCR产物进行交叠PCR,获得去除了yncD基因的序列。将PCR产物连接到T载体上,再经NdeⅠ酶切后,连接到同样酶切并去磷酸化的敲除载体pYG4,将连接产物转化大肠埃希菌S17-1/λpir,卡那霉素平板上筛选阳性克隆,对获得的阳性克隆进行酶切、PCR测序鉴定。将载体通过电转方式转移到受体菌中,采用卡那平板(含有卡那霉素100μg/ml)进行筛选。将获得的整合体细菌在无选择培养基中扩大培养,然后将培养液经适当稀释后涂布于含有5%蔗糖的LB平板进行第2次筛选,对获得的重组菌株进行PCR测序鉴定(SEQIDNO:5和SEQIDNO:6),筛选敲除株。aroC敲除株的构建:aroC基因编码的分支酸合酶,在芳香族氨基酸合成途径中有十分重要的作用。该基因的突变株对某些芳香族化合物,如色氨酸,酪氨酸,苯丙氨酸,对氨基苯甲酸和2,3-二羟基苯甲酸等表现出营养缺陷,在缺乏芳香族化合物的环境,比如人体中,其生长会受到抑制而导致毒力下降。因此,申请人选择在yncD基因敲除株的基础上,敲除aroC基因,以进一步降低敲除株的毒力,增加敲除株的安全性。aroC基因敲除技术路线同yncD基因,只是所用引物序列不同,其序列分别为SEQIDNO:7-10和SEQIDNO:11-12。首先设计两对引物,其序列如SEQIDNO:7-10所示,分别扩增aroC基因上游和下游各750bp片段,由于两对引物中的内侧引物(序列SEQIDNO:8和SEQIDNO:9)有21个碱基的重叠,故可以采用2个外侧引物(序列SEQIDNO:7和SEQIDNO:10),两端设计了ApaⅠ和SacⅠ酶切位点,对前述PCR产物进行交叠PCR,获得去除了aroC基因的序列。将PCR产物连接到T载体上,再经ApaⅠ和SacⅠ双酶切后,连接到同样双酶切并去磷酸化的敲除载体pYG4,将连接产物转化大肠埃希菌S17-1/λpir,卡那霉素平板上筛选阳性克隆,对获得的阳性克隆进行酶切、PCR测序鉴定。将载体通过电转方式转移到受体菌中,采用卡那平板(含有卡那霉素100μg/ml)进行筛选。将获得的整合体细菌在无选择培养基中扩大培养,然后将培养液经适当稀释后涂布于含有5%蔗糖的LB平板进行第2次筛选,对获得的重组菌株进行PCR测序鉴定(SEQIDNO:11和SEQIDNO:12),筛选敲除株。本发明采用的自杀载体是pYG4,载体包含以下成分:来源于R6K质粒的π蛋白依赖性复制起点oriR6K,使质粒只能在表达π蛋白的菌株中复制,对于其它菌株(包括沙门菌)则是自杀性的。卡那霉素抗性片段,用于正向选择。反向选择标志为sacB基因,是来自于枯草芽孢杆菌的果聚糖蔗糖转移酶基因,表达该蛋白的革兰阴性菌在含有蔗糖的培养基中生长时,会将蔗糖分解为对细菌有毒的产物,从而杀死表达该蛋白的细菌,因此可以用于反向选择。由于敲除后不会加入抗性片段,因此适用于疫苗株的突变操作,采用同一载体也可以多次累加敲除。本发明中采用同源重组方式对甲型副伤寒沙门菌的yncD基因和aroC基因进行敲除,从而获得yncD-aroC双基因敲除的甲型副伤寒沙门菌减毒疫苗株。相比yncD单基因敲除株,双基因敲除株由于毒力有了进一步降低,同时最大程度的避免了单基因敲除株的毒力回复突变,因此具有更佳的安全性,免疫效果测定也证明本发明所构建的疫苗株具有很好的免疫原性,对接种过疫苗株的小鼠具有良好的保护效果附图说明图1为aroC基因敲除的过程示意图;(yncD和aroC两者敲出过程相同,只是所使用的引物序列不同,图中所标注p1,p2,p3,p4是代表所敲基因的上下游序列引物)图2为野生株、yncD突变株、yncD-aroC双突变株生长曲线;图3为yncD基因序列进行相似度比对。具体实施方式试剂材料:一.试验中所用试剂除DNA连接酶和试剂盒外,均为takara公司产品;LB培养基:液体:氯化钠1%,胰蛋白胨1%,酵母提取物0.5%;固体:氯化钠1%,胰蛋白胨1%,酵母提取物0.5%,琼脂糖1.4%,121℃高压蒸汽灭菌;а-MEM培养基:根据需要,分别加入终浓度10uM的FeSO4和FeCl3;DNA连接酶promega公司产品胶回收试剂盒omega公司产品质粒提取试剂盒omega公司产品引物制作和DNA片段测序由上海英俊公司完成卡那霉素培养基即在普通LB培养基基础上加入终浓度100ug/ml的卡那霉素;氨苄青霉素培养基:即在普通LB培养基基础上加入终浓度100ug/ml的氨苄青霉素。二.所用主要仪器:凝胶成像仪:Bio-rad-T2a电转仪:Bio-radgene-pluserII分光光度计:Bio-radsmartspeec3000电泳仪::Bio-rad72BR5008PCR仪::Bio-rad580B5502水浴:上海生共HH-2A金属浴:杭州博日CHB-A4-HH摇床:zhichengZHWY-100B恒温摇床离心机:ThermoLEGENDMicro21实施例1甲型副伤寒沙门菌yncD敲除株的构建参见图1,采用交叠PCR方式获得拼接在一起的yncD基因上下游序列(中间去掉yncD基因序列,同时保证阅读框不发生变化),连接到pYG4(pYG4质粒的构建过程请参考“第三军医大学学报”,2009年12月,第31卷第23期第2299-2301页。pYG4质粒序列见CN102172399B的发明专利所述。)中构建成敲除载体pYG4-△yncD,转化到大肠埃希菌S17-1/λpir中复制,进行酶切、PCR及测序鉴定。(1)交叠PCR:根据yncD基因上下游序列分别制作两组PCR引物Dk1和Dk2、Dk3和Dk4(SEQIDNO:1-4,其中,Dk2和Dk3有一段互补序列,Dk1和Dk4内分别含有限制性内切酶Ndel和EcooR1酶切位点)。然后分别做PCR。yncD基因序列如SEQIDNO:14所示。PCR方法如下反应条件:将两组PCR产物跑凝胶电泳后做胶回收,标记为pcr1和pcr2。由于Dk2和Dk3有部分互补序列,所以,再用pcr1和pcr2为模板做交叠PCR,方法如下:反应条件:(2)将所得PCR产物电泳做胶回收,标记为pcr3。用pcr3和PYG4分别做双酶切条件:37℃水浴过夜。将两个酶切产物跑凝胶电泳后胶回收,分别标记为L1和L2。用L1和L2做连接反应构建敲除载体,记为pYG4-△yncD,条件如下:反应条件:16℃金属浴过夜。(3)将pYG4-△yncD电转入大肠埃希菌S17-1/λpir中复制、扩增、提取质粒(卡那霉素抗性),进行酶切、PCR及测序鉴定。反应条件:37℃水浴过夜②PCR体系:电转条件:应在冰上进行,0.1cm电转杯,1.8KV,25uf,200殴。③送英骏公司测序,对测序获得的序列与网上公布的相应序列对比。参见图1第二步,将经测序鉴定的敲除载体通过电转方式转移到甲型副伤寒沙门菌标准株(购自中国医学微生物保藏中心),该野生株不能在含有卡那霉素的平板上生长,同时载体pYG4无法在甲型副伤寒沙门菌中复制,因此只有当载体整合到野生株的染色体上,该菌株才能在抗性平板上生长,挑选单菌落进行PCR测序鉴定。鉴定正确后菌株记为整合株。(2)挑选单菌落做PCR鉴定,并同时送测序:PCR体系:参见图1,为了进一步获得敲除株,将鉴定后的整合菌株置于无抗生素的LB培养基中培养,培养后筛选发生染色体内同源重组的突变株,由于sacB片段的反向选择作用,可用含有5%蔗糖的平板进行筛选,带有sacB片段的整合株无法在该平板上生长,而长出来的菌落都是发生了染色体内重组,从而去掉了载体序列(包括sacB片段)的重组菌株。重组有两种情况,其中50%的可能性产生敲除株,可通过PCR方式筛选鉴定获得敲除株。所得凝胶电泳条带可与野生株相比较,DNA大小差别正好等于yncD大小。获得的重组菌株进行PCR测序鉴定,引物Dk5、Dk6(SEQIDNO:5和SEQIDNO:6)分别为Dk1和Dk4(SEQIDNO:1和SEQIDNO:4)外围引物:所得凝胶电泳条带可与野生株相比较,DNA大小差别正好等于yncD大小。实施例2甲型副伤寒沙门菌yncD-aroC敲除株的构建aroC基因敲除与yncD基因基因敲除方法相同。采用交叠PCR方式获得拼接在一起的aroC基因上下游序列(中间去掉aroC基因序列,同时保证阅读框不发生变化),连接到pYG4中构建成敲除载体pYG4-△aroC,转化到大肠埃希菌S17-1/λpir中复制,进行酶切、PCR及测序鉴定。(1)交叠PCR:根据aroC基因上下序列分别制作两组PCR引物Ck1和Ck2、Ck3和Ck4(SEQIDNO:7-10,其中,Ck2和Ck3有一段互补序列,Ck1和Ck4内分别含有限制性内切酶Apa1和sac1酶切位点)。然后分别做PCR。aroC基因序列如SEQIDNO:13所示。PCR方法如下:将两组PCR产物跑凝胶电泳后做胶回收,标记为pcr1和pcr2.。由于Ck2和Ck3有部分互补序列,所以,再用pcr1和pcr2为模板做交叠PCR,方法如下:(2)将所得PCR产物电泳做胶回收,标记为pcr3。用pcr3和PYG4分别做双酶切条件:37℃水浴过夜将两个酶切产物跑凝胶电泳后胶回收,分别标记为L1和L2。用L1和L2做连接反应构建敲除载体,记为pYG4-△aroC,条件如下:反应条件:16℃金属浴过夜。(3)将pYG4-△aroC电转入大肠埃希菌S17-1/λpir中复制、扩增、提取质粒(卡那霉素抗性),进行酶切、PCR及测序鉴定。反应条件:37℃水浴过夜③电转条件:应在冰上进行,0.1cm电转杯,1.8KV,25uf,200殴。(3)将经鉴定的敲除载体通过电转方式转移到甲型副伤寒沙门菌yncD敲除株(选择对卡那霉素敏感的菌株),该yncD敲除株不能在含有卡那霉素的平板上生长,同时载体pYG4无法在该yncD敲除株中复制,因此只有当载体整合到yncD敲除株的染色体上,该菌株才能在抗性平板上生长,挑选单菌落进行PCR测序鉴定。鉴定正确后,菌株记为整合株。①将鉴定后的pYG4-△aroC质粒点转入yncD敲除株,用含卡那霉素的固体培养基筛选。培养条件:37℃过夜②挑选单菌落做PCR鉴定,并同时送测序:PCR体系:③为了进一步获得yncD-aroC敲除株,将鉴定后的整合菌株置于无抗生素的LB培养基中培养,培养后筛选发生染色体内同源重组的突变株,由于sacB片段的反向选择作用,可用含有5%蔗糖的平板进行筛选,带有sacB片段的整合株无法在该平板上生长,而长出来的菌落都是发生了染色体内重组,从而去掉了载体序列(包括sacB片段)的重组菌株。④重组有两种情况,其中50%的可能性产生敲除株,可通过PCR方式筛选鉴定获得敲除株。所得凝胶电泳条带可与野生株相比较,DNA大小差别正好等于aroC大小。⑤获得的重组菌株进行PCR测序鉴定,引物Ck5、Ck6(SEQIDNO:11和SEQIDNO:12)分别为Ck1和Ck4(SEQIDNO:7和SEQIDNO:10)外围引物:所得凝胶电泳条带可与野生株相比较,DNA大小差别正好等于aroC大小。实施例3yncD敲除株、yncD-aroC双敲除株与野生株生物学性状的比较以LB培养基分别培养yncD敲除株、yncD-aroC双敲除株(疫苗株)与野生株,绘制生长曲线,比较三者在培养基中的生长性状的差异。参见图2,yncD单基因敲除株生长较野生株缓慢,进入对数期以及稳定期的时间均较野生株延迟4小时左右,但经过16小时培养后可以达到与野生株基本相同的菌液浓度;双基因敲除株生长速率减低更为明显,其进入对数期时间较野生株延迟近8小时,进入稳定期的时间较野生株则延迟近12小时,并且其最终的菌液浓度A600值也较野生株明显减低。实施例4疫苗株与野生株毒力的比较采用评价沙门菌毒力通常使用的粘蛋白增毒小鼠模型。收集对数生长期的野生株和疫苗株细菌经PBS洗后重悬,悬液中加入终浓度10%的猪胃粘蛋白,根据预实验结果设置5个浓度梯度组,每组3只BALB/c小鼠。细菌悬液注射至小鼠腹腔,注射后观察72小时内小鼠死亡情况,经统计学处理计算半数致死量LD50。实施例4疫苗株与野生株毒力的比较采用评价沙门菌毒力通常使用的粘蛋白增毒小鼠模型。分别收集对数生长期细菌经PBS洗后重悬,悬液中加入终浓度10%的猪胃粘蛋白,设置5个梯度,分别为2.5×102,2.5×103,2.5×104,2.5×105,2.5×106CFU,每组3只BALB/c小鼠,并设3只PBS空白对照小鼠。将细菌悬液注射至小鼠腹腔,注射后观察72小时内小鼠死亡情况,经统计学处理计算半数致死量LD50。结果见表1,疫苗株的毒力发生显著性降低。其毒力降低的原因:一是yncD编码外膜转运蛋白,突变后对于靶物质的转运能力下降,而导致其在基本培养基中的生长能力下降,虽然目前尚未确认其转运物质,但以上实验可以证实其功能的重要性及其与致病性的密切关系。二是由于aroC基因和细菌合成芳香族氨基酸有关,该基因敲除后细菌的芳香族氨基酸合成收到影响,造成细菌的生长进一步受到影响。表1.甲型副伤寒沙门菌LD50菌株描述接种途径LD50YGC101野生株腹腔注射7.9×101YGC102疫苗株腹腔注射3.33×106实施例5疫苗株免疫效果收集对数生长期野生株细菌经PBS洗涤重悬,悬液中加入终浓度10%的猪胃粘蛋白,根据预实验结果设置3个浓度梯度组,分别为2.5×104,2.5×105,2.5×106CFU,每组3只经疫苗株免疫30天的BALB/c小鼠,对照组3只小鼠注射剂量为2.5×103CFU。细菌悬液注射至小鼠腹腔,注射后观察72小时内小鼠死亡情况。如表2所示,经过疫苗株的免疫接种以后,BALB/c小鼠对野生株的毒力攻击耐受有了很大的提高,证明疫苗株的免疫接种对小鼠有良好的免疫保护效果。表2甲型副伤寒沙门菌疫苗株免疫效果实施例6yncD基因在沙门菌中的相似度分析对已测序的沙门菌的yncD基因序列,并进行相似度比对。结果参见图3。分析表明,yncD基因序列在沙门菌中有高度的保守性,相似度大于98%,高度的保守性表明其基因功能在不同沙门菌的血清型中是一致的。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1
- 用CRISPR/Cas9技术易于筛选获得目的基因敲除细胞系的方法及产品与制造工艺
- 一种Candida amazonensis的FLP/FRT基因敲除方法与制造工艺
- 玉米低镁条件下特异性转运镁离子基因ZmMGT10及其应用的制造方法与工艺
- CRISPR‑Cas9特异性敲除猪SLA‑3基因的方法及用于特异性靶向SLA‑3基因的sgRNA与制造工艺
- 利用CRISPR/Cas9敲除人PD‑1基因构建靶向CD19CAR‑T细胞的方法与制造工艺
- 一种构建基因替代小鼠研究野生型和突变体非肌性肌球蛋白重链II功能的方法与制造工艺
- 构建在海马体区域特异性敲除IKKα基因的小鼠模型的方法及打靶载体和试剂盒与制造工艺
- 猪ApoE基因敲除打靶载体及其构建方法和应用与制造工艺
- 一种血小板减少症的斑马鱼模型的制造方法与工艺
- 基于CRISPR技术敲除FUT8基因的方法与制造工艺