用于捕获α地中海贫血相关基因拷贝数检测试剂盒的制作方法
本发明属于体外诊断领域,具体涉及用于捕获α地中海贫血相关基因拷贝数检测试剂盒。
背景技术:
地中海贫血是严重威胁人类健康的致死致残的常染色体隐性遗传性血液病。全球约2%的人为地中海贫血基因的携带者,在我国南方高发病地区的人群携带率为3%-24%。地中海贫血是由于珠蛋白基因发生缺陷,导致肽链合成减少或缺失的遗传性溶血性血红蛋白病。临床上地中海贫血又分为轻型(地中海贫血携带者),中间型和重型。临床上表现出特殊的地中海贫血面容—头颅增大、颧骨突出、眼距增宽、鼻梁低平,根据病情的轻重表现为小细胞低色素,溶血性贫血伴黄疸,疲乏无力,肝脾肿大,不定期或定期输血,水肿胎或过多铁沉积导致心力衰竭致死。
α-地中海贫血主要由α-珠蛋白基因发生缺失所引起,并且缺失范围具有很大的异质性,可以是仅涉及hba1和hba2基因附近的几千个碱基对的缺失,更多的是包含2个hba1和hba2基因在内的涉及几万到几十万碱基对的缺失,甚至整个α珠蛋白基因簇的丢失。此外,还有只包含α基因上游调控区的片段的缺失。目前全球已鉴定的α地中海贫血缺失超过20种,不同种族的α-珠蛋白基因的缺失谱不同,中国人中最常见的是sea、3.7kb和4.2kb这三种缺失类型,在广西地区突变携带率分别为7.84%,4.78%和1.61%。
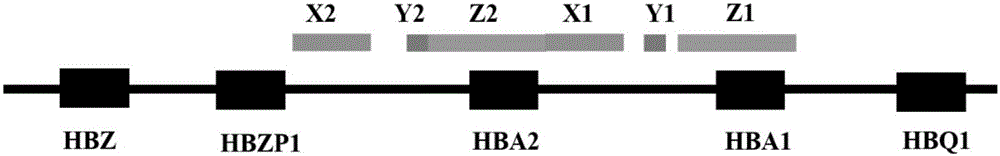
如图1所示,hba1和hba2处于高度同源的两个重复单元中,这些单元包括了三个同源片段(x、y和z盒),其间被三个非同源区所分隔。hba1和hba2基因结构极其相似,其中编码区序列完全相同(5’端非编码区以及intron区有些微差异,3’端非编码区有部分碱基差异),这极大地造成了分析的难度,这也是罕有利用二代测序检测α-地中海贫血的重要原因。
目前检测地中海贫血的方法主要有gap-pcr以及mlpa(多重连接探针扩增),其中,gap-pcr往往是根据已知缺失类型进行引物设计,仅可检测已知缺失突变,检测范围十分有限,而且不能检测其它未知罕见的缺失类型,极有可能造成漏诊,导致重症患儿出生。此外,对于α地中海贫血缺失突变也可利用多重连接探针扩增(mlpa)技术对其进行检测,虽然相较于gap-pcr而言,mlpa不但可以检测α-地中海贫血常见、非常见以及未知罕见缺失突变,但mlpa技术仍存在一定局限性,需要精确测量dna的浓度、样本容易被污染且存在一定假阳性的可能,尤其对于单个外显子缺失突变的情况,需要利用pcr的方法进行进一步的验证实验。总之,目前尚无一种快速、低成本、大通量和自动化检测α-地中海贫血缺失突变的方法。
目前公开文献中也有关于检测α-地中海贫血缺失突变的相关产品,下面是检索到的相关的专利文献:有专利申请(专利申请公开号cn107267648a)采用多重pcr的方法设计引物并提供快速检测地中海贫血常见突变基因的试剂盒,该方法有pcr偏好性,相对于二代测序技术而言,通量低价格昂贵,并且只能检测已知的常见的突变类型。专利申请(专利申请公开号cn107475410a)采用基因芯片技术进行alpha-地中海贫血的缺失或重复突变的检测,基因芯片技术虽然是新一代测序技术,但是相对于二代测序技术背景噪音大,精度明显受探针设计影响并且价格昂贵。专利申请(专利申请公开号cn107841545a)虽采用二代测序技术检测地中海贫血基因型,但是仅描述了常见的二代测序的步骤以及分析流程,并没有针对地中海贫血基因设计探针,更没有针对地中海贫血基因的分析方法,实际操作较难实现。专利申请(专利申请公开号cn106591441a)发明了一种基于全基因捕获测序的alpha/beta地中海贫血突变的检测探针、方法、芯片,该发明虽然针对alpha/beta基因区域设计了相关探针并且提供了探针序列,但是该发明没有考虑hba1以及hba2基因的高度同源性,没有针对hba1以及hba2设计探针,也没有针对高度同源基因的分析方法,这会导致捕获序列特异性差,同时也会影响分析结果的准确性,此外,该发明没有针对hba1/2基因的调控区设计探针,极有可能造成漏诊,导致重症患儿出生。
技术实现要素:
本发明要解决的技术问题是提供一种基于基因捕获和二代测序技术的用于捕获α地中海贫血相关基因拷贝数检测试剂盒,用于解决现有方法错误率较高、稳定性不好、通量较小、特异性差,尤其是对hba1和hba2基因的高度同源性欠缺考虑以及缺失类型种类多且难以区分等问题。
为解决上述技术问题,本发明采用如下技术方案:
在本发明的一方面,提供本发明公开了一种基于基因捕获和二代测序技术的用于捕获α地中海贫血相关基因拷贝数检测试剂盒,包括捕获探针,所述捕获探针由以下17个主要探针和对照探针组成;17个主要探针见表1:
表1
所述捕获探针对目标区域进行捕获,测序,通过数据分析估算hba1基因和hba2基因外显子1和外显子3的拷贝数,并对缺失类型进行基因分型。
作为本发明优选的技术方案,所述主要探针为针对α珠蛋白基因簇上x、y、z同源盒之间的非同源区设计,并且包含所有用于估算拷贝数的特征位点;此外充分考虑hba1基因和hba2基因的高度同源性,其中针对hba1基因和hba2基因区间所设计的捕获探针(见表1,探针序号1-4)至少包含一个能够区分hba1基因和hba2基因的差异位点。
作为本发明的优选技术方案,所述差异位点是用于区分hba1基因和hba2基因的位点,具体为:
染色体chr16,坐标222869,hba2基因,外显子1上游23bp,核苷酸g;
染色体chr16,坐标226673,hba1基因,外显子1上游23bp,核苷酸c;
染色体chr16,坐标223447,hba2基因,外显子3上游24bp,核苷酸g;
染色体chr16,坐标227258,hba1基因,外显子3上游24bp,核苷酸c;
染色体chr16,坐标223614,hba2基因,外显子3下游14bp,核苷酸a;
染色体chr16,坐标227425,hba1基因,外显子3下游14bp,核苷酸g;
染色体chr16,坐标223618,hba2基因,外显子3下游18bp,核苷酸g;
染色体chr16,坐标227429,hba1基因,外显子3下游18bp,核苷酸a;
染色体chr16,坐标223620,hba2基因,外显子3下游20bp,核苷酸t;
染色体chr16,坐标227431,hba1基因,外显子3下游20bp,核苷酸g;
染色体chr16,坐标223622,hba2基因,外显子3下游22bp,核苷酸c;
染色体chr16,坐标227433,hba1基因,外显子3下游22bp,核苷酸t;
染色体chr16,坐标223625,hba2基因,外显子3下游25bp,核苷酸c;
染色体chr16,坐标227436,hba1基因,外显子3下游25bp,核苷酸t;
染色体chr16,坐标223631-223632,hba2基因,外显子3下游31-32bp,核苷酸gc;
染色体chr16,坐标227442-227443,hba1基因,外显子3下游31-32bp,核苷酸ct;
染色体chr16,坐标223643-223644,hba2基因,外显子3下游43-44bp,核苷酸aa;
染色体chr16,坐标227454-227455,hba1基因,外显子3下游43-44bp,核苷酸cc;
染色体chr16,坐标223646,hba2基因,外显子3下游46bp,核苷酸g;
染色体chr16,坐标223457,hba1基因,外显子3下游46bp,核苷酸a;
染色体chr16,坐标223648,hba2基因,外显子3下游48bp,核苷酸g;
染色体chr16,坐标223459,hba1基因,外显子3下游48bp,核苷酸c。
作为本发明优选的技术方案,所述特征位点经过如下步骤筛选得到;包括:(1)挑选用于区分hba1基因和hba2基因外显子1和外显子3的差异位点,其中位点区域如图2所示;(2)挑选并加入hba1基因以及hba2基因上其它位点(除hba1基因和hba2基因差异位点外的其它位点),所挑选位点避开多态位点、常见snp(单核苷酸多态性)变异位点等;(3)挑选并加入hbz、hbq1、hbzp1基因外显子区域上的位点,所挑选位点均匀分布各基因外显子区域,并避开高度同源区、回文系列区域、多态位点、常见snp变异位点等;(4)根据训练集进一步筛选步骤(1)(2)(3)所选位点,其中所选位点在训练集中经过两步标准化之后的覆盖深度的标准差要小于0.18(将经过这四步筛选得到的位点定义为特征位点)。所选77个特征位点如下所示:
染色体chr16,坐标203900,核苷酸a
染色体chr16,坐标203950,核苷酸g
染色体chr16,坐标204000,核苷酸a
染色体chr16,坐标204050,核苷酸g
染色体chr16,坐标204400,核苷酸c
染色体chr16,坐标214480,核苷酸g
染色体chr16,坐标214500,核苷酸a
染色体chr16,坐标214505,核苷酸a
染色体chr16,坐标214600,核苷酸g
染色体chr16,坐标214605,核苷酸t
染色体chr16,坐标219818,核苷酸t
染色体chr16,坐标219853,核苷酸c
染色体chr16,坐标219922,核苷酸g
染色体chr16,坐标221062,核苷酸c
染色体chr16,坐标221082,核苷酸a
染色体chr16,坐标221108,核苷酸c
染色体chr16,坐标221128,核苷酸c
染色体chr16,坐标223383,核苷酸c
染色体chr16,坐标223447,核苷酸c
染色体chr16,坐标223614,核苷酸g
染色体chr16,坐标223618,核苷酸t
染色体chr16,坐标223620,核苷酸c
染色体chr16,坐标223622,核苷酸t
染色体chr16,坐标223625,核苷酸t
染色体chr16,坐标223631,核苷酸c
染色体chr16,坐标223632,核苷酸t
染色体chr16,坐标223643,核苷酸a
染色体chr16,坐标223644,核苷酸c
染色体chr16,坐标223646,核苷酸g
染色体chr16,坐标223648,核苷酸c
染色体chr16,坐标223661,核苷酸c
染色体chr16,坐标223663,核苷酸t
染色体chr16,坐标223670,核苷酸g
染色体chr16,坐标223673,核苷酸c
染色体chr16,坐标223678,核苷酸t
染色体chr16,坐标225100,核苷酸a
染色体chr16,坐标225120,核苷酸g
染色体chr16,坐标225141,核苷酸g
染色体chr16,坐标225161,核苷酸c
染色体chr16,坐标225181,核苷酸c
染色体chr16,坐标225735,核苷酸t
染色体chr16,坐标225745,核苷酸c
染色体chr16,坐标225755,核苷酸a
染色体chr16,坐标225765,核苷酸g
染色体chr16,坐标225780,核苷酸t
染色体chr16,坐标226043,核苷酸t
染色体chr16,坐标226220,核苷酸c
染色体chr16,坐标226221,核苷酸c
染色体chr16,坐标226673,核苷酸c
染色体chr16,坐标227187,核苷酸c
染色体chr16,坐标227250,核苷酸c
染色体chr16,坐标227253,核苷酸g
染色体chr16,坐标227258,核苷酸c
染色体chr16,坐标227425,核苷酸g
染色体chr16,坐标227429,核苷酸t
染色体chr16,坐标227431,核苷酸c
染色体chr16,坐标227433,核苷酸t
染色体chr16,坐标227436,核苷酸t
染色体chr16,坐标227442,核苷酸t
染色体chr16,坐标227443,核苷酸t
染色体chr16,坐标227454,核苷酸c
染色体chr16,坐标227455,核苷酸c
染色体chr16,坐标227457,核苷酸g
染色体chr16,坐标227459,核苷酸c
染色体chr16,坐标227472,核苷酸c
染色体chr16,坐标227474,核苷酸t
染色体chr16,坐标227481,核苷酸g
染色体chr16,坐标227484,核苷酸c
染色体chr16,坐标227487,核苷酸c
染色体chr16,坐标227488,核苷酸c
染色体chr16,坐标227490,核苷酸t
染色体chr16,坐标230700,核苷酸c
染色体chr16,坐标230750,核苷酸a
染色体chr16,坐标231000,核苷酸t
染色体chr16,坐标231050,核苷酸t
染色体chr16,坐标231100,核苷酸c
染色体chr16,坐标231150,核苷酸t
作为本发明优选的技术方案,所述对照探针影响覆盖深度的标准化效果,进而影响拷贝数估算结果。其中对照探针捕获区域经如下步骤筛选:(1)对照探针以安捷伦clearseq探针组(一种捕获试剂)为备选,并从中挑选合适的探针区间;(2)挑选gc含量与所述主要探针相近的区间;(3)挑选训练集中覆盖深度与所述主要探针覆盖深度相关性大于0.8的区间;对照探针序列如下表2所示:
表2
作为本发明优选的技术方案,所述数据分析为结合捕获探针所开发的独特的针对高度同源区的拷贝数估算方法,包括将测序读段使用比对软件比对到人参考基因组、去除重复序列、去除比对结果不可信的读段,标准化覆盖深度,利用极大似然估计估算α地中海贫血相关基因的拷贝数以及缺失类型分型。
作为本发明优选的技术方案,所述去除重复序列具体使用picard去除pcr扩增过程中产生的重复序列。
作为本发明的优选技术方案,所述比对结果不可信的读段为不涵盖任何一个如上所述差异位点的读段。
作为本发明优选的技术方案,所述标准化覆盖深度具体包括如下步骤:
步骤1,计算覆盖深度:计算目标捕获区域每个碱基的覆盖深度;所述覆盖深度是指分配至所述碱基的读段数目;
步骤2,计算特征位点(特征位点如上所述)的覆盖深度;
步骤3,标准化特征位点覆盖深度:
覆盖深度的标准化主要分为两个维度:首先,“单样本多位点标准化”,该步骤是相对于同一样本所有特征位点,即通过所有特征位点的覆盖深度和相关对照探针捕获区间的平均覆盖深度进行计算的,公式如下:
其中,readdepthofuniquesitei为特征位点的覆盖深度,sample’savg.depth指每个样本对照探针的覆盖深度。
其次,“多样本单位点标准化”,该步骤是相对于同一特征位点所有样本进行计算的,所述所有样本指已有训练集中的所有样本,公式如下:
其中,site’savg.depth指每个所选特征位点在训练集中经过第一步标准化之后的平均覆盖深度。
作为本发明的优选技术方案,所述利用极大似然估计估算α地中海贫血相关基因的拷贝数以及缺失类型分型具体包括如下步骤:
(1)假设所选取的特征位点之间相互独立
(2)计算z-score:对于一批样本中的每个样本每个特征位点,按如下公式计算z-score:
其中,zi,j表示第j个样本的特征位点i的z-score值,normrd′i,j为第j个样本特征位点i经步骤3计算得到的标准化覆盖深度,expectedcopynumber为相对于某一种缺失类型在该位点的期望拷贝数,sd(normrd′i)为该特征位点矫正后覆盖深度的标准差;
(3)极大似然估计:把每个位点相对于某一种基因型计算的z-score转化为概率值,对于某一个样本某一种假设缺失类型,将所有位点的概率值相乘,最大概率值所对应的基因型即为该样本所对应的基因型。
作为本发明的优选技术方案,所述捕获探针对目标区域进行捕获后,采用磁珠分离富集,pcr进行扩增,构建测序文库,再进行测序,并利用结合所述捕获探针所开发的独特的针对高度同源区的拷贝数估算方法估算hba1基因和hba2基因外显子1和外显子3的拷贝数,并对缺失类型进行基因分型。
与现有技术相比,本发明的有益效果在于:
1)本发明充分利用基因捕获(capture-based)的技术克服扩增子测序(ampliconsequencing)所带来的测序不均一的问题。
2)本发明采用了illumina二代测序平台,使得获得的目标基因序列信息错误率更低、稳定性更好、通量更大。
3)针对高度同源基因hba1和hba2差异位点设计探针,捕获序列特异性高,读段能够准确地匹配到hba1或hba2,hba1/2的拷贝数估算结果精度高。
4)针对x、y、z同源盒之间的非同源区以及所选特征位点设计捕获探针,有利于进一步确定缺失区域,从而进行更为精确地基因分型。
5)本发明结合设计的捕获探针所开发的独特的针对高度同源区的拷贝数估算方法具有灵敏度高、特异度高等特点。
经实验验证,本发明试剂盒能准确检测捕获α地中海贫血相关基因拷贝数并进行缺失类型分型,检测灵敏度为100%,特异性为99.89%,精度为96.55%,准确性为99.90%。解决了现有方法错误率较高、稳定性不好、通量较小、特异性差,尤其是对hba1和hba2基因的高度同源性欠缺考虑以及缺失类型种类多且难以区分等问题。
附图说明
图1是α珠蛋白基因簇上两个高度同源的重复单元中包含的x、y和z同源盒示意图。显示α珠蛋白基因簇中同源盒x、y、z所在位置。
图2是hba1基因和hba2基因的差异位点所在区域示意图。
具体实施方式
以下通过实施例对本发明进行更加具体的说明。应当理解,此处所描述的实施例是用于解释本发明,而非用于限定本发明。
实施例1:
本发明实施例提供了一种基于二代测序技术的α-地中海贫血突变的检测方法,本实施例对986个样本进行了分析,其中980个样本的α-地中海贫血检测结果用mlpa方法进行了验证,操作步骤具体包括:
1.目标基因和参考序列的设计及合成
在本实施例中,针对hbz/hbzp1/hba2/hba1/hbq1/hs-40基因区域设计了捕获探针,同时添加了对照探针。
2.从样本中提取dna和打断;
提取986个样本的dna3ug,该986样本中包含血液样本以及唾液样本。采用dna提取、纯化,采用超声波对提取的dna进行打断,末端补平并磷酸化,两侧加上接头。
3.目标区域捕获:
本方法使用专门设计的捕获探针对目标区域进行捕获、磁珠分离富集、构建测序文库。此处,目标区域主要包括hbz/hbzp1/hba2/hba1/hbq1/hs-40基因外显子及其上下游一定区域,此外还包括特定的对照探针的捕获区域。hbz/hbzp1/hba2/hba1/hbq1/hs-40的目标捕获区域和相应探针序列为针对α珠蛋白基因簇上x、y、z同源盒之间的非同源区设计(如表1所述),并且包含所有用于估算拷贝数的特征位点(特征位点通过已有数据训练得到);对照探针捕获区域通过训练集筛选:(1)对照探针以安捷伦clearseq探针组(一种捕获试剂)为备选,并从中挑选合适的探针区间;(2)挑选gc含量与所述主要探针相近的区间;(3)挑选训练集中覆盖深度与所述主要探针覆盖深度相关性大于0.8的区间;对照探针序列如下表2所示:
表1
表2
4.测序
测序文库的dna片段被杂交到测序仪的流动槽(flowcell)上并以之为模板生长dna簇,然后在illuminahiseq平台(illumina高通量测序平台)上进行150bp双端测序。
5.数据分析
步骤a,将测序读段(reads)使用比对软件(bwa软件,版本:0.7.12)比对到人参考基因组(hg19)。
步骤b,去除重复序列。使用picard(一种基本序列处理工具,版本2.8)去除pcr扩增过程产生的重复序列(pcrduplicatereads)。
步骤c,去除比对结果不可信的读段。此处,比对结果不可信是指:由于hba1和hba2高度同源,虽然比对软件将一个读段分配到其中一个基因的外显子,但实际上该读段既可以被分配到hba1,也可以分配到hba2。对于被分配到hba1和hba2外显子1和外显子3的读段,本发明剔除那些不涵盖任何一个如下所述用于区分hba1和hba2的外显子1和外显子3的位点的读段:
染色体chr16,坐标222869,hba2基因,外显子1上游23bp,核苷酸g;
染色体chr16,坐标226673,hba1基因,外显子1上游23bp,核苷酸c;
染色体chr16,坐标223447,hba2基因,外显子3上游24bp,核苷酸g;
染色体chr16,坐标227258,hba1基因,外显子3上游24bp,核苷酸c;
染色体chr16,坐标223614,hba2基因,外显子3下游14bp,核苷酸a;
染色体chr16,坐标227425,hba1基因,外显子3下游14bp,核苷酸g;
染色体chr16,坐标223618,hba2基因,外显子3下游18bp,核苷酸g;
染色体chr16,坐标227429,hba1基因,外显子3下游18bp,核苷酸a;
染色体chr16,坐标223620,hba2基因,外显子3下游20bp,核苷酸t;
染色体chr16,坐标227431,hba1基因,外显子3下游20bp,核苷酸g;
染色体chr16,坐标223622,hba2基因,外显子3下游22bp,核苷酸c;
染色体chr16,坐标227433,hba1基因,外显子3下游22bp,核苷酸t;
染色体chr16,坐标223625,hba2基因,外显子3下游25bp,核苷酸c;
染色体chr16,坐标227436,hba1基因,外显子3下游25bp,核苷酸t;
染色体chr16,坐标223631-223632,hba2基因,外显子3下游31-32bp,核苷酸gc;
染色体chr16,坐标227442-227443,hba1基因,外显子3下游31-32bp,核苷酸ct;
染色体chr16,坐标223643-223644,hba2基因,外显子3下游43-44bp,核苷酸aa;
染色体chr16,坐标227454-227455,hba1基因,外显子3下游43-44bp,核苷酸cc;
染色体chr16,坐标223646,hba2基因,外显子3下游46bp,核苷酸g;
染色体chr16,坐标223457,hba1基因,外显子3下游46bp,核苷酸a;
染色体chr16,坐标223648,hba2基因,外显子3下游48bp,核苷酸g;
染色体chr16,坐标223459,hba1基因,外显子3下游48bp,核苷酸c。
hba1和hba2基因具有高度同源性,能否准确区分这两个基因对于拷贝数估算结果至关重要,本发明根据上述特定差异位点设计的探针均能直接用于区分hba1和hba2基因,解决了本领域的技术难题。这样的设计,使本发明试剂盒在实际估算样本拷贝数时能够直接估算样本拷贝数并且能够处理更多的情况(比如hba1和hba2的某些特定位点发生突变时),从而具有决定性的优势,达到了本领域所预料不到的技术效果。
步骤d,标准化覆盖深度,具体包括:步骤1,计算覆盖深度:将目标捕获区域划分为固定长度的区间,并计算每个区间的平均覆盖深度;所述覆盖深度是指分配至所述区间的读段数目与该区间大小的比值;
步骤2,挑选差异位点,差异位点通过已有训练集挑选,包括如下步骤;(1)挑选能够区分hba1基因以及hba2基因的差异位点,其中位点区域如图2所示;(2)挑选hba1基因以及hba2基因上其它位点(除hba1/hba2差异位点外的其它位点),所挑选位点需避开多态位点、常见snp(单核苷酸多态性)变异位点;(3)挑选hbz、hbq1、hbzp1基因外显子区域上的位点,所挑选位点均匀分布各基因外显子区域,并避开高度同源区、回文序列区域、多态位点、常见snp(单核苷酸多态性)变异位点等;(4)根据训练集进一步筛选步骤(1)(2)(3)所选位点,其中所选位点在训练集中经过两步标准化之后的覆盖深度的标准差要小于0.18(将经过这四步筛选所得到的位点定义为特征位点)。所选77个特征位点,如下所示:
染色体chr16,坐标203900,核苷酸a
染色体chr16,坐标203950,核苷酸g
染色体chr16,坐标204000,核苷酸a
染色体chr16,坐标204050,核苷酸g
染色体chr16,坐标204400,核苷酸c
染色体chr16,坐标214480,核苷酸g
染色体chr16,坐标214500,核苷酸a
染色体chr16,坐标214505,核苷酸a
染色体chr16,坐标214600,核苷酸g
染色体chr16,坐标214605,核苷酸t
染色体chr16,坐标219818,核苷酸t
染色体chr16,坐标219853,核苷酸c
染色体chr16,坐标219922,核苷酸g
染色体chr16,坐标221062,核苷酸c
染色体chr16,坐标221082,核苷酸a
染色体chr16,坐标221108,核苷酸c
染色体chr16,坐标221128,核苷酸c
染色体chr16,坐标223383,核苷酸c
染色体chr16,坐标223447,核苷酸c
染色体chr16,坐标223614,核苷酸g
染色体chr16,坐标223618,核苷酸t
染色体chr16,坐标223620,核苷酸c
染色体chr16,坐标223622,核苷酸t
染色体chr16,坐标223625,核苷酸t
染色体chr16,坐标223631,核苷酸c
染色体chr16,坐标223632,核苷酸t
染色体chr16,坐标223643,核苷酸a
染色体chr16,坐标223644,核苷酸c
染色体chr16,坐标223646,核苷酸g
染色体chr16,坐标223648,核苷酸c
染色体chr16,坐标223661,核苷酸c
染色体chr16,坐标223663,核苷酸t
染色体chr16,坐标223670,核苷酸g
染色体chr16,坐标223673,核苷酸c
染色体chr16,坐标223678,核苷酸t
染色体chr16,坐标225100,核苷酸a
染色体chr16,坐标225120,核苷酸g
染色体chr16,坐标225141,核苷酸g
染色体chr16,坐标225161,核苷酸c
染色体chr16,坐标225181,核苷酸c
染色体chr16,坐标225735,核苷酸t
染色体chr16,坐标225745,核苷酸c
染色体chr16,坐标225755,核苷酸a
染色体chr16,坐标225765,核苷酸g
染色体chr16,坐标225780,核苷酸t
染色体chr16,坐标226043,核苷酸t
染色体chr16,坐标226220,核苷酸c
染色体chr16,坐标226221,核苷酸c
染色体chr16,坐标226673,核苷酸c
染色体chr16,坐标227187,核苷酸c
染色体chr16,坐标227250,核苷酸c
染色体chr16,坐标227253,核苷酸g
染色体chr16,坐标227258,核苷酸c
染色体chr16,坐标227425,核苷酸g
染色体chr16,坐标227429,核苷酸t
染色体chr16,坐标227431,核苷酸c
染色体chr16,坐标227433,核苷酸t
染色体chr16,坐标227436,核苷酸t
染色体chr16,坐标227442,核苷酸t
染色体chr16,坐标227443,核苷酸t
染色体chr16,坐标227454,核苷酸c
染色体chr16,坐标227455,核苷酸c
染色体chr16,坐标227457,核苷酸g
染色体chr16,坐标227459,核苷酸c
染色体chr16,坐标227472,核苷酸c
染色体chr16,坐标227474,核苷酸t
染色体chr16,坐标227481,核苷酸g
染色体chr16,坐标227484,核苷酸c
染色体chr16,坐标227487,核苷酸c
染色体chr16,坐标227488,核苷酸c
染色体chr16,坐标227490,核苷酸t
染色体chr16,坐标230700,核苷酸c
染色体chr16,坐标230750,核苷酸a
染色体chr16,坐标231000,核苷酸t
染色体chr16,坐标231050,核苷酸t
染色体chr16,坐标231100,核苷酸c
染色体chr16,坐标231150,核苷酸t
步骤3,标准化覆盖深度:
覆盖深度的标准化主要分为两个维度:首先,“单样本多位点标准化”,该步骤是相对于同一样本所有特征位点,即所有特征位点的覆盖深度和相关对照探针捕获区间的覆盖深度进行计算的,公式如下:
其中,readdepthofuniquesitei为特征位点的覆盖深度,sample’savg.depth指每个样本对照探针的覆盖深度。
其次,“多样本单位点标准化”,该步骤是相对于同一特征位点所有样本(所有样本指已有训练集中的所有样本)进行计算的,公式如下:
其中,site’savg.depth指每个所选特征位点在训练集中经过第一步标准化之后的平均覆盖深度。
步骤e,极大似然估计:
(1)假设所选取的特征位点之间相互独立
(2)计算z-score:对于一批样本中的每个样本每个差异位点,按如下公式计算z-score:
其中,zi,j表示第j个样本的特征位点i的z-score值,normrd′i,j为第j个样本特征位点i经步骤3计算得到的标准化覆盖深度,expectedcopynumber为相对于某一种缺失类型在该位点的拷贝数,sd(normrd′i)为在训练集中该特征位点矫正后覆盖深度的标准差。
(3)极大似然估计:把每个位点相对于某一种基因型计算的z-score转化为概率值,对于某一个样本某一种假设缺失类型,将所有位点的概率值相乘,最大概率值所对应的基因型即为该样本所对应的基因型。
6.实验结果
表3.980个样本的实验结果(所有样本均用mlpa进行过验证)
表3的检测结果表明,在980个进行过mlpa验证的样本中,一共有28个携带者(hba1/hba2基因存在缺失,经过mlpa验证),对于这28个样本,本发明全部准确检测出来,灵敏度为100%,特异性为99.89%,精度为96.55%,准确性为99.90%。本发明所检测的所有真阳性样本的缺失类型如下表4所示,其中有6个样本为--sea/αα缺失类型,20个样本为-α3.7/αα缺失类型,2个样本为-α4.2/αα缺失类型。
表4.本方法检测的阳性结果与mlpa结果的比较(29个样本)
以上仅是本发明的具体应用范例,对本发明的保护范围不构成任何限制;对于所属领域的普通技术人员来说,在上述说明的基础上还可以做出其它不同形式的变化或变动。这里无需也无法对所有的实施方式予以列举说明。凡采用等同变换或者等效替换而形成的类似此种的技术方案,均落在本发明权利保护范围之内。
序列表
<110>明码(上海)生物科技有限公司
<120>用于捕获α地中海贫血相关基因拷贝数检测试剂盒
<130>wh-yxsc-np-18-100070c
<160>88
<170>siposequencelisting1.0
<210>1
<211>240
<212>dna
<213>人工序列(未知)
<400>1
atgagcgccgcccggccgggcgtgcccccgcgccccaagcataaaccctggcgcgctcgc60
ggcccggcactcttctggtccccacagactcagagagaacccaccatggtgctgtctcct120
gccgacaagaccaacgtcaaggccgcctggggtaaggtcggcgcgcacgctggcgagtat180
ggtgcggaggccctggagaggtgaggctccctcccctgctccgacccgggctcctcgccc240
<210>2
<211>240
<212>dna
<213>人工序列(未知)
<400>2
caggcggcggctgcgggcctgggccctcggccccactgaccctcttctctgcacagctcc60
taagccactgcctgctggtgaccctggccgcccacctccccgccgagttcacccctgcgg120
tgcacgcctccctggacaagttcctggcttctgtgagcaccgtgctgacctccaaatacc180
gttaagctggagcctcggtggccatgcttcttgccccttgggcctccccccagcccctcc240
<210>3
<211>240
<212>dna
<213>人工序列(未知)
<400>3
atgagcgccgcccggccgggcgtgcccccgcgccccaagcataaaccctggcgcgctcgc60
gggccggcactcttctggtccccacagactcagagagaacccaccatggtgctgtctcct120
gccgacaagaccaacgtcaaggccgcctggggtaaggtcggcgcgcacgctggcgagtat180
ggtgcggaggccctggagaggtgaggctccctcccctgctccgacccgggctcctcgccc240
<210>4
<211>240
<212>dna
<213>人工序列(未知)
<400>4
tgtagcgcaggcggcggctgcgggcctgggccgcactgaccctcttctctgcacagctcc60
taagccactgcctgctggtgaccctggccgcccacctccccgccgagttcacccctgcgg120
tgcacgcctccctggacaagttcctggcttctgtgagcaccgtgctgacctccaaatacc180
gttaagctggagcctcggtagccgttcctcctgcccgctgggcctcccaacgggccctcc240
<210>5
<211>180
<212>dna
<213>人工序列(未知)
<400>5
cctgagcaggcccaactccagtgcagctgcccaccctgccgccatgtctctgaccaagac60
tgagaggaccatcattgtgtccatgtgggccaagatctccacgcaggccgacaccatcgg120
caccgagactctggagaggtgagtgtcagacgggactgccagagggactgggtgggaggc180
<210>6
<211>240
<212>dna
<213>人工序列(未知)
<400>6
ccccgccccccgtcccaggctcttcctcagccacccgcagaccaagacctacttcccgca60
cttcgacctgcacccggggtccgcgcagttgcgcgcgcacggctccaaggtggtggccgc120
cgtgggcgacgcggtgaagagcatcgacgacatcggcggcgccctgtccaagctgagcga180
gctgcacgcctacatcctgcgcgtggacccggtcaacttcaaggtgcgcggggcgcggtg240
<210>7
<211>240
<212>dna
<213>人工序列(未知)
<400>7
gccgggcggggcccgggctaggccccgccccctcactgagccgcccccgcccccagctcc60
tgtcccactgcctgctggtcaccctggccgcgcgcttccccgccgacttcacggccgagg120
cccacgccgcctgggacaagttcctatcggtcgtatcctctgtcctgaccgagaagtacc180
gctgagcgccgcctccgggacccccaggacaggctgcggcccctcccccgtcctggaggt240
<210>8
<211>221
<212>dna
<213>人工序列(未知)
<400>8
caggcgcagcggggtcgcagggcgcggcgggttccagcgcggggatggcgctgtccgcgg60
aggaccgggcgctggtgcgcgccctgtggaagaagctgggcagcaacgtcggcgtctaca120
cgacagaggccctggaaaggtgcggcaggctgggcgcccccgcccccaggggccctccct180
ccccaagccccccggacgcgcctcacccacgttcctctcgc221
<210>9
<211>241
<212>dna
<213>人工序列(未知)
<400>9
acccacgttcctctcgcaggaccttcctggctttccccgccacgaagacctacttctccc60
acctggacctgagccccggctcctcacaagtcagagcccacggccagaaggtggcggacg120
cgctgagcctcgccgtggagcgcctggacgacctaccccacgcgctgtccgcgctgagcc180
acctgcacgcgtgccagctgcgagtggacccggccagcttccaggtgagcggctgccgtg240
c241
<210>10
<211>240
<212>dna
<213>人工序列(未知)
<400>10
gtgcggggggcgtgcggggcgggtgcaggcgagtgagccttgagcgctcgccgcagctcc60
tgggccactgcctgctggtaaccctcgcccggcactaccccggagacttcagccccgcgc120
tgcaggcgtcgctggacaagttcctgagccacgttatctcggcgctggtttccgagtacc180
gctgaactgtgggtgggtggccgcgggatccccaggcgaccttccccgtgtttgagtaaa240
<210>11
<211>120
<212>dna
<213>人工序列(未知)
<400>11
cctgagcaggcccaactccagtgcagccgcccaccctgccgccatgtctctgaccaagac60
ttaggggaccatcattgtgtccatgtgggccaagatctccacgcaggccgacaccatcgg120
<210>12
<211>240
<212>dna
<213>人工序列(未知)
<400>12
ccccgccccccgtcccaggctcttcctcagccacccgcagaccaagacctacttcccgca60
cttcgacctgcacccggggtccgcgcagttgcgcgcgcacggctccaaggtggtggccgc120
cgtgggcgacgcggtgaagagcatcgacgacatcggcggcgccctgtccaagctgagcga180
gctgcacgcctacatcctgcgcgtggacccggtcaacttcaaggtgcgcggggcgcggtg240
<210>13
<211>180
<212>dna
<213>人工序列(未知)
<400>13
gcggggcggggcccgggctaggccccgcccccgcactgagccgcccccgcccccagctcc60
tgtcccactgcctgctggtcaccctggccgcgcgcttccccgccgacttcacggccgagg120
cccacgccgcctgggccaagttcctatcggtcgtatcctctgtcctgaccgagaagtacc180
<210>14
<211>200
<212>dna
<213>人工序列(未知)
<400>14
ccaagggtggaggcatgcagctgtgggggtctgtgaaaacacttgagggagcagataact60
gggccaaccatgactcagtgcttctggaggccaacaggactgctgagtcatcctgtgggg120
gtggaggtgggacaagggaaaggggtgaatggtactgctgattacaacctctggtgctgc180
ctccccctcctgtttatctg200
<210>15
<211>160
<212>dna
<213>人工序列(未知)
<400>15
catactgtagatacctgtgtacaacttcctattctcagtgaagtgtctcccctgcatccc60
tttcagccagttcattcagctctgcgccattccacagtctcactgattattactatgttt120
ccatcatgatccccccaaaaaatcatgactttattttttt160
<210>16
<211>101
<212>dna
<213>人工序列(未知)
<400>16
cctcccagaggaggttgaatgctccagccggttccagctattgctttgtttacctgttta60
accagtatttacctagcaagtcttccatcagatagcatttg101
<210>17
<211>201
<212>dna
<213>人工序列(未知)
<400>17
atgaggtctgcacctctgtgtgtacttgtgtgatggttagaggactgcctacctcccaga60
ggaggttgaatgctccagccggttccagctattgctttgtttacctgtttaaccagtatt120
tacctagcaagtcttccatcagatagcatttggagagctgggggtgtcacagtgaaccac180
gacctctaggccagtgggaga201
<210>18
<211>120
<212>dna
<213>人工序列(未知)
<400>18
ggtctcacccagctcagctgaggtaactcattttgccatttcttcatttttaggacaact60
gtcatctgggaagtaacaacgcaggatgccccctggggtggactgccccatggaattctg120
<210>19
<211>120
<212>dna
<213>人工序列(未知)
<400>19
cattaaattccagtgccaggattccagaagcactgtgacaatgctgattgggagctctct60
ctcttacctctctggaaggacttgaaaactccatcccctcagggtcattagatgaagaga120
<210>20
<211>120
<212>dna
<213>人工序列(未知)
<400>20
ggcccagcttctgattctttaaaaaccccagggatgccagaggggaacaggaggtgacac60
ttgggaactgcagctgctctattctagtccgtactctccagtcacatgtctcaacccctt120
<210>21
<211>120
<212>dna
<213>人工序列(未知)
<400>21
tccaagctcactgttctcaccacacggccccactccggtttcctggaggctcccccagaa60
gcatttgggggacatagtccaggcaggtgcctcctcccaggtggggcaagcacctggcat120
<210>22
<211>120
<212>dna
<213>人工序列(未知)
<400>22
ataaatatcttttgaatgattgaaggacctaaagccaccctccccctactagggtgacat60
ctatgtgctgtcttctctggaccgggagaagaaggaccactatatcctgactgccttggc120
<210>23
<211>120
<212>dna
<213>人工序列(未知)
<400>23
cctttgatggattgttcagctctggctgtgtccccgagccacactctttccatttccttc60
cttttgctcagtatttgtggacttttttctacttcctctttcacacttttcctttggatg120
<210>24
<211>120
<212>dna
<213>人工序列(未知)
<400>24
ggtctccgcacaccgcccccgcccacagaacccaggggagccgactgcagccactactca60
ctggtccaggtggacctgccagtaggccttgcgggtgacattcaggtaggacagagaacc120
<210>25
<211>120
<212>dna
<213>人工序列(未知)
<400>25
agctgctgtatcccaccctatgaggctgcaaagatgggatggagagggagagagtagcaa60
gttgcagaaagccataggtaaaatccaatcagtagtcagcagagggcaagaaacgcccag120
<210>26
<211>120
<212>dna
<213>人工序列(未知)
<400>26
cgcggggcacggcgagggcagtgcagggtcgaggcgcccggtccaggctgcggcgccgct60
actcacctctccgaactggaaggcggagacgatggtgacaacgacgcccaagaagcacac120
<210>27
<211>120
<212>dna
<213>人工序列(未知)
<400>27
gggcaagctcctctctccacggccatcgccatggagggatcctccaagctcccactttct60
gtgatttcagagtccctcagactcggccgatgggtcatttttcatgtatattttctcttg120
<210>28
<211>120
<212>dna
<213>人工序列(未知)
<400>28
tttggaagaactttaaaatggtgtatgccatgagtatttctttgcatttttcacctgtcc60
ccaccctacctttgtctcccgttttggtgtaaatcttggggatcctgggtgtcttcgagg120
<210>29
<211>120
<212>dna
<213>人工序列(未知)
<400>29
gggatggaggggtcccctcaagggaaggctctgactgtacccccatgtttaggaggcagc60
catggccaagctggccgcctcggaggccgcgaccgccatcagccaccaggtgagtgtcca120
<210>30
<211>120
<212>dna
<213>人工序列(未知)
<400>30
tgggatagaggttaactcggttgtgaaataatggccaaagcggtgcactcttaataactt60
ttttgtagccctggagaaagcttctggattgactgtgagaagcttggcagaaggtgctgc120
<210>31
<211>120
<212>dna
<213>人工序列(未知)
<400>31
gccccacttacaaatgcaggcagcagccagcaggcggctggcagcataaggggcgaagca60
gctcgggaagagaggctgtaccatgtagttggcaaaggtgatggcaatgatggcctggct120
<210>32
<211>120
<212>dna
<213>人工序列(未知)
<400>32
agcactgctgcccaacagtggcccgccctccacctgctctgtctcccaagtccaggtggg60
tggcactgacctgtcaacatgcttgagaagagcatggctgggaagtagtggtggaagtag120
<210>33
<211>120
<212>dna
<213>人工序列(未知)
<400>33
actataacatttctggtttagtgtggccccagggttcagtcagtccatttcaagtcagga60
agccttactcctctcattcccatgtagctgtgatgctcatatttatccccaggcttctgg120
<210>34
<211>120
<212>dna
<213>人工序列(未知)
<400>34
gggcggccctcacctctgcttggggtccttcttgagcagccctgaaagcaaggacttggc60
ctcgggaccaagcgtgcgcgggaagcggatctcctccatgaggatgagctcaaaaagctt120
<210>35
<211>120
<212>dna
<213>人工序列(未知)
<400>35
gagggctccagccaaccccccaaatctgaatcccgagaggccaaggggatacttacgcgc60
cacagagaagttgttgaggggagcctcacgttggtccacatcctgcggccgctccttgta120
<210>36
<211>120
<212>dna
<213>人工序列(未知)
<400>36
tacaagatggcactattttaaactcacctcccttttgtgaggaatgaagcaaactcctgg60
cctgcaggagcccaagagctctgcccggcagcccccctggggcaggagtgcgaggggtca120
<210>37
<211>120
<212>dna
<213>人工序列(未知)
<400>37
tctgggtagtggagatgctgtctgcagtggcatctgtttacctctctcctattagcttcg60
tcagaccatggctaagttccttcaggagcacctggcccccaaggcccaggagatcgatcg120
<210>38
<211>120
<212>dna
<213>人工序列(未知)
<400>38
tccccaaccctgcccctacttaccaggccgagggggatatggccaccgccaatgtggggc60
aagttccctctgccaagcagacctccacgtcgaacaccagggcccgctcctcggggatgg120
<210>39
<211>120
<212>dna
<213>人工序列(未知)
<400>39
cctggggccacgtggcagggacagagatgggtccatggcagtgtctgctcttctctgtga60
aggcaaagggcctctgagggagtattatagccgcctcatccaccagaagcacttccagca120
<210>40
<211>120
<212>dna
<213>人工序列(未知)
<400>40
tgtctgacctgtccctctgcccacaggtgaatgtgacctgcgtcctgaggatcccgagta60
tcaggagagctggcagaggaagtcatgcagaggcaaagccaggagccccgacagggagat120
<210>41
<211>120
<212>dna
<213>人工序列(未知)
<400>41
ccccacatggcacggagccgggctgggatttgatgagaaaggcctgagcccacctgccac60
cctccctgcagtagtaggaatagacaaggttgatgcagccgcccagagtcagggctggaa120
<210>42
<211>120
<212>dna
<213>人工序列(未知)
<400>42
tgttacccattagagtaaccacttcaccgactggcaagcgagggaggggagaccaagggc60
tctgagcagcccccaaagctccttgtccctcagggtggctatgtggggagcggcctacct120
<210>43
<211>120
<212>dna
<213>人工序列(未知)
<400>43
agctagatgccagcggggtcctcggtaactgtacgtggcatcggattgaacctcagtctt60
cctaacaggttgtggtgcctcctggagtgacaaactcctcttttcaagtgacatctcaaa120
<210>44
<211>120
<212>dna
<213>人工序列(未知)
<400>44
caccccacctaccggacagatgaacagtttctgctgcagcggctggcagacggggccatc60
gacctctatgccatggtggtggttctctcgaggtgaggaggcaggcagggaatgcctgag120
<210>45
<211>120
<212>dna
<213>人工序列(未知)
<400>45
gctccccactagggtgttccctcaggtcaatgtcacgaagatgggcagttggggccactt60
taactgttcctactcctgctccttccttctggctccggaagaccccatattccccatcat120
<210>46
<211>120
<212>dna
<213>人工序列(未知)
<400>46
acctcctgggctctggcctggcctctgtgcctcaccccacgccctgtcctcggcacgggg60
tcctcacctgtgcgcacaccagcttggctgagcagcagcggcaggctccgcaccttgtcg120
<210>47
<211>120
<212>dna
<213>人工序列(未知)
<400>47
aaaaaaaaaaaggaagcaacacaaagggacatgttcaggtagccagctccttctttctcc60
aggctcagcagactacaggactggtaggattgaaagcataattacctaagaaaaatgcta120
<210>48
<211>120
<212>dna
<213>人工序列(未知)
<400>48
gtgacttatttcttcaaacagcaggttacctggtgcagtatctgtatgtcaagcggaggt60
ccaaagggtacatcagctcccgaagggtatggctggtaaatgtgtttgtcagtgagaggg120
<210>49
<211>120
<212>dna
<213>人工序列(未知)
<400>49
gagatgggatcaggatcaggagaaaaggagagcgagagaaacaggaggcatcacccctgc60
tctcaagttccaatggacacatgtaataatgactaagctcattgccagagagacgcccca120
<210>50
<211>120
<212>dna
<213>人工序列(未知)
<400>50
ctctcctattcccacagagaccgagcggcttctgacccccaaccccgggtatgggaccca60
ggcggggccttcaccggcccctccgacacccccagaagaggaagaccttcgccgtcgtct120
<210>51
<211>120
<212>dna
<213>人工序列(未知)
<400>51
tgggtgggctgcagagagcgggccggactcacaggccctccccttctctgcccacagtac60
ctggcgttgcctgacgtgtcactgggccggtatgcgtatgtccgtggtgggggtgaccct120
<210>52
<211>120
<212>dna
<213>人工序列(未知)
<400>52
ggccttggaggttgggagccactttcaggctgagcctcccggcttctctccccagcatcc60
cggcggcgcaggcgcagaggagagcgagctgcaggcctacatcgcacagtgccaggacag120
<210>53
<211>120
<212>dna
<213>人工序列(未知)
<400>53
gtgagccaccctgcctggctgtttttaaagtgtgccccaaacataatcccggactattcc60
ttaccttcaagagatgggtcatcatcatagattggttttgctgaaccagaaaagaagagt120
<210>54
<211>120
<212>dna
<213>人工序列(未知)
<400>54
gtccggggcgaggcggatgcaggggaggcctggcgttcctccgcggttcctgtcacaaag60
gcgacgacaagtcccgggtccccggagccgcctccgcgacatacacgagtcgccctccgt120
<210>55
<211>120
<212>dna
<213>人工序列(未知)
<400>55
caccaccctcctcccgagcagggatgagcaagggggcacagggagggctgaccacagcta60
cctgtttgcccagcagcaccaggtccaccttctccttctctgccagcttggccaggaccc120
<210>56
<211>120
<212>dna
<213>人工序列(未知)
<400>56
atgaggcctgctcactggtagaacttcttgttagggctgttagcatggtcagctagcagc60
tggcatggggtgaactgttttccataggcagcttcatatttcttgagccggtccactatc120
<210>57
<211>120
<212>dna
<213>人工序列(未知)
<400>57
ggccaggccctccagtggccccctgcctcacctgctgccctccattctgtcccccacctc60
tccaggtacaagtgcctgctcctggtggattcggtggcatccctgggcgggacccccctt120
<210>58
<211>120
<212>dna
<213>人工序列(未知)
<400>58
tggactggcctgccctgaggtgggactcacccgtcccgagcaaaccacccatctacaggc60
atcgacatcctgtactcgggctcccagaaggccctgaacgcccctccagggacctcgctc120
<210>59
<211>120
<212>dna
<213>人工序列(未知)
<400>59
gaacccggacaggactcctctgcggaggataccggcctgtggtcagagagcctgtgctgt60
tggggctggggcagacagagctaatgccccatctgcccccaaccctgcccatggtgctgg120
<210>60
<211>120
<212>dna
<213>人工序列(未知)
<400>60
gctgtcggtcagggtcaggcaggtcccaggcgggaggctgacgtcagcccgccctgtgcc60
ccccaggtgctgcggatcggcctgctgggctgcaatgccacccgcgagaatgtggaccgc120
<210>61
<211>120
<212>dna
<213>人工序列(未知)
<400>61
aaaactccatgcaccagatgacaatactgaaagagacttggcagagctgtggaggcaggt60
gtgggatatattaaggacataccttgagaacttttgttccaggcaagagggatctaaggg120
<210>62
<211>120
<212>dna
<213>人工序列(未知)
<400>62
tggtcttcttggaaggaataaatgtaaaaatgttgctcagccccacagagttggggtgac60
tccacagggtgaaggcttggaggagtggcgtcttcagaggttctgccctgctcgttggtt120
<210>63
<211>120
<212>dna
<213>人工序列(未知)
<400>63
cagggagcaggcggccaggcgggccagcacatgcggctgcagctcagccatcccccccgg60
gtcccggcaggctcggcatgggtaggggacagccagccctggccaccccctctgggcctg120
<210>64
<211>120
<212>dna
<213>人工序列(未知)
<400>64
atggaagtgaactagctgggaaaacacacaagcaagcctcttacattaaaatcggatcct60
tttaccagaaagcatgcccactcacctctgcccaaaccatccccagcaaaagggaggccc120
<210>65
<211>120
<212>dna
<213>人工序列(未知)
<400>65
tcgacctgggctacagaccctgagggaagtggcctgggggaggggcccacactcacacag60
tccaccaccatcttgcccagttccttggccccaaaaacagtcttggccagctcccagggg120
<210>66
<211>120
<212>dna
<213>人工序列(未知)
<400>66
atcttcaatttaatcacctgaggcaatccttgtcagatcccctacatcaaatcagccacc60
aaatctcaagattctaatctatttctcctctctatccccaatgacaggttgccacacgct120
<210>67
<211>120
<212>dna
<213>人工序列(未知)
<400>67
tgggccactcacctccccagcttccccccaccccacaggcttctcacctccctggctggg60
cacccccgtgggccactcacctccccagtgccccctccccctgcaggccactcacctccc120
<210>68
<211>120
<212>dna
<213>人工序列(未知)
<400>68
agagcacgtgccggcgagcttgggcactggcagaggcgtggaggaagctgcttacagagc60
ctgcagcacagcgcaagtaatccatctcagccgggaacacgttccccaggtacagcgaaa120
<210>69
<211>120
<212>dna
<213>人工序列(未知)
<400>69
tgagaggtcctcacggataaccaccccagacttctcccagaaagccttaccacaggaact60
cgcggggaactgggcccatacacagattccccgtaggaagggttattcacattcatagtg120
<210>70
<211>120
<212>dna
<213>人工序列(未知)
<400>70
ttgtgcttggaggacctgatatttttattgaggcctattttcactacctcaaggttactt60
actttctgccccaacattccaggaaatcctgggaaaccctgtgaaaagagcaacaaataa120
<210>71
<211>120
<212>dna
<213>人工序列(未知)
<400>71
cccggttccccgccagcctgtcccctagcaatgcctccccgtacccgggtcccgcagact60
tacgcgcaagccgcgcgtagggcccagaagcagcagaaccagcaacagagggaggatgcg120
<210>72
<211>120
<212>dna
<213>人工序列(未知)
<400>72
ctctcaccccacccgcccgtgatgtcctctcaccctgcccgcccgtgatgccctcttgcc60
ccgcctgcccgcagtgcataggttccatgtgacagtggctccaggcaccaagttggagag120
<210>73
<211>120
<212>dna
<213>人工序列(未知)
<400>73
cggtcaggaacatggagcgggaacttccaaggtgcaggagaggcggagtggggaggaggg60
gctaggcagggtgggcagctctttactgcacaatctttgagatctgtgcccctctcggtc120
<210>74
<211>120
<212>dna
<213>人工序列(未知)
<400>74
gctcctacccgagggcacccctggaggaaattctcgaggtgacctggacagggcgggctg60
ggcgaggacgctcccggggcctggaggcggagtcgggggcgggcgcgcgcagtcatctga120
<210>75
<211>120
<212>dna
<213>人工序列(未知)
<400>75
attaaaaataaaataaaaattaaaaaatgagtgccttcaggcagtgttccaagcattggt60
gaacacaatggtggggctaatgcagaaaagagtatcatttttgcacacatttctctgggt120
<210>76
<211>120
<212>dna
<213>人工序列(未知)
<400>76
gctctgtgggcctctgagggcggcatgcgggactacgacgaggtgaccgccttcctgggc60
gagtgggggcccttccagcgcctcatcttcttcctgctcagcgccagcatcatccccaat120
<210>77
<211>120
<212>dna
<213>人工序列(未知)
<400>77
gaataaacaaaagtcagtgactgattgagatggcggggtgggcaatggggaagggatgta60
taggtagactcttaagtcagaagccttactgaaatcagccagaaagagctgggatcttct120
<210>78
<211>120
<212>dna
<213>人工序列(未知)
<400>78
cagtagaactggcacattttcaaagtgaaatggcgagagctggctagacaacccaaacac60
tcacatcagcatttgaaaagggccaaaataaatgtttgacaagaattcctttaaaaagag120
<210>79
<211>120
<212>dna
<213>人工序列(未知)
<400>79
agctccccactagctttttggttgacctctcagaccggcaagtggctcaccttcctccag60
gtcatgaaccctgcgtttgagggcagctgtcatagcatcagagcagagagagtagaggtc120
<210>80
<211>120
<212>dna
<213>人工序列(未知)
<400>80
agagagggagaacatgagctggggaggggaagtagctggggttctactctccagagaccc60
tcaccttcagtaagtggagcccaaggtgactacaggcagcagcaactactgtggtcttcc120
<210>81
<211>120
<212>dna
<213>人工序列(未知)
<400>81
cagtgagccacgagcctccgccaggcaggtgctcactgacactgtccccatgggtcgggc60
ttcattagaaaccacagacctcagaatgaataaccccctctcacactgccaggcaaatca120
<210>82
<211>120
<212>dna
<213>人工序列(未知)
<400>82
ccctgtatggtcaggctgggtggggatgggagaggcctggtgactgggaaccttttctcc60
cagccgagttcctgttctgggcttcgctgtgcatgacccatctcagcaggatggccgagg120
<210>83
<211>120
<212>dna
<213>人工序列(未知)
<400>83
aggcaatggaggcagatcagggcatggagaaacctgcctcagcgccatcttcctccctgg60
cacccagatgccattccgccaggcccacgaggcctccgggaaagctgtgttcatggccga120
<210>84
<211>120
<212>dna
<213>人工序列(未知)
<400>84
gcaaaaccagatttttagttaataataggttctggtttaattctacagtagataagtgtt60
tattactttattacttaaataaaagtcatctttttagaccgtgttagtgagtctcccgtc120
<210>85
<211>120
<212>dna
<213>人工序列(未知)
<400>85
aggactcctttggaatatagctgtggacaatgaatcctgcgagcgatgggggcacatcag60
agagcatttttgacctggactatgcatcctgggggatccgctccacgctgatggtcgctg120
<210>86
<211>120
<212>dna
<213>人工序列(未知)
<400>86
caggagagagggagctagagagctctgaggactgatcttgactgtctgcccccagaccat60
cagcatatccgctacaacccgctgcaggatgagtgggtgctggtgtcagctcaccgcatg120
<210>87
<211>120
<212>dna
<213>人工序列(未知)
<400>87
ccacagggatagtgaacctccttctgggtcatatcccaccaagctttttggtcccctagg60
gtgggccttccctactcccttgtagcctgtccagtctttgaagcccaccaggtaactggt120
<210>88
<211>120
<212>dna
<213>人工序列(未知)
<400>88
tccgccgttcccggcctcggcttcctgctgcctacaccgccctagcccaggcccccttag60
tgagcgcttggggagagaatttccccggacgcccccctgggcgtggcctcagccccattt120
- 还没有人留言评论。精彩留言会获得点赞!