基于Xgboost的药物靶点识别方法与流程
本发明涉及基于xgboost的药物靶点识别方法,属于药物靶点识别领域。
背景技术:
药物和生物大分子之间的结合位点是药物靶点。药物靶点涉及受体,酶,离子通道,转运蛋白,免疫系统,基因等。超过50%的现有药物以受体为靶标,受体成为主要和最重要的靶点。由于药物靶向研究是现代药物研究的源泉,它可以为重大疾病的预防和治疗提供重要信息,使基于新目标的新药开发具有重大的社会和经济效益。因此,药物靶标成为医学领域的热点。
大多数蛋白质药物是g蛋白偶联受体(gpcr)(23%)和酶(50%)。一些研究人员预测,有超过2000种蛋白质药物。然而,据报道只有数百种药物靶点。临床验证药物目标的数量仍然很少。部分原因是随着冗余数据的积累,简单的分析方法已不能满足大规模高通量数据分析的需要。但是,由于吞吐量,精度和成本的限制,实验方法,应用难以广泛开展。作为处理大量数据的快速且低成本的方法,基于机器学习的药物目标预测越来越受到关注。
黄晨等结合蛋白质的基本序列,两阶段结构和亚细胞定位,通过svm预测离子通道中的潜在药物靶标。hopkinsal等人基于序列同源性和结构域分析已知药物靶标并将其应用于寻找新靶点。基于蛋白质的3d结构,kinningssl等研究可以与药物化合物结合的结合区域。campillosm基于副作用的相似性预测潜在的药物靶标。郑等人发现药物结合位点始终具有一定的结构和理化性质。此外,
尽管研究人员在鉴定药物靶标方面取得了巨大成就,但鉴定庞大而复杂的酸序列需要一种具有高计算效率和高识别准确度的算法。chent在2004年提出了一种名为极限梯度增强(xgboost)的新方法,他改进了boost算法,它的多线程并行和正则化术语不仅提高了算法的准确性,而且缩短了运行时间。因此,xgboost是一种解决药物靶标识别问题的合适算法。
技术实现要素:
本发明的目的是为了解决上述现有技术存在的问题,进而提供基于xgboost的药物靶点识别方法。
本发明的目的是通过以下技术方案实现的:
基于xgboost的药物靶点识别方法,所述基于xgboost的药物靶点识别方法具体步骤为:
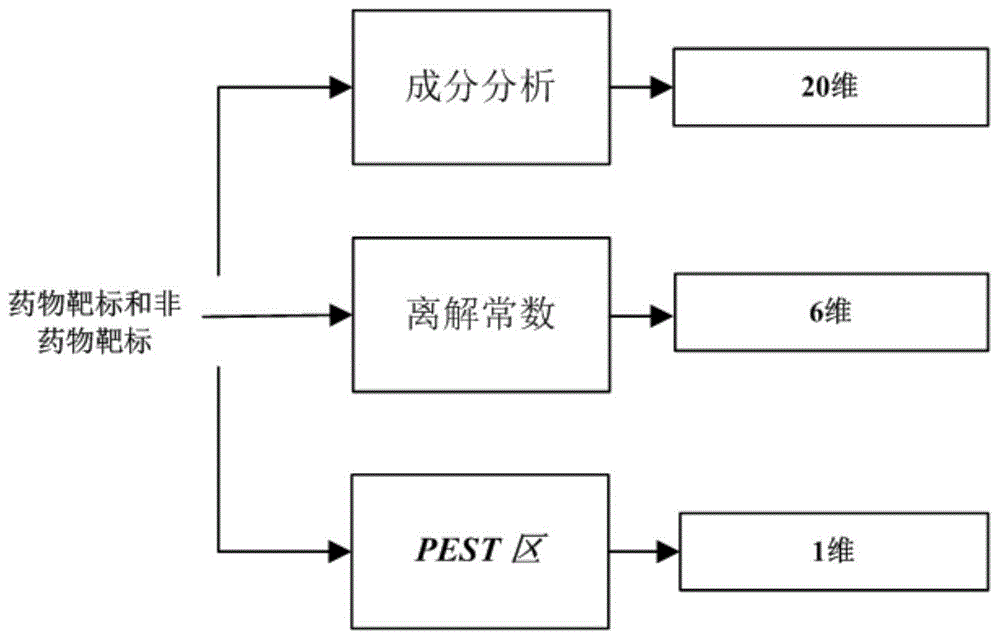
步骤一:成分分析:计算药物靶标和非药物靶标在20种氨基酸中每种氨基酸的平均百分比;
步骤二:离解常数:将20种氨基酸根据其各自的亲水性将氨基酸分成6类小群;
步骤三:pest区:根据epestfind程序识别氨基酸中潜在的pest蛋白区域;
步骤四:根据步骤一、步骤二和步骤三提取出药物靶标的3种特征;
步骤五:利用xgboost算法对步骤四中的提取出的特征进行药物靶点的识别。
本发明基于xgboost的药物靶点识别方法,所述xgboost算法的具体为:
目标函数包括损失函数和正则化项:
obj(θ)=l(θ)+ω(θ)
其中,l(θ)是损失函数,ω(θ)为正则化项;
按照以下公式构建t树的模型为:
xgboost的基本分类器是cart,目标函数可以如下:
目标是获取每个树的参数fi,根据之前的(t-1)树训练了第t树
因此,第t个目标函数为
将损失函数l(θ)进行二阶泰勒展开
将决策树定义为:
ft(x)=wq(x),w∈rm,q:rd→{1,2,…,m};
w记录每个叶节点的分数,q是一个函数,决定每个输入样本最终落在哪个节点上;
在xgboost中,将正则化参数定义为:
λ和γ都是控制模型复杂度的参数;
所以第t个树的目标函数为:
定义gj=∑gi和hj=∑hi,然后可以得到:
这里,wj独立于其他项,第j个节点和最优obj的最优分数为:
最后,按照一定的规则分割树;
本发明基于xgboost的药物靶点识别方法,可以高速、高效、低成本的识别潜在药物靶点;发现潜在的药物靶点不仅可以推动疾病作用机制和药理学研究,还可以为药物潜在的副作用和药品的商业化提供指导信息。
附图说明
图1为本发明的特征提取框图。
图2为药物靶点与非药物靶点的氨基酸组成。
图3为准确率曲线。
具体实施方式
下面将结合附图对本发明做进一步的详细说明:本实施例在以本发明技术方案为前提下进行实施,给出了详细的实施方式,但本发明的保护范围不限于下述实施例。
实施例一:如图1-2所示,本实施例所涉及的基于xgboost的药物靶点识别方法,所述基于xgboost的药物靶点识别方法具体步骤为:
步骤一:成分分析:计算药物靶标和非药物靶标在20种氨基酸中每种氨基酸的平均百分比;
步骤二:离解常数:将20种氨基酸根据其各自的亲水性将氨基酸分成6类小群;
步骤三:pest区:根据epestfind程序识别氨基酸中潜在的pest蛋白区域;
步骤四:根据步骤一、步骤二和步骤三提取出药物靶标的3种特征;
步骤五:利用xgboost算法对步骤四中的提取出的特征进行药物靶点的识别。
成分分析:由于真正的药物靶标的构成与非药物靶标的构成完全不同,因此这些靶标中所有20种氨基酸的出现频率可能大不相同。为了找出药物靶标和非药物靶标之间的差异,绘制平均氨基酸组成的图片,如图1所示。因此,计算药物靶标和非药物靶标中每种氨基酸的平均百分比。
计算了2596种药物靶标和非药物靶标的平均氨基酸组成。正如图2中所看到的,药物目标在'l'中最丰富,并且'g'、'a'、'v'、'e'、's'的组成非常高。
总之,药物靶标的组成与非药物靶标之间存在显着差异。因此,将其用作识别药物目标的功能。
离解常数:疏水性残基和亲水性残基的形态对于确定蛋白质结构非常重要。由于氨基酸的亲水性范围较广,可根据其各自的亲水性将氨基酸分成小群,因此在药物靶标和非药物靶标上必须有很大差异。表1显示了20个氨基酸中的六组。
表1.氨基酸被分为6类
因此,每个药物靶标的序列可以转移到这6组中。每个维度是这六个组之一的平均组成。
pest区:1986年,rechsteinerm和rogerssw做出了假设,即'p'、'e'、's'和't'的氨基酸可以作为蛋白水解信号。现在越来越多的报道证实含有pest区域的序列可以导致蛋白质的快速降解。epestfind程序可用于识别所有不良和潜在的pest蛋白质序列。仅将潜在的pest蛋白区域作为鉴定药物靶标的特征。计算了每个序列中潜在有害生物区域的数量。
因此,我们提取了3种特征,即27维来确定非药物目标的药物目标。
目前合适的药物靶点的数量仍然有限。相对于未知的药物靶点来说,已知的药物靶点只不过是冰山一角。靶点的选择在整个药物研发过程中起着至关重要的作用。现代药物研究中,新靶点的建立往往是新药创新的前提和保障。随着现代分子生物学技术的发展和人类基因组计划的完成,出现了大量可供治疗干预的新型分子靶点,但并不是所有的靶点都能够成为与疾病有关的有效靶点,因此对新型靶点进行发现和验证便成为非常重要的工作。传统使用生物实验的方法不仅成本高昂而且效率低下,本发明开发的xgboost识别药物靶点方法,可以高速、高效、低成本的识别潜在药物靶点。发现潜在的药物靶点不仅可以推动疾病作用机制和药理学研究,还可以为药物潜在的副作用和药品的商业化提供指导信息。
实施例二:如图1所示,本实施例所涉及的基于xgboost的药物靶点识别方法,所述xgboost算法的具体为:
目标函数包括损失函数和正则化项:
obj(θ)=l(θ)+ω(θ)
其中,l(θ)是损失函数,ω(θ)为正则化项;
按照以下公式构建t树的模型为:
xgboost的基本分类器是cart,目标函数可以如下:
目标是获取每个树的参数fi,根据之前的(t-1)树训练了第t树
因此,第t个目标函数为
将损失函数l(θ)进行二阶泰勒展开
将决策树定义为:
ft(x)=wq(x),w∈rm,q:rd→{1,2,…,m};
w记录每个叶节点的分数,q是一个函数,决定每个输入样本最终落在哪个节点上;
在xgboost中,将正则化参数定义为:
λ和γ都是控制模型复杂度的参数;
所以第t个树的目标函数为:
定义gj=∑gi和hj=∑hi,然后可以得到:
这里,wj独立于其他项,第j个节点和最优obj的最优分数为:
最后,按照一定的规则分割树;
extremegradi-entboosting(xgboost)改进了传统的梯度提升决策树(gbdt)。传统的gbdt算法在优化时仅使用损失函数的第一个导数信息。xgboost对损失函数执行二阶泰勒展开,并使用一阶和二阶导数的信息。此外,xgboost可以在openmp的帮助下自动使用cpu。cpu的多核并行计算,大大提高了运行速度。其次,与gbdt算法不同,xgboost支持稀疏矩阵输入。xgboost定义了一个新的数据矩阵dmatrix,训练集将在训练开始时进行预处理,因此可以提高训练过程每次迭代的效率,减少模型训练时间。
gbdt的流程如下:
目标函数通常用于衡量不同模型的质量。它总是由两个部分组成:损失函数和正则化项。
obj(θ)=l(θ)+ω(θ)
l(θ)是损失函数。如果我们只使用损失函数来评估模型的质量,那么模型很容易过度拟合。因此,应考虑正则化参数。它代表了模型的复杂性。因此,最终模型应该在损失函数和正则化项之间取得平衡。
如果训练了t树,可以按照以下方式构建模型:
xgboost和gbdt的基本分类器都是cart,因此目标函数可以如下
目标是获取每个树的参数fi我们根据之前的(t-1)树训练了第t树。
因此,第t个目标函数是
然后,将损失函数进行二阶泰勒展开
然后,我们需要计算正则化项。首先,我们将决策树定义为:
ft(x)=wq(x),w∈rm,q:rd→{1,2,…,m}
w记录每个叶节点的分数。q是一个函数,可以决定每个输入样本最终落在哪个节点上。在xgboost中,我们将正则化参数定义如下:
λ和γ都是控制模型复杂度的参数。所以第t个树的目标函数如下:
我们可以定义gj=∑gi和hj=∑hi,然后我们可以得到:
这里,wj独立于其他项,我们可以得到第j个节点和最优obj的最优分数。
最后,我们应该按照一定的规则分割树。
我们可以看到,如果分裂后的增益小于γ,最好不要添加分支。
实施例三:如3所示,本实施例所涉及的基于xgboost的药物靶点识别方法,所述基于xgboost的药物靶点识别方法的实验验证过程为,我们获得了2596个真正的药物目标,并且我们产生了2596个伪药物目标。为了验证xgboost在鉴定药物靶标方面的有效性,我们进行了十次交叉验证。
我们将这5192个序列随机分成10组。对于每个组,我们选择519个序列作为测试集,其余4673个序列作为训练集。所以,我们总共进行了十次实验。此外,每个序列都成为训练集和测试集。将xgboost的参数设置为表2所述。
表2.xgboost的参数设置
我们使用四种评估方法来评估xgboost在鉴定药物靶标方面的表现。我们将十个实验的结果放在表3中。测试了总共5190个序列。
表3.10次实验的结果
然后可以计算出accuracy=99.13%,precision=99.04%,recall=99.23%,specificity=99.04%;在本研究中,假药物靶标为0,药物靶标为1。10个实验的准确率曲线如图2所示。
以上所述,仅为本发明较佳的具体实施方式,这些具体实施方式都是基于本发明整体构思下的不同实现方式,而且本发明的保护范围并不局限于此,任何熟悉本技术领域的技术人员在本发明揭露的技术范围内,可轻易想到的变化或替换,都应涵盖在本发明的保护范围之内。因此,本发明的保护范围应该以权利要求书的保护范围为准。
- 还没有人留言评论。精彩留言会获得点赞!