环状RNAcirc-Ankib1在制备促进神经再生和修复神经损伤药物中的应用的制作方法
本发明属于神经再生和修复技术领域,具体涉及一种环状rnacirc-ankib1在制备促进神经再生和修复神经损伤药物中的应用。
背景技术:
周围神经损伤通常由拉伸性、缺血性、穿透性、挤压性等创伤性损伤引起。与中枢神经系统不同,周围神经系统损伤后轴突具有一定的内在再生能力。schwann细胞是周围神经系统中重要的胶质细胞,在神经系统中发挥重要作用。周围神经损伤后,schwann细胞增殖迁移形成细胞索带,同时会通过合成分泌神经营养因子,激活免疫反应等为损伤后的神经再生提供适宜的微环境。从分子的角度研究神经再生过程中的关键因素和途径,有利于发现新的治疗靶点。
环状rnacircularrnas(circrnas)是一类不具有5'末端帽子和3'末端poly(a)尾巴、并以共价键形成环形结构的rna.。circrnas能通过作为microrna的分子海绵、剪接和转录调节因子等方式调节基因表达,参与多种生物学过程。
目前关于circrnas的研究主要集中在癌症方面,是一种非常有前景的生物标记物甚至是治疗靶点。在神经系统发育中circrnas也发挥着重要的作用,可以影响神经元的迁移和轴突生长。但到目前为止circrnas在周围神经系统损伤修复中的作用鲜有报道,尤其是对schwann细胞的表型调节。
技术实现要素:
本发明目的在于提供一种新的大鼠源的环状rnacirc-ankib1及其用途,可用于制备促进神经再生和修复神经损伤药物。
本发明具体技术方案如下:
一种环状rnacirc-ankib1,所述circrna的cdna核苷酸序列如seqidno:1所示,首尾连接成环状结构。
本发明另一目的在于提供上述环状rnacirc-ankib1在制备促进神经再生和修复神经损伤药物中的应用,
所述神经损伤为周围神经系统坐骨神经损伤。
可根据circ-ankib1核苷酸序列设计干预circ-ankib1表达的物质,选自化合物、蛋白、多肽、多糖、糖蛋白、糖肽、核酸中的一种或几种。
优选的,所述干预circrna表达的物质,选自与circ-ankib1环化位点处的序列互补的sirna。
本发明另一目的在于提供一种干预上述circ-ankib1表达的sirna,核苷酸序列如下:
circ-ankib1sirna1
正义链:5’-cacgaaugugaaacauguudtdt-3’(seqidno:7)
反义链:5’-aacauguuucacauucgugdtdt-3’(seqidno:8)
circ-ankib1sirna2
正义链:5’-gcucacgaaugugaaacaudtdt-3’(seqidno:9)
反义链:5’-auguuucacauucgugagcdtdt-3’(seqidno:10)。
本发明研究结果表明,所述sirna能够干扰circ-ankib1的表达,促进schwann细胞增殖。
本发明另一目的在于提供上述干预circ-ankib1表达的sirna在制备促进神经再生和修复神经损伤药物中的应用。
本发明构建大鼠坐骨夹伤模型,取损伤不同时间点的坐骨神经组织,通过rna-seq及生物信息学分析,挑选损伤后表达变化明显的circrnas。针对筛选到的circrnas设计特异性的背向引物,再通过pcr验证以及测序鉴定,获得一种新的环状rnacirc-ankib1。在原代schwann细胞中,设计sirna干扰circ-ankib1的表达,可以促进schwann细胞增殖。
附图说明
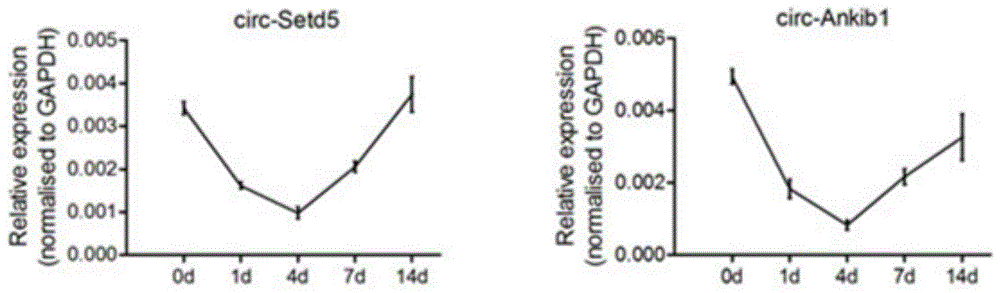
图1为实施例1所述circ-ankib1和circ-setd5在大鼠坐骨神经损伤后坐骨神经组织中不同时间点的表达变化(以gapdh为内参)。
图2为实施例2所述circ-ankib1和circ-setd5的sirna对schwann细胞中circ-ankib1和circ-setd5表达的影响(以gapdh为内参)。
图3为实施例2所述circ-ankib1和circ-setd5的sirna干扰circ-ankib1和circ-setd5对schwann细胞增殖的影响。
具体实施方式
以下通过实施例说明本发明的具体步骤,但不受实施例限制。
在本发明中所使用的术语,除非另有说明,一般具有本领域普通技术人员通常理解的含义。
下面结合具体实施例并参照数据进一步详细描述本发明。应理解,这些实施例只是为了举例说明本发明,而非以任何方式限制本发明的范围。
在以下实施例中,未详细描述的各种过程和方法是本领域中公知的常规方法。
实施例1:circ-ankib1的筛选与鉴定
对sd(spraguedawley)大鼠坐骨神经夹伤后不同时间点(0d、1d、4d、7d、14d)的坐骨神经进行rna-seq。根据测序结果挑选全部由外显子组成且环>500nt的表达差异明显的circrnas。测序结果分析筛选出来的circrna,进一步通过pcr验证circrna的存在。根据circrnas引物设计原则对这些circrnas设计背向引物,每个circrna使用ncbi设计引物。根据引物信息选择不同的退火温度进行pcr扩增,再通过琼脂糖凝胶电泳选出条带大小与预期相符的circrnas,送公司测序,并将测序结果与原始序列比对验证其是否跨越环化位点。最终得到与预期条带大小一致且测序鉴定过环化位点的circrnas,并挑选其中表达量较高的circ-ankib1和circ-setd5进行进一步研究,circ-ankib1的cdna序列如seqidno:1所示,circ-setd5的cdna序列如seqidno:2所示。
一、坐骨神经组织rna提取
取大鼠坐骨神经夹伤后0d、1d、4d、7d、14d近端的组织按
二、rna逆转录合成cdna
使用逆转录试剂盒(takararr037a),将500ng的rna逆转录为cdna。
(1)circrna的逆转
冰上操作,每个反应体系10μl,如下:
(2)mrna的逆转
冰上操作,每个反应体系10μl,如下:
反应程序为:37℃45min,85℃5min,4℃∞。
三、qrt-pcr
根据circrnas引物设计原则设计circrnasqrt-pcr引物序列。
circ-ankib1qrt-pcr引物
circ-ankib1-f5’-agaccgcagacatgctcc-3’(seqidno:3)
circ-ankib1-r5’-agtccctaatatcctattcattcca-3’(seqidno:4)
circ-setd5qrt-pcr引物
circ-setd5-f5’-tactcggcggtcttcc-3’(seqidno:5)
circ-set5-r5’-ctccatctccagctcttt-3’(seqidno:6)
将逆转录反应得到的cdna,按照1∶10稀释,进行如下qrt-pcr反应。
(1)按下列组份配制qrt-pcr反应液:
(2)反应液混匀,real-timepcr仪反应程序如下:
(3)反应设置3个复孔,内参为gapdh,程序结束后,查看溶解曲线与扩增曲线,舍去误差较大的实验数据,根据具体实验要求,对数据进行统计分析。
考察上述circrna在大鼠坐骨神经损伤后坐骨神经组织中不同时间点的表达变化,结果如图1所示。结果表明circ-setd5和circ-ankib1在坐骨神经损伤后表达呈现先下调再上调的状态。
实施例2:circrna的功能验证
一、原代schwann细胞的培养
取出生1d的sd大鼠的坐骨神经,用显微剪剪碎,加入3mg/ml的胶原酶1ml,37℃消化30min,每10min摇晃一次;1200rpm,室温离心5min,弃除胶原酶,加入胰酶1ml,置于37℃培养箱中消化8min左右,加入完全培养基3ml终止消化,过200目筛网,1200rpm,室温离心5min;再加入完全培养基2ml洗1-2遍;将细胞种在用左旋多聚赖氨酸(pll)包被好的培养皿中培养(5%co2,37℃);第二天,换成含有ara-c(10μm)的完全培养基,抑制成纤维细胞的快速增殖;第四天,换成含有hrg(50ng/ml)和forskolin(2μm)的完全培养基刺激细胞的生长。待细胞长到密度为90%以上后,用补体将细胞纯化。先用胰酶将细胞从培养皿中消化下来并转移到离心管中,室温离心1200rpm,5min;弃上清,用含有1μlanti-thy1.1(1∶1000)的1ml完全培养基将细胞沉淀重悬,置冰上孵育2h;1200rpm,室温离心5min,弃掉管中液体,加入rabbitcomplement(250ulrabbitcomplement+750uldmem),37℃,孵育1h;dmem洗三遍;将细胞种在用pll包被好的培养皿中培养;第二天换液培养(含有hrg和forskolin),待细胞长满,纯度达95%以上,用于后续实验。二、schwann细胞转染
转染特异性针对circ-ankib1和circ-setd5的sirna:本实验中,特异性针对circ-ankib1sirna和circ-setd5sirna(由广州市锐博生物科技有限公司制备,终浓度:100nm)及其对照sirnanegativecontrol(nc),用lipofectaminetmrnaimax(invitrogen)转入schwann细胞,第二天换成完全培养基,根据需要进行后续实验。
circ-ankib1sirna和circ-setd5sirna如下所示:
circ-ankib1sirna1:
正义链:5’-cacgaaugugaaacauguudtdt-3’(seqidno:7)
反义链:5’-aacauguuucacauucgugdtdt-3’(seqidno:8)
circ-ankib1sirna2:
正义链:5’-gcucacgaaugugaaacaudtdt-3’(seqidno:9)
反义链:5’-auguuucacauucgugagcdtdt-3’(seqidno:10)。
circ-ankib1sirna3:
正义链:5’-ucgagcucacgaaugugaadtdt-3’(seqidno:11)。
反义链:5’-uucacauucgugagcucgadtdt-3’(seqidno:12)。
circ-setd5sirna1:
正义链:5’-cuucccaugcuggguaauudtdt-3’(seqidno:13)。
反义链:5’-aauuacccagcaugggaagdtdt-3’(seqidno:14)。
circ-setd5sirna2:
正义链:5’-caugcuggguaauuacaaadtdt-3’(seqidno:15)
反义链:5’-uuuguaauuacccagcaugdtdt-3’(seqidno:16)
circ-setd5sirna3:
正义链:5’-uggguaauuacaaaguggadtdt-3’(seqidno:17)。
反义链:5’-uccacuuuguaauuacccadtdt-3’(seqidno:18)。
三、schwann细胞rna提取、逆转录及qrt-pcr
原代schwann细胞分别转染特异性针对circ-ankib1sirna及其对照sirnanegativecontrol(nc),48h后,收细胞,按
四、edu细胞增殖实验
原代schwann细胞分别转染特异性针对circ-ankib1和circ-setd5的sirna(circ-ankib1sirna1、circ-ankib1sirna2、circ-setd5sirna2、circ-setd5sirna3)及其对照(nc),48h后,根据cell-lighttmedu
序列表
<110>南通大学
<120>环状rnacirc-ankib1在制备促进神经再生和修复神经损伤药物中的应用
<160>18
<170>siposequencelisting1.0
<210>1
<211>875
<212>dna
<213>人工序列(artificialsequence)
<400>1
aatgtgaaacatgttgtagaagtactccagaagtggctgaaggaagaaagaaagtgccac60
tgcctatcagaaaataaaacaaaacatgggaaatacaaccaccaaattccgcaaagcact120
catcaatggtgatgaaaacctggcttgccaaatctatgaaaataatcctcagctaaaaga180
atcccttgatccaaatgtatcttatggagagccctatcagcacaacactccattacacta240
tgctgctagacatggaatgaataggatattagggacttttctttttggtagagatggaaa300
cccaaataaacggaatgtgcacaatgaaacatctatgcatttgttgtgtatgggacctca360
aatcatgatatctgaaggaacccttcatcctcgcttagcacggcctgtggaagatgattt420
cagaagggcagattgtctgcagatgatcttacgatggaaaggagcaaaacttgaccaggg480
tgaatatgagagagcggctattgatgcagttgataacaaaaagaacacaccccttcacta540
tgctgcagcctcagggatgaaagcctgtgtggagcttttagtaaaacacggaggagactt600
gtttgctgaaaatgaaaataaagatactccttgtgattgtgcggaaaagcaacaccacaa660
agacctggccctcagtctggagtctcagatggtgttctctcgggacccagaggctgaaga720
aatagaagctgagtatgctgcactggacaagcgagagccatatgaaggactaaggcccca780
ggatcttcgtaggttaaaagatatgcttattgtggagaccgcagacatgctccaggcccc840
tctgtttactgctgaggcactgcttcgagctcacg875
<210>2
<211>589
<212>dna
<213>人工序列(artificialsequence)
<400>2
taattacaaagtggactgtgcttgtcacaagggaaaccggaattgccccatacagaaaag60
aaatcccaatgctgcagaattgccactcccacctcctagctttcccaccattggagcaga120
gaccagacgtagaaaagcacggcggaaagagctggagatggagcagcaaaatgaggttcc180
agaagagaatcatgacccacaaccacaagaagttccagaaaaagtaactgtatccagtga240
gcatgaggaagttgacaatccagaagaaaaaccggaagaagaaaaagaagaggccacaga300
cgaccaggagaactcagctcatagcagaaggactcgggaagataggaaggttgaagccat360
catgcatgcttttgaaactttagagaagagaaagaaacggcgggatcagcctgtagagca420
gagcaactcagacgtagagattactactagcagttcagagataggagttggagaagagac480
aaaaactgcagcccccgagtcagaagttaacaaccctgttacaaacattgccatcccaag540
catcccacagagcactggtgtgaatactcggcggtcttcccatgctggg589
<210>3
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>3
agaccgcagacatgctcc18
<210>4
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>4
agtccctaatatcctattcattcca25
<210>5
<211>16
<212>dna
<213>人工序列(artificialsequence)
<400>5
tactcggcggtcttcc16
<210>6
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>6
ctccatctccagctcttt18
<210>7
<211>21
<212>dna/rna
<213>人工序列(artificialsequence)
<400>7
cacgaaugugaaacauguutt21
<210>8
<211>21
<212>dna/rna
<213>人工序列(artificialsequence)
<400>8
aacauguuucacauucgugtt21
<210>9
<211>21
<212>dna/rna
<213>人工序列(artificialsequence)
<400>9
gcucacgaaugugaaacautt21
<210>10
<211>21
<212>dna/rna
<213>人工序列(artificialsequence)
<400>10
auguuucacauucgugagctt21
<210>11
<211>21
<212>dna/rna
<213>人工序列(artificialsequence)
<400>11
ucgagcucacgaaugugaatt21
<210>12
<211>21
<212>dna/rna
<213>人工序列(artificialsequence)
<400>12
uucacauucgugagcucgatt21
<210>13
<211>21
<212>dna/rna
<213>人工序列(artificialsequence)
<400>13
cuucccaugcuggguaauutt21
<210>14
<211>21
<212>dna/rna
<213>人工序列(artificialsequence)
<400>14
aauuacccagcaugggaagtt21
<210>15
<211>21
<212>dna/rna
<213>人工序列(artificialsequence)
<400>15
caugcuggguaauuacaaatt21
<210>16
<211>21
<212>dna/rna
<213>人工序列(artificialsequence)
<400>16
uuuguaauuacccagcaugtt21
<210>17
<211>21
<212>dna/rna
<213>人工序列(artificialsequence)
<400>17
uggguaauuacaaaguggatt21
<210>18
<211>21
<212>dna/rna
<213>人工序列(artificialsequence)
<400>18
uccacuuuguaauuacccatt21
- 还没有人留言评论。精彩留言会获得点赞!