基于深度学习和基因表达数据的化合物肝毒性早期预测方法与流程
本发明涉及计算机辅助药物筛选领域,具体地说是涉及一种基于深度学习和基因表达数据的化合物肝毒性的早期预测方法,适用于根据基因表达数据对化合物肝毒性进行早期预测。
背景技术:
药物肝毒性是导致新药研究失败和临床药物撤市的一个重要因素。据统计在新药研发过程中因候选药物肝毒性而导致失败的比例为37%,在临床应用中因药物肝毒性而导致药物撤市的比例为18%,因此,在药物研发早期以及临床使用中对药物肝毒性进行预测对于提高研发成功率和合理用药具有重要意义。由于药物肝毒性发生机制复杂,如何提高药物肝毒性预测的准确性以及适用性,特别是对迟发性药物肝毒性的预测依然面临重大挑战。
传统的药物肝毒性预测方法主要是基于体内外实验模型对药物的毒性安全进行评估,该方法需要大量的活体动物进行实验基础,具有实验周期长、耗费高等缺点。随着安全、环保、动物保护等方面的法规要求日益严格,全球化的市场竞争也要求药物开发周期大幅缩减,因此如何克服传统药物肝毒性预测方法所存在的缺点,开发高效的药物肝毒性评估方法具有重要意义。随着信息技术的发展,国内外研究开始尝试利用机器学习与化合物结构特征建立计算模型来进行药物肝毒性预测,但其往往面临以下问题:1)易受单一化合物结构的限制,对于结构多样性的化合物的肝毒性预测往往准确率较低;2)预测结果缺乏生物学意义,无法从生物作用机制系统地解释预测结果;3)无法对迟发性的药物肝毒性进行早期预测;4)传统的机器学习方法无法从大数据中自动学习特征信息,需要大量的人工特征挑选。因此,针对以上方法的局限性,本发明提供了一种基于深度学习和基因表达数据的化合物肝毒性预测方法,能够通过基因水平系统地对化合物迟发性肝毒性进行早期预测。
技术实现要素:
本发明克服现有技术存在的不足,公开了一种基于深度学习算法(deeplearningalgorithm,dl)和基因表达数据的化合物肝毒性的早期预测方法,本方法将药物基因组学与人工智能深度学习算法充分结合,克服了传统化合物肝毒性预测方法的局限性,实现了通过基因水平系统地对化合物迟发性肝毒性进行早期预测,从而为新药研发过程中的毒性安全评估及临床合理用药提供一种高效、准确、快速的化合物肝毒性预测方法。
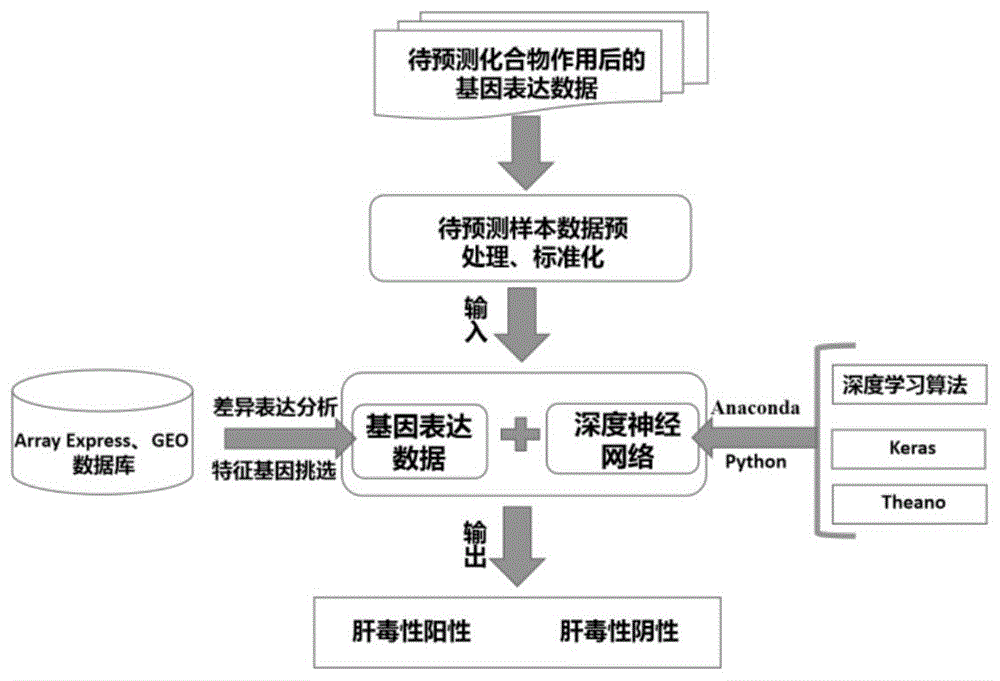
本发明的目的可以通过以下技术路线(图1)来实现:
1.一种基于深度学习和基因表达数据的化合物肝毒性早期预测方法,其特征在于,包括如下步骤:
步骤一:通过从公共生物医学数据库中对化合物作用后的基因表达数据进行挖掘,将得到的原始数据进行数据清洗与标准化,构建建模数据样本;
步骤二:通过基因差异表达分析与特征权重计算筛选肝毒性特征基因,作为最终模型样本特征;
步骤三:基于深度学习算法构建预测模型,将筛选得到的肝毒性特征基因的表达数据用于模型的训练与学习;
步骤四:通过网格搜索算法与交叉验证对模型的关键参数进行优化,提高模型的预测性能。
2.所述步骤一包括如下步骤:
1.1通过从公共生物医学数据库中收集来自同一基因芯片测定的化合物作用后的基因表达数据;
1.2基因表达数据样本根据肝毒性病变级别按5分法分为severe(严重,病变范围为[75%,100%]),moderatelysevere(中度严重,病变范围为[50%,75%)),moderate(中度,病变范围为[25%,50%)),slight(轻度,病变范围为[1%,25%)),minimal(轻微,病变范围为[0%,1%));
1.3将出现任意一次毒性级别为severe、moderatelysevere、moderate和slight毒性的化合物标记为阳性化合物,其所有时间点、所有剂量下的用药组样本均为肝毒性阳性样本;毒性病变级别minimal的化合物的基因表达数据样本和所有对照组样本作为肝毒性阴性样本。
1.4基于r语言及bioconductorr包,对收集的肝毒性阳性样本和阴性样本的基因表达数据进行预处理。其中,通过impute包对基因表达的缺失值及无效值进行填充,通过limma包对基因表达数据进行标准化;
1.5按照基因芯片的注释文件,将标准化的基因表达数据的探针id与相应的genesymbol进行逐一匹配;
1.6将所有建模样本数据按照80%:20%随机分为训练集和测试集,训练集用于模型的训练学习,测试集用于模型的性能评估。
步骤1.1中,所述公共生物医学数据库包括arrayexpress、geneexpression和omnibus(geo)。
所述步骤二包括如下步骤:
2.1基于贝叶斯算法的limmar包对预处理后的基因表达数据进行差异表达分析,选取其中差异表达倍数的绝对值大于或等于2并且adjust-p值小于或等于0.05的基因作为特征基因;
2.2进一步通过深度学习算法对特征基因的权重进行计算并保留特征权重值大于0.1的基因作为最终模型构建的特征基因,即肝毒性特征基因。
所述步骤三包括如下步骤:
3.1模型选取序贯(sequential)模型接口,模型结构包括输入层(inputlayer)、隐藏层(hiddenlayer)以及输出层(outputlayer),其中隐藏层包括全连接层(denselayer)和dropout层(dropoutlayer);
3.2模型的输入为基因表达数据,其中每个特征基因都作为输入层的一个节点;
3.3模型的输出为二分类结果0和1,其中0代表肝毒性阴性,1代表肝毒性阳性;
3.4模型的隐藏层中,其通过rectifiedlinearunit(relu)激活函数来激活输入层的值进而传入全连接层,该激活函数的公式为:
y=relu(wx+b)
其中,x为输入数据的值,y为数据激活后的值,w为权重矩阵,b为偏差;
3.5模型的输出层中,其通过sigmoid激活函数来激活隐藏层的值进而传出为最终的输出结果,该激活函数的公式为:
z=sigmoid(w′y+b′)
其中,y为隐藏层传出的激活后的值,z为模型输出结果,w′为转置权重矩阵,b′为转置偏差;
3.6模型的训练过程中,采用compile模块对模型的学习过程进行配置,其参数分别设置为:优化器(optimizer)设为rootmeansquareprop(rmsprop),指标列表(metrics)设为accuracy以及损失函数(lossfunction)设为binary_crossentropy,其中该损失函数的计算公式为:
其中,lh(x,z)为预测值与真实值的差异大小(即损失),x为样本对应的真实值,z为样本对应的预测值,d为epoch数。
3.7将步骤二处理后最终得到的肝毒性特征基因表达数据作为模型的输入,其中80%作为训练集用来训练模型,20%作为测试集用来测试模型性能;
所述步骤四包括如下步骤:
4.1设置参数寻优范围,其中epochnumber为[10,50,100,200,500],batchsize为[10,32,64,128],learningrate为[0.01,0.001,0.00001],dropoutrate为[0,0.2,0.5],nodenumber为[50,100,300,500,1000];
4.2通过网格搜索算法对构建的900(5×4×3×3×5)个模型进行寻优;
4.3通过10折交叉验证模式及评价指标对模型的预测性能进行评价,其中性能评价指标包括:敏感度(sensitivity,sen);特异性(specificity,spe);准确性(accuracy,acc);马修斯相关系数(matthewscorrelationcoefficient,mcc);roc曲线下面积(theareaunderthereceiveroperatingcharacteristic(roc)curve,auc)。其中,敏感度、特异性和准确性越接近于100%,马修斯相关系数及roc曲线下面积越接近于1,表明该模型预测性能越好;相反,敏感度、特异性和准确性越接近于0,马修斯相关系数及roc曲线下面积越接近于0.5,表明该模型预测性能越差。
其中,tp代表真阳性;tn代表真阴性;fp代表假阳性;fn代表假阴性;
与现有技术相比,采用本发明的优点如下:
1.能够通过基因水平系统、准确地对迟发性药物肝毒性进行早期预测;
2.相比于传统基于化合物结构特征的预测方法,本预测方法可以准确预测不同结构化合物的肝毒性,具有较好的鲁棒性;
3.本方法采用的深度学习算法具有较强的自动学习特征能力,能够从大数据中自动学习重要特征信息,避免了大量人为特征挑选;
4.本方法构建的化合物肝毒性预测模型具有较优的预测性能,其预测准确性相比于传统的预测方法有了大幅度提升;
5.该方法的适用性较好,由于其具有早期预测的特点,能够为新药研发的临床前毒性安全评价和临床合理用药提供理论依据和技术支撑。
附图说明:
图1是本发明基于深度学习和基因表达数据的化合物肝毒性早期预测方法的总流程图;
图2是本发明方法化合物肝毒性预测模型的结构示意图;
图3是基于深度学习和基因表达数据的化合物肝毒性早期预测方法的预测结果图;
图4是基于深度学习和基因表达数据的化合物肝毒性早期预测方法的预测性能图;
具体实施方式:
为了使本发明的目的、技术方案及优点更加清楚明白,下面结合附图对本发明的技术方案再作进一步的说明。
一种基于深度学习和基因表达数据的化合物肝毒性早期预测方法的具体技术方案是:
1.通过从arrayexpress数据库中收集来自affymetrixgenechipratgenome2302.0芯片测定的87个化合物作用下的基因表达数据。所搜集的基因表达数据样本根据毒性病变级别按5分法分为(严重,病变范围为[75%,100%]),moderatelysevere(中度严重,病变范围为[50%,75%)),moderate(中度,病变范围为[25%,50%)),slight(轻度,病变范围为[1%,25%)),minimal(轻微,病变范围为[0%,1%))。为使所构建的模型能早期预测迟发性毒性,并具有较高的适用性,本研究将出现任意一次毒性级别为severe、moderatelysevere、moderate和slight毒性的化合物标记为阳性化合物,其所有时间点、所有剂量下的用药组样本均为阳性样本;毒性病变级别minimal的化合物的基因表达数据样本和所有对照组样本作为毒性阴性样本。通过整理,最终获得988个基因样本数据,其中阳性样本496个,阴性样本492个。将所有建模样本数据按照80%:20%随机分为训练集和测试集,790个样本数据作为训练集用于模型的训练学习,198个样本数据作为测试集用于模型的性能评估。
2.基于r语言及bioconductorr包对原始基因表达数据进行一系列统计学数据清洗,构建最终基因表达谱作为建模数据。其中,首先通过impute包对基因表达的缺失值及无效值进行填充,通过limma包对基因表达数据进行标准化,进一步按照基因芯片的注释文件,将探针id与genesymbol进行匹配,最后通过deseq2包对基因表达数据进行差异表达分析,构建最终的特征基因表达矩阵。其中,首先通过基于贝叶斯算法的limmar包对基因表达数据进行差异表达分析,选取其中差异表达倍数的绝对值大于或等于2,adjust-p值小于或等于0.05的基因作为初步筛选得到的特征基因,进一步通过深度学习算法对初步筛选得到的特征基因进行相应的权重计算并保留特征权重值大于0.1的基因,最终筛选得到1574个特征基因用于最终模型的构建。
3.本方案以anaconda5.1的python3.6为平台,借助基于python和theano的深度学习框架keras搭建深度学习预测模型。该模型选取序贯(sequential)模型接口,搭建二分类预测模型。模型结构包括输入层(inputlayer)、隐藏层(hiddenlayer)以及输出层(outputlayer),其中隐藏层包括全连接层(denselayer)和dropout层(dropoutlayer)(图2)。模型的隐藏层中,其通过rectifiedlinearunit(relu)激活函数来激活输入层的值进而传入全连接层,该激活函数的公式为:
y=relu(wx+b)
其中,x为输入数据的值,y为数据激活后的值,w为权重矩阵,b为偏差;
模型的输出层中,其通过sigmoid激活函数来激活隐藏层的值进而传出为最终的输出结果,该激活函数的公式为:
z=sigmoid(w′y+b′)
其中,y为隐藏层传出的激活后的值,z为模型输出结果,w′为转置权重矩阵,b′为转置偏差;
模型的训练过程中,采用compile模块对模型的学习过程进行配置,其参数分别设置为:优化器(optimizer)设为rootmeansquareprop(rmsprop),指标列表(metrics)设为accuracy以及损失函数(lossfunction)设为binary_crossentropy,其中该损失函数的计算公式为:
其中,lh(x,z)为预测值与真实值的差异大小(即损失),x为样本对应的真实值,z为样本对应的预测值,d为epoch数;
然后将处理得到的特征基因表达数据作为模型的输入,其中80%作为训练集用来训练模型,20%作为测试集用来测试模型性能。
4.通过使用建模数据中的测试集样本对训练好的模型的预测性能进行评估,其中分别采用敏感度(sensitivity,sen);特异性(specificity,spe);准确性(accuracy,acc);马修斯相关系数(matthewscorrelationcoefficient,mcc);roc曲线下面积(theareaunderthereceiveroperatingcharacteristiccurve,auc)等相关评价指标进行性能评判。进一步,通过使用网格搜索算法与十折交叉验证对构建的900(5×4×3×3×5)个模型的关键参数(epochnumber、batchsize、learningrate、dropoutrate和nodenumber)进行优化,从而使模型获得最佳预测性能。
其中,tp代表真阳性;tn代表真阴性;fp代表假阳性;fn代表假阴性;
最终,通过参数寻优,最优模型的具体参数设定为隐藏层为2层且每层为50个节点,dropoutrate设定为0.5来避免模型的过拟合,learningrate为0.001,batchsize为128,epochnumber为50。通过测试集对最优模型的性能考察,其预测准确率为97.1%,auc为0.989,敏感性为97.4%,特异性为96.8%,马修斯相关系数为0.942,相比于国内外大多数基于传统机器学习的化合物肝毒性预测模型,该模型具有较优的预测性能(图3、图4)。
上述实例仅仅是本发明的一个具体实施方式,对其的简单变换、替换等也均在发明的保护范围内。
- 还没有人留言评论。精彩留言会获得点赞!