基于人工智能与图形学结合的染色体图像处理方法与流程
本发明涉及染色体处理技术,具体涉及一种基于人工智能与图形学结合的染色体图像处理方法。
背景技术:
染色体图像几乎是确保胎儿基因是否正常的唯一手段,可以在孕期中及时发现胎儿的各种基因问题和后天疾病隐患。在基因学相关的研究背景下,这种能够从基因角度分析胎儿情况的手段具有很强的指导意义,而其图像的制作难度也可想而知。据了解,在纯手工进行染色体图像分析的时期,每一幅图像(即每一个样本)大概需要40分钟以上的处理时间,而每一个胎儿的最终结果则需要从几十个样本中统计出正常和异常的比率(根据正常样本的数量,更改需要额外统计的样本数),其效率之低可见一斑。
目前,染色体图像的成像过程转为仪器操作,人工对胎儿细胞制片后放至仪器下进行成像即可,这一进步减少了人工对每个样本观察显微镜的过程,且在计算机上能够实现基础的图像处理辅助功能,使得工作效率大大提升。但在如今的人口压力下,这样的速度也已经越来越难以满足医疗的需求,因为成像后的图片仍需人工进行后续处理,人力成本很高。
在当前人工智能蓬勃发展的大背景下,如果能够将人工智能应用于医学领域的染色体图像处理上,尽量用机器运算来代替人工过程,则可以极大地提升工作效率。
深度学习以其在人工智能上的广泛应用在近年来备受各界的关注,可见采用深度学习的方法让计算机解决一些智能化的问题是人工智能发展的一个主流方向。深度学习可以结合的领域多种多样,在计算机图形学上也有很不错的应用前景,因此本发明将使用深度学习作为处理计算机图形学问题的主要工具,相信二者的结合对相关领域的研究有着不小的推动作用。
对染色体图像处理过程中的难点问题是染色体图像的分割和识别工作,目前国内外虽然对此问题做了不少的研究,但究其实用性,还存在着几点不足之处:
①如何改进现有的算法体系,以达到人工成本降低的目的:目前无论是基于传统图形学还是基于深度学习的算法架构,都需要大量的人力进行参与。这是由于目前的研究工作都是在单一的条件下进行的,或是针对单一问题,或是面向单一的数据集。这样的研究条件决定了短时间内无法用于实装的处理系统;
②如何解决分割处理中的团簇或掩盖的情况:目前的传统算法研究上,针对分割的文献很少,足见其难度。在深度学习上,u-net的方法值得借用,但明确针对染色体图像的成果同样很少。至于团簇和掩盖的情况,定然需要研究一些相应的处理手段;
③如何设计染色体图像自动处理后的评判标准:目前市面上对于人工智能处理的需求是“全自动”,全过程的自动处理定然会遇到很多特殊的情况,需要少量的人工辅助,那么怎样才可以核定某一幅图像是需要进行人工辅助的,也需要更具创新性的理论进行定义。
技术实现要素:
本发明所要解决的技术问题是:提出一种基于人工智能与图形学结合的染色体图像处理方法,快速、准确地对染色体图像的分割和分类识别进行处理,提高工作效率,降低人力成本,并保证处理的质量。
本发明解决上述技术问题采用的技术方案是:
基于人工智能与图形学结合的染色体图像处理方法,包括以下步骤:
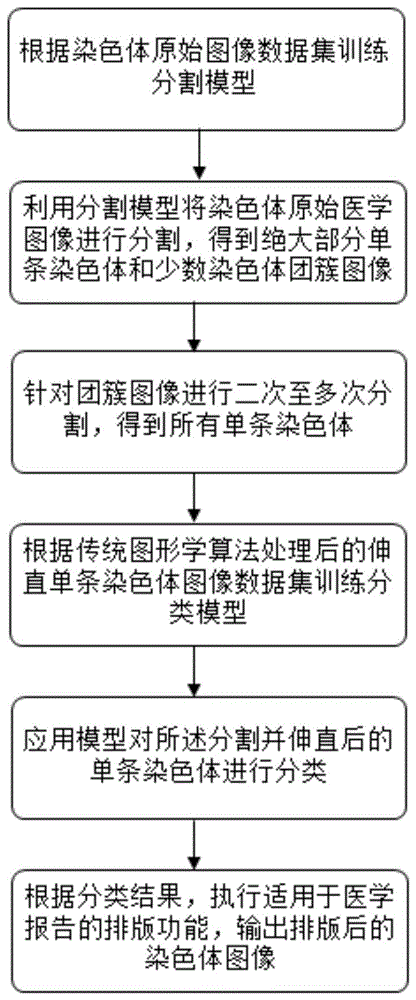
a.根据染色体原始图像数据集训练分割模型;
b.在进行染色体图像处理时,利用分割模型将待处理的染色体原始医学图像进行分割;
c.针对步骤b中未完全分割的染色体团簇图像进行二次至多次分割,得到单条染色体;
d.根据图形学算法处理后的伸直单条染色体图像数据集训练分类模型;
e.应用分类模型对经过分割并伸直后的单条染色体进行分类;
f.根据分类结果,执行适用于医学报告的排版功能,输出排版后的染色体图像。
作为进一步优化,步骤a中,在训练分割模型之前,首先进行染色体原始图像数据集的制作,具体包括:通过数据标注工具将获取到的染色体原始图像进行标注,为图像上每一个可见的染色体标注一个包围框,进而生成二值化的掩膜,并输出成卷积网络便于处理的json格式数据,随后对每一幅图像包括其掩膜图像进行旋转、位移、缩放变换。
作为进一步优化,所述获取的染色体原始图像中染色体的数量无限制。
作为进一步优化,步骤a中,所述分割模型采用基于深度学习的u-net模型框架,利用制作的染色体原始图像数据集划分训练集和验证集,采用训练集对模型进行训练,采用验证集对训练的模型进行验证测试,最终获得满足要求的分割模型。
作为进一步优化,步骤b中,在利用分割模型将待处理的染色体原始医学图像进行分割后,获得分割的染色体图像的二值化掩膜,根据所述二值化掩膜在待处理的染色体原始医学图像上提取分割出来的染色体图像;所述分割出来的染色体图像中包括绝大部分的单条染色体和少部分的未完全分割的染色体团簇图像。
作为进一步优化,步骤c中,针对步骤b中未完全分割的染色体团簇图像若在一定次数分割仍未成功时,调用人工辅助接口给出待人工辅助处理的提示,由操作者通过勾选条件或者直接划线、描点操作辅助程序完成分割任务。
作为进一步优化,在调用人工辅助接口之前先对未完全分割的染色体团簇图像进行灰度均衡化的预处理。
作为进一步优化,步骤d中,所述图形学算法处理包括:
d1.预处理过程:对单条染色体的图像进行灰度均衡化处理;
d2.中线提取及首尾切割:在中轴片段的获取上,采用德劳内三角形方法取出染色体的中轴,用于像素关联处理;
d3.方向匹配及像素关联:对于中轴上的每一像素点,取其关联中轴曲线的法线,沿着法线通过的每一个有效像素点均为与该中轴像素点所关联的像素;在方向匹配上,通过对中轴像素3*3邻域的像素分布,确定出适应16个不同方向的近似法线;此外,针对中轴弯曲处的像素点进行二次补点操作,即在该点的弯曲趋势上,前后各选取一个点,按照原斜率关联像素后按顺序添加进待伸直队列当中;
d4.角度校正及首尾贴合:首先需要衡量待伸直队列的平均长度,以此判定某队列是否需要进行校正处理;如果需要校正,针对该队列找回原图像中的中轴点位置,并抛弃其当前角度信息,用定义的16种角度对像素进行重新关联,直到找到一条在长度上最为合适的队列;如果新队列的角度信息需要补点,则进行补点处理,然后把所有新产生的队列统一替换到伸直图像中;
角度校正过程完成后,需要进行首尾切割部分的整合,使用边界膨胀法进行处理:从切割线开始,为每一个有效像素点提供一个十字型1像素的膨胀模板,膨胀后将获得的切割部分像素贴合到伸直好的中轴关联部分的首尾端,下一轮则使用新获得的像素作为膨胀的起点,如此循环直至不再获得新的像素点;
经过步骤d1-d4的处理后获得伸直后的单条染色体图像。
作为进一步优化,步骤d中,所述分类模型采用单输入、24类输出的卷积网络结构,对伸直单条染色体图像数据集中的每一幅单条染色体图像进行学习。
作为进一步优化,步骤f中,所述输出的排版后的染色体图像为从染色体原始医学图像中分割出来的未经伸直处理的单条染色体图像。
本发明的有益效果是:
(1)本发明对图形学算法做了自动化的改进,使得阈值化处理、灰度均衡化处理等预处理过程可以完全脱离人工操作。避免了因个人特性造成的误差,会给图像带来满足人眼视觉的提升。同时,本发明也支持人工进行再次调整,以满足人工纠错和特异性需求,并且不会影响其最终处理的质量。
(2)本发明引入了对图像分割分类都较为有效的人工智能算法——深度学习,可以完全顾及到染色体的条带特征以及像素信息,准确率高达95%以上,经测试,即使没有处理过的原始染色体图像,在人眼完全无法分辨的时候,网络得出的结果依然十分准确;
(3)本发明首次采用非端到端的深度学习网络模型进行图像处理,即在分割和分类工作之间,加入图形学算法染色体伸直的操作,旨在增加分类网络的准确性。该操作排除了染色体形态对分类工作产生的影响,经验证其可以起到良好的效果。
(4)本发明整体的自动化程度很高,无需过多的人工参与,输入原始图像即可产生最终结果,因此,极大地提高了工作效率,降低人工成本。
附图说明
图1为本发明中的基于人工智能与图形学结合的染色体图像处理方法流程图。
具体实施方式
本发明旨在提出一种基于人工智能与图形学结合的染色体图像处理方法,快速、准确地对染色体图像的分割和分类识别进行处理,提高工作效率,降低人力成本,并保证处理质量。
针对国内外目前对染色体图像的分割和识别工作的研究成果存在的不足之处,本发明通过相应手段进行了逐一解决:
①针对如何改进算法体系,降低人工成本的问题:本发明采用了临床的原始图像对深度学习模型进行训练,或是将原始图像统一经过图形学方法进行预处理后,再送入网络中学习。这样的操作使训练得到的模型具有对大多数染色体图像的适用性,这是由于原始图像的质量千差万别,并非精心挑选的具有某一特征的训练集。此外,大量人力问题的解决主要是体现在数据标注上,本方案采取的方法是对原始染色体图像进行变换,通过旋转、位移、缩放等操作实现一图多变,极大程度上扩充了数据的容量,并且我们借鉴了在其他领域仅需要少量数据便可达到高准确度的网络结构,更可以在保证质量的前提下减少人力成本。
②针对如何解决分割处理中的团簇或掩盖的问题:由于团簇和掩盖现象在实践中是十分常见的,这与制片操作员的水平有很大关系,但有少量该现象并不会影响医生的诊断,实际的操作中也是将重叠部分直接套选取用。因此本方案要对这种现象进行单独处理,如果一次分割后的结果出现了无法识别的部分,则对这一部分进行二次甚至多次分割,确保输出结果为单条染色体,同时利用u-net网络的分割特性,以满足重叠区域的套选。
③针对如何设计染色体图像自动处理后的评判标准的问题:在图像分割时,如果存在始终无法成功分离的团簇或掩盖现象,则一定需要人工进行辅助。而在最后的分类任务中,本方案利用了深度学习输出的置信度,如果在所设置的阈值范围之内,则无需进行人工干预,而阈值是系统中可以进行调整的,并且系统可以支持对每一幅图像的再处理(自动或人工)。此外,系统会根据处理过程中的情况,输出具有指示性的log信息(如检测时少了一条染色体等)供操作者随时查看,以决定是否需要人工干预。
在具体实现上,如图1所示,本发明中的基于人工智能与图形学结合的染色体图像处理方法,包括以下实现步骤:
1.根据染色体原始图像数据集训练分割模型;
本步骤中,训练分割模型之前,还需要制作染色体原始图像数据集:通过数据标注工具(如labelme)将获取到的染色体原始图像进行标注,无需限制一张图中染色体的数量,保证样本的随机性,为图像上每一个可见的染色体标注一个包围框,进而生成二值化的掩膜,并输出成网络便于处理的json格式数据。随后对每一幅图像包括其掩膜图像进行旋转、位移、缩放等变换,可以成指数倍地增加其数据量。
所述训练分割模型,主要采用基于深度学习的u-net模型框架,扫描染色体图像,通过全卷积的网络结构,分析图像的每一个像素,较浅的层次解决像素定位问题,较深的层次则是用于像素的分类(是否属于掩膜部分)。最终网络会输出图像的分割情况,并由此生成所需的掩膜。
2.在进行染色体图像处理时,利用分割模型将待处理的染色体原始医学图像进行分割;
本步骤中,在利用分割模型将待处理的染色体原始医学图像进行分割后,获得分割的染色体图像的二值化掩膜,根据所述二值化掩膜在待处理的染色体原始医学图像上提取分割出来的染色体图像;所述分割出来的染色体图像中包括绝大部分的单条染色体和少部分的未完全分割的染色体团簇图像。
3.针对步骤2中未完全分割的染色体团簇图像进行二次至多次分割,得到单条染色体;
本步骤中,针对少部分的未完全分割的染色体团簇图像,可以通过二次分割或者三次分割获得单条染色体图像,而如果分割次数达到预定的阈值了,则需要人工干预,则此时可以通过调用人工辅助接口给出待人工辅助处理的提示,由操作者通过勾选条件或者直接划线、描点操作辅助程序完成分割任务。但是为了便于人眼观察,在调用人工辅助接口之前先对未完全分割的染色体团簇图像进行灰度均衡化的预处理。
4.根据图形学算法处理后的伸直单条染色体图像数据集训练分类模型;
本步骤中,所述图形学算法处理主要包括:
(1)预处理过程:对单条染色体的图像进行灰度均衡化处理,以突出其像素的特性及像素间的关联,便于在步骤5中进行分类处理,同时也满足最终结果输出的可视化;
(2)中线提取及首尾切割:在中轴片段的获取上,采用德劳内三角形方法取出染色体的中轴,用于像素关联处理。由于获得的中轴片段并不能关联到染色体的所有像素点,又希望避免对中轴进行扩展处理,因此对匹配到中轴长度外的染色体部分进行切割,即先不对这些部分进行处理,在最后将中轴部分伸直完成后再将这些部分进行贴合。
(3)方向匹配及像素关联:对于中轴上的每一像素点,取到其关于中轴曲线的法线,而沿着法线通过的每一个有效像素点均为与该中轴像素点所关联的像素。关于方向的匹配,则是借鉴了计算机图形学中屏幕画线的理论,设计了类似的逆过程,通过对中轴像素3*3邻域的像素分布,确定出适应16个不同方向的近似法线。此外,还需要考虑因曲线外部弯曲处像素密度小于中轴上像素密度而造成的像素丢失。由于图像中像素和线的关系,可以确定只有中轴弯曲处某一点的法线斜率为1或-1时,会造成比较大的像素丢失情况。因此针对这样的中轴像素点需要进行二次补点操作,即在该点的弯曲趋势上,前后各选取一个点,按照原斜率关联像素后按顺序添加进待伸直队列当中,可以保证丢失的像素更少。
(4)角度校正及首尾贴合:首先需要衡量待伸直队列的平均长度,并以此作为对于某一个队列来说是否需要进行校正的判定。如果需要处理,针对该队列找回原图像中的中轴点位置,并抛弃其当前角度信息,用前面定义的16种角度对像素进行重新关联,直到找到一条在长度上最为合适的队列(如果所有的队列都不能满足条件,则将该点作为无效中轴点放弃)。如果新队列的角度信息需要补点,则进行补点处理,然后把所有新产生的队列统一替换到伸直图像中。角度校正过程完成后,就需要进行首尾切割部分的整合,使用边界膨胀法进行处理。从切割线开始,为每一个有效像素点提供一个十字型1像素的膨胀模板,膨胀后将获得的切割部分像素贴合到伸直好的中轴关联部分的首尾端,下一轮则使用新获得的像素作为膨胀的起点,如此循环直至不再获得新的像素点。
经过以上图形学算法的应用,便可以获得伸直后的单条染色体图像。当然原始的单条图像依然需要保留,这是作为最后输出报告的可视图像。
在训练分类模型时,设计单输入—24类输出的网络结构,对数据集中每一幅单条染色体图像进行学习。而数据集的制作,则是前序过程的中间结果,直接应用即可。
5.应用分类模型对经过分割并伸直后的单条染色体进行分类;
本步骤中,在对分割出来的单条染色体进行伸直处理后就可以应用训练好的分类模型进行分类。
6.根据分类结果,执行适用于医学报告的排版功能,输出排版后的染色体图像。
本步骤中,所述输出的排版后的染色体图像为从染色体原始医学图像中分割出来的未经伸直处理的单条染色体图像。
- 还没有人留言评论。精彩留言会获得点赞!