密码子优化的制作方法
以ascii文本文件提交序列表
下面提交的ascii文本文件的内容以引用方式整体并入本文:序列表的计算机可读形式(crf)(文件名:759892000440seqlist.txt,记录日期:2018年7月25日,大小:4kb)。
发明领域
本公开总体上涉及优化技术,且更具体地涉及用于优化在宿主中表达蛋白质的序列(例如,核酸序列)的系统和方法。
背景技术:
密码子简并性是指遗传密码的冗余性,其以氨基酸可以由不同的同义密码子指定的现象表现出来。得注意的是,发现这些同义密码子在大多数经测序的基因组中以不相等的频率被使用。这种现象被称为密码子使用偏好性。
因为生物医学和生物技术研究及工业生产需要具有正确折叠和修饰的高质量蛋白质,所以如何探索和总结反映高度表达的基因的密码子使用偏好性的潜在有益规则和模式对于提高蛋白质的表达水平至关重要。然而,蛋白质表达是多步骤的过程,其涉及在转录、mrna周转、翻译和翻译后修饰的水平上的调控,从而使得能够形成稳定的产物。即使是单个同义密码子替换也可以使转基因的表达增加超过1,000倍。因此,密码子优化为合成基因在重组宿主中的最佳表达奠定了基础。
技术实现要素:
本文提供了使用多目标优化算法考虑和平衡多种因素的增强密码子优化的系统和方法。根据一些实施方案,密码子优化尤其基于三个目标:(i)首先如何分配某些氨基酸的同义密码子的计数,(ii)如何将同义密码子置于其最合适的位置,以及(iii)如何减少不利但意外产生的子序列和/或基序。在一些实施方案中,这三个目标被量化为协调指数、密码子背景指数和离群指数。在优化过程中,使用多目标算法如非支配排序遗传算法iii(nsga-iii)或其变型来考虑目标。具体而言,对于给定的候选核酸序列,可以参考高度表达的基因的已知特征来计算目标。在一些实施方案中,在基因合成和蛋白质表达之前,从一个或多个优化的序列中去除各种已知的不利基序和/或特征(例如,从文献中鉴定的)。
因此,本发明提供了一种系统方法,由此优选影响蛋白质表达的所有或大部分参数和因素(包括但不限于密码子协调性、密码子使用(例如同义密码子分布)、密码子背景指数、顺式作用mrna去稳定基序、rna酶剪接位点、gc含量、核糖体结合位点(rbs)、基因的mrna二级结构(例如mrna自由能)和重复元件)都被考虑到,以改进和优化核酸序列,从而促进基因在表达系统,诸如在包括真核细胞和原核细胞(如哺乳动物、昆虫、酵母、细菌、藻类)在内的表达宿主细胞中,以及在无细胞表达系统中的蛋白质表达。
在一些实施方案中,提供了用于优化在宿主中表达蛋白质的核酸序列的计算机实现方法,所述计算机实现方法包括:a)接收初始群体集,其中初始群体集包含能够表达蛋白质的多个初始候选核酸序列;和b)基于初始群体集,使用计算机辅助的nsga-iii算法或其变型进行协调指数、密码子背景指数和离群指数的优化,从而获得能够表达蛋白质的多个优化的核酸序列,其中候选核酸序列的协调指数指示多个高度表达的基因和候选核酸序列之间的同义密码子的使用频率分布的一致性,其中候选核酸序列的密码子背景指数是用于将同义密码子置于合适位置的量度,并且其中候选核酸序列的离群指数是多个预定序列特征对候选核酸序列的负面影响的量度。
在一些实施方案中,该方法还包括提供指示多个优化的核酸序列中的至少一个优化的核酸序列的输出。
在一些实施方案中,接收初始群体集包括:接收蛋白质序列;基于接收的蛋白质序列产生初始群体集。
在一些实施方案中,接收初始群体集包括:接收核酸序列;将接收的核酸序列翻译成蛋白质序列;基于蛋白质序列产生初始群体集。
在一些实施方案中,初始群体集具有预定大小。
在一些实施方案中,初始群体集包括多个初始候选核酸序列的二进制表示。
一些实施方案中,执行协调指数、密码子背景指数和离群指数的优化包括:最大化协调指数;最大化密码子背景指数;和最小化离群指数。
在一些实施方案中,执行协调指数、密码子背景指数和离群指数的优化包括:针对初始群体集的每个初始候选核酸序列,计算针对各自的初始候选核酸序列的各自的协调指数值、各自的密码子背景指数值和各自的离群指数值;基于该计算,分配对应于多个初始候选核酸序列的多个适应度值;基于该多个适应度值,对多个初始候选核酸序列进行分选;以及将经分选的多个初始候选核酸序列的子集纳入在后续群体集中。在一些实施方案中,多个适应度值包括候选核酸序列的协调指数、密码子背景指数和离群指数。
在一些实施方案中,该方法还包括基于初始群体产生后代群体;以及将后代群体纳入在后续群体集中。
在一些实施方案中,后代群体通过二进制锦标赛(binarytournament)选择、交叉/重组、突变或其任何组合产生。
在一些实施方案中,初始群体集和后续群体集具有相同的大小。
在一些实施方案中,执行协调指数、密码子背景指数和离群指数的优化包括多次迭代,其中多次迭代的第i次迭代包括:接收对应于第(i-1)次迭代的核酸序列的群体集;将对应于第(i-1)次迭代的群体集的每个核酸序列与非支配水平相关联;基于相关联的非支配水平,对对应于第(i-1)次迭代的群体集中的核酸序列进行分选;产生对应于第i次迭代的群体集,其中对应于第i次迭代的群体集包括对应于第(i-1)次迭代的经分选的核酸序列的子集和基于对应于第(i-1)次迭代的经分选的核酸序列产生的后代群体;以及基于一个或多个终止条件,确定是否使用对应于第i次迭代的群体集继续进行第(i+1)次迭代。
在一些实施方案中,将每个核酸序列与非支配水平相关联包括:针对对应于第(i-1)次迭代的群体集的每个核酸序列,计算各自的协调指数值、各自的密码子背景指数值和各自的离群指数值。
在一些实施方案中,产生对应于第i次迭代的群体集包括:将对应于第(i-1)次迭代的经分选的核酸序列中的至少一个核酸序列与多个预定参考点中的一个相关联。
在一些实施方案中,所述一个或多个终止条件包括:达到固定迭代数、最佳适应度达到平稳期且未产生更好的结果、一些解满足近最优解的最小标准,或其任何组合。
在一些实施方案中,候选核酸序列的协调指数基于下式计算:h=1-d(fhs,fts),其中d()指示距离函数;其中fhs包括包含多个高度表达的基因内的多个氨基酸的同义密码子的频率的矢量;并且其中fts包括包含候选核酸序列的编码基因内的多个氨基酸的同义密码子的频率的矢量。
在一些实施方案中,d()指示测量两个矢量之间距离的函数。在一些实施方案中,d()是距离函数,其包括但不限于:两个矢量的欧几里德距离、余弦距离、曼哈顿距离或闵可夫斯基距离。
在一些实施方案中,多个高度表达的基因或候选核酸序列的同义密码子的频率被定义为:
在一些实施方案中,候选核酸序列的密码子背景指数基于下式计算:cc=1-d(fhcc,ftcc),其中d()指示距离函数;其中fhcc包括包含多个高度表达的基因内两个连续氨基酸的同义密码子对的频率的矢量;并且其中ftcc包括包含候选核酸序列的编码基因内两个连续氨基酸的同义密码子对的频率的矢量。
在一些实施方案中,d()指示测量两个矢量之间距离的函数。在一些实施方案中,d()是距离函数,其包括但不限于:两个矢量的欧几里德距离、余弦距离、曼哈顿距离或闵可夫斯基距离。
在一些实施方案中,多个高度表达的基因或候选核酸序列的同义密码子对的频率被定义为:
在一些实施方案中,离群指数基于下式计算:
在一些实施方案中,多个预定特征包括:gc含量值、cis元件、重复元件、rna剪接位点、核糖体结合序列、mrna的最小自由能或其任何组合。
在一些实施方案中,所述多个预定特征基于选择的表达系统来鉴定。
在一些实施方案中,nsga-iii算法的变型包括elitensga-iii算法或基于nsga-ii的免疫算法。
在一些实施方案中,执行协调指数、密码子背景指数和离群指数的优化包括:按照协调指数的降序、然后按照密码子背景指数的降序、然后按照离群指数的升序对多个优化的核酸序列进行排名;选择一个或多个排名最高的优化的核酸序列用于合成。
在一些实施方案中,所述方法还包括:c)从多个优化的核酸序列中的优化的核酸序列中去除预定的不利子序列或基序。
在一些实施方案中,预定的不利子序列或基序基于对多个文本部分的分析来鉴定。
在一些实施方案中,去除预定的不利子序列或基序包括:鉴定优化的核酸序列中的预定的不利子序列或基序;基于所鉴定的预定的不利子序列或基序鉴定多个同义密码子;从多个同义密码子中选择同义密码子,以替换优化的核酸序列中所鉴定的预定的不利子序列。
在一些实施方案中,协调指数、密码子背景指数和离群指数中的至少一者基于来自一个或多个数据库的多个高度表达的基因的一个或多个特征来计算。
在一些实施方案中,所述一个或多个特征包括密码子频率、同义密码子频率、密码子对频率或其组合。
在一些实施方案中,所述方法还包括设置一个或多个参数,其中所述一个或多个参数包括群体集的大小、划分数目、模拟二进制交叉的分布指数、模拟二进制交叉的交叉率、比特翻转突变的突变率、比特翻转突变的分布指数,或其任何组合。
在一些实施方案中,提供了存储一个或多个程序的非暂时性计算机可读存储介质,所述一个或多个程序包括指令,所述指令在由电子设备的一个或多个处理器执行时使电子设备实施本文所述的任何方法。
在一些实施方案中,提供了用于优化在宿主中表达蛋白质的核酸序列的系统,系统包括:一个或多个处理器;内存;以及一个或多个程序,其中所述一个或多个程序存储在内存中并且被配置为由所述一个或多个处理器执行,所述一个或多个程序包括用于实施本文所述的任何方法的指令。
在一些实施方案中,提供了用于优化在宿主中表达蛋白质的核酸序列的电子设备,该设备包括用于实施本文所述的任何方法的工具(means)。
在一些实施方案中,提供了存储在可记录介质上的用于优化在宿主中表达蛋白质的核酸序列的程序产品,该程序产品包括用于实施本文所述的任何方法的计算机软件。
在一些实施方案中,提供了分离的核酸分子,所述分离的核酸分子包含从本文所述的任何方法获得的优化的核酸序列。
在一些实施方案中,提供了包含上述分离的核酸分子的载体。
在一些实施方案中,提供了包含上述分离的核酸分子或上述载体的重组宿主细胞。
在一些实施方案中,提供了用于使蛋白质在宿主细胞中表达的方法,该方法包括:(a)使用本文所述的任何方法获得在宿主细胞中表达所述蛋白质的优化的核酸序列,(b)合成包含优化的核酸序列的核酸分子;(c)将核酸分子引入到宿主细胞中以获得重组宿主细胞;和(d)在使得所述蛋白质能够从优化的核酸序列表达的条件下培养重组宿主细胞。
附图说明
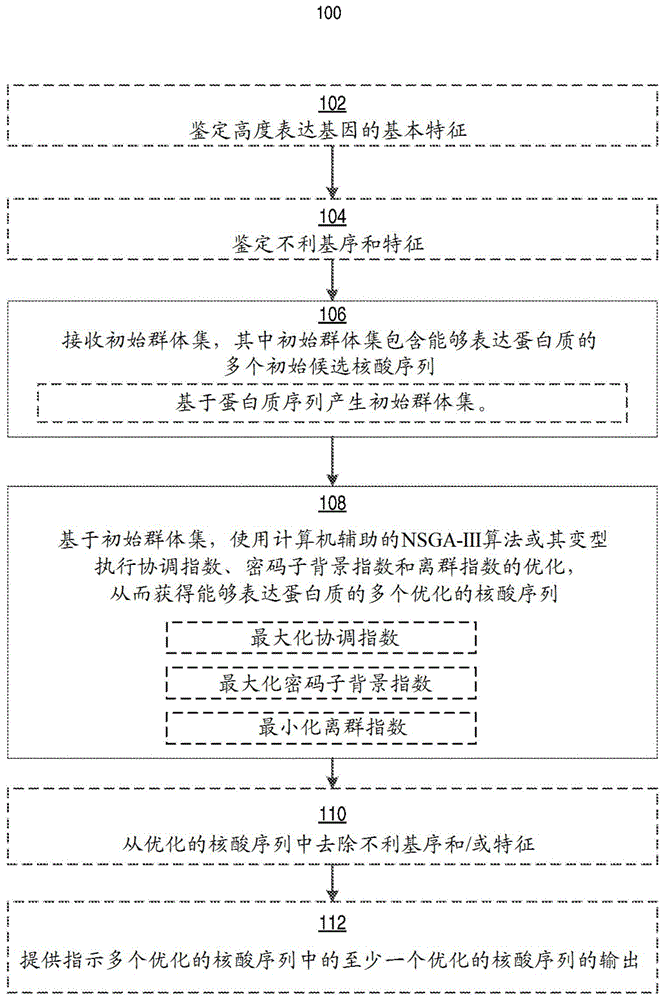
图1描绘了根据一些实施方案的密码子优化的示例过程的框图。
图2a描绘了根据一些实施方案的用于构建和执行用于优化在宿主中表达蛋白质的序列(例如,核酸序列)的算法的示例性流水线。
图2b描绘了根据一些实施方案的遗传算法的示例性一般工作流程。
图3描述了根据一些实施方案的优化的gfp和jnk3a1相对于其野生型的蛋白质印迹结果。
图4描绘了根据一些实施方案的示例性电子设备。
具体实施方式
本发明提供了用于改善基因在各种宿主中的重组表达的增强的密码子优化,所述宿主包括但不限于大肠杆菌、cho、hek293、酵母、昆虫、无细胞表达系统等。根据本发明的示例性系统收集表达系统的高度表达的基因,提取基本序列特征,复制目标序列(例如,核酸序列)中的有益综合模式,并去除不利特征以改善靶基因在表达系统中的表达。
目前,多种密码子优化工具已被开发并汇总在下表1中。这些工具已经考虑了多个,优选大多数或所有的参数和因素,包括密码子使用(例如密码子适应指数[cai]、密码子的有效数目[enc]、相对同义密码子使用[rscu]和同义密码子使用顺序[scuo])、密码子对、trna使用(例如trna适应指数[tai])、gc含量、核糖体结合位点(rbs)、隐藏终止密码子、基序避免、限制性位点去除、基因的mrna二级结构(例如mrna自由能)和亲水性指数优化,以便在细菌、酵母、昆虫和哺乳动物细胞的密码子优化过程中促进表达。
表1
然而,由于有这么多因素可以视为关键点,所以如何平衡它们仍然是一个挑战,因为这是一个多目标优化问题,但目标可能会相互冲突。另一方面,在考虑时忽略一个或多个因素或参数可能会导致靶基因在表达系统中的低表达或无表达。
本文提供了使用多目标优化算法考虑和平衡多种因素的增强密码子优化的系统和方法。根据一些实施方案,密码子优化尤其基于三个目标:(i)首先如何分配某些氨基酸的同义密码子的计数,(ii)如何将同义密码子置于其最合适的位置,以及(iii)如何减少不利但意外产生的子序列和/或基序。在一些实施方案中,这三个目标被量化为协调指数、密码子背景指数和离群指数。在优化过程中,使用多目标算法如非支配排序遗传算法iii(nsga-iii)或其变型来考虑目标。具体而言,对于给定的候选核酸序列,可以参考高度表达的基因的已知特征来计算目标。在一些实施方案中,在基因合成和蛋白质表达之前,从一个或多个优化的序列中去除各种已知的不利基序和/或特征(例如,从文献中鉴定的)。
因此,本发明提供了系统方法,由此优选影响蛋白质表达的所有或大部分参数和因素(包括但不限于密码子协调性、密码子使用(例如同义密码子分布)、密码子背景指数、顺式作用mrna去稳定基序、rna酶剪接位点、gc含量、核糖体结合位点(rbs)、基因的mrna二级结构(例如mrna自由能)和重复元件)都被考虑到,以改进和优化核酸,从而促进基因在表达系统中,诸如在包括真核和原核细胞(如哺乳动物、昆虫、酵母、细菌、藻类)在内的表达宿主细胞中,以及在无细胞表达系统中的蛋白质表达。
因此,本发明在一个方面中提供了使用nsga-iii算法或其变型优化多个(例如,多于2个)目标的改善重组蛋白表达的序列优化方法。在另一方面,提供了用于在基因合成和蛋白质表达之前(例如,在完成nsga-iii算法的迭代之后)从核酸序列中去除不利基序和特征的方法。还提供了用于量化和计算优化算法中的多个目标的方法,以及用于鉴定要减少或去除的不利基序和特征的方法。
还提供了系统、非暂时性计算机可读存储介质、电子设备和程序产品,其用于存储用于实施本文所述的方法的任何一个或多个步骤的一个或多个程序。还提供了包含从本文所述的方法获得的优化的核酸序列的分离的核酸分子;包含所述分离的核酸分子的载体;包含所述分离的核酸分子或所述载体的重组宿主细胞。还提供了用于使蛋白质在宿主细胞中表达的方法,其涉及本文所述的任何方法。
应当理解,本文描述的本发明的实施方案包括"由“实施方案“组成"和/或"基本上由”实施方案“组成"。
本文中提及"约"值或参数包括(且描述了)针对该值或参数本身的变化。例如,提及"约x"的描述包括对"x"的描述。
如本文所用,提及“非”值或参数一般意指且描述了“除值或参数以外”。例如,所述方法不用来治疗x型癌症意指所述方法用于治疗除x以外的类型的癌症。
如本文和所附权利要求书中所使用的,除非上下文另有明确指示,否则单数形式"一"、"或"和"所述"包括复数指代物。
如本文和所附权利要求书中所使用的,除非上下文另有明确指示,否则"集"是指一个或多个指代物。
密码子优化方法
本发明在一方面了提供用于优化在宿主中表达蛋白质的核酸序列的方法(例如,计算机实现或计算机辅助方法)。与这些方法相关的是用于在基因合成和蛋白质表达之前(例如,在完成nsga-iii算法的迭代之后)从核酸序列中去除不利基序和特征的方法。也与这些方法相关的是用于量化和计算优化算法中的多个目标的方法,以及用于鉴定要减少或去除的不利基序和特征的方法。
图1示出密码子优化的示例性过程100,其中虚线框表示任选步骤。虽然过程100的部分在本文中被描述为由特定设备执行,但是应当理解,过程100不限于此。在其他实例中,仅使用单个电子设备(例如,电子设备400)或使用多个电子设备来执行过程100。在过程100中,一些框被任选地组合,一些框的顺序被任选地改变,且一些框被任选地省略。在一些实例中,可以与过程100组合执行附加步骤。
在框106处,电子设备接收初始群体集,其中初始群体集包含能够表达蛋白质的多个初始候选核酸序列。在一些实施方案中,初始群体集随机产生。在一些实施方案中,初始群体集具有预定大小(例如,由用户确定)。
在一些实施方案中,如框106所示,接收初始群体集包括基于蛋白质序列产生初始群体集。例如,接收初始群体集可以包括:接收蛋白质序列(例如,作为来自用户的输入的蛋白质序列);以及基于接收的蛋白质序列产生初始群体集。作为另一实例,接收初始群体集可以包括:接收核酸序列(例如,作为来自用户的输入的核酸序列);将接收的核酸序列翻译成蛋白质序列;基于蛋白质序列产生初始群体集。
在一些实施方案中,初始群体集包括多个初始候选核酸序列的二进制表示(例如,二进制串)。一般而言,选择二进制串而不是密码子列表/阵列/矢量作为表示编码基因的数据结构,并且除了选择之前对基因的适应度评价之外,包括群体初始化、交叉/重组、突变、选择在内的遗传算法的所有操作对象都是二进制串。如下文进一步描述的,在一些实施方案中,当在选择之前需要对整个群体的每个个体评估适应度函数(即三个指数函数)时,应暂时将二进制表示转换回密码子串。
在框108处,电子设备基于初始群体集,使用计算机辅助的nsga-iii算法或其变型执行协调指数、密码子背景指数和离群指数的优化,从而获得能够表达蛋白质的多个优化的核酸序列。
候选核酸序列的协调指数总是或在一些实施方案中指示多个高度表达的基因和候选核酸序列(即在优化期间编码候选蛋白质的基因)之间的同义密码子的使用频率分布的一致性,这有助于解决如何分配某些氨基酸的同义密码子的计数。候选核酸序列的密码子背景指数是用于将同义密码子置于合适位置的量度。候选核酸序列的离群指数是多个预定序列特征对候选核酸序列的负面影响的量度。
在一些实施方案中,如框106所示,执行协调指数、密码子背景指数和离群指数的优化包括:最大化协调指数;最大化密码子背景指数;和最小化离群指数。
可以通过使用多目标遗传算法来执行优化,三个目标是最大化协调指数;最大化密码子背景指数;和最小化离群指数。在一些实施方案中,使用nsga-iii算法或变型。与传统的遗传算法不同,nsga-iii中群体成员间的多样性的维持是通过提供并适应性地更新许多分布良好的预定义参考点来辅助的,因此nsga-iii在其选择算子上具有显著变化。进一步地,相对于其他遗传算法如nsga-ii,nsga-iii展现出它在解决三个目标至15个目标优化问题方面的功效。nsga-iii算法的变型包括elitensga-iii算法、基于nsga-ii的免疫算法、mam-moia或mola。elitensga-iii算法在aminibrahim等人于2016年发表的标题为"elitensga-iii:animprovedevolutionarymany-objectiveoptimizationalgorithm"的出版物中有所描述,该出版物以引用的方式并入本文。各种免疫算法描述于例如guan-chunluh等人于2010年9月发表的标题为"moia:multi-objectiveimmunealgorithm"的出版物、felipecampelo等人于2007年发表的标题为"overviewofartificialimmunesystemsformulti-objectiveoptimization"的出版物、zhi-huahu于2010年4月发表的标题为"amultiobjectiveimmunealgorithmbasedonamultiple-affinitymodel"的出版物和2017年7月25日提交的中国专利申请第201710611752.5号中,这些出版物以引用的方式并入本文。
根据nsga-iii算法(或类似遗传算法)的操作,执行协调指数、密码子背景指数和离群指数的优化包括:针对初始群体集的每个初始候选核酸序列,计算各自的初始候选核酸序列的各自的协调指数值、各自的密码子背景指数值和各自的离群指数值;基于该计算,分配对应于多个初始候选核酸序列的多个适应度值;基于该多个适应度值,对多个初始候选核酸序列进行分选;以及将经分选的多个初始候选核酸序列的子集纳入在后续群体集中(即,以供在第2次迭代中使用)。
根据nsga-iii算法(或类似遗传算法)的操作,该方法还包括基于初始群体产生后代群体;以及将后代群体纳入在后续群体集中(即,以供在第2次迭代中使用)。在一些实施方案中,后代群体通过二进制锦标赛选择、交叉/重组、突变或其任何组合产生。
在一些实施方案中,初始群体集和后续群体集(即,以供在第2次迭代中使用)具有相同的大小。
根据nsga-iii算法(或类似遗传算法)的操作,执行协调指数、密码子背景指数和离群指数的优化包括多次迭代。多次迭代中的第i次迭代(其中i可以是2、3、4、5、6……n)包括:接收对应于第(i-1)次迭代的核酸序列的群体集;将对应于第(i-1)次迭代的群体集的每个核酸序列与非支配水平相关联;基于相关联的非支配水平,对对应于第(i-1)次迭代的群体集中的核酸序列进行分选;产生对应于第i次迭代的群体集,其中对应于第i次迭代的群体集包括对应于第(i-1)次迭代的经分选的核酸序列的子集和基于对应于第(i-1)次迭代的经分选的核酸序列产生的后代群体;以及基于一个或多个终止条件,确定是否使用对应于第i次迭代的群体集继续进行第(i+1)次迭代。
在一些实施方案中,将每个核酸序列与非支配水平相关联包括:针对对应于第(i-1)次迭代的群体集的每个核酸序列,计算各自的协调指数值、各自的密码子背景指数值和各自的离群指数值。
根据nsga-iii算法的操作,在一些实施方案中,产生对应于第i次迭代的群体集包括:将对应于第(i-1)次迭代的经分选的核酸序列中至少一个核酸序列与多个预定参考点中的一个相关联。
在一些实施方案中,所述一个或多个终止条件包括:达到固定迭代数、最佳适应度达到平稳期且未产生更好的结果、一些解满足近最优解的最小标准,或其任何组合。
在一些实施方案中,该方法还包括为优化算法设置一个或多个参数,其中所述一个或多个参数包括群体集的大小、划分数目、模拟二进制交叉的分布指数、模拟二进制交叉的交叉率、比特翻转突变的突变率、比特翻转突变的分布指数,或其任何组合。
在一些实施方案中,在优化期间,协调指数、密码子背景指数和离群指数中的至少一者基于来自一个或多个数据库的多个高度表达的基因的一个或多个特征来计算。在一些实施方案中,所述一个或多个特征包括密码子频率、同义密码子频率、密码子对频率或其组合。对于给定的候选核酸序列,高度表达的基因的这些特征可用于计算协调指数、密码子背景指数和离群指数,如下式所示。
在一些实施方案中,如框102所指示,基于私有或公共数据库鉴定高度表达的基因的这些特征。例如,该一个或多个数据库可以是专有数据库,其包含从公司的订单系统收集的先前成功优化的订单。作为另一个实例,该数据可以通过对各种培养条件下的rna-seq数据进行数据挖掘来获得,所述rna-seq数据可以是公共信息。进行数据处理的目的是获取高度表达的基因的基本信息,包括密码子频率、同义密码子频率和密码子对频率。
在一些实施方案中,候选核酸序列的协调指数基于下式计算:h=1-d(fhs,fts),其中d()指示距离函数;其中fhs包括包含多个高度表达的基因内的多个氨基酸的同义密码子的频率的矢量;并且其中fts包括包含候选核酸序列的编码基因内的多个氨基酸的同义密码子的频率的矢量。
在一些实施方案中,d()指示测量两个矢量之间距离的函数。在一些实施方案中,d()是距离函数,其包括但不限于:两个矢量的欧几里德距离、余弦距离、曼哈顿距离或闵可夫斯基距离。
在一些实施方案中,多个高度表达的基因或候选核酸序列的同义密码子的频率被定义为:
在一些实施方案中,候选核酸序列的密码子背景指数基于下式计算:cc=1-d(fhcc,ftcc),其中d()指示距离函数;其中fhcc包括包含多个高度表达的基因内两个连续氨基酸的同义密码子对的频率的矢量;并且其中ftcc包括包含候选核酸序列的编码基因内两个连续氨基酸的同义密码子对的频率的矢量。
在一些实施方案中,d()指示测量两个矢量之间距离的函数。在一些实施方案中,d()是距离函数,其包括但不限于:两个矢量的欧几里德距离、余弦距离、曼哈顿距离或闵可夫斯基距离。
在一些实施方案中,多个高度表达的基因或候选核酸序列的同义密码子对的频率被定义为:
在一些实施方案中,离群指数基于下式计算:
在一些实施方案中,多个预定特征包括:gc含量值、cis元件、重复元件、rna剪接位点、核糖体结合序列、mrna的最小自由能或其任何组合。
在一些实施方案中,所述多个预定特征基于选择的表达系统来鉴定。对于各种表达系统,不利因素的目录(catalogue)可能发生变化,其影响或权重也不相等。
在一些实施方案中,执行协调指数、密码子背景指数和离群指数的优化包括:按照协调指数的降序、然后按照密码子背景指数的降序、然后按照离群指数的升序对多个优化的核酸序列进行排名;选择一个或多个排名最高的优化的核酸序列用于合成。
在框110处,该方法任选地还包括:c)从多个优化的核酸序列中的优化的核酸序列中去除预定的不利子序列或基序。在一些实施方案中,去除预定的不利子序列或基序包括:鉴定优化的核酸序列中的预定的不利子序列或基序;基于所鉴定的预定的不利子序列或基序鉴定多个同义密码子;从多个同义密码子中选择同义密码子,以替换优化的核酸序列中所鉴定的预定的不利子序列。
在一些实施例中,如方框104所指示,预定的不利子序列或基序基于对多个文本部分的分析(例如,自动文本挖掘或文献的手动检查)来鉴定。
在一些实施方案中,该方法还包括提供指示多个优化的核酸序列中的至少一个优化的核酸序列的输出。
在一些实施方案中,提供了存储一个或多个程序的非暂时性计算机可读存储介质,所述一个或多个程序包括指令,所述指令在由电子设备的一个或多个处理器执行时使电子设备实施本文所述的任何方法。
在一些实施方案中,提供了用于优化在宿主中表达蛋白质的核酸序列的系统,系统包括:一个或多个处理器;内存;以及一个或多个程序,其中所述一个或多个程序存储在内存中并且被配置为由所述一个或多个处理器执行,所述一个或多个程序包括用于实施本文所述的任何方法的指令。
在一些实施方案中,提供了用于优化在宿主中表达蛋白质的核酸序列的电子设备,该设备包括用于实施本文所述的任何方法的工具。
在一些实施方案中,提供了存储在可记录介质上的用于优化在宿主中表达蛋白质的核酸序列的程序产品,该程序产品包括用于实施本文所述的任何方法的计算机软件。
在一些实施方案中,提供了分离的核酸分子,所述分离的核酸分子包含从本文所述的任何方法获得的优化的核酸序列。
在一些实施方案中,提供了包含上述分离的核酸分子的载体。
在一些实施方案中,提供了包含上述分离的核酸分子或上述载体的重组宿主细胞。
在一些实施方案中,提供了用于使蛋白质在宿主细胞中表达的方法,该方法包括:(a)使用本文所述的任何方法获得在宿主细胞中表达所述蛋白质的优化的核酸序列,(b)合成包含优化的核酸序列的核酸分子;(c)将核酸分子引入到宿主细胞中以获得重组宿主细胞;和(d)在使得所述蛋白质能够从优化的核酸序列表达的条件下培养重组宿主细胞。
图2a示出了根据本发明的一些实施方案的用于构建和执行用于优化在宿主中表达蛋白质的序列(例如,核酸序列)的算法的示例性流水线200。过程200例如使用图4所示的一个或多个电子设备来执行。在一些实例中,使用客户端-服务器系统执行过程200,并且以任何方式在服务器和客户端设备之间划分过程200的框。在其他实例中,过程200的框在服务器和/或多个客户端设备之间进行划分。因此,虽然过程200的部分在本文中被描述为由特定设备进行,但是将理解,过程200不限于此。在其他实例中,仅使用单个电子设备(例如,电子设备400)或使用多个电子设备来进行过程200。在过程200中,一些框被任选地组合,一些框的顺序被任选地改变,且一些框被任选地省略。在一些实例中,可以与过程200组合执行附加步骤。
数据收集与文献综述
参考图2a,在框202处,可以从一个或多个数据库中鉴定多个高度表达的基因。该数据库可以是公共的或私有的。例如,该一个或多个数据库可以是专有数据库,其包含从公司的订单系统收集的先前成功优化的订单。作为另一个实例,该数据可以通过对各种培养条件下的rna-seq数据进行数据挖掘来获得,所述rna-seq数据可以是公共信息。
在框204处,鉴定高度表达的基因的基本特征。在示例性实现方式中,按照illumina推荐的标准样品的mrna-seq工作流程执行mrna-seq实验和数据分析。在该过程中,truseqstrandedmrna文库制备套件可用于文库制备,并且nextseq的pe300可用于测序。随后,可应用通过tophat、cufflinks和自制脚本进行的数据处理,目的是获取高度表达的基因的基本信息,包括密码子频率、同义密码子频率和密码子对频率。
在框206和208处,示例性系统还可以鉴定要避免的任何报告的和验证的不利特征,以便保持已确立的优势。为了发现可能导致蛋白表达减少的负面因素,该系统可以进行文献综述。例如,通过自动文本挖掘和/或手动检查,可以针对各种宿主鉴定所报告的与表达相关的不利基序和mrna特征。
优化算法的关键因素/适应度函数
编码基因的表达具有多个步骤,这取决于转录、mrna更新、翻译(包括起始、启动子逃逸、延伸和终止)和翻译后修饰的水平。然而,密码子优化可以被简化为组合问题,并被分组为三个直观的操纵:(i)首先如何分配某些氨基酸的同义密码子的计数,(ii)如何将同义密码子置于其最合适的位置,以及(iii)如何减少不利但意外产生的子序列和/或基序。
根据本发明的一些实施方案,下面提供分别与三种上述操纵匹配并且与蛋白质表达高度相关的三个关键因素:协调指数、密码子背景指数和离群指数。如下文所讨论的,这三个指数基于从各种数据源收集的上述基础数据来计算。
参考图2a,在框210处,执行包括两个步骤212和214的优化程序。在框212中所示的步骤1处,该系统基于nsga-iii算法或其变型执行多目标密码子优化,这涉及最大化协调指数、最大化密码子背景指数和最小化离群指数。
1.协调指数
协调指数表示高度表达的基因和候选核酸序列之间同义密码子使用频率分布的一致性。候选核酸序列是指在优化算法的至少一次迭代中评估的编码候选蛋白质的基因,其在标题"多目标优化算法"下有详细描述。在一些实施方案中,协调指数被定义为:
h=1-d(fhs,fts)
在上式中,h为协调指数,d()为两个矢量之间的距离函数,该距离函数可以是但不限于:欧几里德距离、余弦距离、曼哈顿距离或闵可夫斯基距离。fhs是包含高度表达的基因中18个氨基酸(除met/m和trp/w外)的同义密码子的频率的矢量,并且由于从64个密码子中去除了3个终止密码子(即taa、tag和tga)、氨基酸met/m的密码子(即atg)和氨基酸trp/w的密码子(即tgg)而具有59个元件。fts是包含等待密码子优化的候选蛋白质的编码基因(即候选核酸序列)内的18个氨基酸的同义密码子的频率的矢量。
相对于密码子适应指数(cai),协调指数专注于同义密码子的分布(即使用平衡/负载平衡),但并不总是旨在通过唯一地选择最频繁出现的排名第1的同义密码子来最大化cai。
在一些实施方案中,在计算协调指数期间使用的高度表达的基因或候选核酸序列的某些同义密码子的频率被定义为:
虽然协调指数考虑了密码子的使用,但它只关心同义密码子的频率分布,而它们在18个氨基酸之一的不同基因座处的分配仍然是一个问题(即,相同氨基酸的同义密码子的排序设置)。因此,需要下面描述的密码子背景指数,以通过同义密码子配对来解决此瓶颈,从而为同义密码子选择近似最佳的排名。
2.密码子背景指数
候选核酸序列的密码子背景指数是用于将同义密码子置于合适位置的量度。在一些实施方案中,密码子背景指数被定义为:
cc=1-d(fhcc,ftcc)。
在上式中,cc代表密码子背景指数,d()是两个矢量之间的距离函数,该距离函数可以是但不限于:欧几里德距离、余弦距离、曼哈顿距离或闵可夫斯基距离。fhcc是包含高度表达的基因内所有种类的两个连续氨基酸的同义密码子对的频率的矢量。例如,氨基酸phe/f具有两个同义密码子,即ttt和ttc;氨基酸lys/k也具有aaa和aag作为密码子;它们的同义密码子对应为2×2组合,包括tttaaa、tttaag、ttcaaa和ttcaag。由于蛋氨酸/m和色氨酸/w这两种氨基酸的排列(即mm、mw、ww和wm)不存在同义密码子对,因此cc的长度为61乘61减4,最终等于3717。ftcc是包含候选蛋白质的编码基因(即候选核酸序列)内所有种类的两个连续氨基酸的同义密码子对的频率的矢量,其长度也是3717。
在密码子背景指数的计算期间使用的高度表达的基因或候选核酸序列的某些同义密码子对的频率被定义为:
3.离群指数
离群指数是通过加权函数计算的用于评估所鉴定的多个序列特征对蛋白质表达的负面影响的量度。在一些实施方案中,离群指数被定义为:
在上式中,n是所鉴定的多个序列因素的数目,且n>1。fi(x)指示所鉴定的n个序列特征的第i个序列因素的罚分函数;而wi指示给予fi(x)的相对权重。因此,优化的基因应尽可能具有低的离群指数值。
在一些实施方案中,所述多个序列因素可以通过图2a中所示的步骤202、204和208中的一个或多个来鉴定。在一些实施方案中,所述多个序列因素含有但不限于下文详细描述的gc含量、cis元件、重复元件、rna剪接位点、核糖体结合序列、mrna的最小自由能。
3(a).mrna的最小自由能(mfe)
mrna的位于起始密码子下游的潜在的强茎环二级结构可能阻碍核糖体复合体的运动,从而减缓翻译并降低翻译效率。mrna的稳定二级结构甚至可导致核糖体复合体从mrna上脱落,并导致翻译的过早终止。存在多种自由能计算和二级结构预测方法,包括mfold、rnafold和rnastructure。根据本发明的实施方案,具有低自由能(△g<-18kcal/mol)或长互补茎(>10bp)的mrna的局部二级结构被界定为太稳定而无法进行有效翻译。基因序列优选被优化为使局部结构不那么稳定。对于mrna结构自由能计算和二级结构预测,优选考虑mrna的5'-utr和3'-utr。
在一些实施方案中,被认为太稳定的二级结构与较高的罚分相关联。用于给出较高罚分的权重是灵活的。
3(b).gc含量
还优选考虑mrna的gc含量。gc%的理想范围为大约30-70%。高gc含量将使mrna形成强的茎环二级结构。这也将给pcr扩增和基因克隆带来问题。优选使用密码子简并性来使靶序列的高gc含量突变(例如,在包括二进制串的交叉和突变在内的nsga-iii算法的操作期间)至约50-60%。
gc%有两种不同的测量。一个是沿整个序列取平均值的全局gc%;另一个更有用,它是在固定大小(例如,60bp)的移位"窗口"内计算的局部gc%。根据本发明的实施方案,局部gc%被优化至35-65%左右。
3(c).不稳定因素(如顺式作用mrna去稳定基序、rna酶剪接位点和重复元件等)
为了减少或最小化mrna降解或增加mrna的稳定性从而减少mrna的周转时间,优选使顺式作用mrna去稳定基序(包括但不限于富含au的元件(are)以及rna酶识别和切割位点)从基因序列中突变或缺失。具有auuua(seqidno:1)核心基序的富含au的元件(are)通常存在于mrna的3'非翻译区。mrna顺式元件的另一实例由序列基序tgyygatgyyyyy(seqidno:2)组成,其中y代表t或c。rna酶识别序列包括但不限于rna酶e识别序列。rna酶缺乏的宿主菌株也可用于蛋白质表达。
rna酶剪接位点可导致rna剪接以产生不同的mrna,并从而降低原始mrna水平。rna酶剪接位点也优选突变为非功能性的以维持mrna水平。
为了产生高水平的mrna,优选在基因序列中使用最佳的转录启动子序列。对于原核宿主如大肠杆菌,强启动子之一是t7rna聚合酶的t7启动子(t7rnap)。长串联或短串联简单重复序列(ssr)的一些碱基优选使用密码子简并性进行突变以破坏重复序列,从而减少聚合酶滑移,从而减少蛋白质或蛋白质过早突变。
还存在影响mrna翻译和所得的蛋白表达水平的另外的因素和参数。这些因素影响从翻译起始到翻译终止的翻译。核糖体在核糖体结合位点(rbs)结合mrna以启动翻译。由于核糖体不与双链rna结合,因此此区域周围的局部mrna结构优选为单链,且不形成任何稳定的二级结构。优选将诸如大肠杆菌的原核细胞的共有rbs序列aggagg(seqidno:3)(也称为shine-dalgarnon序列)置于刚好在待表达基因中的翻译起始位点之前的几个碱基处。然而,优选使内部核糖体进入位点(ires)发生突变,以防止核糖体结合,从而避免非特异性翻译启动。
上述因素的描述可见于,例如,saeidkadkhodaei等人于2018年5月发表的标题为“cis/transgeneoptimization:systematicdiscoveryofnovelgeneexpressionusingbioinformaticsandcomputationalbiologyapproaches”的出版物,timothyjgingerich等人于2014年7月发表的标题为“au-richelementsandthecontrolofgeneexpressionthroughregulatedmrnastability”的出版物,以及talabakheet于2017年10月发表的标题为“ared-plus:anupdatedandexpandeddatabaseofau-richelement-containingmrnasandpre-mrnas”的出版物,shuangzhang等人于1995年发表的标题为“identificationandcharacterizationofasequencemotifinvolvedinnonsense-mediatedmrnadecay”的出版物,jiongma等人于2002年发表的标题为"correlationsbetweenshine-dalgarnosequencesandgenefeaturessuchaspredictedexpressionlevelsandoperonstructures"的出版物,esthery.c.koh等人于2013年12月发表的标题为"aninternalribosomeentrysite(ires)mutantlibraryfortuningexpressionlevelofmultiplegenesinmammaliancells"的出版物,这些出版物以引用的方式整体并入本文。
对于各种表达系统,不利因素的目录可变化,其影响或权重也不相等。因此,可针对各种表达系统动态修改fi(x)及其权重。例如,在设置gc含量和mfe的允许范围后,“超出范围”的程度将按比率进行罚分。同样,不稳定因素的出现次数也可以直接记录为罚分。
应该认识到,即使候选核酸序列的离群指数高,候选序列仍可能有一些机会在迭代中存留下来,以便保持整个群体的多样性。换句话说,通过离群指数进行的不利基序/特征过滤不是强制性的,因为较高的离群指数(即罚分)可能只会导致较低的存留率。相反,在nsga-iii算法的迭代完成之后的不利基序/特征去除(即,在图1中的步骤110或图2中的步骤214中)是强制性的。
总之,本发明不仅试图通过最大化协调指数和密码子背景指数的值来促进正面影响,而且还通过最小化离群指数来尽可能避免不利影响。
多目标(如超过2个目标)优化算法
由于本发明是三个综合目标的优化任务,因此可以使用多目标遗传算法。在一些实施方案中,可以使用nsga-iii算法或其变型如elitensga-iii(也由k.deb提出),因为它们在通过在遗传算法的经典框架的选择操纵期间维持群体多样性来解决多目标优化问题上具有优势。
nsga-iii由kalyanmoydeb和himanshujain于2014年提出。它是遵循nsgaii框架的基于参考点的多目标进化算法,该算法强调非支配的但接近所提供一组参考点的群体成员。相对于其他遗传算法如nsga-ii,nsga-iii展现出其在解决三个目标至15个目标优化问题方面的功效。与传统的遗传算法不同,nsga-iii中群体成员间的多样性的维持是通过提供并适应性地更新许多分布良好的预定义参考点来辅助的,因此nsga-iii在其选择算子上具有显著变化。
nsga-iii算法在kalyanmoydeb等人于2014年8月发表的标题为"anevolutionarymany-objectiveoptimizationalgorithmusingreference-point-basednondominatedsortingapproach,parti:solvingproblemswithboxconstraints"的出版物中有描述,该出版物以引用方式整体并入本文。相关的nsga-ii算法在kalyanmoydeb等人于2002年8月发表的标题为“afastandelitistmultiobjectivegeneticalgorithm:nsga-ii”的出版物中有描述,该出版物以引用的方式整体并入本文。
在nsga-iii的实现过程中,选择二进制串而不是密码子列表/阵列/矢量作为代表核酸序列的数据结构,并且包括群体初始化、交叉/重组、突变在内的通用遗传算法的所有通用操纵对象都是二进制串,因为相对于密码子列表/阵列/矢量作为数据结构,二进制串需要更小的计算机内存并且实现更快的操纵速度。在一些实施方案中,三个连续比特用于表示一个位置处的密码子,因为三个比特的所有组合的数目足以匹配某些氨基酸的同义密码子的所有可能候选者。例如,三个比特具有8种组合,例如000、001、010、011、100、101、110和111,其计数大于任何氨基酸,甚至分别拥有6个同义密码子的氨基酸l、r和s的同义密码子的数目。
因此,3个比特串中的每一个代表给定氨基酸的同义密码子。在适应度计算(例如,协调指数、密码子背景指数和离群指数的计算)期间,代表群体的单个候选者的二进制串被转换回编码测序(codingsequencing)(即dna)。另一方面,如上所讨论的,遗传算法的操作(包括交叉、突变、选择)的对象都是二进制串,因此该转换是临时性的。因此,适应度计算基于序列,而所有其他操作都基于二进制串以提高效率和速度。
在开始nsga-iii之前,需要设置多个参数,包括群体大小、划分数目、所模拟二进制交叉的分布指数、所模拟二进制交叉的交叉率、比特翻转突变的突变率、比特翻转突变的分布指数。nsga-iii的作者针对多目标问题的划分提出了一种两层途径,其中指定了外部和内部划分数目。为了使用两层途径,我们可以用外部划分数目和内部划分数目替代划分数目。每个个体的初始化过程都是随机的,且交叉和突变操纵与如图2b所示的经典遗传算法没有很大区别。
图2b描绘了遗传算法的示例性一般工作流程,其包括生物启发的算子如交叉、突变和群体进化选择。在本发明的实现过程中,二进制串表示序列,因此所有以上算子的对象都是二进制串。
当在选择之前需要对整个群体的每个个体评估适应度函数(即前面所示的三个指数函数)时,二进制串将被暂时地转换回密码子串。在许多进化世代和进化终止之后,最终产生的密码子串将被串联并作为用于重组表达的最佳基因输出。
在一些实施方案中,终止条件包括但不限于:达到固定的世代数、最佳适应度达到平稳期且未产生更好的结果、一些解满足近最优解的最低标准。
根据nsga-iii算法的教导,这些最佳基因应该是位于三维空间的帕累托表面上的解并且应被等同地处理。出于实际目的,由于用于基因合成和表达测试的资源有限,我们首先按照协调指数的降序,然后按照密码子背景指数的降序并最后按照离群指数的升序对它们进行排名。鉴于配额仅是一个序列,可以选择排名第1者进行合成和异源表达。假设没有严格的成本控制,建议测试其中在帕累托表面具有足够间隔的几个,例如,一个具有最高协调指数的候选者,一个具有最高密码子背景指数的候选者和一个具有最低离群指数的候选者。在本发明中,初步的最佳基因没有终止密码子,因此可以在编码序列的3′末端附加两个连续的终止密码子。
用于分子克隆的特定子序列去除
参考图2a,在框214处,优化程序包括基序避免和限制性位点去除的步骤。为了促进分子克隆的便利性,在基因合成和蛋白质表达之前,从一个或多个优化的序列中去除一些不利基序和限制位点(例如,客户不喜欢的基序和限制位点)。过程包括:
步骤1:定位必须避免的所有子序列。
步骤2:列出可用于在子序列内进行替换的所有同义密码子。
步骤3:在我们应保持没有新的子序列在同一时间出现的条件下,高度表达的基因内更频繁使用的同义密码子具有更高的选择优先级。
步骤4:使用步骤2-3迭代地处理每个找到的子序列。
在一些实施方案中,如框206和208所指示,通过文本挖掘和文献综述,针对各种宿主分别鉴定不利基序和特征。
示例性实现方式
本文描述的示例性实现方式通过优化和表达cho3e7细胞系中两个基因(jnk3a1和gfp)来举例说明了本发明对密码子优化的效率,这两个基因的基本信息概述在下文。由于应用flag标签的抗体执行蛋白质印迹以便评估表达水平,所以在两个蛋白的c末端附加flag标签,同时使用β-肌动蛋白作为加载对照。每个表达实验重复两次。
根据illumina推荐的经典mrna-seq方案,执行在包括freestylecho表达培养基和cdcho培养基(thermofish)在内的几种培养基中培养的cho3e7的mrna-seq。与本公司成功优化的部分订单整合,共500个序列被定义为cho3e7细胞系的高度表达的基因。在文献综述后,将以下子序列分组为不利基序,不利基序的出现导致罚分(即离群指数增加)。合适的局部(60bp滑动窗口)和全局gc含量为35-65%左右,mrna二级结构的可接受的最小mfe△g为-18kcal/mol,这些参数的离群值导致了罚分。
1)剪接位点:ggtaag,ggtgat
2)富含at的元件:atttta,attttta,atttttta
3)核糖体结合位点:accaccatgg(seqidno:4),gccaccatgg(seqidno:5)
4)抗病毒基序:tgtgt,aacgtt,cgttcg,agcgct,gacgtc,gacgtt
5)cpg岛:cgcgcgcg
6)聚合酶滑移位点:gggggg,cccccc
7)淀粉样前体蛋白3主要稳定元件:tctctttacattttggtctctatactaca(seqidno:6)
8)k-盒:ctgtgata
9)brd-盒:agcttta
在通过nsga-iii进行密码子优化期间,群体大小被设置为100,个体被二进制编码并随机产生,其长度等于蛋白质氨基酸数目的3倍,进化世代数等于200,000,划分数目取决于适应度函数的数目,所模拟二进制交叉的分布指数为15.0,所模拟二进制交叉的单点交叉率为0.9,比特翻转突变的突变率为1.0/l,比特翻转突变的分布指数为20.0。
在最大化协调指数和密码子背景指数并最小化离群指数后,每个蛋白质具有几个输出最佳编码基因,其中只有一个具有最大协调指数的基因被选择用于随后的表达测试。由于ecori和hindiii酶被用于载体构建和克隆,因此通过密码子替换避免了gaattc和aagctt。
本文以ascii文本文件提交的序列表包括两种蛋白质gfp_flag(seqidno:7)和jnk3_flag(seqidno:8)的优化序列。
下面描述用于评价优化基因相对于相同基因的野生型的性能的实验的详细步骤。
步骤1:瞬时转染和细胞培养
1.使用ecori和hindiii酶将合成的基因克隆到ptt5载体中。在freestylecho表达培养基中培养cho3e7细胞,并使用标准分子生物学技术以合适的细胞-载体比率(即,相对于1ug/ml的载体浓度,细胞密度为1-1.2×106/ml)完成载体的瞬时转染。
2.瞬时转染后,需要将cho3e7细胞在37℃下在5%co2下悬浮培养,持续48小时。
步骤2:细胞破碎
1.从上游获取培养的细胞,在4℃下离心(10,000xg)2min。弃去上清液。
2.添加1ml1*pbs以重悬在eppendorf管底部的细胞。然后在4℃离心(10,000xg)2min,弃去上清液。
3.每1*106个细胞向eppendorf管中添加200μl裂解缓冲液(低渗缓冲液[10mmtris、1.5mmmgcl2、10mmkcl,ph7.9]+0.5%ddm、pmsf[终浓度1mm]、核酸酶混合液)。用移液器重悬细胞。
4.将细胞置于杯状超声细胞破碎器中进行细胞破碎(4℃,3s超声,1s间隔,共10min)。
5.破碎后,在4℃下离心(12,000xg)20min。回收上清液。
步骤3:样品处理
1.使用bca法测量上清液的浓度。
2.用上样缓冲液处理部分上清液。
步骤4:电泳和蛋白质印迹
1.根据sop加载经处理的样品进行sds-page。(每个样品8μg)
2.电泳后,根据sop完成蛋白质印迹实验:
1)转移:在sds-page后取出凝胶,并将蛋白质从凝胶转移至pvdf膜上(转移缓冲液:将200ml5x转移溶液添加到150ml绝对乙醇中并稀释至1l,并转移1h)。
2)封闭:转移后,用快速封闭溶液将pvdf封闭10min。
3)孵育:封闭后,与5%牛奶和相应的标记的抗体一起孵育45min。(flag标签:以1:5000稀释的小鼠-抗-flagmabgenscript(目录号a00187),并添加1:1000稀释的thetmβ肌动蛋白抗体mab小鼠genscript(目录号a00702)1h,然后添加1:2500稀释的标记的二抗山羊抗小鼠igg-hrpgenscript(目录号a00160))。
4)暴露:抗体孵育后,使用chemidoctm接触成像系统执行暴露成像,并将图像保存到指定位置进行编辑。
5)使用imagelab进行蛋白质定量分析。
图3是蛋白质印迹结果,其阐明了根据本公开的实施方案的cho3e7细胞系中两个基因(即gfp和jnk3a1)的优化序列与野生型之间的表达比较,其中仅测试每个基因中具有最高协调指数的优化解以进行表达比较。显然证明,相对于几乎未改变的内部对照β-肌动蛋白,本发明对于密码子优化是有效的并且促进了表达。左侧泳道始终是梯形标记物,单个质粒的每次表达均重复两次。根据粗略的定量分析,估计本发明的密码子优化后,gfp的表达提高了约6.2倍,而jnk3的表达提高了约2.4倍。
示例性电子设备
图4示出了根据一个实施方案的计算设备的实例。设备400可以是连接到网络的主计算机。设备400可以是客户端计算机或服务器。如图4所示,设备400可以是任何合适类型的基于微处理器的设备,例如个人计算机、工作站、服务器或手持计算设备(便携式电子设备),例如电话或平板电脑。该设备可以包括例如处理器410、输入设备420、输出设备430、存储设备440和通信设备460中的一个或多个。输入设备420和输出设备430一般可以对应于上述那些设备,并且可以与计算机连接或集成。
输入设备420可以是提供输入的任何合适的设备,如触摸屏、键盘(keyboard)或小键盘(keypad)、鼠标或语音识别设备。输出设备430可以是提供输出的任何合适的设备,如触摸屏、触觉设备或扬声器。
存储器440可以是提供存储的任何合适的设备,如包括ram、高速缓存、硬盘驱动器或可移动存储盘在内的电、磁或光学内存。通信设备460可以包括能够通过网络发送和接收信号的任何合适的设备,如网络接口芯片或设备。计算机的组件可以以任何适当的方式连接,例如经由物理总线连接或无线地连接。
可存储在存储器440中并由处理器410执行的软件450可包括,例如,体现本公开的功能性的编程(例如,如体现在上文所描述的设备中的编程)。
软件450还可以在任何非暂时性计算机可读存储介质内存储和/或传输,以供指令执行系统、装置或设备,例如上面所述的可从该指令执行系统、装置或设备提取与该软件相关联的指令并执行该指令的那些系统、装置或设备使用或者与其结合使用。在本公开的上下文中,计算机可读存储介质可以为可含有或存储供指令执行系统、装置或设备使用或与其结合使用的编程的任何介质,例如存储器440。
软件450还可以在任何传输介质内传播,以供指令执行系统、装置或设备,例如上面所述的可从该指令执行系统、装置或设备提取与该软件相关联的指令并执行该指令的那些系统、装置或设备使用或者与其结合使用。在本公开的上下文中,传输介质可为可通信、传播或传输编程以供指令执行系统、装置或设备使用或与其结合使用的任何介质。传输可读介质可以包括但不限于电子、磁、光学、电磁或红外有线或无线传播介质。
设备400可连接到网络,该网络可以是任何合适类型的互连通信系统。该网络可以实现任何合适的通信协议,并且该网络的安全可以由任何合适的安全协议来保护。该网络可以包括具有可实现网络信号的传送和接收的任何适当的布置的网络链路,如无线网络连接、t1或t3线路、有线网络、dsl或电话线。
设备400可以实现适于在网络上操作的任何操作系统。软件450可以用任何合适的编程语言(例如c、c++、java或python)编写。在各种实施方案中,体现本公开的功能性的应用软件可以以不同的配置(诸如以客户端/服务器布置的方式)或者通过web浏览器作为例如基于web的应用程序或web服务来进行部署。
尽管已经参考附图充分描述了本公开和实例,但是应当注意,各种改变和修改对于本领域技术人员来说是显而易见的。此类改变和修改应当被理解为包括在由权利要求书限定的本公开和实例的范围内。
为了解释的目的,已经参考特定实施方案描述了前述说明书。然而,以上说明性讨论并非旨在穷举或将本发明限制为所公开的精确形式。鉴于上述教导,许多修改和变化是可能的。选择和描述实施方案是为了最好地解释该技术的原理及其实际应用。因此,本领域的其他技术人员能够利用如适合于所考虑的特定用途的各种修改来最佳地利用该技术和各种实施方案。
序列表
<110>南京金斯瑞生物科技有限公司(nanjinggenscriptbiotechco.,ltd.)
<120>密码子优化
<130>75989-20004.40
<140>尚未分配
<141>同时随同提交
<160>8
<170>fastseqforwindowsversion4.0
<210>1
<211>5
<212>rna
<213>人工序列(artificialsequence)
<220>
<223>合成构建体
<400>1
auuua5
<210>2
<211>13
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>合成构建体
<400>2
tgyygatgyyyyy13
<210>3
<211>6
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>合成构建体
<400>3
aggagg6
<210>4
<211>10
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>合成构建体
<400>4
accaccatgg10
<210>5
<211>10
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>合成构建体
<400>5
gccaccatgg10
<210>6
<211>29
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>合成构建体
<400>6
tctctttacattttggtctctatactaca29
<210>7
<211>738
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>合成构建体
<400>7
atgtctaagggagaagagctgtttaccggcgtggtccctatcctggtggagctggacggc60
gatgtgaacggccagaaattcagcgtgtccggcgagggcgaaggcgacgccacctacggc120
aagctgacactgaagttcatctgcaccaccggcaagctgcctgtcccttggccaacactg180
gtgaccaccttcagctacggagtgcaatgcttctccagataccctgaccacatgaagcag240
cacgatttctttaaatctgccatgcccgagggctacgtgcaggaacggaccatcttctac300
aaggacgacggaaattacaagaccagagccgaggtgaagttcgagggcgacaccctggtg360
aaccggatcgagctgaagggcatcgacttcaaagaggatggcaacatcctgggccacaag420
atggaatacaactacaactcccacaacgtgtacatcatggccgacaagcctaagaacggc480
atcaaggtgaacttcaagatcagacacaacatcaaggacggctctgtgcagctggccgac540
cactaccagcagaacacccccatcggcgacggccctgtgctgctgcctgataaccactat600
ctgtctacacagtccgctctgtccaaagatcctaacgagaagcgggaccacatgatcctg660
ctggagttcgtgaccgccgctggcatcacccatggcatggacgagctgtacaaggactac720
aaggacgatgacgacaag738
<210>8
<211>1290
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>合成构建体
<400>8
atgtccctgcactttctgtactactgctctgagcctaccctggacgtgaagatcgccttc60
tgtcagggcttcgataagcaggtggacgtctcctatatcgctaagcactacaacatgagc120
aaatccaaggtggacaaccagttctactctgtcgaggtgggcgactctaccttcaccgtg180
ctgaagagataccagaacctgaaacccatcggctccggcgctcagggcatcgtgtgcgcc240
gcttacgacgccgtgctggatagaaacgtggccatcaagaagctgagccggcctttccag300
aaccagacacacgctaagcgggcctacagagagctggtcctgatgaagtgcgtgaaccac360
aagaacatcatctccctgctgaatgtgttcacccctcagaaaaccctggaagagttccag420
gatgtgtacctggtgatggaactgatggacgccaacctgtgccaggtgatccagatggaa480
ctggaccacgagcggatgtcctacctgctgtaccagatgctgtgtggcatcaagcacttg540
catagcgctggcatcatccacagagatctgaaaccttctaacatcgtggtgaagtccgac600
tgcaccctgaagatcctggacttcggcctggccagaaccgctggcacctctttcatgatg660
acaccctacgtggtgaccagatactaccgggcccctgaagtgatcctgggcatgggctac720
aaggagaacgtggacatctggtccgtgggatgcatcatgggcgagatggtcagacacaag780
atcctgttccccggaagagattacatcgaccagtggaacaaggtgatcgagcagctgggc840
accccttgtcctgagttcatgaagaaactgcagcctaccgtgcggaactacgtggaaaac900
cggcctaagtacgccggcctgacctttccaaagctgttccctgactctctgttccccgct960
gacagcgagcacaacaagctgaaagcctctcaggccagagatctgctgtccaagatgctg1020
gtgatcgaccctgctaagagaatctccgtggacgatgccctgcagcacccctacatcaac1080
gtgtggtacgaccctgctgaggtggaagcccctccacctcagatctacgacaagcagctg1140
gacgaaagagagcacaccatcgaggagtggaaggagctgatctataaagaagtgatgaac1200
tccgaggaaaagaccaagaacggcgtggtcaagggccagccttccccctctgctcaggtg1260
cagcaagactacaaggacgatgatgacaag1290
- 还没有人留言评论。精彩留言会获得点赞!