一种肿瘤外显子测序数据分析方法与流程
本发明涉及生物信息分析领域,特别是涉及一种肿瘤外显子测序数据分析方法。
背景技术:
外显子虽然只占约1%人类基因组大小,但多数疾病都是由编码基因的低频或罕见突变引起,同时外显子测序容易实现>100x的深度测序,是一种高效的发现人类疾病信息的研究手段。目前,一般都是通过探针杂交富集外显子,然后利用二代测序获得其序列信息,最后结合公共数据库的外显子数据,解释变异与疾病之间的关联。
肿瘤发病机制十分复杂,传统的病理组织学分型并不能完全满足治疗需要,外显子测序技术由于相于全基因组测序和基因分型芯片,具有极佳的检测功效和性价比,可以从基因水平对肿瘤进行精确的研究,在肿瘤分子分型中的应用有助于对肿瘤的认识、肿瘤患者治疗和预后判断等。
基于以上所述,对肿瘤进行外显子测序,而后进行生物信息分析有这重要的生物学意义和临床价值。但目前外显子测序数据分析方法杂多且单一,未形成统一、全面丰富的方法。相对于国内发明专利201610316928.x(肿瘤外显子组测序分析系统及方法),本发明公开的方法,具有更全面的分析内容包括对体细胞突变更全面系统的分析、比对数据库更全面的优点。
技术实现要素:
本发明的目的在于克服上述现有技术的不足之处而提供一种肿瘤外显子测序数据分析方法。
为实现上述目的,本发明采取的技术方案为:一种肿瘤外显子测序数据分析方法,具体步骤如下:
s1:体细胞变异检测,并对somaticsnv/indel进行整体统计和注释,以及somaticcnv分析;
s2:突变全局展示,针对覆盖深度、somaticsnv、indel以及cnv,进行全面分析;
s3:对突变特异性的频谱和特征进行分析;
s4:筛选出肿瘤样本中的已知驱动基因,并构建背景突变率模型,预测和寻找可能的驱动突变;
s5:寻找肿瘤高频突变,并采用卷积检验方法对各个高频突变类型进行统计检验。
s6:分析高频somaticcnv在样本中的分布热图、高频cnv染色体分布峰图、高频cnv染色体分布峰图。
s7:计算基因组肿瘤样本的纯度和倍性;
s8:进行肿瘤异质性、克隆以及进化树分析,展示克隆突变,进行pca主成分分析。
优选地,所述对somaticsnv/indel进行整体统计使用mutect软件,使用mutect软件来寻找somaticsnv和indel位点;所述对somaticsnv/indel进行注释使用annovar软件,利用annovar软件将所检测到snp以及indel等基因组变异与外部数据库进行注释分析,以确定与人类疾病高度相关变异的基因组位置、变异频率、蛋白有害性、基因型杂合性以及所在的功能通路等信息;所述somaticcnv分析是使用varscan2对tumor及normal成对样本检测somaticcnv,获得somaticcnv集合。
优选地,外部数据库包括hapmap、cosmic70、esp6500、exac;优选地,可采用sift、polyphen突变危害性软件进行分析。
优选地,所述体细胞变异全局图共分为6圈,第一圈为染色体的外框、第二圈为肿瘤样本的测序覆盖度图、第三圈为正常样本的测序覆盖度图、第四圈为圆点表示snpindel的密度、第五圈为cnv结果展示拷贝数增加、第六圈为cnv结果展示拷贝数缺失。
优选地,所述对突变特异性的特征进行分析采用以下方法实现:
1)以6种碱基突变类型为中心,各取5’和3’各一个碱基形成多种组合,该组合有96种类型;
2)以这96种突变类型为基础,确定肿瘤基因组的突变特征;
3)通过nmf算法对肿瘤样本发生的96种突变类型进行聚类,得到对应的突变特征,统计各突变特征在每个样本中的分布情况;
4)计算新发现的突变特征与cosmic中已知的突变特征之间的相关性,确定这些新特征的生物学意义。
优选地,所述使用的已知驱动基因的数据库包括但不限于cgc、mdg125、smg127、cdg291。
优选地,所述各个高频突变类型进行统计检验包括高频基因统计、突变基因go和kegg富集分析。
优选地,所述肿瘤异质性、克隆分析通过以下方法实现:利用软件工具基于样本的snv数据,计算每个样本中的克隆数目情况,同时统计每个肿瘤细胞的突变占比,将肿瘤组织内遗传突变信息相似的肿瘤细胞进行聚类,按以下参数进行cluster过滤:
1)只保留包含突变数>=5的cluster;
2)只保留meancellularprevalence值大于0.05的cluster(大于5%的细胞比例)。
优选地,所述进化树分析通过以下方法实现:获得所有肿瘤样本的突变数据后,采用mega7的“branchandbound”模式绘制肿瘤进化树。
优选地,所述展示克隆突变是使用热图展示每个克隆的聚类情况,每个分支中包含基因列表;所述主成分分析是采用pca进行主成分分析。
与现有技术相比本发明具有以下的有益效果:
(1)分析内容更全面:在市面上常规分析的基础上,增加更全面、系统的体细胞突变分析,对研究肿瘤的形成、耐药性、复发性等各项复杂机制有重要的意义;特别是其中,突变特异性、高频somaticcnv分析、肿瘤纯度和倍性、肿瘤异质性及进化分析,与研究肿瘤的复发、转移、治疗和预后有密切相关性。
(2)比对数据库更全面:突变检测是外显子检测的核心步骤,数据库质量的好坏能够保证突变检测结果的完整性、可靠性。本发明采用多个国际认可的外显子数据库进行变异注释,能够筛选低频、新生突变;同时,使用sift、polyphen等主流突变危害性软件对突变危害进行评估,保证样本中变异信息的全面挖掘。
附图说明
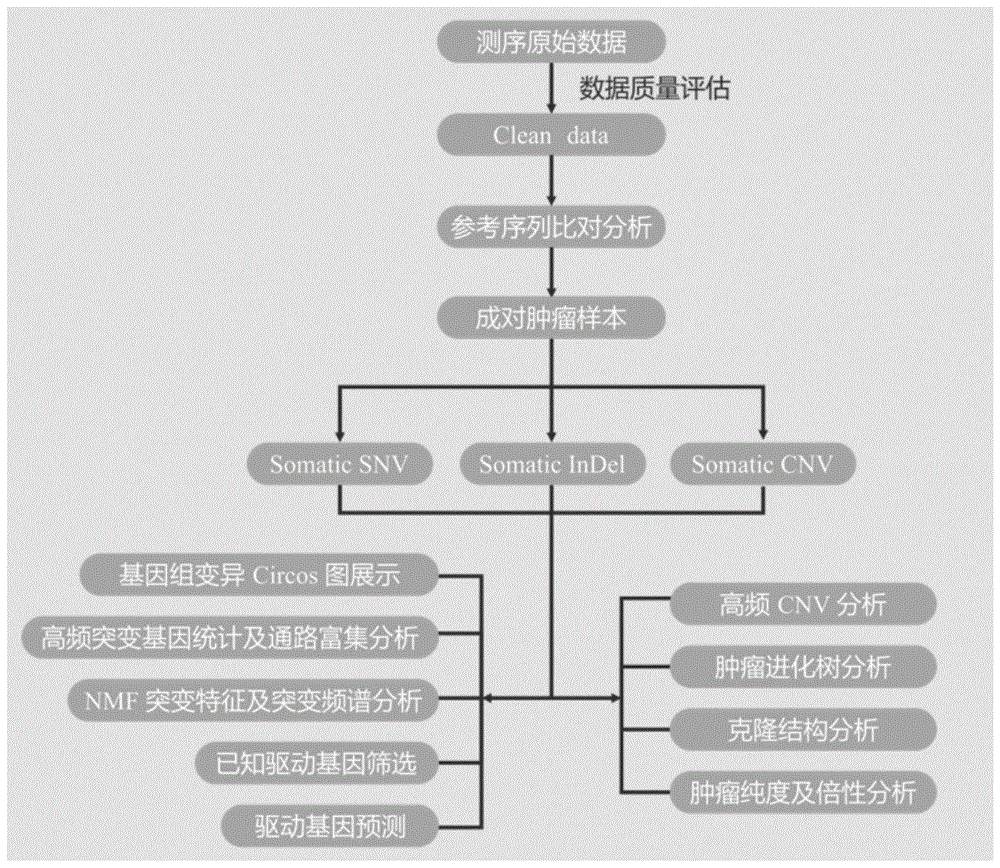
图1是本发明实施例提供的一种肿瘤外显子测序数据分析方法的流程图。
图2是基本发明的全局展示图。
图3是本发明实施例中的癌症样本突变特征图;左侧纵坐标从上到下为染色体,右侧为显著候选驱动基因,下横坐标为gistic计算的每个峰的q值对应的-log值,绿色线为阈值线(q=0.25)。
图4是本发明实施例中的高频cnv(扩增,gain)染色体分布峰图;左侧纵坐标从上到下为染色体,右侧为显著候选驱动基因,下横坐标为gistic计算的每个峰的q值对应的-log值,绿色线为阈值线(q=0.25)。
图5是本发明实施例中的样本肿瘤细胞突变比例展示图。
图6是本发明实施例中的肿瘤进化树示意图。
具体实施方式
为了更加简洁明了的展示本发明的技术方案、目的和优点,下面结合具体实施例及其附图对本发明做进一步的详细描述。
本发明提供一种肿瘤外显子测序数据分析方法,具体步骤如下:
s1:体细胞变异检测
s1.1somaticsnv/indel整体统计
体细胞单核苷酸变异(snv)、插入缺失(indel)是肿瘤基因组的重点研究对象,通过对肿瘤样本somaticsnv与indel信息的挖掘,可以进一步了解其形成机制、肿瘤异质性等信息。本发明使用mutect软件来寻找somaticsnv和indel位点。
s1.2somaticsnv/indel注释
人类基因组存在多个变异位点,当中有的变异极为常见,并且已被证明不影响人类正常生存,然而有的变异则与人类疾病高度相关。为了寻找更有价值的疾病变异,本发明利用annovar软件将所检测到snp以及indel等基因组变异与外部数据库进行注释分析,以确定变异的基因组位置、变异频率、蛋白有害性、基因型杂合性以及所在的功能通路等信息。
本发明中的外部数据库包括hapmap、cosmic70、esp6500、exac等;优选的可采用sift、polyphen等突变危害性软件进行分析。
hapmap:人类基因组单体型数据库,用于发现与人类健康、疾病以及对药物和环境因子的个体反应差异相关的基因;
cosmic70:癌症体细胞突变数据库(catalogueofsomaticmutationsincancer),观察到的次数,以及观察到的癌组织,包括非编码突变;
esp6500:国家心肺和血液研究所外显子组测序计划的外显子测序数据库,包含snp变异、indel变异和y染色体上的变异的所有个体中,突变碱基的等位基因频率;
exac:整合了60706个无亲缘关系个体的数据,这些个体来源于大量疾病研究和群体遗传学研究,能够用做严重疾病研究的参考数据库;
sift:sortingintolerantfromtolerant,是用来预测氨基酸改变是否影响功能,分值越小越可能“有害”;
polyphen:基于humandiv数据库和humanvar数据库用来预测非同义突变造成的氨基酸改变是否影响功能,数值越大越“有害”。
s1.3somaticcnv分析
拷贝数变异(copynumbervariation,cnv)表现为基因组片段的拷贝数增加或者减少,是基因组结构变异(structuralvariation,sv)的重要组成部分。癌症的发生除了受基因组的碱基点突变影响外,还受cnv和sv等大型突变事件影响。本发明主要使用varscan2对tumor及normal成对样本检测somaticcnv,获得somaticcnv集合。优选的,如果原始somaticcnv结果较多,可通过log值以及长度进行后续筛选。
s2突变全局展示
针对覆盖深度、somaticsnv、indel以及cnv进行分析,本发明中使用circos工具绘制肿瘤样本所发现的体细胞变异全局图。体细胞变异全局图共分为6圈,第一圈为染色体的外框、第二圈为肿瘤样本的测序覆盖度图、第三圈为正常样本的测序覆盖度图、第四圈为圆点表示snpindel的密度、第五圈为cnv结果展示拷贝数增加、第六圈为cnv结果展示拷贝数缺失(图2)。
s3突变特异性
对体细胞snv的变异进行了多个角度的分析,包括突变频谱(mutationspectrum)和突变特征(mutationsignature)。从这些结果中,可以清晰地了解到肿瘤发生在点突变水平上的特征。通过突变频谱分析可以得知各个肿瘤样本各种类型突变(如c>a/g>t)的数量及样本是否有某种类型突变的偏好性;通过分析体细胞突变频谱以及突变特征,可以研究不同癌种的体细胞点突变特点。
s3.1突变频谱
通过突变频谱分析可以得知各个肿瘤样本各种类型突变(如c:g>t:a)的数量及样本是否有某种类型突变的偏好性。通过分析体细胞突变率以及突变频谱等,可以研究不同癌种的体细胞突变特征。
s3.2突变特征
以6种碱基突变类型为中心,各取5’和3’各一个碱基形成多种组合,这些组合类型有96种结果(5’碱基4种类型,中心碱基6种类型,3’碱基4种类型,4x6x4=96)。以这96种突变类型为基础,通过不同组合,确定肿瘤基因组的突变特征(mutationsignature)。通过nmf(nonnegativematrixfactorization)算法对肿瘤样本发生的96种突变类型进行聚类,得到对应的突变特征。同时,统计各突变特征在每个样本中的分布情况。最后,通过nmf算法,计算新发现突变特征与cosmic中已知的30个突变特征(http://cancer.sanger.ac.uk/cosmic/signatures)之间相关性,确定这些新特征的生物学意义(图3)。
s4驱动基因分析
肿瘤组织一般存在高达数千个基因突变,但只有其中很少一部分会真正导致疾病,这一类能够诱发癌症的突变基因就称为驱动基因(cancerdrivergenes)。驱动基因主导了肿瘤的发生,有利于肿瘤的生长扩散,对癌症形成起到主要的作用。
s4.1已知驱动基因
将体细胞变异信息之后,通过在已报道驱动基因数据进行查找,筛选出该肿瘤样本中已知的驱动基因。
优选的,所述已报到的驱动基因数据包括但不限于以下4项:
(1)cgc:cancergenecensus(http://cancer.sanger.ac.uk/census)列表里的所列出的699个驱动基因;
(2)mdg125:已发表文章所总结的125个突变驱动基因(mut-drivergene),分为tsg和oncogene两种;
(3)smg127:已发表的pan-cancer数据找出的127个显著突变基因,数值表示在pan-caner的12类肿瘤的所有样本里的突变频率;
(4)cdg291:已发表的高可信度癌症驱动基因(high-confidencecancerdrivergenes)数。该数据包含12种癌症的3205例样本,通过oncodriveclust等多个软件找找出了291非常可信的癌症驱动基因以及144个其他candidatedrivers。
s4.2驱动基因预测
肿瘤细胞往往存在大量突变,但这些突变更多突变位置是随机、无偏好性的,均匀分布于整个基因上的同义突变。只有受选择的功能获得性突变(gain-of-functionmutation)才有可能使癌细胞获得了生长优势,而具有此突变的基因则有可能形成真正的驱动基因。功能获得性突变往往在蛋白的某些特定区域聚集形成突变簇,因此本发明利用oncodriveclust软件全面地考虑到驱动突变在位点分布上具有形成突变簇的偏好性,利用同义突变无偏分布的特点构建背景突变率模型,寻找可能的驱动突变。
s5肿瘤高频突变
高频突变基因(significantlymutatedgenes,smg)是指突变频率显著高于背景突变频率的基因,一般综合考虑了体细胞snv和indel等变异。本发明利用music软件寻找在肿瘤样本中(相比于对照样本)突变频率更高的基因,并对各个突变类型进行统计检验(smgtest),检验方法为卷积检验(convolutiontest,简称ct)。包括高频基因统计、突变基因go和kegg富集分析。
s6高频somaticcnv分析
拷贝数变异(copynumbervariation,cnv)表现为基因组片段的拷贝数增加或者减少,是基因组结构变异(structuralvariation,sv)的重要组成部分。癌症的发生除了受基因组的碱基点突变影响外,还受cnv和sv等大型突变事件影响。本发明的分析包括但不限于高频cnv在样本中的分布热图、高频cnv(扩增,gain)染色体分布峰图(图4)、高频cnv(缺失,loss)染色体分布峰图。
s7肿瘤纯度和倍性
当dna是从癌细胞和正常细胞的混合组织中提取和测序的时候,癌细胞的比例和癌细胞中的基因组的拷贝数信息是不能直接得到的。本发明利用absolute软件依据肿瘤样本基因组的拷贝数和体细胞突变点的等位基因频率,可以计算出肿瘤样本的纯度(purity)和倍性(ploidy)。
s8肿瘤异质性及进化分析
s8.1肿瘤异质性和克隆分析
肿瘤异质性主要是肿瘤在生长过程中,经过多次分裂增殖,其子细胞呈现出基因方面的改变,从而使肿瘤细胞的生长速度、侵袭能力、对药物的敏感性、预后等各方面产生差异。对肿瘤组织进行克隆结构分析正是基于肿瘤异质性,通过信息分析手段,将肿瘤组织内遗传突变信息相似的肿瘤细胞进行聚类。本发明利用pyclone软件,基于样本的snv数据,计算每个样本中的克隆数目情况。同时统计每个肿瘤细胞的突变占比(图5)。根据结果,可选择以下参数进行cluster过滤:
1.只保留包含突变数>=5的cluster
2.只保留meancellularprevalence值大于0.05的cluster(大于5%的细胞比例)
s8.2克隆进化树分析
获得所有肿瘤样本的突变数据后,利用mega7的“branchandbound“模式绘制肿瘤进化树(maximum-parsimonytree),以了解肿瘤内部异质性(或者原发灶与复发灶等)的来源情况(图6)。
s8.3克隆突变展示
基于进化树的聚类结果,利用热图展示每个克隆的聚类情况。同时为方便客户了解克隆的进化历程,本发明提供每个分支中所包含的基因列表。通过查找列表中的基因,发现潜在的进化驱动基因。
s8.4主成分分析
基于所有肿瘤样本的突变数据(vaf),进行pca(principalcomponentanalysis)主成分分析,以了解同一病人来源的肿瘤样本聚类情况。
以上所述实施例仅表达了本发明的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。因此,本发明专利的保护范围应以所附权利要求为准。
- 还没有人留言评论。精彩留言会获得点赞!