用于早期诊断心脑血管疾病的甲基化标志物的制作方法
1.本发明涉及医学领域,特别涉及用于早期诊断心脑血管疾病的甲基化标志物。
背景技术:
2.心脑血管疾病是心脏血管和脑血管疾病的统称,泛指由于高脂血症、血液黏稠、动脉粥样硬化、高血压等所导致的心脏、大脑及全身组织发生的缺血性或出血性疾病。心脑血管疾病是一种严重威胁人类,特别是50岁以上中老年人健康的常见病,具有高患病率、高致残率和高死亡率的特点。目前,全世界每年死于心脑血管疾病的人数高达1500万人。我国心脑血管疾病的发病率和死亡率总体呈上升趋势,每年死于心脑血管疾病的人数有350万,占各种死亡原因的首位。
3.冠心病是指由于冠状动脉粥样硬化使管腔狭窄、痉挛或阻塞,导致心肌缺血、缺氧或坏死而引发的心脏病,统称为冠状动脉性心脏病或冠状动脉疾病。根据病变部位、范围和程度等临床特征将冠心病分为5种类型:(1)隐匿型或无症状性心肌缺血:无症状,但在静息、动态或负荷心电图下显示心肌缺血改变,或放射性核素心肌显像提示心肌灌注不足,无组织形态改变;(2)心绞痛:由心肌供血不足引起发作性胸骨后疼痛;(3)心肌梗死:缺血症状严重,因冠状动脉闭塞导致心肌急性缺血坏死;(4)缺血性心肌病:长期慢性心肌缺血或坏死导致心肌纤维化,表现为心脏增大、心力衰竭和心律失常;(5)猝死:突发心搏骤停引起的死亡,多为缺血心肌局部发生电生理紊乱引起的严重心律失常所致。近10余年冠心病发病率在我国呈明显上升趋势,冠心病发病率一般以心肌梗死发病率为代表。目前冠心病主要的诊断方法如下:(1)临床特征:一般结合检查者的病史和身体检查状况,用于初步诊断,但特异性很低;(2)影像学方法:心电图、超声心动图和冠状动脉血管造影,但往往受医生经验和仪器设备的影响;(3)目前最常用的冠心病标志物要有以下几类:心肌损伤标志物、炎症因子及黏附分子和细胞因子标志物、血浆脂蛋白及载脂蛋白标志物和凝血相关蛋白标志物等。因为某个标志物只反映疾病某个疾病机理导致这些标志物在临床并意义未得到广泛认同。
4.脑卒中俗称中风,是一种急性脑血管疾病,包括缺血性脑卒中和出血性脑卒中。缺血性脑卒中占所有脑卒中的60%~70%,主要是由于动脉粥样硬化引起脑部血管狭窄或者闭塞,从而引起脑组织缺血、缺氧,进一步导致局限性脑组织的缺血性坏死或软化,患者多在40岁以上,男性较女性多,严重者可引起死亡。出血性脑卒中分为脑出血和蛛网膜下腔出血,主要是由于长期的高血压、动脉瘤或脑血管先天比较脆弱等原因,导致脑血管破裂出血,这些血液在脑中压迫正常的脑组织,使大脑无法发挥正常的功能,也就是常说的“脑溢血”,且死亡率比较高。目前常使用影像学方法来进行脑卒中的诊断,例如ct和核磁共振检查,ct对于出血性脑卒中的灵敏度较高,但对于缺血性脑卒中的灵敏度只有16%,由于辐射的原因不宜频繁使用;核磁共振检查对缺血性脑卒中的灵敏度高于ct且没有辐射影响,但其缺点是较低的可行性、实用性和可及性(设备以及训练有素的人员)。
5.冠心病和脑卒中都属于心脑血管疾病。大多数心血管疾病是可以预防和治疗的,
一般通过普及知识提高意识、避免外源性刺激因素和合理膳食适度运动来进行预防,其治疗效果很大程度上依赖于早期诊断及相应的干预措施。目前,临床上关于冠心病和脑卒中疾病诊断标志物的灵敏度和特异性很有限,尤其是缺乏早期诊断的标志物,因此更为敏感、特异的早期分子标记物亟待发掘。dna甲基化是基因上重要的一种化学修饰,影响着基因转录的调控过程和细胞核结构。本研究通过飞行时间质谱dna甲基化分析技术在多组样品中进行分析,发现了心脑血管疾病和健康对照组的血液dna甲基化存在显著差异。因此,血液异常的dna甲基化信号有可能为心脑血管疾病体外早期诊断带来突破。此外,血液容易收集,dna甲基化常温较稳定的特点也让其在临床应用中独具优势。因此,探索和开发适用于临床检测需要的灵敏和特异血液dna甲基化诊断技术对提高心脑血管疾病早期诊疗效果和降低死亡率均有重要的科学意义和临床应用价值。
技术实现要素:
6.本发明的目的是提供红褐蛋白环指1(mahogunin ring finger-1,mgrn1)基因甲基化水平在辅助诊断心脑血管疾病中的应用。
7.第一方面,本发明要求保护甲基化mgrn1基因作为标志物在制备产品中的应用;所述产品的用途为如下中的至少一种:
8.(1)辅助诊断心脑血管疾病或在临床症状之前预警心脑血管疾病;
9.(2)辅助诊断冠心病或在临床症状之前预警冠心病;
10.(3)辅助诊断脑卒中或在临床症状之前预警脑卒中;
11.(4)辅助区分冠心病和脑卒中;
12.(5)辅助诊断不同临床特征的冠心病或在临床症状之前预警不同临床特征的冠心病;
13.(6)辅助诊断不同临床特征的脑卒中或在临床症状之前预警不同临床特征的脑卒中。
14.进一步地,(1)中所述辅助诊断心脑血管疾病具体可体现为如下中的至少一种:辅助区分冠心病患者和健康对照、脑卒中患者和健康对照。其中,所述健康对照可理解为现在及曾经均没有患过心脑血管疾病和癌症且血常规指标都在参考范围内。
15.在本发明的具体实施方式中,(5)中所述辅助诊断不同临床特征的冠心病或在临床症状之前预警不同临床特征的冠心病具体体现为如下中的至少一种:可辅助区分隐匿型或无症状性心肌缺血患者和健康对照、可辅助区分心绞痛患者和健康对照、可辅助区分心肌梗死患者和健康对照、可辅助区分缺血性心肌病患者和健康对照、可辅助区分猝死患者和健康对照。其中,所述健康对照可理解为现在及曾经均没有患过心脑血管疾病和癌症且血常规指标都在参考范围内。
16.在本发明的具体实施方式中,(6)中所述辅助区分不同临床特征的脑卒中或在临床症状之前预警不同临床特征的脑卒中具体体现为如下中的至少一种:可辅助区分缺血性脑卒中和健康对照、可辅助区分出血性脑卒中和健康对照。所述健康对照可理解为现在和曾经均没有患过心脑血管疾病和癌症且血常规指标都在参考范围内。
17.在上述(1)-(6)中,所述心脑血管疾病可为能够引起机体内mgrn1基因甲基化水平改变的疾病,如冠心病和脑卒中等。所述在临床症状之前为早于临床发病时间2年内或者1
年内。
18.第二方面,本发明要求保护用于检测mgrn1基因甲基化水平的物质在制备产品中的应用。所述产品的用途可为前文(1)-(6)中的至少一种。
19.第三方面,本发明要求保护用于检测mgrn1基因甲基化水平的物质和储存有数学模型建立方法和/或使用方法的介质在制备产品中的应用。所述产品的用途可为前文(1)-(6)中的至少一种。
20.所述数学模型可按照包括如下步骤的方法获得:
21.(a1)分别检测n1个a类型样本和n2个b类型样本的mgrn1基因甲基化水平(训练集);
22.(a2)取步骤(a1)获得的所有样本的mgrn1基因甲基化水平数据,按照a类型和b类型的分类方式,通过二分类逻辑回归法建立数学模型,确定分类判定的阈值。
23.其中,(a1)中的n1和n2均可为50以上的正整数。
24.所述数学模型的使用方法包括如下步骤:
25.(b1)检测待测样本的mgrn1基因甲基化水平;
26.(b2)将步骤(b1)获得的所述待测样本的mgrn1基因甲基化水平数据代入所述数学模型,得到检测指数;然后比较检测指数和阈值的大小,根据比较结果确定所述待测样本的类型是a类型还是b类型。
27.在本发明的具体实施方式中,所述阈值设为0.5。大于0.5归为一类,小于0.5归为另外一类,等于0.5作为不确定的灰区。其中a类型和b类型为相对应的两分类,二分类的分组,哪一组是a类型,哪一组是b类型,要根据具体的数学模型来确定,无需约定。
28.在实际应用中,所述阈值也可根据最大约登指数确定(具体可为最大约登指数对应的数值)。大于阈值归为一类,小于阈值归为另外一类,等于阈值作为不确定的灰区。其中a类型和b类型为相对应的两分类,二分类的分组,哪一组是a类型,哪一组是b类型,要根据具体的数学模型来确定,无需约定。
29.所述a类型样本和所述b类型样本可为如下中的任一种:
30.(c1)未来2年内发生冠心病的潜在患者和健康对照;
31.(c2)未来2年内发生脑卒中的潜在患者和健康对照;
32.(c3)未来2年内发生冠心病的潜在患者和未来2年内发生脑卒中的潜在患者;
33.(c4)未来2年内发生不同临床特征冠心病的潜在患者和健康对照;
34.(c5)未来2年内发生不同临床特征脑卒中的潜在患者和健康对照;
35.(c6)未来1年内发生冠心病的潜在患者和健康对照;
36.(c7)未来1年内发生脑卒中的潜在患者和健康对照;
37.(c8)未来1年内发生冠心病的潜在患者和未来1年内发生脑卒中的潜在患者;
38.(c9)未来1年内发生不同临床特征冠心病的潜在患者和健康对照;
39.(c10)未来1年内发生不同临床特征脑卒中的潜在患者和健康对照。
40.其中,所述健康对照可理解为现在和曾经均没有患过心脑血管疾病和癌症且血常规指标都在参考范围内。
41.第四方面,本发明要求保护前文第三方面中所述的“储存有数学模型建立方法和/或使用方法的介质”在制备产品中的应用。所述产品的用途可为前文(1)-(6)中的至少一
种。
42.第五方面,本发明要求保护一种试剂盒。
43.本发明所要求保护的试剂盒包括用于检测mgrn1基因甲基化水平的物质。所述试剂盒的用途可为前文(1)-(6)中的至少一种。
44.进一步地,所述试剂盒中还可含有前文第三方面或第四方面中所述的“储存有数学模型建立方法和/或使用方法的介质”。
45.第六方面,本发明要求保护一种系统。
46.本发明所要求保护的系统,包括:
47.(d1)用于检测mgrn1基因甲基化水平的试剂和/或仪器;
48.(d2)装置,所述装置包括单元x和单元y;
49.所述单元x用于建立数学模型,包括数据采集模块、数据分析处理模块和模型输出模块;
50.所述数据采集模块用于采集(d1)检测得到的n1个a类型样本和n2个b类型样本的mgrn1基因甲基化水平数据;
51.其中,n1和n2均可为50以上正整数。
52.所述数据分析处理模块能够基于所述数据采集模块采集的n1个a类型样本和n2个b类型样本的mgrn1基因甲基化水平数据,按照a类型和b类型的分类方式,通过二分类逻辑回归法建立数学模型,确定分类判定的阈值;
53.所述模型输出模块用于输出所述数据分析处理模块建立的数学模型;
54.所述单元y用于确定待测样本类型,包括数据输入模块、数据运算模块、数据比较模块和结论输出模块;
55.所述数据输入模块用于输入(d1)检测得到的待测者的mgrn1基因甲基化水平数据;
56.所述数据运算模块用于将所述待测者的mgrn1基因甲基化水平数据代入所述数学模型,计算得到检测指数;
57.所述数据比较模块用于将所述检测指数与阈值进行比较;
58.所述结论输出模块用于根据所述数据比较模块的比较结果输出所述待测样本的类型是a类型还是b类型的结论;
59.所述a类型样本和所述b类型样本为如下中的任一种:
60.(c1)未来2年内发生冠心病的潜在患者和健康对照;
61.(c2)未来2年内发生脑卒中的潜在患者和健康对照;
62.(c3)未来2年内发生冠心病的潜在患者和未来2年内发生脑卒中的潜在患者;
63.(c4)未来2年内发生不同临床特征冠心病的潜在患者和健康对照;
64.(c5)未来2年内发生不同临床特征脑卒中的潜在患者和健康对照;
65.(c6)未来1年内发生冠心病的潜在患者和健康对照;
66.(c7)未来1年内发生脑卒中的潜在患者和健康对照;
67.(c8)未来1年内发生冠心病的潜在患者和未来1年内发生脑卒中的潜在患者;
68.(c9)未来1年内发生不同临床特征冠心病的潜在患者和健康对照;
69.(c10)未来1年内发生不同临床特征脑卒中的潜在患者和健康对照。
70.其中,所述健康对照可理解为现在和曾经均没有患过心脑血管疾病和癌症且血常规指标都在参考范围内。
71.在本发明的具体实施方式中,所述阈值设为0.5。大于0.5归为一类,小于0.5归为另外一类,等于0.5作为不确定的灰区。其中a类型和b类型为相对应的两分类,二分类的分组,哪一组是a类型,哪一组是b类型,要根据具体的数学模型来确定,无需约定。
72.在实际应用中,所述阈值也可根据最大约登指数确定(具体可为最大约登指数对应的数值)。大于阈值归为一类,小于阈值归为另外一类,等于阈值作为不确定的灰区。其中a类型和b类型为相对应的两分类,二分类的分组,哪一组是a类型,哪一组是b类型,要根据具体的数学模型来确定,无需约定。
73.在前文各方面中,所述mgrn1基因甲基化水平可为mgrn1基因中如下(e1)-(e4)所示片段中全部或部分cpg位点的甲基化水平。所述甲基化mgrn1基因可为mgrn1基因中如下(e1)-(e4)所示片段中全部或部分cpg位点甲基化。
74.(e1)seq id no.1所示的dna片段或与其具有80%以上同一性的dna片段;
75.(e2)seq id no.2所示的dna片段或与其具有80%以上同一性的dna片段;
76.(e3)seq id no.3所示的dna片段或与其具有80%以上同一性的dna片段;
77.(e4)seq id no.4所示的dna片段或与其具有80%以上同一性的dna片段。
78.进一步地,所述“全部或部分cpg位点”可为seq id no.1所示的dna片段中所有cpg位点(见表1)、seq id no.2所示的dna片段中所有cpg位点(见表2)、seq id no.3所示的dna片段中所有cpg位点(见表3)、seq id no.4所示的dna片段中所有cpg位点(见表4)。
79.或,所述“全部或部分cpg位点”可为所述seq id no.3所示的dna片段中的全部(见表3)和所述seq id no.1所示的dna片段中的全部(见表1)。
80.或,所述“全部或部分cpg位点”可为所述seq id no.3所示的dna片段中的全部(见表3)和所述seq id no.2所示的dna片段中的全部(见表2)。
81.或,所述“全部或部分cpg位点”可为所述seq id no.3所示的dna片段中的全部(见表3)和所述seq id no.4所示的dna片段中的全部(见表4)。
82.或,所述“全部或部分cpg位点”可为所述seq id no.3所示的dna片段中的全部或任意52个或任意51个或任意50个或任意49个或任意48个或任意47个或任意46个或任意45个或任意44个或任意43个或任意42个或任意41个或任意40个或任意39个或任意38个或任意37个或任意36个或任意35个或任意34个或任意33个或任意32个或任意31个或任意30个或任意29个或任意28个或任意27个或任意26个或任意25个或任意24个或任意23个或任意22个或任意21个或任意20个或任意19个或任意18个或任意17个或任意16个或任意15个或任意14个或任意13个或任意12个或任意11个或任意10个或任意9个或任意8个或任意7个或任意6个或任意5个或任意4个或任意3个或任意2个或任意1个。
83.或,所述“全部或部分cpg位点”可为seq id no.3所示的dna片段中如下二十五项所示cpg位点的全部或任意24项或任意23项或任意22项或任意21项或任意20项或任意19项或任意18项或任意17项或任意16项或任意15项或任意14项或任意13项或任意12项或任意11项或任意10项或任意9项或任意8项或任意7项或任意6项或任意5项或任意4项或任意3项或任意2项或任意1项:
84.(f1)seq id no.3所示的dna片段自5’端第389-390位所示cpg位点(mgrn1_c_17);
85.(f2)seq id no.3所示的dna片段自5’端第403-404位所示cpg位点(mgrn1_c_18);
86.(f3)seq id no.3所示的dna片段自5’端第424-425位所示cpg位点(mgrn1_c_19);
87.(f4)seq id no.3所示的dna片段自5’端第518-519位所示cpg位点(mgrn1_c_20);
88.(f5)seq id no.3所示的dna片段自5’端第524-525位所示cpg位点(mgrn1_c_21);
89.(f6)seq id no.3所示的dna片段自5’端第539-540位所示cpg位点(mgrn1_c_22);
90.(f7)seq id no.3所示的dna片段自5’端第546-547位所示cpg位点(mgrn1_c_23);
91.(f8)seq id no.3所示的dna片段自5’端第563-564位和565-566位和570-571位和572-573位所示cpg位点(mgrn1_c_24.25.26.27);
92.(f9)seq id no.3所示的dna片段自5’端第583-584位所示cpg位点(mgrn1_c_28);
93.(f10)seq id no.3所示的dna片段自5’端第591-592位所示cpg位点(mgrn1_c_29);
94.(f11)seq id no.3所示的dna片段自5’端第594-595位所示cpg位点(mgrn1_c_30);
95.(f12)seq id no.3所示的dna片段自5’端第603-604位所示cpg位点(mgrn1_c_31);
96.(f13)seq id no.3所示的dna片段自5’端第612-613位所示cpg位点(mgrn1_c_32);
97.(f14)seq id no.3所示的dna片段自5’端第648-649位所示cpg位点(mgrn1_c_33);
98.(f15)seq id no.3所示的dna片段自5’端第653-654位所示cpg位点(mgrn1_c_34);
99.(f16)seq id no.3所示的dna片段自5’端第662-663位和664-665位和669-670位所示cpg位点(mgrn1_c_35.36.37);
100.(f17)seq id no.3所示的dna片段自5’端第692-693位和695-696位所示cpg位点(mgrn1_c_38.39);
101.(f18)seq id no.3所示的dna片段自5’端第704-705位所示cpg位点(mgrn1_c_40);
102.(f19)seq id no.3所示的dna片段自5’端第713-714位和718-719位所示cpg位点(mgrn1_c_41.42);
103.(f20)seq id no.3所示的dna片段自5’端第724-725位所示cpg位点(mgrn1_c_43);
104.(f21)seq id no.3所示的dna片段自5’端第729-730位所示cpg位点(mgrn1_c_44);
105.(f22)seq id no.3所示的dna片段自5’端第741-742位所示cpg位点(mgrn1_c_45);
106.(f23)seq id no.3所示的dna片段自5’端第771-772位所示cpg位点(mgrn1_c_46);
107.(f24)seq id no.3所示的dna片段自5’端第794-795位所示cpg位点(mgrn1_c_47);
108.(f25)seq id no.3所示的dna片段自5’端第825-826位所示cpg位点(mgrn1_c_48)。
109.在本发明的具体实施方式中,有些相邻的甲基化位点在利用飞行时间质谱进行dna甲基化分析时由于几个cpg位点位于一个甲基化片段上,峰图无法区分(无法区分的位点在表6中有记载),因而在进行甲基化水平分析、以及构建和使用相关数学模型时将其按照一个甲基化位点进行处理。前文所述的(f8)、(f16)、(f17)和(f19)便是这种情况。
110.在上述各方面中,所述用于检测mgrn1基因甲基化水平的物质可包含(或为)用于扩增mgrn1基因全长或部分片段的引物组合。所述用于检测mgrn1基因甲基化水平的试剂可包含(或为)用于扩增mgrn1基因全长或部分片段的引物组合;所述用于检测mgrn1基因甲基化水平的仪器可为飞行时间质谱检测仪。当然所述用于检测mgrn1基因甲基化水平的试剂中还可包含进行飞行时间质谱所用的其他常规试剂。
111.进一步地,所述部分片段可为如下中至少一个片段:
112.(g1)seq id no.1所示的dna片段或其包含的dna片段;
113.(g2)seq id no.2所示的dna片段或其包含的dna片段;
114.(g3)seq id no.3所示的dna片段或其包含的dna片段;
115.(g4)seq id no.4所示的dna片段或其包含的dna片段;
116.(g5)与seq id no.1所示的dna片段或其包含的dna片段具有80%以上同一性的dna片段;
117.(g6)与seq id no.2所示的dna片段或其包含的dna片段具有80%以上同一性的dna片段;
118.(g7)与seq id no.3所示的dna片段或其包含的dna片段具有80%以上同一性的dna片段;
119.(g8)与seq id no.4所示的dna片段或其包含的dna片段具有80%以上同一性的dna片段。
120.在本发明中,所述引物组合具体可为引物对a和/或引物对b和/或引物对c和/或引物对d;
121.所述引物对a为引物a1和引物a2组成的引物对;所述引物a1具体可为seq id no.5或seq id no.5的第11-35位核苷酸所示的单链dna;所述引物a2具体可为seq id no.6或seq id no.6的第32-56位核苷酸所示的单链dna;
122.所述引物对b为引物b1和引物b2组成的引物对;所述引物b1具体可为seq id no.7或seq id no.7的第11-35位核苷酸所示的单链dna;所述引物b2具体可为seq id no.8或seq id no.8的第32-56位核苷酸所示的单链dna;
123.所述引物对c为引物c1和引物c2组成的引物对;所述引物c1具体可为seq id no.9或seq id no.9的第11-35位核苷酸所示的单链dna;所述引物c2具体可为seq id no.10或seq id no.10的第32-56位核苷酸所示的单链dna;
124.所述引物对d为引物d1和引物d2组成的引物对;所述引物d1具体可为seq id no.11或seq id no.11的第11-35位核苷酸所示的单链dna;所述引物d2具体可为seq id no.12或seq id no.12的第32-56位核苷酸所示的单链dna。
125.另外,本发明还要求保护一种区分待测样本为a类型样本还是b类型样本的方法。
该方法可包括如下步骤:
126.(a)可按照包括如下步骤的方法建立数学模型:
127.(a1)分别检测n1个a类型样本和n2个b类型样本的mgrn1基因甲基化水平(训练集);
128.(a2)取步骤(a1)获得的所有样本的mgrn1基因甲基化水平数据,按照a类型和b类型的分类方式,通过二分类逻辑回归法建立数学模型,确定分类判定的阈值。
129.其中,(a1)中的n1和n2均可为50以上的正整数。
130.(b)可按照包括如下步骤的方法确定所述待测样本为a类型样本还是b类型样本:
131.(b1)检测所述待测样本的mgrn1基因甲基化水平;
132.(b2)将步骤(b1)获得的所述待测样本的mgrn1基因甲基化水平数据代入所述数学模型,得到检测指数;然后比较检测指数和阈值的大小,根据比较结果确定所述待测样本的类型是a类型还是b类型。
133.在本发明的具体实施方式中,所述阈值设为0.5。大于0.5归为一类,小于0.5归为另外一类,等于0.5作为不确定的灰区。其中a类型和b类型为相对应的两分类,二分类的分组,哪一组是a类型,哪一组是b类型,要根据具体的数学模型来确定,无需约定。
134.在实际应用中,所述阈值也可根据最大约登指数确定(具体可为最大约登指数对应的数值)。大于阈值归为一类,小于阈值归为另外一类,等于阈值作为不确定的灰区。其中a类型和b类型为相对应的两分类,二分类的分组,哪一组是a类型,哪一组是b类型,要根据具体的数学模型来确定,无需约定。
135.所述a类型样本和所述b类型样本为中的任一种:
136.(c1)未来2年内发生冠心病的潜在患者和健康对照;
137.(c2)未来2年内发生脑卒中的潜在患者和健康对照;
138.(c3)未来2年内发生冠心病的潜在患者和未来2年内发生脑卒中的潜在患者;
139.(c4)未来2年内发生不同临床特征冠心病的潜在患者和健康对照;
140.(c5)未来2年内发生不同临床特征脑卒中的潜在患者和健康对照;
141.(c6)未来1年内发生冠心病的潜在患者和健康对照;
142.(c7)未来1年内发生脑卒中的潜在患者和健康对照;
143.(c8)未来1年内发生冠心病的潜在患者和未来1年内发生脑卒中的潜在患者;
144.(c9)未来1年内发生不同临床特征冠心病的潜在患者和健康对照;
145.(c10)未来1年内发生不同临床特征脑卒中的潜在患者和健康对照。
146.其中,所述健康对照可理解为现在和曾经均没有患过心脑血管疾病和癌症且血常规指标都在参考范围内。
147.在实际应用中,以上任一所述数学模型可能会根据dna甲基化的检测方法以及拟合方式不同有所改变,要根据具体的数学模型来确定,无需约定。
148.在本发明的实施例中,所述模型具体为log(y/(1-y))=b0+b1x1+b2x2+b3x3+
…
.+bnxn,其中y为因变量即将待测样品的一个或者多个甲基化位点的甲基化值代入模型以后得出的检测指数,b0为常量,x1~xn为自变量即为该待测样品的一个或者多个甲基化位点的甲基化值(每一个值为0-1之间的数值),b1~bn为模型赋予每一个位点甲基化值的权重。
149.在本发明的实施例中,所述模型的建立还可酌情加入年龄、性别、白细胞计数等已
知参数来提高判别效率。本发明的实施例中建立的二个具体模型用于辅助区分未来2年内发生心脑血管疾病的潜在患者(冠心病和脑卒中)和健康对照。所述模型一具体为:log(y/(1-y))=2.502+2.261*mgrn1_c_17-2.754*mgrn1_c_18+2.724*mgrn1_c_19+1.261*mgrn1_c_20-7.112*mgrn1_c_21+7.724*mgrn1_c_22+3.856*grn1_c_23+7.726*mgrn1_c_24.25.26.27+2.646*mgrn1_c_28+2.753*mgrn1_c_29+1.764*mgrn1_c_30-5.113*mgrn1_c_31+3.657*mgrn1_c_32-0.899*mgrn1_c_33+2.439*mgrn1_c_34-15.631*mgrn1_c_35.36.37-3.271*mgrn1_c_38.39-6.357*mgrn1_c_40+4.057*mgrn1_c_41.42-2.636*mgrn1_c_43-2.546*mgrn1_c_44+4.199*mgrn1_c_45+2.436*mgrn1_c_46-2.261*mgrn1_c_47-2.724*mgrn1_c_48-0.031*年龄+0.542*性别(男性赋值为1,女性赋值为0)+0.013*白细胞个数。所述模型一的阈值为0.5。通过模型计算的检测指数大于0.5的患者候选为未来2年内发生冠心病的潜在患者,小于0.5的患者候选为健康对照。所述模型二具体为:log(y/(1-y))=-0.538+4.261*mgrn1_c_17-3.754*mgrn1_c_18+3.547*mgrn1_c_19+0.483*mgrn1_c_20-2.849*mgrn1_c_21-1.190*mgrn1_c_22-12.243*grn1_c_23+23.270*mgrn1_c_24.25.26.27+3.248*mgrn1_c_28+3.754*mgrn1_c_29+0.248*mgrn1_c_30-14.153*mgrn1_c_31-0.452*mgrn1_c_32+0.273*mgrn1_c_33-9.080*mgrn1_c_34-27.488*mgrn1_c_35.36.37+11.935*mgrn1_c_38.39+14.335*mgrn1_c_40-12.863*mgrn1_c_41.42+3.261*mgrn1_c_43-3.546*mgrn1_c_44-2.249*mgrn1_c_45-3.248*mgrn1_c_46-3.261*mgrn1_c_47-4.261*mgrn1_c_48+0.010*年龄-0.053*性别(男性赋值为1,女性赋值为0)+0.123*白细胞个数。所述模型二的阈值为0.5。通过模型计算的检测指数大于0.5的患者候选为未来2年内发生脑卒中的潜在患者,小于0.5的患者候选为健康对照。所述模型一和所述模型二中,所述mgrn1_c_17为seq id no.3所示的dna片段自5’端第389-390位所示cpg位点的甲基化水平;所述mgrn1_c_18为seq id no.3所示的dna片段自5’端第403-404位所示cpg位点的甲基化水平;所述mgrn1_c_19为seq id no.3所示的dna片段自5’端第424-425位所示cpg位点的甲基化水平;所述mgrn1_c_20为seq id no.3所示的dna片段自5’端第518-519位所示cpg位点的甲基化水平;所述mgrn1_c_21为seq id no.3所示的dna片段自5’端第524-525位所示cpg位点的甲基化水平;所述mgrn1_c_22为seq id no.3所示的dna片段自5’端第539-540位所示cpg位点的甲基化水平;所述mgrn1_c_23为seq id no.3所示的dna片段自5’端第546-547位所示cpg位点的甲基化水平;所述mgrn1_c_24.25.26.27为seq id no.3所示的dna片段自5’端第563-564位和565-566位和570-571位和572-573位所示cpg位点的甲基化水平;所述mgrn1_c_28为seq id no.3所示的dna片段自5’端第583-584位所示cpg位点的甲基化水平;所述mgrn1_c_29为seq id no.3所示的dna片段自5’端第591-592位所示cpg位点的甲基化水平;所述mgrn1_c_30为seq id no.3所示的dna片段自5’端第594-595位所示cpg位点的甲基化水平;所述mgrn1_c_31为seq id no.3所示的dna片段自5’端第603-604位所示cpg位点的甲基化水平;所述mgrn1_c_32为seq id no.3所示的dna片段自5’端第612-613位所示cpg位点的甲基化水平;所述mgrn1_c_33为seq id no.3所示的dna片段自5’端第648-649位所示cpg位点的甲基化水平;所述mgrn1_c_34为seq id no.3所示的dna片段自5’端第653-654位所示cpg位点的甲基化水平;所述mgrn1_c_35.36.37为seq id no.3所示的dna片段自5’端第662-663位和664-665位和669-670位所示cpg位点的甲基化水平;所述mgrn1_c_38.39为seq id no.3所示的dna片段自5’端第692-693位和695-696位所示cpg位
点的甲基化水平;所述mgrn1_c_40为seq id no.3所示的dna片段自5’端第704-705位所示cpg位点的甲基化水平;所述mgrn1_c_41.42为seq id no.3所示的dna片段自5’端第713-714位和718-719位所示cpg位点的甲基化水平;所述mgrn1_c_43为seq id no.3所示的dna片段自5’端第724-725位所示cpg位点的甲基化水平;所述mgrn1_c_44为seq id no.3所示的dna片段自5’端第729-730位所示cpg位点的甲基化水平;所述mgrn1_c_45为seq id no.3所示的dna片段自5’端第741-742位所示cpg位点的甲基化水平;所述mgrn1_c_46为seq id no.3所示的dna片段自5’端第771-772位所示cpg位点的甲基化水平;所述mgrn1_c_47为seq id no.3所示的dna片段自5’端第794-795位所示cpg位点的甲基化水平;所述mgrn1_c_48为seq id no.3所示的dna片段自5’端第825-826位所示cpg位点的甲基化水平。
150.在上述各方面中,所述检测mgrn1基因甲基化水平为检测血液中mgrn1基因甲基化水平。
151.在上述各方面中,当所述a类型样本或所述b类型样本为(c4)和(c9)中不同临床特征的冠心病患者,所述a类型样本或所述b类型样本具体可为无症状性心肌缺血样本、心绞痛样本、心肌梗死患者样本、缺血性心肌病患者样本、猝死样本中的任意一种。
152.在上述各方面中,当所述a类型样本或所述b类型样本为(c5)和(c10)中不同临床特征的脑卒中患者,所述a类型样本或所述b类型样本具体可为缺血性脑卒中和出血性脑卒中样本中的任意一种。
153.以上任一所述mgrn1基因具体可包括genbank登录号:nm_015246.4(gi:1519311542),转录物变体1、genbank登录号:nm_001142289.2(gi:334883177),转录物变体2、genbank登录号:nm_001142290.2(gi:334883179),转录物变体3、genbank登录号:nm_001142291.2(gi:334883181),转录物变体4、genbank登录号:nr_102267.1(gi:456367266),转录物变体5。
154.本发明提供了冠心病血液中mgrn1基因的高甲基化和脑卒中血液中低甲基化现象。实验证明,以血液为样本就能够区分心脑血管疾病(冠心病和脑卒中)和健康对照、区分不同临床特征的冠心病患者和健康对照、区分不同临床特征的脑卒中患者和健康对照。本发明对于提高心脑血管疾病早期诊疗效果和降低死亡率均有重要的科学意义和临床应用价值。
附图说明
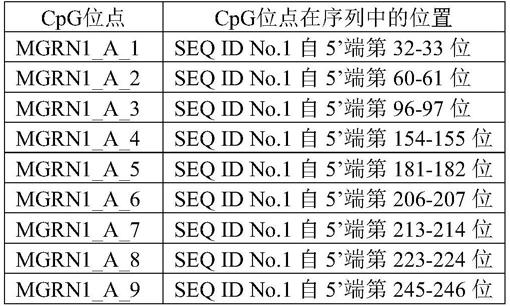
155.图1为数学模型示意图。
156.图2为未来2年内发生冠心病数学模型举例说明。
157.图3为未来2年内发生脑卒中数学模型举例说明。
具体实施方式
158.下述实施例中所使用的实验方法如无特殊说明,均为常规方法。
159.下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
160.实施例1、用于检测mgrn1基因甲基化位点的引物设计
161.经过大量序列和功能分析,选择了红褐蛋白环指1(mahogunin ring finger-1,mgrn1)基因中的四个片段(mgrn1_a片段、mgrn1_b片段、mgrn1_c片段、mgrn1_d片段)进行甲
基化水平和心脑血管疾病相关性分析。
162.mgrn1_a片段(seq id no.1)位于hg19参考基因组chr16:4699588-4700441,反义链。
163.mgrn1_b片段(seq id no.2)位于hg19参考基因组chr16:4713936-4714859,正义链。
164.mgrn1_c片段(seq id no.3)位于hg19参考基因组chr16:4730091-4731004,反义链。
165.mgrn1_d片段(seq id no.4)位于hg19参考基因组chr16:4732525-4733325,反义链。
166.mgrn1_a片段中的cpg位点信息如表1所示。
167.mgrn1_b片段中的cpg位点信息如表2所示。
168.mgrn1_c片段中的cpg位点信息如表3所示。
169.mgrn1_d片段中的cpg位点信息如表4所示。
170.表1 mgrn1_a片段中cpg位点信息
[0171][0172][0173]
表2 mgrn1_b片段中cpg位点信息
[0174][0175][0176]
表3 mgrn1_c片段中cpg位点信息
[0177]
[0178][0179]
表4 mgrn1_d片段中cpg位点信息
no.5、seq id no.7、seq id no.9、seq id no.11中自5’第1至10位为非特异标签,第11至35位为特异引物序列;seq id no.6、seq id no.8、seq id no.10和seq id no.12中自5’第1至31位为非特异标签,第32至56位为特异引物序列。引物序列中不包含snp和cpg位点。
[0183]
表5 mgrn1甲基化引物序列
[0184][0185]
实施例2、mgrn1基因甲基化检测及结果分析
[0186]
一、研究样本
[0187]
研究样本采用流行病学整群抽样方法,通过2年的时间对某市18岁以上社区人群进行随访调查。本研究通过伦理委员会审查,所有调查对象均签署了知情同意书。每年通过当地医院、疾控中心慢病管理系统、社区卫生服务中心和工作站慢病常规登记项目、社保中心报销数据记录心脑血管疾病和癌症发病信息。队列开始时间为基线调查日期,结局变量为心脑血管发病,对于失访研究对象的随访时间,统一按照随访结束时间的一半来计算。截止随访日期2018年7月,共计心脑血管疾病发病620人,本发明选择了队列入组后2年内新发心脑血管疾病患者作为病例组,其中342例为冠心病患者,278例为脑卒中患者。经年龄和性别匹配后,选择曾经及随访期间未发生心脑血管疾病和癌症且血常规指标都在参考范围内的人群作为健康对照,共计612例。
[0188]
所有患者离体血液样本都是发病前即入组时收集的。患病情况在后续发病时都经过影像学和病理确诊。
[0189]
入组后2年以内发生冠心病的342例患者按照临床分型划分:隐匿型或无症状性心肌缺血45例,心绞痛64例,心肌梗死83例,缺血性心肌病74例、猝死76例。其中,137例是在入组后1年以内发生冠心病的,其中包括隐匿型或无症状性心肌缺血20例,心绞痛21例,心肌梗死33例,缺血性心肌病30例、猝死33例。
[0190]
入组后2年以内发生脑卒中的278例患者按照临床分型划分:出血性脑卒中112例,缺血性脑卒中166例。其中,110例是在入组后1年以内发生脑卒中的,其中包括出血性脑卒中49例,缺血性脑卒中61例。
[0191]
健康对照、冠心病和脑卒中患者各自年龄的中位数分别为65、64和65岁,且这3种群体中各自的男女比例都约为1:1。入组后1年以内发病的冠心病和脑卒中患者各自年龄的中位数分别为65和64岁,且群体中各自的男女比例都约为1:1。
[0192]
二、甲基化检测
[0193]
1、提取血液样本的总dna。
[0194]
2、将步骤1制备的血液样本总dna进行重亚硫酸盐处理(参照qiagen的dna甲基化试剂盒说明书操作)。重亚硫酸盐处理后,未发生甲基化的胞嘧啶(c)被转化成尿嘧啶(u),而甲基化的胞嘧啶保持不变,即原来cpg位点的c碱基经重亚硫酸盐处理后转化为c或u。
[0195]
3、以步骤2经过重亚硫酸盐处理的dna为模板,采用表5中的4对特异引物对通过dna聚合酶按照常规pcr反应要求的反应体系进行pcr扩增,4对引物都采用相同的常规pcr体系,且4对引物都按照以下程序进行扩增。
[0196]
pcr反应程序为:95℃,4min
→
(95℃,20s
→
56℃,30s
→
72℃,2min)45个循环
→
72℃,5min
→
4℃,1h。
[0197]
4、取步骤3的扩增产物,通过飞行时间质谱进行dna甲基化分析,具体方法如下:
[0198]
(1)向5μl pcr产物中加入2μl虾碱性磷酸盐(sap)溶液(0.3ml sap[0.5u]+1.7ml h2o)然后按照以下程序在pcr仪中孵育(37℃,20min
→
85℃,5min
→
4℃,5min);
[0199]
(2)取出2μl步骤(1)得到的sap处理后的产物,根据说明书加入5μl t-cleavage反应体系中,然后在37℃孵育3h;
[0200]
(3)取步骤(2)的产物,加入19μl去离子水,再用6μg resin在旋转摇床进行去离子化孵育1h;
[0201]
(4)2000rpm室温离心5min,将微量上清由nanodispenser机械手臂上样384spectrochip;
[0202]
(5)飞行时间质谱分析;获得的数据用spectroacquire v3.3.1.3软件收集,通过massarray epityper v1.2软件实现可视化。
[0203]
上述飞行时间质谱检测使用的试剂均来试剂盒(t-cleavage masscleave reagent auto kit,货号:10129a);上述飞行时间质谱检测使用的检测仪器为massarray
○
r analyzer chip prep module 384,型号:41243;上述数据分析软件为检测仪器自带软件。
[0204]
5、对步骤4得到的数据进行分析。
[0205]
数据统计分析由spss statistics 23.0进行。
[0206]
非参数检验用于两组之间的比较分析。
[0207]
多个cpg位点的组合对于不同样品分组的鉴别效果通过逻辑回归和受试者曲线的统计学方法得以实现。
[0208]
所有的统计检验都是双侧的,p值<0.05被认为具有统计学意义。
[0209]
通过质谱实验,共获得128个可以区别的g峰图。采用spectroacquire v3.3.1.3软件根据含g峰和a峰面积比较,计算甲基化水平(spectroacquire v3.3.1.3软件可自动通过计算峰面积得到每个样本在每个cpg位点的甲基化水平)。
[0210]
三、结果分析
[0211]
1、健康对照、冠心病和脑卒中患者血液中mgrn1基因甲基化水平差异(早于临床发病时间2年)
[0212]
以342位冠心病患者、278位脑卒中患者和612名健康对照的血液为研究材料进行分析mgrn1基因中所有cpg位点的甲基化水平(表6),其中冠心病和脑卒中患者在入组时均无症状,入组后2年以内发病。结果表明,健康对照的mgrn1基因的甲基化水平中位数为0.42(iqr=0.30-0.54),脑卒中mgrn1基因的甲基化水平中位数为0.40(iqr=0.28-0.51),冠心
病患者甲基化水平中位数为0.47(iqr=0.34-0.60)。通过比较分析三者间的mgrn1基因的甲基化水平,结果发现脑卒中患者的mgrn1基因中所有cpg位点甲基化水平显著低于健康对照(p<0.05,表6),冠心病患者的mgrn1基因中所有cpg位点甲基化水平显著高于健康对照(p<0.05,表6)。此外,冠心病患者的mgrn1基因中所有cpg位点甲基化水平显著高于脑卒中患者(p<0.05,表6)。因此,mgrn1基因的甲基化水平可以在人群中用于筛选在未来2年时间内将会爆发脑卒中和冠心病的潜在患者,是非常有临床价值的分子标志物。
[0213]
2、健康对照、冠心病和脑卒中患者血液中mgrn1基因甲基化水平差异(早于临床发病时间1年)
[0214]
以137位冠心病患者、110位脑卒中患者和612名健康对照的血液为研究材料进行分析三者之间mgrn1基因中所有cpg位点的甲基化水平差异(表7),其中冠心病和脑卒中患者在入组时均无症状,入组后1年以内发病。结果表明,健康对照的mgrn1基因的甲基化水平中位数为0.42(iqr=0.30-0.54),脑卒中mgrn1基因的甲基化水平中位数为0.40(iqr=0.29-0.52),冠心病患者甲基化水平中位数为0.46(iqr=0.33-0.60)。通过比较分析三者的mgrn1基因的甲基化水平,结果发现脑卒中患者的mgrn1基因中所有cpg位点甲基化水平显著低于健康对照(p<0.05,表7),冠心病患者的mgrn1基因中所有cpg位点甲基化水平显著高于健康对照(p<0.05,表7)。此外,冠心病患者的mgrn1基因中所有cpg位点甲基化水平显著高于脑卒中患者(p<0.05,表7)。因此,mgrn1基因的甲基化水平可以在人群中用于筛选在1年时间内将会爆发脑卒中和冠心病的潜在患者,是非常有临床价值的分子标志物。
[0215]
3、健康对照与不同临床特征的冠心病、脑卒中之间的甲基化水平差异(早于临床发病时间2年)
[0216]
我们比较分析具有不同临床特征的342名冠心病患者、278名脑卒中患者和612名健康对照的mgrn1基因的甲基化水平差异,其中冠心病和脑卒中患者在入组时均无症状,入组后2年以内发病。342名冠心病患者按照临床特征划分:隐匿型或无症状性心肌缺血45例,心绞痛64例,心肌梗死83例,缺血性心肌病74例、猝死76例。278例脑卒中患者按照临床分型划分:出血性脑卒中112例,缺血性脑卒中166例。通过比较分析具有不同临床特征的342名冠心病患者和612名健康对照的mgrn1基因的甲基化水平,结果发现不同临床特征的冠心病患者(隐匿型或无症状性心肌缺血、心绞痛、心肌梗死、缺血性心肌病、猝死)中mgrn1基因所有cpg位点的甲基化水平都与健康对照有显著性差异(p<0.05,表8)。此外,我们发现具有不同临床特征的脑卒中患者(出血性脑卒中、缺血性脑卒)mgrn1基因中所有cpg位点的甲基化水平都与健康对照有显著性差异(p<0.05,表8)。
[0217]
4、健康对照与不同临床特征的冠心病、脑卒中之间的甲基化水平差异(早于临床发病时间1年)
[0218]
我们比较分析具有不同临床特征的137名冠心病患者、110名脑卒中患者和612名健康对照的mgrn1基因的甲基化水平差异,其中冠心病和脑卒中患者在入组时均无症状,入组后1年以内发病。137名冠心病患者按照临床特征划分:隐匿型或无症状性心肌缺血20例,心绞痛21例,心肌梗死33例,缺血性心肌病30例、猝死33例。110例脑卒中患者按照临床分型划分:出血性脑卒中49例,缺血性脑卒中61例。通过比较分析具有不同临床特征的137名冠心病患者和612名健康对照的mgrn1基因的甲基化水平,结果发现不同临床特征的冠心病患者(隐匿型或无症状性心肌缺血、心绞痛、心肌梗死、缺血性心肌病、猝死)中mgrn1基因所有
cpg位点的甲基化水平都与健康对照有显著性差异(p<0.05,表9)。此外,我们发现具有不同临床特征的脑卒中患者(出血性脑卒中、缺血性脑卒)mgrn1基因中所有cpg位点的甲基化水平都与健康对照有显著性差异(p<0.05,表9)。因此,mgrn1基因的甲基化水平可以用于预测在1年时间内爆发不同临床特征的冠心病和脑卒中疾病的可能性。
[0219]
5、用于辅助心脑血管疾病诊断的数学模型的建立
[0220]
本发明建立的数学模型可以用于达到如下目的:
[0221]
(1)在临床发病以前,对人群中有冠心病发病风险的个体进行预警。
[0222]
(2)在临床发病以前,对人群中有冠心病发病风险的个体进行预警,并且适用于各种类型的冠心病。
[0223]
(3)在临床发病以前,对人群中有脑卒中发病风险的个体进行预警。
[0224]
(4)在临床发病以前,对人群中有脑卒中发病风险的个体进行预警,并且适用于各种类型的脑卒中。
[0225]
(5)在临床发病以前,对人群中有脑卒中和冠心病发病风险的个体进行预警,并且区分冠心病患者和脑卒中患者。
[0226]
其中,所述冠心病发病风险个体者具体可为早于临床发病时间2年内或者1年内的冠心病潜在患者(即2年内或者1年内会被临床确诊为冠心病)。所述脑卒中发病风险个体具体可为早于临床发病时间2年内或者1年内的脑卒中潜在患者(即2年内或者1年内会被临床确诊为脑卒中)。
[0227]
数学模型的建立方法如下:
[0228]
(a)数据来源:步骤一中列出的342例冠心病患者,278例脑卒中患者和612例健康对照的离体血液样本的目标cpg位点(表1-表4中的一种或多种的组合)甲基化水平(检测方法同步骤二)。
[0229]
数据可根据实际需要加入年龄、性别、白细胞计数等已知参数来提高判别效率。
[0230]
(b)模型建立
[0231]
根据需要选取任意两类不同类型患者数据即训练集,(例如:未来2年内发生冠心病的潜在患者和健康对照、未来2年内发生脑卒中的潜在患者和健康对照、未来2年内发生冠心病的潜在患者和未来2年内发生脑卒中的潜在患者、未来2年内发生隐匿型或无症状性心肌缺血的潜在患者和健康对照、未来2年内发生心绞痛的潜在患者和健康对照、未来2年内发生心肌梗死的潜在患者和健康对照、未来2年内发生缺血性心肌病的潜在患者和健康对照、未来2年内发生猝死的潜在患者和健康对照、未来2年内发生出血性脑卒中的潜在患者和健康对照、未来2年内发生缺血性脑卒中的潜在患者和健康对照,未来1年内发生冠心病的潜在患者和健康对照、未来1年内发生脑卒中的潜在患者和健康对照、未来1年内发生冠心病的潜在患者和脑卒中患者、未来1年内发生隐匿型或无症状性心肌缺血的潜在患者和健康对照、未来1年内发生心绞痛的潜在患者和健康对照、未来1年内发生心肌梗死的潜在患者和健康对照、未来1年内发生缺血性心肌病的潜在患者和健康对照、未来1年内发生猝死的潜在患者和健康对照、未来1年内发生出血性脑卒中的潜在患者和健康对照、未来1年内发生缺血性脑卒中的潜在患者和健康对照;其中,所述健康对照可理解为现在和曾经均没有患过心脑血管疾病和癌症且血常规指标都在参考范围内)作为用于建立模型的数据,使用sas,r,spss等统计软件使用二分类逻辑回归的统计方法通过公式建立数学模型。
数学模型公式计算出的最大约登指数对应的数值为阈值或直接设定0.5为阈值,待测样品经过测试和代入模型计算后得到的检测指数大于阈值归为一类(b类),小于阈值归为另外一类(a类),等于阈值作为不确定的灰区。在对新的待测样品进行预测来判断属于哪一类时,首先通过dna甲基化的测定方法检测该待测样品mgrn1基因上一个或者多个cpg位点的甲基化水平,然后将这些甲基化水平的数据代入上述数学模型(如果构建模型时纳入了年龄、性别、白细胞计数等已知参数,则该步骤同时向模型公式中代入该待测样品的相应参数的具体数值),计算得到所述待测样本对应的检测指数,然后比较所述待测样本对应的检测指数和阈值的大小,根据比较结果确定所述待测样本属于哪一类样本。
[0232]
举例:如图1所示,将训练集中mgrn1基因单个cpg位点的甲基化水平或者多个cpg位点组合的甲基化水平的数据通过sas、r、spss等统计软件使用二分类逻辑回归的公式建立用于区分a类和b类的数学模型。该数学模型在此为二类逻辑回归模型,具体为:log(y/(1-y))=b0+b1x1+b2x2+b3x3+
…
.+bnxn,其中y为因变量即将待测样品的一个或者多个甲基化位点的甲基化值代入模型以后得出的检测指数,b0为常量,x1~xn为自变量即为该测试样品的一个或者多个甲基化位点的甲基化值(每一个值为0~1之间的数值),b1~bn为模型赋予每一个位点甲基化值的权重。具体应用时,先根据训练集中已经检测的样本的一个或者多个dna甲基化位点的甲基化程度(x1~xn)及其已知的分类情况(a类或者b类,分别对y赋值0和1)建立数学模型,由此确定该数学模型的常量b0以及各个甲基化位点的权重b1~bn,并由该数学模型计算出的以最大约登指数对应的数值为阈值或直接设定0.5为划分的阈值。待测样品经过测试和代入模型计算后得到的检测指数即y值大于阈值归为b类,小于阈值归为a类,等于阈值作为不确定的灰区。其中a类和b类为相对应的两分类(二分类的分组,哪一组是a类,哪一组是b类,要根据具体的数学模型来确定,在此不做约定),比如:未来2年内发生冠心病的潜在患者和健康对照、未来2年内发生脑卒中的潜在患者和健康对照、未来2年内发生冠心病的潜在患者和未来2年内发生脑卒中的潜在患者、未来2年内发生隐匿型或无症状性心肌缺血的潜在患者和健康对照、未来2年内发生心绞痛的潜在患者和健康对照、未来2年内发生心肌梗死的潜在患者和健康对照、未来2年内发生缺血性心肌病的潜在患者和健康对照、未来2年内发生猝死的潜在患者和健康对照、未来2年内发生出血性脑卒中的潜在患者和健康对照、未来2年内发生缺血性脑卒中的潜在患者和健康对照,未来1年内发生冠心病的潜在患者和健康对照、未来1年内发生脑卒中的潜在患者和健康对照、未来1年内发生冠心病的潜在患者和脑卒中患者、未来1年内发生隐匿型或无症状性心肌缺血的潜在患者和健康对照、未来1年内发生心绞痛的潜在患者和健康对照、未来1年内发生心肌梗死的潜在患者和健康对照、未来1年内发生缺血性心肌病的潜在患者和健康对照、未来1年内发生猝死的潜在患者和健康对照、未来1年内发生出血性脑卒中的潜在患者和健康对照、未来1年内发生缺血性脑卒中的潜在患者和健康对照(其中,所述健康对照可理解为现在和曾经均没有患过心脑血管疾病和癌症且血常规指标都在参考范围内)。对受试者的样品进行预测来判断属于哪一类时,首先采集受试者的血液,然后从中提取dna。将提取的dna通过重亚硫酸盐转化后,用dna甲基化的测定方法对受试者的mgrn1基因的单个cpg位点的甲基化水平或者多个cpg位点组合的甲基化水平进行检测,然后将检测得到的甲基化数据代入上述数学模型。如果该受试者的mgrn1基因一个或者多个cpg位点的甲基化水平代入上述数学模型后计算出来的值即检测指数大于阈值,则该受试者判定与训练集中检测指数
大于阈值的归属一类(b类);如果该受试者的mgrn1基因一个或者多个cpg位点的甲基化水平数据代入上述数学模型后计算出来的值即检测指数小于阈值,则该受试者跟训练集中检测指数小于阈值的归属一类(a类);如果该受试者的mgrn1基因一个或者多个cpg位点的甲基化水平数据代入上述数学模型后计算出来的值即检测指数等于阈值,则不能判断该受试者是a类还是b类。
[0233]
举例:图2的示意图举例说明mgrn1_c的优选cpg位点(mgrn1_c_17、mgrn1_c_18、mgrn1_c_19、mgrn1_c_20、mgrn1_c_21、mgrn1_c_22、mgrn1_c_23、grn1_c_24,25,26,27、mgrn1_c_28、mgrn1_c_29、mgrn1_c_30、mgrn1_c_31、mgrn1_c_32、mgrn1_c_33、mgrn1_c_34、mgrn1_c_35,36,37、mgrn1_c_38,39、mgrn1_c_40、mgrn1_c_41,42、mgrn1_c_43、mgrn1_c_44、mgrn1_c_45、mgrn1_c_46、mgrn1_c_47和mgrn1_c_48)的甲基化以及数学建模用于冠心病的判别:将未来2年内发生冠心病的潜在患者(早于临床发病时间≤2年)和健康对照训练集(在此为:342名冠心病患者和612名健康对照)中已经检测的mgrn1_c_17、mgrn1_c_18、mgrn1_c_19、mgrn1_c_20、mgrn1_c_21、mgrn1_c_22、mgrn1_c_23、grn1_c_24,25,26,27、mgrn1_c_28、mgrn1_c_29、mgrn1_c_30、mgrn1_c_31、mgrn1_c_32、mgrn1_c_33、mgrn1_c_34、mgrn1_c_35,36,37、mgrn1_c_38,39、mgrn1_c_40、mgrn1_c_41,42、mgrn1_c_43、mgrn1_c_44、mgrn1_c_45、mgrn1_c_46、mgrn1_c_47和mgrn1_c_48这25个可区分的cpg位点组合的甲基化水平的数据以及患者的年龄、性别(男性赋值为1,女性赋值为0)、白细胞计数通过r软件使用二分类逻辑回归的公式建立用于区分冠心病患者和健康对照的数学模型。该数学模型在此为二类逻辑回归模型,由此确定该数学模型的常量b0以及各个甲基化位点的权重b1~bn,在此例中具体为:log(y/(1-y))=2.502+2.261*mgrn1_c_17-2.754*mgrn1_c_18+2.724*mgrn1_c_19+1.261*mgrn1_c_20-7.112*mgrn1_c_21+7.724*mgrn1_c_22+3.856*grn1_c_23+7.726*mgrn1_c_24.25.26.27+2.646*mgrn1_c_28+2.753*mgrn1_c_29+1.764*m grn1_c_30-5.113*mgrn1_c_31+3.657*mgrn1_c_32-0.899*mgrn1_c_33+2.439*mgrn1_c_34-15.631*mgrn1_c_35.36.37-3.271*mgrn1_c_38.39-6.357*mgrn1_c_40+4.057*mg rn1_c_41.42-2.636*mgrn1_c_43-2.546*mgrn1_c_44+4.199*mgrn1_c_45+2.436*mgrn1_c_46-2.261*mgrn1_c_47-2.724*mgrn1_c_48-0.031*年龄+0.542*性别(男性赋值为1,女性赋值为0)+0.013*白细胞个数,其中y为因变量即将待测样品的25个可区分甲基化位点的甲基化值以及年龄、性别、白细胞计数代入模型以后得出的检测指数。在设定0.5为阈值的情况下,待测样品的mgrn1_c_17、mgrn1_c_18、mgrn1_c_19、mgrn1_c_20、mgrn1_c_21、mgrn1_c_22、mgrn1_c_23、grn1_c_24,25,26,27、mgrn1_c_28、mgrn1_c_29、mgrn1_c_30、mgrn1_c_31、mgrn1_c_32、mgrn1_c_33、mgrn1_c_34、mgrn1_c_35,36,37、mgrn1_c_38,39、mgrn1_c_40、mgrn1_c_41,42、mgrn1_c_43、mgrn1_c_44、mgrn1_c_45、mgrn1_c_46、mgrn1_c_47和mgrn1_c_48这25个可区分cpg位点的甲基化水平经过测试后连同其年龄、性别、白细胞计数的信息代入模型进行计算,得到的检测指数即y值大于0.5归为未来2年内发生冠心病的潜在患者,小于0.5归为健康对照,等于0.5则不确定为未来2年内发生冠心病的潜在患者还是健康对照。此模型的曲线下面积(auc)计算结果为0.75(表14)。
[0234]
举例:图3的示意图举例说明mgrn1_c的优选cpg位点mgrn1_c_17、mgrn1_c_18、mgrn1_c_19、mgrn1_c_20、mgrn1_c_21、mgrn1_c_22、mgrn1_c_23、grn1_c_24,25,26,27、mgrn1_c_28、mgrn1_c_29、mgrn1_c_30、mgrn1_c_31、mgrn1_c_32、mgrn1_c_33、mgrn1_c_34、
mgrn1_c_35,36,37、mgrn1_c_38,39、mgrn1_c_40、mgrn1_c_41,42、mgrn1_c_43、mgrn1_c_44、mgrn1_c_45、mgrn1_c_46、mgrn1_c_47和mgrn1_c_48)的甲基化以及数学建模用于脑卒中的判别:将未来2年内发生脑卒中的潜在患者(早于临床发病时间≤2年)和健康对照训练集(在此为:278名脑卒中患者和612名健康对照)中已经检测的这25个可区分的优选cpg位点组合的甲基化水平的数据以及患者的年龄、性别(男性赋值为1,女性赋值为0)、白细胞计数通过r软件使用二分类逻辑回归的公式建立用于区分脑卒中患者和健康对照的数学模型。该数学模型在此为二类逻辑回归模型,由此确定该数学模型的常量b0以及各个甲基化位点的权重b1~bn,在此例中具体为:log(y/(1-y))=-0.538+4.261*mgrn1_c_17-3.754*mgrn1_c_18+3.547*mgrn1_c_19+0.483*mgrn1_c_20-2.849*mgrn1_c_21-1.190*mgrn1_c_22-12.243*grn1_c_23+23.270*mgrn1_c_24.25.26.27+3.248*mgrn1_c_28+3.754*mgrn1_c_29+0.248*mgrn1_c_30-14.153*mgrn1_c_31-0.452*mgrn1_c_32+0.273*mgrn1_c_33-9.080*mgrn1_c_34-27.488*mgrn1_c_35.36.37+11.935*mgrn1_c_38.39+14.335*mgrn1_c_40-12.863*mgrn1_c_41.42+3.261*mgrn1_c_43-3.546*mgrn1_c_44-2.249*mgrn1_c_45-3.248*mgrn1_c_46-3.261*mgrn1_c_47-4.261*mgrn1_c_48+0.010*年龄-0.053*性别(男性赋值为1,女性赋值为0)+0.123*白细胞个数,其中y为因变量即将待测样品的25个可区分甲基化位点的甲基化值以及年龄、性别、白细胞计数代入模型以后得出的检测指数。在设定0.5为阈值的情况下,待测样品的mgrn1_c_17、mgrn1_c_18、mgrn1_c_19、mgrn1_c_20、mgrn1_c_21、mgrn1_c_22、mgrn1_c_23、grn1_c_24,25,26,27、mgrn1_c_28、mgrn1_c_29、mgrn1_c_30、mgrn1_c_31、mgrn1_c_32、mgrn1_c_33、mgrn1_c_34、mgrn1_c_35,36,37、mgrn1_c_38,39、mgrn1_c_40、mgrn1_c_41,42、mgrn1_c_43、mgrn1_c_44、mgrn1_c_45、mgrn1_c_46、mgrn1_c_47和mgrn1_c_48这25个可区分cpg位点的甲基化水平经过测试后连同其年龄、性别、白细胞计数的信息代入模型进行计算,得到的检测指数即y值大于0.5归为未来2年内发生脑卒中的潜在患者,小于0.5归为健康对照,等于0.5则不确定为未来2年内发生脑卒中的潜在患者还是健康对照。此模型的曲线下面积(auc)计算结果为0.74(表14)。
[0235]
从两位受试者(甲,乙)分别采集血液提取dna,将提取的dna通过重亚硫酸盐转化后,用dna甲基化的测定方法对受试者的mgrn1_c_17、mgrn1_c_18、mgrn1_c_19、mgrn1_c_20、mgrn1_c_21、mgrn1_c_22、mgrn1_c_23、grn1_c_24,25,26,27、mgrn1_c_28、mgrn1_c_29、mgrn1_c_30、mgrn1_c_31、mgrn1_c_32、mgrn1_c_33、mgrn1_c_34、mgrn1_c_35,36,37、mgrn1_c_38,39、mgrn1_c_40、mgrn1_c_41,42、mgrn1_c_43、mgrn1_c_44、mgrn1_c_45、mgrn1_c_46、mgrn1_c_47和mgrn1_c_48这25个可区分的cpg位点的甲基化水平进行检测。然后将检测得到的甲基化水平数据连同受试者的年龄、性别和白细胞计数的信息代入上述数学模型。甲受试者经数学模型计算出来的值为0.81大于0.5,则甲受试者判定为未来2年内发生冠心病或脑卒中的潜在患者(未来2年内临床发病);乙受试者经数学模型计算出来的值为0.12小于0.5,则乙受试者判定为健康对照(未来2年内不会临床发病)。检测结果与实际情况相符。
[0236]
(c)模型效果评价
[0237]
根据上述方法,分别建立用于区分未来2年内发生冠心病的潜在患者和健康对照、未来2年内发生脑卒中的潜在患者和健康对照、未来2年内发生冠心病的潜在患者和未来2年内发生脑卒中的潜在患者、未来2年内发生隐匿型或无症状性心肌缺血的潜在患者和健
康对照、未来2年内发生心绞痛的潜在患者和健康对照、未来2年内发生心肌梗死的潜在患者和健康对照、未来2年内发生缺血性心肌病的潜在患者和健康对照、未来2年内发生猝死的潜在患者和健康对照、未来2年内发生出血性脑卒中的潜在患者和健康对照、未来2年内发生缺血性脑卒中的潜在患者和健康对照,未来1年内发生冠心病的潜在患者和健康对照、未来1年内发生脑卒中的潜在患者和健康对照、未来1年内发生冠心病的潜在患者和脑卒中患者、未来1年内发生隐匿型或无症状性心肌缺血的潜在患者和健康对照、未来1年内发生心绞痛的潜在患者和健康对照、未来1年内发生心肌梗死的潜在患者和健康对照、未来1年内发生缺血性心肌病的潜在患者和健康对照、未来1年内发生猝死的潜在患者和健康对照、未来1年内发生出血性脑卒中的潜在患者和健康对照、未来1年内发生缺血性脑卒中的潜在患者和健康对照(其中,所述健康对照可理解为现在和曾经均没有患过心脑血管疾病和癌症且血常规指标都在参考范围内)的数学模型,并且通过受试者曲线(roc曲线)对其有效性进行评价。roc曲线得出的曲线下面积(auc)越大,说明模型的区分度越好,分子标志物越有效。采用不同cpg位点进行数学模型构建后的评价结果如表10、表11、表12和表13所示。表10、表11、表12和表13中,1个cpg位点代表mgrn1_c扩增片段中任意一个cpg位点的位点,2个cpg位点代表mgrn1_c中任意2个cpg位点的组合,3个cpg位点代表mgrn1_c中任意3个cpg位点的组合,
……
以此类推。表中的数值为不同位点组合评价结果的范围值(即任意个cpg位点组合方式的结果均在此范围内)。
[0238]
上述结果显示,mgrn1基因对于各组的鉴别能力(未来2年内发生冠心病的潜在患者和健康对照、未来2年内发生脑卒中的潜在患者和健康对照、未来2年内发生冠心病的潜在患者和未来2年内发生脑卒中的潜在患者、未来2年内发生隐匿型或无症状性心肌缺血的潜在患者和健康对照、未来2年内发生心绞痛的潜在患者和健康对照、未来2年内发生心肌梗死的潜在患者和健康对照、未来2年内发生缺血性心肌病的潜在患者和健康对照、未来2年内发生猝死的潜在患者和健康对照、未来2年内发生出血性脑卒中的潜在患者和健康对照、未来2年内发生缺血性脑卒中的潜在患者和健康对照,未来1年内发生冠心病的潜在患者和健康对照、未来1年内发生脑卒中的潜在患者和健康对照、未来1年内发生冠心病的潜在患者和脑卒中患者、未来1年内发生隐匿型或无症状性心肌缺血的潜在患者和健康对照、未来1年内发生心绞痛的潜在患者和健康对照、未来1年内发生心肌梗死的潜在患者和健康对照、未来1年内发生缺血性心肌病的潜在患者和健康对照、未来1年内发生猝死的潜在患者和健康对照、未来1年内发生出血性脑卒中的潜在患者和健康对照、未来1年内发生缺血性脑卒中的潜在患者和健康对照;其中,所述健康对照可理解为现在和曾经均没有患过心脑血管疾病和癌症且血常规指标都在参考范围内)随着位点数的增加而增加。
[0239]
除此以外,在表1-表4所示的cpg位点中,还存在少数几个优选位点的组合比多个非优选位点组合的鉴别能力更好的情况。例如表14、表15、表16和表17所示的mgrn1_c_17、mgrn1_c_18、mgrn1_c_19、mgrn1_c_20、mgrn1_c_21、mgrn1_c_22、mgrn1_c_23、grn1_c_24,25,26,27、mgrn1_c_28、mgrn1_c_29、mgrn1_c_30、mgrn1_c_31、mgrn1_c_32、mgrn1_c_33、mgrn1_c_34、mgrn1_c_35,36,37、mgrn1_c_38,39、mgrn1_c_40、mgrn1_c_41,42、mgrn1_c_43、mgrn1_c_44、mgrn1_c_45、mgrn1_c_46、mgrn1_c_47和mgrn1_c_48这25个可区分cpg位点的组合是mgrn1_c中任意二十五个组合的优选位点。
[0240]
综上所述,mgrn1基因上的cpg位点及其各种组合,mgrn1_a片段上的cpg位点及其
各种组合,mgrn1_b片段上的cpg位点及其各种组合,mgrn1_c片段上的cpg位点及其各种组合,mgrn1_c片段上mgrn1_c_17、mgrn1_c_18、mgrn1_c_19、mgrn1_c_20、mgrn1_c_21、mgrn1_c_22、mgrn1_c_23、grn1_c_24,25,26,27、mgrn1_c_28、mgrn1_c_29、mgrn1_c_30、mgrn1_c_31、mgrn1_c_32、mgrn1_c_33、mgrn1_c_34、mgrn1_c_35,36,37、mgrn1_c_38,39、mgrn1_c_40、mgrn1_c_41,42、mgrn1_c_43、mgrn1_c_44、mgrn1_c_45、mgrn1_c_46、mgrn1_c_47和mgrn1_c_48cpg位点及其各种组合,mgrn1_d片段上的cpg位点及其各种组合,以及mgrn1_a、mgrn1_b、mgrn1_c和mgrn1_d上的cpg位点及其各种组合的甲基化水平都对未来2年内发生冠心病的潜在患者和健康对照、未来2年内发生脑卒中的潜在患者和健康对照、未来2年内发生冠心病的潜在患者和未来2年内发生脑卒中的潜在患者、未来2年内发生隐匿型或无症状性心肌缺血的潜在患者和健康对照、未来2年内发生心绞痛的潜在患者和健康对照、未来2年内发生心肌梗死的潜在患者和健康对照、未来2年内发生缺血性心肌病的潜在患者和健康对照、未来2年内发生猝死的潜在患者和健康对照、未来2年内发生出血性脑卒中的潜在患者和健康对照、未来2年内发生缺血性脑卒中的潜在患者和健康对照,未来1年内发生冠心病的潜在患者和健康对照、未来1年内发生脑卒中的潜在患者和健康对照、未来1年内发生冠心病的潜在患者和脑卒中患者、未来1年内发生隐匿型或无症状性心肌缺血的潜在患者和健康对照、未来1年内发生心绞痛的潜在患者和健康对照、未来1年内发生心肌梗死的潜在患者和健康对照、未来1年内发生缺血性心肌病的潜在患者和健康对照、未来1年内发生猝死的潜在患者和健康对照、未来1年内发生出血性脑卒中的潜在患者和健康对照、未来1年内发生缺血性脑卒中的潜在患者和健康对照(其中,所述健康对照可理解为现在和曾经均没有患过心脑血管疾病和癌症且血常规指标都在参考范围内)有判别能力。
[0241]
表6比较健康对照、冠心病和脑卒中患者之间的甲基化水平差异(早于临床发病时间≤2年)
[0242]
[0243]
[0244]
[0245][0246]
表7比较健康对照、冠心病和脑卒中患者之间的甲基化水平差异(早于临床发病时间≤1年)
[0247]
[0248]
[0249]
[0250][0251]
表8比较健康对照与不同临床特征的冠心病、脑卒中之间的甲基化水平差异(早于临床发病时间≤2年)
[0252]
[0253]
[0254]
[0255][0256]
表9比较健康对照和不同临床特征的冠心病、脑卒中之间的甲基化水平差异(早于临床发病时间≤1年)
[0257]
[0258]
[0259]
[0260][0261]
表10 mgrn1_c的cpg位点及其组合用于区分健康对照和脑卒中、健康对照和冠心病、脑卒中和冠心病(早于临床发病时间≤2年)
[0262]
[0263][0264]
表11 mgrn1_c的cpg位点及其组合用于区分健康对照和脑卒中、健康对照和冠心病、脑卒中和冠心病(早于临床发病时间≤1年)
[0265]
[0266]
[0267][0268]
表12 mgrn1_c的cpg位点及其组合用于区分健康对照和不同临床特征的冠心病、脑卒中患者(早于临床发病时间≤2年)
[0269]
[0270][0271]
注:表中数据为曲线下面积(auc)。
[0272]
表13 mgrn1_c的cpg位点及其组合用于区分健康对照和不同临床特征的冠心病、脑卒中患者(早于临床发病时间≤1年)
[0273]
[0274][0275]
注:表中数据为曲线下面积(auc)。
[0276]
表14 mgrn1_c的最佳cpg位点及其组合用于区分区分健康对照和脑卒中、健康对照和冠心病、脑卒中和冠心病(早于临床发病时间≤2年)
[0277]
[0278][0279]
注:表中数据为曲线下面积(auc)。
[0280]
表15 mgrn1_c最佳cpg位点及其组合用于区分区分健康对照和脑卒中、健康对照和冠心病、脑卒中和冠心病(早于临床发病时间≤1年)
[0281]
[0282][0283]
表16 mgrn1_c最佳cpg位点及其组合用于区分健康对照和不同临床特征的冠心病、脑卒中患者(早于临床发病时间≤2年)
[0284]
[0285][0286]
注:表中数据为曲线下面积(auc)。
[0287]
表17 mgrn1_c最佳cpg位点及其组合用于区分健康对照和不同临床特征的冠心病、脑卒中患者(早于临床发病时间≤1年)
[0288][0289][0290]
注:表中数据为曲线下面积(auc)。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1