一种特征mRNA表达谱组合及结肠癌早期预测方法与流程
本发明属于生物技术和医学领域,具体地说,涉及一种特征mrna表达谱组合及结肠癌早期预测方法。
背景技术:
结肠癌(coloncancer)是常见的发生于结肠部位的消化道恶性肿瘤,多发于直肠与乙状结肠交界处。结肠癌男女患病比率为2-3:1,40-50岁人群发病率最高。慢性结肠炎患者、结肠息肉患者、男性肥胖者等为易感人群。结肠癌早期无明显症状,早期诊断较为困难。全球疾病负担(globalburdenofdisease,gbd)数据显示,2017年全球患有结肠直肠癌的人数超过930万,其中中国患病人数高达235万。2017年全球患有结肠直肠癌的死亡人数约为90万,占总死亡人数的1.60%。中国2017年死亡患者数约为19万,占总死亡人数的1.79%。统计结果显示,从1990年到2017年全球结肠直肠癌患病率和死亡率持续增长。中国结肠直肠癌患病率和死亡率在2010年之前低于全球平均水平,2010年后患病率和死亡率急剧增长高于全球平均水平。
支持向量机(supportvectormachine,svm)是一类按监督学习方式对数据进行二元分类的广义线性分类器,其决策边界是对学习样本求解的最大边距超平面。svm模型是将实例表示为空间中的点,这样映射就使得单独类别的实例被尽可能宽的明显的间隔分开。然后,将新的实例映射到同一空间,并基于它们落在间隔的哪一侧来预测所属类别。当训练数据是线性可分时,svm通过硬间隔最大化学习进行分类。当训练数据线性不可分时,svm通过使用核技巧以及软间隔最大化学习进行分类。svm对于特征含义相似的中等大小的数据集很强大,也适用于小型数据集。通常情况下,对样本量小于1万的数据集svm都有很好的预测效果。svm在疾病诊断、肿瘤分类、肿瘤基因识别等有着广泛的应用。
肿瘤早期诊断一直是医学界的难题。现有的早期诊断方法多是观测某一个或一类标志物的表达水平,难以达到理想的诊断效果。由于这些标志物在肿瘤患者和正常人群中的表达分布有部分重叠,难以界定标志物的临界值将肿瘤患者和正常人群较好地分开。因此,利用多个标志物表达特征组合可能是肿瘤早期诊断的一种有效方法。messengerrna(mrna)是由dna的一条链作为模板转录而来的、携带遗传信息的能指导蛋白质合成的一类单链核糖核酸。肿瘤组织与正常组织相比往往表现出大量mrna的失调,研究表明这些失调的mrna跟肿瘤的发生、病理机制和预后状态有密切关系。然而,由于单个mrna分子在肿瘤和正常人群中表达分布有重叠,难以界定早期预测的临界值。
因此,有必要建立一种有助于结肠癌的早期预测的更稳定的多个差异mrna表达特征组合的诊断模型。
技术实现要素:
有鉴于此,本发明针对上述的问题,提供了一种特征mrna表达谱组合及结肠癌早期预测方法,能够准确地进行结肠癌i/ii期预测。
为了解决上述技术问题,本发明公开了一种特征mrna表达谱组合,包括asb8、cited2、gltp、gsn、itm2c、leprot、mbnl1、ndrg2、plpp1、rap1a、rnf11、rnf185、serinc1、sppl2a、stx12、tgfbi、trak2、trib3、txnip和vsir,其核苷酸探针序列如seqidno.1-20所示。
本发明还公开了一种基于上述的特征mrna表达谱组合的结肠癌早期预测方法,包括以下步骤:
步骤1、获取结肠癌早期患者稳定差异表达的特征mrna;
步骤2、选取特征mrna表达数据,对每个样本进行数据标准化;
步骤3、使用支持向量机对标准化后的数据构建早期预测模型;
步骤4、根据患者特征mrna的表达水平进行早期预测;
该方法为非疾病的诊断和治疗目的。
可选地,所述步骤1中的获取结肠癌早期患者稳定差异表达的特征mrna具体为:
步骤1.1、从genomicdatacommonsdataportal数据库中下载结肠癌患者肿瘤组织和癌旁组织转录组数据以及临床数据,获得结肠癌患者肿瘤组织基因表达谱readcounts数值,即为测序读段数值,进行对数转换;
步骤1.2、选取具有一定表达丰度的mrna,即在所有样本中mrna的readcounts大于等于10;再对所有mrna的readcounts取对数,设样本总数为n,筛选后mrna总数为m,v为mrna的readcounts,u为取对数之后的表达值,则有;
uij=log2vij,i∈(1,n),j∈(1,m)(1)
其中,i为样本编号,j为mrna编号,uij为第i个样本、第j个mrna编号取对数之后的表达值,vij为第i个样本、第j个mrna编号的readcounts数值;
步骤1.3、选取疾病分期为i期和ii期的结肠癌患者,将这些患者记为结肠癌早期患者,结肠癌早期患者总数记为n′;
步骤1.4、选取肿瘤和正常样本中稳定表达的mrna,即在肿瘤和正常样本中变异系数均小于0.1的mrna,设μ为所有样本中mrna的表达均值,σ为标准差,变异系数的计算公式为:
其中,j为mrna编号,cv为变异系数,cvj为第j个样本的变异系数,σj为第j个mrna编号的标准差,μj为第j个mrna编号的mrna的表达均值,设m1为稳定表达的mrna总数,则有:
步骤1.5、选取肿瘤和正常样本中差异表达的mrna;使用取对数后的表达值计算肿瘤和正常样本mrna取对数后的倍数变化f,公式为:
其中,j为mrna编号,fj为第j个mrna编号的倍数变化,μ1j为第j个mrna编号的肿瘤样本的表达均值,μ2j为第j个mrna编号的正常样本的表达均值;
然后使用独立样本t检验比较肿瘤和正常样本中mrna的表达差异,独立样本t检验公式为:
其中n1为肿瘤样本数,n2为正常样本数,μ1为肿瘤样本mrna表达均值,μ2为正常样本mrna表达均值,
对所有t检验得出的p值进行错误发现率(falsediscoveryrate,fdr)校正,定义q为fdr校正后的数值,r为p值在m1个mrna中排序后的位置,则有:
其中,j为mrna编号,qj代表第j个mrna编号的fdr校正后的数值,pj代表第j个mrna编号的t检验得出的p值,rj代表第j个mrna编号的p值在m1个mrna中排序后的位置;
最后选取倍数变化f的绝对值大于1且fdr校正后q值小于等于0.05的mrna,记为特征mrna,设特征mrna总数为m2,则有:
m2=m1{|fj|≥1,qj≤0.05},j∈(1,m1)(7)
。
可选地,所述步骤2中的选取特征mrna表达数据,对每个样本进行数据标准化,公式为:
其中i为样本编号,j为特征mrna编号;μi为第i个样本所有特征mrna表达均值,σi为第i个样本所有特征mrna标准差,uij为取对数后的特征mrna表达值,uij′为标准化后的mrna数值。
可选地,所述步骤3中的使用支持向量机对标准化后的数据构建早期预测模型,具体为:
步骤3.1、先对所有样本进行分组:将全部样本中80%划分为训练集+验证集,余下20%划分为测试集;训练集+验证集用于5折交叉验证,即将训练集+验证集分为相等的5组,按顺序将其中一组作为验证集,其余4组作为训练集。给定参数,训练集用于构建模型,验证集用于检验模型精确度;
步骤3.2、最优参数筛选:svm中参数gamma控制高斯核的宽度,c是正则化参数,限制每个点的重要性;参数网格设置为:
gamma=[0.001,0.01,0.1,1,10,100](9)
c=[0.001,0.01,0.1,1,10,100](10)
在交叉验证中,依次使用每两个参数gamma和c的组合构建模型,然后用验证集检验模型精确度;对每个参数组合,5折交叉验证的每次验证产生1个精确度,共进行5次验证即产生5个精确度;选取5次验证的平均精确度最高的参数组合作为最优参数;
步骤3.3、使用最优参数和训练集+验证集的数据构建模型,最后用测试集对模型进行评估。评估指标包括精确度(accuracy)、准确率(precision)、召回率(recall)、特异性(specificity)、f1分数(f1score)、马修斯相关系数(matthewscorrelationcoefficient,mcc)和受试者工作曲线(receiveroperatingcurve,roc)下面积(areaunderthecurve,auc);在测试集中,定义实际为肿瘤且预测为肿瘤计数为truepositive(tp),实际为正常但预测为肿瘤计数为falsepositive(fp),实际为肿瘤但预测为正常为falsenegative(fn),实际为正常且预测为正常为truenegative(tn);以上评估指标计算公式为:
以上评估指标中精确度、准确率、召回率、特异性、f1分数和auc返回介于(0,1)之间的值;精确度越高表示模型总体预测效率越高;准确率越高说明犯i类错误越小;召回率越高说明犯ii类错误越小;特异性高说明在预测为正例的样本中很少有负例混入;f1分数是一个综合指标,为准确率和召回率的调和平均;mcc是观察到的和预测的二元分类之间的相关系数,返回介于(-1,1)之间的值,其中1表示完美预测,0表示不比随机预测好,-1表示预测和观察之间的完全不一致;auc越高表明分类器预测的正实例概率越高;因此,以上指标越接近1表明模型整体的预测效果越好;
步骤3.4、若以上评估指标都大于0.9,说明模型具有较好的预测效果,则使用所有数据,用最优参数组合构建最终预测模型。
可选地,所述步骤4中的根据患者特征mrna的表达水平进行早期预测,具体为:
步骤4.1、对预测样本的特征mrna表达数据进行标准化,设u为预测样本特征mrna表达值,μ为预测样本特征mrna表达均值,σ为预测样本特征mrna标准差,公式为:
其中j为特征mrna编号,uj′为标准化后的mrna数值;
步骤4.2、将预测样本标准化后的mrna数值代入最终预测进行预测;预测结果为1表示患有结肠癌,预测结果为0表示正常。
与现有技术相比,本发明可以获得包括以下技术效果:
1)预测速度快:使用本发明构建的预测模型可以对大规模样本进行快速预测,100个样本的预测时间只需要几秒钟。
2)准确度高:本发明构建的预测模型预测精确度和准确率较高,都达到90%以上,roc曲线下面积auc可达1.000。
3)平台异质性影响较小:由于不同分析平台测定的mrna表达值有较大差异,本发明预测使用标准化后的特征mrna表达值,因此受平台异质性的影响较小。
当然,实施本发明的任一产品并不一定需要同时达到以上所述的所有技术效果。
附图说明
此处所说明的附图用来提供对本发明的进一步理解,构成本发明的一部分,本发明的示意性实施例及其说明用于解释本发明,并不构成对本发明的不当限定。在附图中:
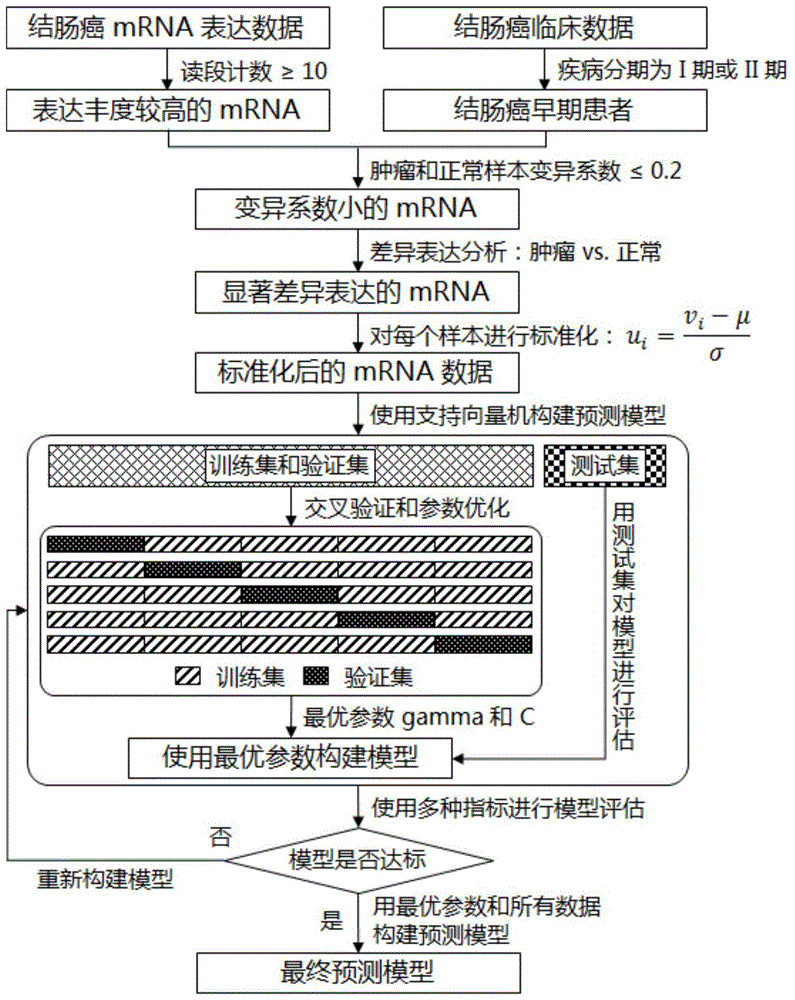
图1是本发明数据筛选和模型构建的流程;
图2是本发明支持向量机模型交叉验证参数优化过程;
图3是本发明支持向量机模型测试集评估指标;
图4是本发明支持向量机模型测试集roc曲线。
具体实施方式
以下将配合实施例来详细说明本发明的实施方式,藉此对本发明如何应用技术手段来解决技术问题并达成技术功效的实现过程能充分理解并据以实施。
一种基于特征mrna表达谱组合的结肠癌早期预测方法,包括以下步骤:
步骤1、获取结肠癌早期患者稳定差异表达的mrna(特征mrna),具体为:
步骤1.1、从genomicdatacommonsdataportal数据库中下载结肠癌患者肿瘤组织和癌旁组织转录组数据以及临床数据,获得结肠癌患者肿瘤组织基因表达谱readcounts数值,即为测序读段数值,进行对数转换;
步骤1.2、选取具有一定表达丰度的mrna,即在所有样本中mrna的readcounts大于等于10。再对所有mrna的readcounts取对数,设样本总数为n,筛选后mrna总数为m,v为mrna的readcounts,u为取对数之后的表达值,则有;
uij=log2vij,i∈(1,n),j∈(1,m)(1)
其中,i为样本编号,j为mrna编号,uij为第i个样本、第j个mrna编号取对数之后的表达值,vij为第i个样本、第j个mrna编号的readcounts数值。
步骤1.3、选取疾病分期为i期和ii期的结肠癌患者,将这些患者记为结肠癌早期患者,结肠癌早期患者总数记为n′;
步骤1.4、选取肿瘤和正常样本中稳定表达的mrna,即在肿瘤和正常样本中变异系数均小于0.1的mrna,设μ为所有样本中mrna的表达均值,σ为标准差,变异系数的计算公式为:
其中,j为mrna编号,cv为变异系数,cvj为第j个样本的变异系数,σj为第j个mrna编号的标准差,μj为第j个mrna编号的mrna的表达均值,设m1为稳定表达的mrna总数,则有:
步骤1.5、选取肿瘤和正常样本中差异表达的mrna。使用取对数后的表达值计算肿瘤和正常样本mrna取对数后的倍数变化f,公式为:
其中j为mrna编号,fj为第j个mrna编号的倍数变化,μ1j为第j个mrna编号的肿瘤样本的表达均值,μ2j为第j个mrna编号的正常样本的表达均值。
然后使用独立样本t检验比较肿瘤和正常样本中mrna的表达差异,独立样本t检验公式为:
其中n1为肿瘤样本数,n2为正常样本数,μ1为肿瘤样本mrna表达均值,μ2为正常样本mrna表达均值,
对所有t检验得出的p值进行错误发现率(falsediscoveryrate,fdr)校正,定义q为fdr校正后的数值,r为p值在m1个mrna中排序后的位置,则有:
其中,j为mrna编号,qj代表第j个mrna编号的fdr校正后的数值,pj代表第j个mrna编号的t检验得出的p值,rj代表第j个mrna编号的p值在m1个mrna中排序后的位置。
最后选取倍数变化f的绝对值大于1且fdr校正后q值小于等于0.05的mrna,记为特征mrna,设特征mrna总数为m2,则有:
m2=m1{|fj|≥1,qj≤0.05},j∈(1,m1)(7)
步骤2、选取特征mrna表达数据,对每个样本进行数据标准化,公式为:
其中i为样本编号,j为特征mrna编号。μi为第i个样本所有特征mrna表达均值,σi为第i个样本所有特征mrna标准差,uij为取对数后的特征mrna表达值,uij′为标准化后的mrna数值。
步骤3、使用支持向量机对标准化后的数据构建早期预测模型,具体为:
步骤3.1、先对所有样本进行分组。将全部样本中80%划分为训练集+验证集,余下20%划分为测试集。训练集+验证集用于5折交叉验证,即将训练集+验证集分为相等的5组,按顺序将其中一组作为验证集,其余4组作为训练集。给定参数,训练集用于构建模型,验证集用于检验模型精确度。
步骤3.2、最优参数筛选。svm中参数gamma控制高斯核的宽度,c是正则化参数,限制每个点的重要性。参数网格设置为:
gamma=[0.001,0.01,0.1,1,10,100](9)
c=[0.001,0.01,0.1,1,10,100](10)
在交叉验证中,依次使用每两个参数gamma和c的组合构建模型,然后用验证集检验模型精确度。对每个参数组合,5折交叉验证的每次验证产生1个精确度,共进行5次验证即产生5个精确度。选取5次验证的平均精确度最高的参数组合作为最优参数。
步骤3.3、使用最优参数和训练集+验证集的数据构建模型,最后用测试集对模型进行评估。评估指标包括精确度(accuracy)、准确率(precision)、召回率(recall)、特异性(specificity)、f1分数(f1score)、马修斯相关系数(matthewscorrelationcoefficient,mcc)和受试者工作曲线(receiveroperatingcurve,roc)下面积(areaunderthecurve,auc)。在测试集中,定义实际为肿瘤且预测为肿瘤计数为truepositive(tp),实际为正常但预测为肿瘤计数为falsepositive(fp),实际为肿瘤但预测为正常为falsenegative(fn),实际为正常且预测为正常为truenegative(tn)。以上评估指标计算公式为:
以上评估指标中精确度、准确率、召回率、特异性、f1分数和auc返回介于(0,1)之间的值。精确度越高表示模型总体预测效率越高;准确率越高说明犯i类错误越小;召回率越高说明犯ii类错误越小;特异性高说明在预测为正例的样本中很少有负例混入;f1分数是一个综合指标,为准确率和召回率的调和平均;mcc是观察到的和预测的二元分类之间的相关系数,返回介于(-1,1)之间的值,其中1表示完美预测,0表示不比随机预测好,-1表示预测和观察之间的完全不一致;auc越高表明分类器预测的正实例概率越高。因此,以上指标越接近1表明模型整体的预测效果越好。
步骤3.4、若以上评估指标都大于0.9,说明模型具有较好的预测效果。则使用所有数据,用最优参数组合构建最终预测模型。
步骤4、根据患者特征mrna的表达水平进行早期预测,具体为:
步骤4.1、对预测样本的特征mrna表达数据进行标准化,设u为预测样本特征mrna表达值,μ为预测样本特征mrna表达均值,σ为预测样本特征mrna标准差,公式为:
其中j为特征mrna编号,uj′为标准化后的mrna数值。
步骤4.2、将预测样本标准化后的mrna数值代入最终预测进行预测。预测结果为1表示患有结肠癌,预测结果为0表示正常。
实施例1
步骤1、获取结肠癌早期患者稳定差异表达的mrna(特征mrna),详细流程见图1。
步骤1.1、从genomicdatacommonsdataportal数据库中下载结肠癌患者肿瘤组织和癌旁组织转录组数据以及临床数据,获得结肠癌患者肿瘤组织基因表达谱readcounts数值,进行对数转换。
步骤1.2、选取具有一定表达丰度的mrna,即在所有样本中mrna的readcounts大于等于10,详见公式(1)。
步骤1.3、选取疾病分期为i期和ii期的结肠癌患者,详见公式(2)-(3),将这些患者记为结肠癌早期患者。
步骤1.4、选取肿瘤和正常样本中稳定表达的mrna,即在肿瘤和正常样本中变异系数均小于0.1的mrna。
步骤1.5、选取肿瘤和正常样本中差异表达的mrna,详见公式(4)-(7)。记为特征mrna。本例中选取前20个结肠癌特征mrna(按fdr校正后p值从小到大排序)进行模型构建,见表1。20个结肠癌特征mrna的核苷酸探针序列见表2。
表1.结肠癌特征mrna
表2.结肠癌特征mrna的核苷酸探针序列
步骤2、对每个样本进行数据标准化,详见公式(8)。
步骤3、使用支持向量机对标准化后的数据构建早期诊断模型。
步骤3.1、先对所有样本进行分组。将全部样本中80%划分为训练集+验证集,余下20%划分为测试集。训练集+验证集用于5折交叉验证,即将训练集+验证集分为相等的5组,按顺序将其中一组作为验证集,其余4组作为训练集。给定参数,训练集用于构建模型,验证集用于检验模型精确度。详见图1。
步骤3.2、最优参数筛选。svm参数网格设置见公式(9)-(10)。在交叉验证中,依次使用每两个参数gamma和c的组合构建模型,然后用验证集检验模型精确度。对每个参数组合,5折交叉验证的每次验证产生1个精确度,共进行5次验证即产生5个精确度。选取5次验证的平均精确度最高的参数组合作为最优参数。图2所示为交叉验证参数优化过程,当参数gamma=0.01,参数c=100时模型交叉验证精确度最高:1.000。因此该模型的最优参数为:gamma=0.01,c=100。
步骤3.3、使用最优参数和训练集+验证集的数据构建模型,最后用测试集对模型进行评估。评估指标包括精确度(accuracy)、准确率(precision)、召回率(recall)、特异性(specificity)、f1分数(f1score)、马修斯相关系数(matthewscorrelationcoefficient,mcc)和受试者工作曲线(receiveroperatingcurve,roc)下面积(areaunderthecurve,auc)。评估指标详见公式(11)-(17)。
步骤3.4、图3所示为以上评估指标中的精确度、准确率、召回率、特异性、f1分数和mcc,这6个指标均为1.000;图4所示为roc曲线和auc,测试集中auc为1.000。说明以上评估指标说明该模型有很好的预测效果。因此使用所有数据,用最优参数组合构建最终预测模型。
步骤4、根据患者特征mrna的表达水平进行早期预测:
步骤4.1、对预测样本的特征mrna表达数据进行标准化,详见公式(18)。本发明随机选取10例样本进行预测,并在构建最终预测模型时将这10例样本剔除。所选取的10例样本编号和标准化后特征mrna数值见表3。
表3.10例样本编号和特征mrna标准化后的数值
步骤4.2、将预测样本标准化后的mrna数值代入最终预测进行预测。预测结果为1表示患有结肠癌,预测结果为0表示正常。10例样本编号,对应的tcga编号,实际状态和预测结果见表4。10例样本预测结果与实际状态完全符合,说明本发明可以对结肠癌进行精确的早期预测。
表4.10例样本编号,对应的tcga编号,实际和预测的状态
综上所述,本发明的特征mrna表达谱组合具有很高的预测准确性,能够有效地进行结肠癌的早期预测。此外,本发明没有平台依赖性,能够对多种来源的数据进行预测。
上述说明示出并描述了发明的若干优选实施例,但如前所述,应当理解发明并非局限于本文所披露的形式,不应看作是对其他实施例的排除,而可用于各种其他组合、修改和环境,并能够在本文所述发明构想范围内,通过上述教导或相关领域的技术或知识进行改动。而本领域人员所进行的改动和变化不脱离发明的精神和范围,则都应在发明所附权利要求的保护范围内。
sequencelisting
<110>广东省第二人民医院
<120>一种特征mrna表达谱组合及结肠癌早期预测方法
<130>2020
<160>20
<170>patentinversion3.3
<210>1
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>1
gctctgtaataacagtaataaatagctctg30
<210>2
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>2
ccacccccatatgtacagatgataataggg30
<210>3
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>3
atcgatgtcatctacgagacgtatacccag30
<210>4
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>4
gtgaagcaaggctttgagcctccctccttt30
<210>5
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>5
cttagcttgtactttggacgcgtttctata30
<210>6
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>6
aaccaggattatcatctatattggaagtca30
<210>7
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>7
atcctttcaaaccctcatgactgacaaaaa30
<210>8
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>8
ggagggtgccctcccgtctcccacaacttc30
<210>9
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>9
agcatgcttcttgctaacatttgggctcag30
<210>10
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>10
gaaatgtcccctcaaactcattgcagcaga30
<210>11
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>11
tgtagtatccatatgttgcttaaatttcct30
<210>12
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>12
ttatgactgcatagtttgtggaaacaaaga30
<210>13
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>13
caggtcagaagaatgatggaatgttttaga30
<210>14
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>14
atgttcagactggttcttcttacatatact30
<210>15
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>15
gcacagtggcccaaagagcagcttcagaga30
<210>16
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>16
agctatgagttgaaatgttctgtcaaatgt30
<210>17
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>17
agaaaatgttgtgctgtatgttcttgattt30
<210>18
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>18
acacttggggtccacaatcccaggtccata30
<210>19
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>19
tccacccttttctgagagttattacagcca30
<210>20
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>20
ctgatcttggcgtgtagtcctgcacctgtt30
- 还没有人留言评论。精彩留言会获得点赞!