基于网络表示学习的计算疾病相似度系统的制作方法
本发明涉及疾病相似度的计算领域,特别涉及一种基于网络表示学习的计算疾病相似度系统。
背景技术:
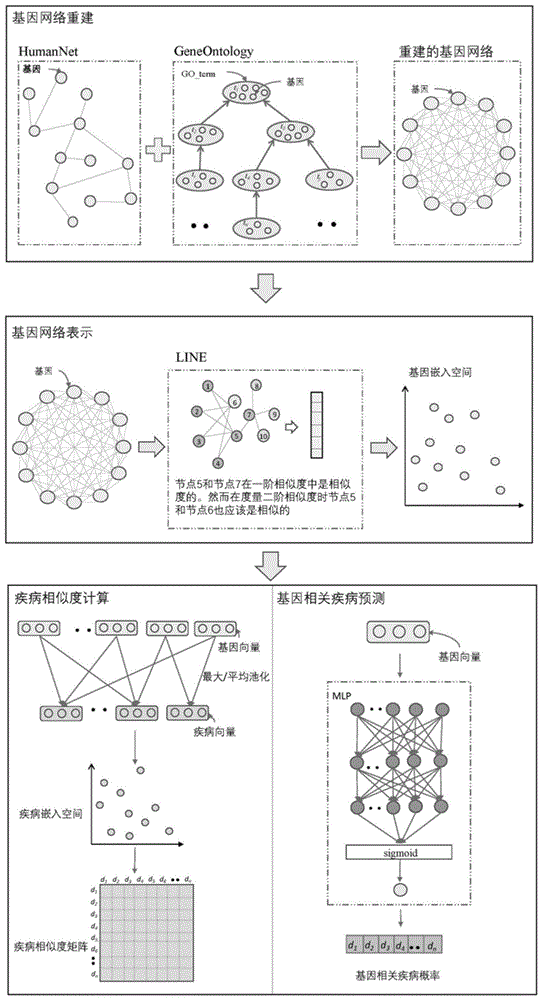
:近年来疾病相似度的研究在生物信息领域受到了大量的关注,随之出现了很多建立疾病之间相似的方法。建立疾病之间的关系有助于增进对疾病生物学的理解,并且,在复杂疾病发病原理的理解、诊断、潜在疾病治疗药物的预测等任务中都起到重要作用。而疾病相似度是对疾病之间关系的量化过程,因此计算疾病的相似度具有重大的生物学和药理学意义。生活中,各种信息网络可能包含大量的节点和边,这会导致直接在网络上进行计算是非常困难且缓慢的。因此,人们提出将图中的节点表示为可以反映图信息的低维的向量后再进行计算。这种将图中节点表示为向量的过程就称为网络表示学习,又叫做网络嵌入、图嵌入。网络学习表示的目的是根据网络中节点之间的相互联系,将网络中的每个节点用低维稠密的向量空间表示(其中向量空间的维度远小于节点的总个数),并且能够保持原有网络的结构与功能,能够支持后续的网络处理和分析任务,如节点分类、节点聚类、网络可视化和链路预测等。目前大多从基于本体计算疾病术语相似度和基于疾病相关基因计算疾病相似度两个角度考虑。基于本体计算疾病术语相似度是根据疾病对信息量最大的共同祖先的信息量来计算疾病术语相似度。目前基于疾病相关基因计算疾病相似度有两种方法,第一种是基于共同的疾病基因(bog),通过统计疾病之间的共同相关基因个数来获得疾病的相似度;另一种时基于过程相似性计算基因相似度从而得到疾病相似度。但以上计算疾病相似度的方法的准确率都不高。技术实现要素:本发明目的是通过网络表示学习的方法解决计算疾病相似度的准确率不高的问题。基于网络表示学习的计算疾病相似度系统,包括:disgenet数据集、geneontlogy数据集、humannet网络、信息融合模块、网络嵌入模块、疾病相似度计算模块、基因与疾病预测模块;所述disgenet数据集用于存储基因和疾病之间的关系;所述geneontlogy数据集用于存储go_term之间的关系以及go_term和基因之间的关系;所述humannet网络用于存储基因之间的关系;所述信息融合模块,用于将基因相似度矩阵进行拉普拉斯平滑得到矩阵r,利用矩阵r计算两个go_term注释的基因集之间相似度,利用go_term注释的基因集之间相似度计算两个go_term之间的相似度,利用两个go_term之间的相似度获得基因之间的相似度;所述网络嵌入模块,基于基因之间的相似度将每个基因转化为向量形式。所述疾病相似度计算模块,基于基因的向量表示和疾病相关基因数据集disgenet将疾病相关基因融合,得到疾病的向量表示,利用疾病向量表示度量疾病的相似性。所述基因与疾病预测模块,实现基于基因的向量表示,结合mlp模型,对基因与疾病之间的关系进行预测的功能。所述go_term为基因本体的数据集;所述go_term注释的基因集是与go_term相关基因集的集合。本发明的有益效果为:本发明提供的基于网络表示学习的计算疾病相似度系统,系统包含网络融合模块、网络嵌入模块、疾病相似度计算模块、基因与疾病预测模块。通过网络表示学习的方式各模块之间相互协作从而提升计算疾病相似度的准确性。附图说明图1为基于网络表示学习的计算疾病相似度整体流程图。具体实施方式具体实施方式一:本实施方式基于网络表示学习的计算疾病相似度系统,包括:disgenet数据集、geneontlogy数据集、humannet网络、信息融合模块、网络嵌入模块、疾病相似度计算模块、基因与疾病预测模块。所述disgenet数据集存储基因和疾病之间的关系;所述geneontlogy数据集用于存储go_term之间的关系以及go_term和基因之间的关系;所述humannet网络存储基因之间的关系。所述信息融合模块,用于将基因相似度矩阵进行拉普拉斯平滑得到矩阵r,利用矩阵r计算两个go_term注释的基因集之间相似度,利用go_term注释的基因集之间相似度从路径相似度和公共父节点的相似度两方面考虑,计算两个go_term之间的相似度,利用两个go_term之间的相似度获得基因之间的相似度;所述网络嵌入模块,基于基因之间的相似度将每个基因转化为向量形式;所述疾病相似度计算模块,基于基因的向量表示和疾病相关基因数据disgenet将疾病相关基因融合,得到疾病的向量表示。利用疾病向量表示度量疾病的相似性,实现输入两个疾病的id,系统输出疾病之间的相似度;所述基因与疾病预测模块,实现基于基因的向量表示,结合mlp模型,对基因与疾病之间的关系进行预测的功能;所述go-term是多个基因的集合;所述go_term注释的基因集是与go_term相关基因集的集合。具体实施方式二:本实施方式与具体实施方式一不同的是:所述信息融合模块,用于将基因相似度矩阵进行拉普拉斯平滑得到矩阵r,利用矩阵r计算两个go_term注释的基因集之间相似度,利用go_term注释的基因集之间相似度从路径相似度和公共父节点的相似度两方面考虑,计算两个go_term之间的相似度,利用两个go_term之间的相似度获得基因之间的相似度矩阵,其具体过程为:步骤二一、提取humannet信息:humannet中很大一部分节点之间是没有边的,这样两个基因之间的相似度就会为零,不便于接下来的计算,为了去除这些零值,将基因相似度矩阵进行拉普拉斯平滑:其中其中,r为拉普拉斯平滑后的相似度矩阵,lambda是拉普拉斯平滑的参数,sum是humannet中所有基因对相似性的总和,n是基因的数量,b是用于将值规格化为(0,1)区间的值的扩展因子,s[i][j]是i,j的相似度矩阵,gi和gj表示疾病基因。步骤二二、计算两个go_term之间的相似度,具体包括以下步骤:步骤二二一、结合来自humannet和go_term的信息计算两个go_term注释的基因集之间的相似度:d(t1,t2)是两个go_term注释的基因集之间的相似度,t1、t2是两个go_term,g1、g2是t1、t2对应的基因集,dij=1-r[i][j]是两个基因之间的距离,|g1∪g2|是与t1、t2两个go_term相关的两个基因的集合g1、g2并集的基因数。步骤二二二、计算两个go-term之间的相似度:其中,h(t1,t2)=d(t1,t2)2*|g|+(1-d(t1,t2)2)*max(|g1|,|g2|)其中,p是距离t1、t2最近的公共父节点,gp是p和t1、t2子节点注释的基因集,g是根节点和t1、t2子节点注释的基因集,f′(t1,t2,p)是t1、t2、p基于路径的相似度,h(t1,t2)是t1、t2基于公共父节点的相似度,是t1注释的基因集、t2注释的基因集、t1和t2的共同亲本p注释的基因集三个基因集并集的基因数目。步骤二三、计算基因之间的相似度:根据步骤二二获得的go_term之间的相似度,获得基因之间的相似度:其中,基于z-score和给定的go_term(一个t代表一个go_term),可以从tj获得两个集合:t′th={t′|(zt,t′≥1.6)}t′tl={t′|(zt,t′<1.6)}如果|t′th|>|t′tl|,那么t′j=t′th,否则t′j=t′tl。其中是相似性标准分数,ti和tj分别是gi和gj注释的术语集,|ti|+|tj|是ti和tj中基因数的和,t′j是tj中部分go_term的集合,t′i是ti中部分go_term语句的集合,y表示i或j,t2是ty中的go_term,所述的注释的术语集是与基因相关的go_term的集合。其他步骤与具体实施方式一相同。具体实施方式三:本实施方式与实施方式一和二不同的是:所述网络嵌入模块,基于基因之间的相似度将每个疾病基因转化为向量形式具体过程为:在这一模块,基于前面获得的基因间相似度,使用网络表示方法—line的二阶相似度方法学习基因的向量表示。所述的line的二阶相似度方法,每个顶点扮演两个角色:顶点和其他顶点的邻居;步骤三一、对于每条有向边(i,j)从基因vi到基因vj之间的联合概率为:其中为基因vi的作为起始节点时的向量表示,为基因vj作为终止节点时的向量表示,t表示向量的转置,v是网络中顶点的总数。步骤三二、在网络结构中vi、vj的经验值定义为:其中,wij表示gensim(gi,gj),d′i是vi所有出度的和,所述经验值为经验概率。步骤三三、调整基因的向量表示使p2、之间的差异最小化,即使目标函数最小化。其中λi为i的权重,d1(·)表示p2、的相对熵。具体实施方式四:本实施方式与实施方式一至三不同的是:所述疾病相似度计算模块,基于基因的向量表示和疾病相关基因数据集disgenet将疾病相关基因融合,采用averagepooling方法得到疾病的向量表示,用两个疾病向量的余弦相似度表示两个疾病的相似度,实现输入两个疾病的id,输出疾病之间的相似度。具体实施方式五:本实施方式与实施方式一至四不同的是:所述基因与疾病预测模块,实现基于基因的向量表示,结合mlp模型,对基因与疾病之间的关系进行预测的功能具体过程为:基于信息融合模块、网络嵌入模块的计算获得基因的向量表示,基因与疾病预测模块将基因的向量表示结合mlp模型,通过训练可选择与任一疾病匹配,输出基因与疾病的相关概率;本系统使用了四层的mlp感知器,并使用梯度下降的方法更新参数。其中,四层感知器为:输入层+隐含层+隐含层+输出层,四个层次整合为:f(x)=softmax(b2+w2(sigmoid(w1x+b1)))其中,w1是权重,b1是偏置,sigmoid(w1x+b1)是第一层隐含层的输出,softmax(w2x1+b2)是输出层的输出,f为softmax函数,输入为基因的向量表示,输出为与基因相关疾病的概率;其他步骤与具体实施方式一或二或三或四相同。实施例按照具体实施方式一至具体实施方式五的技术方案进行,并使用roc曲线和auc面积作为评价指标,评价系统准确性:评价标准方面,由于进行的疾病相似度实验,和基因与疾病关系预测实验从本质上看都属于链路预测的任务,所以使用roc曲线和auc面积作为评价指标。roc曲线图中横坐标表示特异性,纵坐标表示敏感性。roc曲线下方面积用auc表示,auc面积越大表示性能越好。两个实验结果如下表所示:表1疾病相似度计算实验结果表2基因与疾病关系预测实验结果方法auc面积humannet+line0.756go+line0.7625humannet+go+line0.796当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1