一种骨肉瘤转移相关基因signature研究及潜在药物挖掘方法与流程
本发明涉及骨肉瘤转移相关基因技术领域,具体为一种骨肉瘤转移相关基因signature研究及潜在药物挖掘方法。
背景技术:
骨肉瘤(osteosarcoma,os)是恶性异常梭形细胞瘤的统称,它们的共同特征是产生未成熟的骨,也称为类骨质,恶性程度以及因此转移(或扩散)的趋势取决于组织学等级,os是最常见的原发性骨实体瘤,约占原发性骨肉瘤的20%,可能出现在身体的任何骨骼处,但是最常见的部位是手臂或腿部,尤其是膝关节周围,该家族既包含通过手术即可治愈的低级别类型,同时也包含高级别致死性的类型,os主要是发生在年轻人群中,超过25%的病例发生在25岁以下的人群中,通常认为os在男性中更为常见,这可能是由于男性骨骼生长时间较女性更长,在不同种族上,未观察到os的发生有偏好性。
尽管在局部疾病患者中采用多种联合疗法可以使治愈率达到65-70%,但治疗过程可能会是漫长而艰巨的,通常持续一年或更长时间,然而,在目前的治疗方案下,对于发生转移的骨肉瘤患者还没达到生存率接近70%的效果,对预后有重要影响的因素包括肿瘤位置(轴向位置更差)、肿瘤大小、对化疗反应较差以及转移等,其中与临床观察结果最一致的因素是转移,如可切除的肺部病变的患者只有大约30-50%的存活机会,而无法切除的肺转移灶或对化疗无响应的病变或多发性骨病变的患者,无论采取何种治疗方法,其病情都会继续恶化,但现有的研究无法进一步对os的病因和转移机制有更深入了解,在潜在药物挖掘上,也没有能够为os的治疗提供新的可供选择的方案。
技术实现要素:
(一)解决的技术问题
针对现有技术的不足,本发明提供了一种骨肉瘤转移相关基因signature研究及潜在药物挖掘方法,具备利用tcga和geo基因表达数据,鉴定了一组可能与os转移相关的mrs,并对mrs的潜在功能和潜在作用药物进行挖掘等优点,解决了背景技术提出的问题。
(二)技术方案
为实现上述利用tcga和geo基因表达数据,鉴定了一组可能与os转移相关的mrs,并对mrs的潜在功能和潜在作用药物进行挖掘的目的,本发明提供如下技术方案:一种骨肉瘤转移相关基因signature研究及潜在药物挖掘方法,包括以下步骤:
s1、数据下载及预处理:
对os的target数据做以下几步预处理:
4)去掉没有临床信息或者总生存时间<30天的样本;
5)去掉正常组织样本数据;
6)去掉在超过一半样本中表达量为0的基因;
对geo数据做以下几步预处理:
1)去掉正常组织样本数据,只保留肿瘤组织数据;
2)将总生存时间(gse39055为recurrence–freesurvival)为year或month的转成days;
3)利用bioconductor包将芯片探针map到人类基因symbol;
s2、mrs筛选和功能注释:
由于target和gse21257、gse32981、gse39055及gse49003均为芯片数据,下载的表达谱数据均为normalized后的,转移(metastatic)与未转移(non-metastatic)分组样本使用limma工具进行差异表达分析,由于target和gse32981数据的degs数量极少,这里取pvalue<0.01且log2foldchange绝对值>0.5作为degs阈值,对于gse21257和gse49003数据,取fdr<0.05且log2foldchange绝对值>0.5作为degs阈值,将4组数据集得到的degs定义为转移相关基因(metastasesrelatedsignature,mrs),使用rpackageclusterprofiler(v3.8)对mrs进行go、kegg功能注释和富集分析,取qvalue(fdr)<0.05作为显著富集的阈值;
s3、免疫基因的表达谱筛选:
使用单因素cox风险回归模型分析mrs与转移事件的预后关系,取logrankp<0.1为显著性阈值,使用kaplan-meierplotter方法分析并展示分组事件的预后总生存曲线;
s4、潜在药物挖掘:
利用l1000fwd工具挖掘与degs有关的小分子物质,通过比较鉴定出的上调和下调degs与数据库中记录的上调/下调基因,可以反向推出哪些小分子物质可能与问题有关,dgidb数据库记录了超过40000个基因和10000种药物的相互作用信息,与l1000数据库不同,dgidb记录的是特定基因及与其存在相互作用的药物关联信息,根据用户提供的基因,可以找到与该基因存在相互作用的药物,在使用cmap查询之前,使用bioconductorrpackage将4个亚型mrs基因symbol转换为affymetrix探针id,在cmap数据库中,预测具有显著负分数的药物将成为os的新型治疗药物,cmap使用基因集富集分析算法计算连接性分数,平均得分≤0.65用于识别潜在的候选药物,对于cmap提供的每个探针,保留amplitude≤-0.67或>0.67的为显著表达异常探针用于pathway分析(anamplitudeof±0.67representsatwo-foldchangebetweenthetreatmentandthecontrol);
s5、样本信息统计:
经过预处理后的os数据总共得到209个样本,其中target有85个满足条件样本,gse21257、gse32981、gse39055及gse49003分别有53、23、12和36个满足条件样本,从4个数据集的age分布来看,medianage均在20岁以下,alive和dead事件的患者medianoverallsurvivaltime也存在显著差异,比较target和gse21257转移和非转移样本的总生存时间预后情况可以看到二者也存在显著差异,此外转移与非转移样本recurrence预后也存在显著差异,未转移样本的总体生存时间也较转移样本的显著更高;
s6、mrs筛选和功能分析:
使用target和gse21257、gse32981及gse49003数据集,我们分析了转移与未转移样本的差异表达表达基因(degs),这里将degs定义为转移相关signatures(mrs),从上调和下调基因数量看,在非转移性样本中上调基因大于下调基因数,由于gse49003数据集为具有转移能力和无转移能力的细胞系,推测这可能导致其鉴定出的degs与target和gse21257、gse32981的有较大差异;
s7、mrs与预后的关系:
根据target和gse21257数据集预后(总生存时间)信息,使用单因素cox回归分析方法分析target/gse49003和gse21257/gse49003共有的18个mrs与预后的关系,发现其中有15个mrs在target或gse21257数据集上与预后有显著关系,根据hr(hazardratio)>1或<1将这些基因分为不利预后因子和有利预后因子,发现有利的预后基因在转移组上均表现出表达水平下调,而不利的预后基因在转移组上表现出表达水平上调,取在target和gse21257数据集上均与预后存在显著关系的il10ra和tlr7基因,根据表达量中位值将样本分成高表达组(l2)、低表达组(l1),kmploter结果表明高表达组样本预后显著好于低比表达组;
s8、转移相关signature的潜在药物分析;
l1000数据库提供超过16000种药物和小分子物质在大约1000个肿瘤细胞系上诱导的基因表达特征(上调/下调),利用l1000fwd工具对target和gse21257、gse32981及gse49003数据集的degs(包括上调和下调基因)进行反向药物筛选,分别得到51、15、92和64个候选的小分子物质,四组数据集之间共有的小分子物质很少,此外得到一个小分子物质brd-a15079084在3组数据集上均出现;
在dgidb药物与基因相互作用数据库,分析15个与预后存在显著关系的mrs可能相互作用的药物,发现有2个基因tlr7和ifngr1具有潜在的相互作用药物信息,并且这些药物与基因之间的相互作用关系主要为促进激活关系,考虑到tlr7为有利的预后因子,在转移组中表达被抑制,因此这些促进tlr7基因表达活性的药物可能会在治疗osteosarcoma转移上发挥一定作用;
在cmap数据库,看到4组数据集共有的药物也较少,但都注释到了一个共同的药物:紫霉素(viomycin),该物质是一种来自链霉菌的强碱性肽,能够抑制蛋白质合成的化合物,用于治疗结核病,此外另有12个药物:adiphenine、alexidine、camptothecin、cp-320650-01、digoxigenin、genistein、gw-8510、h-7、iopamidol、nadolol、prestwick-691、sulfamonomethoxine在至少3个数据集上出现。
优选的,所述s1中的从target项目下载最新的骨肉瘤(os)临床信息和基因表达数据,共包含89个样本,从geo上下载gse21257、gse32981、gse39055和gse49003芯片数据,分别包含了53、23、37和12个具有转移信息的肿瘤样本或细胞系。
优选的,所述s2中还使用了ranktest对转移(metastatic)与未转移(non-metastatic)分组样本的基因表达水平差异进行了显著性检验,可以看到这些degs的ranktestp均<0.05。
优选的,所述s4中在l1000fwd工具注释的结果中,如果提供的上调基因集/下调基因集与数据库记录的上调基因集/下调基因集一致,则该基因集对应的小分子物质定义为similar,反之在定义为opposite。
优选的,比较4个数据集筛选到的mrs重叠情况,发现4组之间共有的mrs非常少,这反映尽管同为osteosarcoma但是在转移模式上却表现出广泛的异质性,虽然这种共有的基因很少,但在mrs的go富集上却观察到非常高的一致性,这些mrs均显著富集到免疫相关的goterm上。
(三)有益效果
与现有技术对比,本发明具备以下有益效果:
该种骨肉瘤转移相关基因signature研究及潜在药物挖掘方法,分析了转移组与未转移组osteosarcoma样本的总体生存时间发现,发现转移对osteosarcoma的预后有重要影响,比较转移和未转移样本的差异表达基因,筛选出了一组与转移有关的基因,表明免疫系统的特定模式改变与osteosarcoma的转移有重要关联,从基因表达水平变化特征看,免疫相关的基因在转移组普遍表现为下调,推测免疫系统的抑制可能有助于osteosarcoma的转移,进一步说明,一些基因(包含免疫相关基因)的表达抑制,可能有助于osteosarcoma转移,mrs的预后分析进一步鉴定出了一组与预后有显著的关系基因,为进一步挖掘osteosarcoma转移的治疗提供了可能的选择。
附图说明
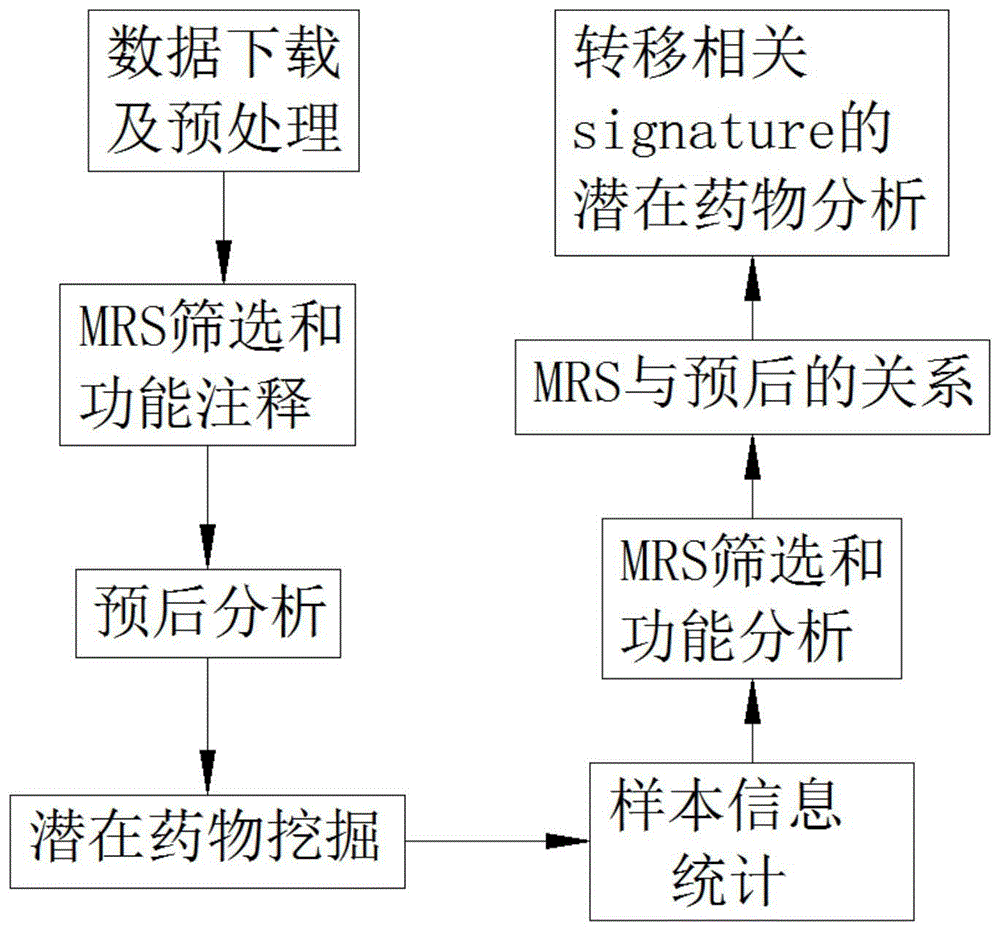
图1为本发明研究方法流程图。
具体实施方式
下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
请参阅图1,一种骨肉瘤转移相关基因signature研究及潜在药物挖掘方法,包括以下步骤:
s1、数据下载及预处理:
对os的target数据做以下几步预处理:
去掉没有临床信息或者总生存时间<30天的样本;
去掉正常组织样本数据;
去掉在超过一半样本中表达量为0的基因;
对geo数据做以下几步预处理:
1)去掉正常组织样本数据,只保留肿瘤组织数据;
2)将总生存时间(gse39055为recurrence–freesurvival)为year或month的转成days;
3)利用bioconductor包将芯片探针map到人类基因symbol;
进一步的,s1中的从target项目下载最新的骨肉瘤(os)临床信息和基因表达数据,共包含89个样本,从geo上下载gse21257、gse32981、gse39055和gse49003芯片数据,分别包含了53、23、37和12个具有转移信息的肿瘤样本或细胞系。
s2、mrs筛选和功能注释:
由于target和gse21257、gse32981、gse39055及gse49003均为芯片数据,下载的表达谱数据均为normalized后的,转移(metastatic)与未转移(non-metastatic)分组样本使用limma工具进行差异表达分析,由于target和gse32981数据的degs数量极少,这里取pvalue<0.01且log2foldchange绝对值>0.5作为degs阈值,对于gse21257和gse49003数据,取fdr<0.05且log2foldchange绝对值>0.5作为degs阈值,将4组数据集得到的degs定义为转移相关基因(metastasesrelatedsignature,mrs),使用rpackageclusterprofiler(v3.8)对mrs进行go、kegg功能注释和富集分析,取qvalue(fdr)<0.05作为显著富集的阈值;
进一步的,s2中还使用了ranktest对转移(metastatic)与未转移(non-metastatic)分组样本的基因表达水平差异进行了显著性检验,可以看到这些degs的ranktestp均<0.05。
s3、免疫基因的表达谱筛选:
使用单因素cox风险回归模型分析mrs与转移事件的预后关系,考虑到具有预后信息的target和gse21257数据集样本均偏少(85/53),这里取logrankp<0.1为显著性阈值,使用kaplan-meierplotter方法分析并展示分组事件的预后总生存曲线,上述分析的r版本为3.5.1;
s4、潜在药物挖掘:
利用l1000fwd工具挖掘与degs有关的小分子物质,l1000数据库记录了超过16000中药物或小分子物质处理cancercelllines后所诱导的上调和下调基因(signatures),通过比较鉴定出的上调和下调degs与数据库中记录的上调/下调基因,可以反向推出哪些小分子物质可能与问题有关(例如,metastatic/primary之间表达差异),dgidb数据库记录了超过40000个基因和10000种药物的相互作用信息,与l1000数据库不同,dgidb记录的是特定基因及与其存在相互作用的药物关联信息,根据用户提供的基因,可以找到与该基因存在相互作用的药物,在使用cmap查询之前,使用bioconductorrpackage将4个亚型mrs基因symbol转换为affymetrix探针id,在cmap数据库中,预测具有显著负分数的药物将成为os的新型治疗药物,cmap使用基因集富集分析算法计算连接性分数,平均得分≤0.65用于识别潜在的候选药物,对于cmap提供的每个探针,保留amplitude≤-0.67或>0.67的为显著表达异常探针用于pathway分析(anamplitudeof±0.67representsatwo-foldchangebetweenthetreatmentandthecontrol);
进一步的,s4中在l1000fwd工具注释的结果中,如果提供的上调基因集/下调基因集与数据库记录的上调基因集/下调基因集一致,则该基因集对应的小分子物质定义为similar,反之在定义为opposite。
s5、样本信息统计:
经过预处理后的os数据总共得到209个样本,其中target有85个满足条件样本,gse21257、gse32981、gse39055及gse49003分别有53、23、12和36个满足条件样本,从4个数据集的age分布来看,medianage均在20岁以下,alive和dead事件的患者medianoverallsurvivaltime也存在显著差异,比较target和gse21257转移和非转移样本的总生存时间预后情况可以看到二者也存在显著差异,此外转移与非转移样本recurrence预后也存在显著差异,未转移样本的总体生存时间也较转移样本的显著更高;
s6、mrs筛选和功能分析:
使用target和gse21257、gse32981及gse49003数据集,我们分析了转移与未转移样本的差异表达表达基因(degs),这里将degs定义为转移相关signatures(mrs),从上调和下调基因数量看,在非转移性样本中上调基因大于下调基因数,这说明转移的发生可能更倾向于涉及到某些通路的活性抑制而非增强,由于gse49003数据集为具有转移能力和无转移能力的细胞系,推测这可能导致其鉴定出的degs与target和gse21257、gse32981的有较大差异;
进一步的,比较4个数据集筛选到的mrs重叠情况,发现4组之间共有的mrs非常少,这反映尽管同为osteosarcoma但是在转移模式上却表现出广泛的异质性,虽然这种共有的基因很少,但在mrs的go富集上却观察到非常高的一致性,这些mrs均显著富集到免疫相关的goterm上,这表明免疫系统的改变(根据up/down的基因推测可能为免疫系统的抑制)与osteosarcoma的转移有密切关系,这与已有的资料也是一致的。
s7、mrs与预后的关系:
根据target和gse21257数据集预后(总生存时间)信息,使用单因素cox回归分析方法分析target/gse49003和gse21257/gse49003共有的18个mrs与预后的关系,发现其中有15个mrs在target或gse21257数据集上与预后有显著关系,根据hr(hazardratio)>1或<1将这些基因分为不利预后因子和有利预后因子,发现有利的预后基因在转移组上均表现出表达水平下调,而不利的预后基因在转移组上表现出表达水平上调,取在target和gse21257数据集上均与预后存在显著关系的il10ra和tlr7基因,根据表达量中位值将样本分成高表达组(l2)、低表达组(l1),kmploter结果表明高表达组样本预后显著好于低比表达组;
s8、转移相关signature的潜在药物分析;
l1000数据库提供超过16000种药物和小分子物质在大约1000个肿瘤细胞系上诱导的基因表达特征(上调/下调),利用l1000fwd工具对target和gse21257、gse32981及gse49003数据集的degs(包括上调和下调基因)进行反向药物筛选,分别得到51、15、92和64个候选的小分子物质,四组数据集之间共有的小分子物质很少,这可能与它们极少的degs有关,此外得到一个小分子物质brd-a15079084在3组数据集上均出现,可能意味着该小分子物质会是一个潜在的药物。
在dgidb药物与基因相互作用数据库,分析15个与预后存在显著关系的mrs可能相互作用的药物,发现有2个基因tlr7和ifngr1具有潜在的相互作用药物信息,并且这些药物与基因之间的相互作用关系主要为促进激活关系,考虑到tlr7为有利的预后因子,在转移组中表达被抑制,因此这些促进tlr7基因表达活性的药物可能会在治疗osteosarcoma转移上发挥一定作用。
在cmap数据库,看到4组数据集共有的药物也较少,但都注释到了一个共同的药物:紫霉素(viomycin),该物质是一种来自链霉菌的强碱性肽,能够抑制蛋白质合成的化合物,用于治疗结核病,此外另有12个药物:adiphenine、alexidine、camptothecin、cp-320650-01、digoxigenin、genistein、gw-8510、h-7、iopamidol、nadolol、prestwick-691、sulfamonomethoxine在至少3个数据集上出现,虽然这些潜在的候选药物已经广泛应用于临床中,然而对于它们可能在其他疾病上的应用仍需要进一步研究。
尽管已经示出和描述了本发明的实施例,对于本领域的普通技术人员而言,可以理解在不脱离本发明的原理和精神的情况下可以对这些实施例进行多种变化、修改、替换和变型,本发明的范围由所附权利要求及其等同物限定。
- 还没有人留言评论。精彩留言会获得点赞!