针对多样本变异图的变体调用的制作方法
1.本公开总体涉及生物信息学,并且更具体地但不排他地涉及处理与人类基因组相关的信息。
背景技术:
2.已经提出了各种方法来转变原始基因组数据。一种方法依赖于使用事实上的参考基因组将读段映射到线性参考人类基因组。然而,事实上的参考基因组仅代表人类群体的微小子集,并且因此不能可靠地反映存在的巨大等位基因多样性。这导致所谓的等位基因偏差,其中,未表示与参考基因组的正常(例如,健康)偏差。这导致不相似的那些样本的不良的读段比对准确性。
3.另一种方法提出通过对样本进行测序并且然后将其与基于图的参考基因组进行比较来变换原始基因组数据。基于图的参考基因组可以将比事实上的参考基因组更多的人类基因组并入单个结构中。然而,基于图的方法也具有显著的缺点。例如,将读段映射到参考图的方式在方法上可以与用于线性参考基因组的现有方法不同。此外,基于图的方法不能充分补偿等位基因偏差,这使得不适合于许多应用。
技术实现要素:
4.下面呈现了各种范例实施例的简要概述。在以下概述中可以做出一些简化和省略,其旨在突出并介绍各种范例实施例的一些方面,但不限制本发明的范围。足以允许本领域的一般技术人员产生且使用发明性概念的范例实施例的详细描述将跟随在稍后的章节中。
5.根据一个或多个实施例,一种用于处理信息的方法包括:对基于图的参考基因组中的节点进行排序;将标识信息分配给经排序的节点;将深度值分配给所述经排序的节点中的相应节点;确定参考基因组路径和一条或多条变异路径(variation path);并且基于分配给所述一条或多条变异路径上的节点的所述深度值,确定所述基于图的参考基因组中的一个或多个变体。可以所述节点在通过所述基于图的参考基因组的预定方向上被拓扑地排序。
6.分配所述深度值可以包括将初始值分配给所述节点中的第一节点,并且针对所述节点中的每个后续节点,对从所述每个后续节点到所述第一节点的节点数量进行计数,沿着所述参考基因组路径、所述变异路径中的一个或多个、或所述参考基因组路径与所述变异路径中的一个或多个的组合采取返回所述第一节点的最直接路径。如果前趋集合不为空,那么可以采取到第一节点的直接路径。否则,可以取公式4的条件4(下面讨论)(例如,其所有后继中的最小深度值减1)。
7.确定所述参考基因组路径和所述一条或多条变异路径可以包括执行贯穿所述基于图的参考基因组的节点的全局搜索以确定所述参考基因组路径;并且对沿着所述参考基因组路径的节点执行局部搜索以确定变异路径,所述变异路径中的每个包括一个或多个局
部路径。所述一个或多个局部路径中的每个将所述参考基因组路径上的节点中的至少一个连接到所述参考基因组路径之外的节点中的至少一个,或连接所述参考基因组路径之外的节点中的至少两个。
8.所述一个或多个变体可以包括以下中的至少一个:到所述基于图的参考基因组中的插入;所述基于图的参考基因组中的缺失;或所述基于图的参考基因组中的替换。所述方法还可以包括基于所述一个或多个变体确定模式,其中,所述模式对应于对象感染疾病的倾向或用于执行针对药物批准的临床试验的指南。
9.根据一个或多个其他实施例,一种用于处理信息的系统包括存储器和处理器,所述存储器被配置为存储指令,所述处理器被配置为执行所述指令以:(a)对基于图的参考基因组中的节点进行排序,(b)将标识信息分配给经排序的节点,(c)将深度值分配给所述经排序的节点中的相应节点,(d)确定参考基因组路径和一条或多条变异路径,并且(e)基于分配给所述一条或多条变异路径上的节点的所述深度值,确定所述基于图的参考基因组中的一个或多个变体。可以所述节点在通过所述基于图的参考基因组的预定方向上被拓扑地排序。
10.所述处理器可以通过以下操作来分配所述深度值:将初始值分配给所述节点中的第一节点;并且针对所述节点中的每个后续节点,对从所述每个后续节点到所述第一节点的节点数量进行计数,沿着所述参考基因组路径、所述变异路径中的一个或多个、或所述参考基因组路径与所述变异路径中的一个或多个的组合采取返回所述第一节点的最直接路径。
11.所述处理器可以通过以下操作来确定所述参考基因组路径和所述一条或多条变异路径:执行贯穿所述基于图的参考基因组的节点的全局搜索以确定所述参考基因组路径;以及执行针对沿着所述参考基因组路径的节点的局部搜索以确定变异路径,所述变异路径中的每个包括一个或多个局部路径。所述局部路径中的每个可以将所述参考基因组路径上的节点中的至少一个连接到所述参考基因组路径之外的节点中的至少一个,或者连接所述参考基因组路径之外的节点中的至少两个。所述一个或多个变体可以包括以下中的至少一个:到所述基于图的参考基因组中的插入;所述基于图的参考基因组中的缺失;或所述基于图的参考基因组中的替换。
12.根据一个或多个其他实施例,一种存储指令的非瞬态计算机可读介质,所述指令用于引起处理器执行操作,所述操作包括对基于图的参考基因组中的节点进行排序,将标识信息分配给经排序的节点,将深度值分配给所述经排序的节点中的相应节点,确定参考基因组路径和一条或多条变异路径,并且基于分配给所述一条或多条变异路径上的节点的所述深度值,确定所述基于图的参考基因组中的一个或多个变体。可以所述节点在通过所述基于图的参考基因组的预定方向上被拓扑地排序。
13.分配所述深度值可以包括将初始值分配给所述节点中的第一节点,并且针对所述节点中的每个后续节点,对从所述每个后续节点到所述第一节点的节点数量进行计数,沿着所述参考基因组路径、所述变异路径中的一个或多个、或所述参考基因组路径和所述变异路径中的一个或多个的组合采取返回所述第一节点的最直接路径。
14.确定所述参考基因组路径和所述一条或多条变异路径可以包括执行贯穿所述基于图的参考基因组的节点的全局搜索以确定所述参考基因组路径;以及执行针对沿着所述
参考基因组路径的节点的局部搜索以确定变异路径,所述变异路径中的每个包括一个或多个局部路径。所述一个或多个局部路径中的每个可以将所述参考基因组路径上的所述节点中的至少一个连接到所述参考基因组路径之外的节点中的至少一个,或者连接所述参考基因组路径之外的节点中的至少两个。所述一个或多个变体可以包括以下中的至少一个:到所述基于图的参考基因组中的插入;所述基于图的参考基因组中的缺失;或所述基于图的参考基因组中的替换。所述方法还可以包括基于所述一个或多个变体确定模式,其中,所述模式对应于对象感染疾病的倾向或用于执行针对药物批准的临床试验的指南。
附图说明
15.附图与下文的详细描述一起并入在说明书中且形成说明书的一部分,并且用以进一步示出包括所主张发明的概念的实施例且阐释那些实施例的各种原理和优点,在附图中,在单独的视图中相同的附图标号始终指代相同或功能上相似的元件。
16.在以下说明书中更充分地公开这些和其他更详细和具体的特征,其中,参考了附图,在附图中:
17.图1图示了用于调用遗传信息中的变体的方法的实施例;
18.图2图示了基于图的参考基因组的示例;
19.图3图示了可以如何将深度值分配给图中的节点的示例;
20.图4图示了图中的参考基因组路径的示例;
21.图5a至5e图示了确定变异路径的局部搜索的示例;
22.图6图示了包括插入的变异路径的示例;
23.图7图示了包括缺失的变异路径的示例;
24.图8图示了包括替换的变异路径的示例;并且
25.图9图示了用于从基因组数据确定变体的系统的实施例。
具体实施方式
26.应理解,附图仅为示意性的并且不按比例绘制。还应理解,贯穿附图使用的相同附图标记表示相同或相似的部件。
27.描述和附图示出各种示例实施例的原理。将了解,本领域技术人员将能够设计各种布置,尽管本文中未明确地描述或示出所述布置,但其体现本发明的原理且包括于本发明的范围内。此外,本文中所述的所有示例主要明确地意在用于教学目的以辅助读者理解本发明的原理及由(一个或多个)发明人所提供的概念,从而深化本领域,且所有示例不应解释为限于此类特定所述示例及条件。此外,如本文中所使用,术语“或”指代非排他性或(即,和/或),除非另外指明(例如,“否则”或“或在替代方案中”)。并且,本文所描述的各种示例实施例不一定相互排斥,因为一些示例实施例可与一个或多个其他示例实施例组合从而形成新的示例实施例。例如“第一”、“第二”、“第三”等描述词不旨在限制所讨论的元素的次序,且用于区分一个元素与下一元素,并且通常可互换。诸如最大值或最小值的值可以被预先确定,并基于应用而被设置为不同的值。
28.示例实施例包括用于对遗传信息执行变体调用的系统和方法,其涉及确定已经并且到基于图的基因组中的样本上的变体的存在和(一种或多种)类型。这些实施例中的一个
或多个包括:对基于图的参考基因组中的节点进行排序,将标识信息分配给经排序的节点,将深度值分配给所述经排序的节点中的相应节点,确定参考基因组路径和一条或多条变异路径,并且基于分配给所述一条或多条变异路径上的节点的所述深度值,确定所述基于图的参考基因组中的一个或多个变体。在至少一个实施例中,所述系统和方法可以以减少或解决存在于用于转变原始基因组数据的现有方法中的等位基因偏差问题的方式实施。实施例还可以适用于许多研究应用,并且特别是需要识别新颖的高影响变体的研究应用。
29.系统和方法实施例可以通过在基于图的参考基因组上识别一个或多个变体来实现这种改善的性能,所述基于图的参考基因组是从并入到单个结构中的一组(例如,数千或数百万)健康和多样化人类基因组构建的。这种基于图的基因组可以表示人类基因组的多样性的更完整的表示。在一个实施例中,可以仅识别一种类型的感兴趣变体。在另一实施例中,可以识别多种变体类型。(一种或多种)变体类型可以包括例如基于图的参考基因组中的表型的插入、缺失(deletion)和替换。当共同考虑时,可以分析变体以找出指示人在他或她的一生中发展一种或多种疾病(例如,癌症、精神疾病等)的倾向的趋势和模式。为了获得新药的批准的目的,变体的确定也可用于管理临床试验。
30.图1图示了用于调用遗传信息中的变体(包括可以在参考基因组中找到的变体)的方法的实施例。虽然许多实施例是关于人类基因组描述的,但是可以应用其他实施例来确定动物基因组中的变体。参考基因组可以基于数千或数百万个样本生成,其中,后者针对提供表示一个或多个一般或特定感兴趣群体的遗传信息的扩展指示的目的是优选的。
31.在110处,该方法包括获得待分析的参考基因组。参考基因组可以是例如基于de bruijn图技术、非循环图技术、smith-waterman技术、或另一技术或方法生成的基于图的人类参考基因组。这样的图包括多个节点,每个节点对应于基因组中的不同遗传信息。该图可以包括表示基因组中的不同节点或片段之间的关系的边。根据本文中的实施例,可以分析节点和路径以调用变体。
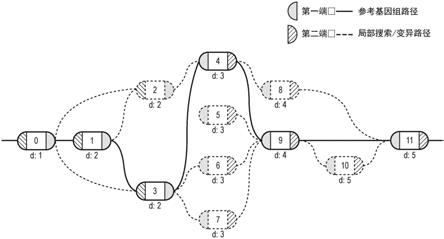
32.图2图示了包括编号为0至11的十二个节点的基于图的参考基因组的示例。虽然在图1的图中仅图示了十二个节点,但是应当理解,系统和方法实施例可以应用于具有不同数量的节点(例如,小于或大于十二个)的图。在一个实施例中,图可以具有数百或数千个节点,或者系统和方法实施例可以仅局部地聚焦于这样的图中的减少数量的节点。
33.参考基因组中的节点0至11可以对应于(或指示)相应数量的表型,并且由两种类型的路径中的至少一种连接。第一类型的路径是指示表示参考群体的节点(或表型)的连接的参考基因组路径。沿着参考基因组路径连接的节点被认为对应于一般或预定群体中的对象的那些表型。例如,群体可以包括医学或生物学标准认为是正常和健康群体的群体或由其组成。在另一实施方式中,群体可以包括具有遗传性状和/或其他感兴趣特征(无论是否被认为是正常的)的特定集合的对象或由其组成。
34.第二类型的路径被认为是变体路径(variant path),并且可以对应于不被包括在参考基因组路径中的所有路径。每个变体路径可以图形地显示为直接或间接地连接到沿着参考基因组路径的至少一个节点。该连接可以涉及变体路径从参考基因组路径上的节点射出,从另一节点(在参考路径或变体路径上)进入参考基因组路径上的节点,或连接在参考基因组路径上的两个节点之间。如下面将更详细描述的,变体路径上的每个节点也可以连接到上述类型的多个节点。就遍历图而言,参考基因组和变体路径可以是双向的或单向的、
或在图的不同部分处的两者的组合。为了图示性目的,图中的所有路径将被讨论为双向的,从左到右以及从右到左遍历十二个节点。因而,基于图的参考基因组中的每一节点可以包括在节点的相应侧中的两个末端。
35.在采集基于图的参考基因组之后,可以实施系统和方法实施例以标记图中的变体。待标记的变体可以是预定类型或多种类型。可以在图中标记(或调用)的变体的示例包括但不限于插入、缺失和替换,如下面更详细描述的。
36.为了识别变体的类型,可以应用该系统和方法来解决以下问题:给定多样本变异图g=(v,e)(其包括节点的有限集合v={v1,v2,...,vn}和边的集合),提取相对于参考基因组路径的变异。在图2的该示例图中,n等于12,对应于十二个节点。就边e而言,在图论中,图的节点的度是入射到节点的边的数量。如果不存在从当前节点开始的变异,则可以满足以下引理:如果内部节点(例如,除了参考基因组路径上的开始节点和结束节点之外)具有最大度≤2,则该节点可以不用作变异路径的开始索引或结束索引。这可以如下地解释。
37.在图2中,图g被构建为具有沿着参考基因组路径的线性连接,并且沿着参考基因组路径布置的节点可以不被认为具有任何变异。在一个实施例中,沿着参考基因组路径的节点可以如下地定义。内部节点可以是与前一节点和下一节点连接的节点。因此,每个内部节点可以具有2的最大度。对于参考基因组路径的开始节点和结束节点中的每个,度可以是1。在图2中,开始节点是节点0,结束节点是节点11。考虑到这种理解,该方法可以继续如下。
38.在120处,对图g中的节点执行排序操作。在一个实施例中,图g中的所有节点可以相对于参考基因组路径被拓扑地排序(例如,开始节点、内部节点和结束节点)。排序可以在预定方向上执行。如果参考基因组图是双向的,那么可以在正向方向或反向方向上执行排序操作。如果参考基因组图是单向的,那么可以在图的唯一有效方向上执行排序操作。在图2中图示了排序操作的示例。
39.在130处,将标识号(节点-id)分配(或重新分配)给每个节点,使得在预定方向上遍历图,相对于给定节点i的所有前续节点具有小于i的节点-id,并且相对于节点i的所有后继节点具有大于i的节点-id。可以对开始节点、内部节点和结束节点执行该操作。结果是在预定方向上以升序将节点-id分配给图中的节点。在图2中图示了节点分配操作的示例。
40.在140处,一旦节点已经被拓扑排序并且节点-id已经被分配(或重新分配)给图g中的节点,就可以将深度值分配给每个节点。这可以被实现如下。假设以下公式(1)至(3)为真:
41.r={r|r∈v并且r∈参考路径}(1)
42.pi={p|p∈v并且p是节点的前导}(2)
43.si={s|s∈v并且s是节点i的后继}(3)
44.在以上公式中,v对应于节点的有限集合,r对应于位于参考路径中的节点的集合,pi对应于为节点i的前续的节点的集合,si对应于为节点i的后继的节点的集合,并且r、p和s是变量。基于这些假设,可以基于方程组(4)将深度值分配给图g的每个节点。
[0045][0046]
在方程(4)中,i表示节点标识号(节点-id),并且j和l是变量。表达式min
j depth(j)意味着在为节点集合v的一部分的所有节点j上最小化。表达式min
k depth(k)意味着在节点k上最小化。表达式max
l
depth(l)意味着在节点l上最大化。表达式j《l,k《i并且l》i是对上述公式的约束。例如,j《l意味着集合v中的节点-id小于i的所有节点。
[0047]
图3图示了如何使用方程组(4)中阐述的规则将深度值(d)分配给图1中的节点0至11的示例。计算并分配给节点的深度值也可以被理解为节点序列中的计数值,采取可能返回开始节点的最直接路径——无论最直接路径是仅沿着参考基因组路径、仅沿着一条或多条变异路径、还是参考基因组路径和一条或多条变异路径的片段的组合。换句话说,深度值可以是从开始节点移除的节点的数量,其中,基因组参考路径中的开始节点被分配为第一节点。因此,可以如下分配图3的图g中的节点。
[0048]
第一节点(i=节点-id=0)被分配深度值1(d:1),因为该节点是沿着图g中的参考基因组路径布置的节点序列(计数值=1)中的第一节点(开始节点)。
[0049]
第二节点(i=节点-id=1)被分配深度值2(d:2),因为该节点是从开始节点开始的节点序列(计数值=2)中的第二节点,采取最直接路径返回开始节点。在这种情况下,返回开始节点的最直接路径是处于沿着参考基因组路径的路径210。
[0050]
第三节点(i=节点-id=2)被分配深度值2(d:2),因为该节点是从开始节点开始的节点序列(计数值=2)中的另一个第二节点,采取最直接路径返回开始节点。在这种情况下,返回开始节点的最直接路径是通过变异路径220(其绕过第二节点1)。
[0051]
第四节点(i=节点-id=3)被分配深度值2(d:2),因为该节点是从开始节点开始的节点序列(计数值=2)中的另一个第二节点,采取最直接路径返回开始节点。在这种情况下,返回开始节点的最直接路线由是通过变异路径225。
[0052]
第五节点(i=节点-id=4)被分配深度值3(d:3),因为该节点是从开始节点开始的节点序列(计数值=3)中的第三节点,采取最直接路径返回开始节点。在这种情况下,返回开始节点的最直接路线由是通过变异路径220和230。该路线经过内部节点2。
[0053]
第六节点(i=节点-id=5)被分配深度值3(d:3),因为该节点不具有经由其前续(其前续集为空)到开始节点的直接路径。基于公式4,条件4,该节点的深度值必须经由路径240从其后继节点计算。其后继是节点9,并且节点9具有深度值4。因此,其深度值为3。
[0054]
第七节点(i=节点-id=6)被分配深度值3(d:3),因为该节点是从开始节点开始的节点序列(计数值=3)中的第三节点,采取最直接路径返回开始节点。在这种情况下,返回开始节点的最直接路线由是通过变异路径225和255。该路线经过内部节点3。
[0055]
第八节点(i=节点-id=7)被分配深度值3(d:3),因为该节点是从开始节点开始的节点序列(计数值=3)中的第三节点,采用返回开始节点的最直接路径。在这种情况下,返回开始节点的最直接路径是通过变异路径220和260。该路线经过内部节点3。
[0056]
第九节点(i=节点-id=8)被分配深度值4(d:4),因为该节点是从开始节点开始的节点序列(计数值=4)中的第四节点,采取最直接路径返回开始节点。在这种情况下,返
回开始节点的最直接路线由是通过变异路径220、230和265。该路线经过内部节点2和内部节点4。
[0057]
第十节点(i=节点-id=9)被分配深度值4(d:4),因为该节点是从开始节点开始的节点序列(计数值=4)中的第四节点,采取最直接路径返回开始节点。在这种情况下,返回开始节点的最直接路线是通过参考基因组路径的部分250和变异路径220和230。该路线经过内部节点2和内部节点4。
[0058]
第十一节点(i=节点-id=10)被分配深度值5(d:5),因为该节点是从开始节点开始的节点序列(计数值=5)中的第五节点,采取最直接路径返回开始节点。在这种情况下,返回开始节点的最直接路线是通过变异路径220、230和270以及参考基因组路径的部分250。该路线经过内部节点9、内部节点4和内部节点2。
[0059]
第十二节点(i=节点-id=11)是结束节点,并且被分配深度值5(d:5),因为该节点是从开始节点开始的节点序列(计数值=5)中的第五节点,采取最直接路径返回开始节点。在这种情况下,返回开始节点的最直接路线是通过变异路径220和230以及参考基因组路径250和275的部分。该路线经过内部节点2、内部节点4和内部节点9。(存在也将为节点11产生该深度值的其他路线)。
[0060]
使用这种方法和方程组(4),可以如表1所示的那样计算和分配基于图的基因组中的节点的深度值。
[0061][0062]
表1
[0063]
在150处,可以使用在两阶段循环搜索来确定基于图的参考基因组中的参考基因组路径和变异路径。在一个实施例中,可以在将深度值分配给图中的节点的操作140之后执行操作150。在另一实施例中,操作150可以与操作140同时执行。
[0064]
在执行两阶段循环搜索时,可以执行第一类型的搜索以在图中定位全局搜索路径,其对应于参考基因组路径。这可以解释如下。最初,图构造从参考基因组开始,并且然后执行多次迭代。在与图对准的不同参考基因组样本的每次迭代中,将变异添加到图。因此,参考基因组图在每次迭代中变得丰富。在添加这些参考节点时,将标志物分配给节点,以便将来可以将其识别为参考基因组的一部分。
[0065]
在图3的图中,参考基因组路径是连接节点0
→1→3→4→9→
11的路径,如图4所强调的。为了阐明,由全局搜索产生的参考基因组(或全局搜索)路径是遍历上述节点的隔离路径。全局和局部搜索可能不是不相交的操作。例如,通过沿着参考基因组路径遍历来执行全局搜索。如果遇到节点的最大度》2(例如,对于参考路径的开始和结束节点,最大度》1),则可以启动局部搜索。
[0066]
一旦执行全局搜索以确定参考基因组路径,就可以执行两阶段循环搜索的第二类型的搜索。执行第二类型的搜索以确定图中的变异路径。在执行此搜索时,应理解,多条变异路径可以穿过位于沿着参考基因组路径或变异路径的单个节点或与其相关联。因此,可以在第二阶段搜索中提取的每个路径可以被认为是有效的变异。
[0067]
在执行这种搜索时,从左到右遍历图,当在全局搜索期间找到任何内部节点的最大度≥2(或参考基因组路径上的开始节点和结束节点的最大度≥1)时,然后可以执行局部搜索。因为图g已经被拓扑排序并且节点-id已经被分配(例如,以递增的顺序),所以如果节点具有严格大于当前节点i的节点-id(使用先前执行的节点-id的重新分配),则可以访问该节点。当发现沿着参考基因组路径的节点时,可以停止局部搜索,并且可以添加(一个或多个)相关联的遍历路径作为(一个或多个)变异。因此,可以对至少一些节点执行多个局部搜索。
[0068]
如前所述,操作140和150可以同时执行。在这种情况下,在确定参考基因组路径之后,以沿着参考基因组路径的第一节点(其为开始节点0)开始,在预定搜索方向(例如,从左到右)上遍历图。此时,计算开始节点0的深度值。然后,相对于节点0执行局部搜索以确定源自节点0的(一个或多个)变异路径。然后计算沿着那些(一个或多个)变异路径的节点的深度值。随后,计算沿着参考基因组路径的下一个节点的深度值。然后相对于下一个节点执行局部搜索,以确定源自该节点的(一个或多个)变异路径。然后计算沿着那些(一个或多个)变异路径的节点的深度值。该过程针对沿着参考基因组路径的后续节点继续,直到针对所有节点计算深度值并且已经确定图中的所有变异路径。
[0069]
图5a至5e图示了可以执行以确定基于图的参考基因组中的变异路径的局部搜索的示例。在图5a至5e中,在已经计算了图中的所有节点的深度值与全局搜索的性能相结合之后,执行这些局部搜索。在全局搜索期间,如果沿着参考基因组(或全局)路径的内部节点的深度值相对于开始节点(或结束节点,如果在相反方向上遍历图)》2,那么针对该内部节点发起局部搜索。如果内部节点被认为是相对于不是开始节点或结束节点的节点,那么如果节点的度》1,则执行局部搜索。
[0070]
参考图5a,确定节点4在全局路径搜索期间具有3的深度值。因此,已经满足了对节点4执行局部搜索的前提条件。因此,从节点0(开始节点)执行局部搜索,并且涉及从该节点遍历局部路径。当节点被识别为在全局路径中时,终止局部搜索。在当前情况下,通过局部搜索找到两条变异路径。第一变异路径在节点0处开始,并且分别沿着局部路径220和局部路径230穿过节点2到节点4。第一变异路径可以表示为0
→2→
4。第二变异路径从节点0开始并沿着局部路径225传递到节点3。第二变异路径可以表示为0
→
3。在该示例中,沿着两个局部路径的变体对应于不同的类型,例如,将第一变异路径确定为对应于替换或结构变异,并且将第二变异路径确定为对应于缺失。
[0071]
参考图5b,还可以从节点1开始执行局部搜索。这样的搜索将识别包括沿着局部路
径230和280的节点1、2和4的变异路径。该变异路径可以表示为1
→2→
4。尽管开始节点(节点1)和结束节点(节点4)之间的深度值差为1,但是节点3具有位于开始节点和结束节点(1和4)之间的重复深度值2。如下面更详细地解释的,变异路径1
→2→
4可以被分类为替换或结构变异。
[0072]
参考图5c,还可以从节点3开始执行局部搜索。这样的搜索确定包括沿着局部路径255和285的节点3、6和9的第一变异路径以及包括沿着局部路径260和290的节点3、7和9的第二变异路径。在这两种情况下,开始节点(节点3)和结束节点(节点9)的深度值之间的差》1。如下面更详细地讨论的,在这种情况下,两条变异路径可以被分类为相同类型的变体,即替换或结构变异。
[0073]
参考图5d,还可以从节点4开始执行局部搜索。这样的搜索确定包括沿着局部路径265和295的节点4、8和11的变异路径。该变异路径可以表示为4
→8→
11。如下面将更详细讨论的,与该路径相关联的变体是替换或结构变异。
[0074]
参考图5e,还可以从节点9开始执行局部搜索。这样的搜索确定包括沿着局部路径270和299的节点9、10和11的变异路径。该变异路径可以表示为9
→
10
→
11。开始节点(节点9)和结束节点(节点11)的深度值之间的差值≤1。因此,在该开始节点和结束节点之间的全局搜索路径中没有具有重复深度值的节点。如下面更详细讨论的,与变异路径9
→
10
→
11相关联的变体是插入变异。
[0075]
在160处,一旦已经分配了深度值并且确定了变异路径,就可以确定对应于连接到沿着参考基因组路径的节点的节点的变体的类型。在一个实施例中,图可以仅包括一种类型的变体。在其他实施例中,图可以包括多种类型的变体。在图3的示例中,该图包括三种类型的变体:插入、缺失和替换。
[0076]
图6图示了变体节点10已经插入到节点9和11之间的参考基因组路径中(例如,作为分支或偏离)的示例。在一个实施例中,确定基于图的人类基因组g中插入的存在可以涉及检查沿着参考基因组路径定位的相邻节点。
[0077]
更具体地,可以执行搜索以定位沿着参考基因组路径的相邻节点,所述相邻节点连接到不在参考基因组路径上的节点,并且不在基因组路径上的节点通过一条或多条变异路径连接到相邻节点,所述一条或多条变异路径也可以被称为局部搜索路径。当找到这样的一对相邻节点时,当相邻节点之间的深度值的差≤1时,可以确定存在节点的插入。在这种情况下,局部搜索路径包括相邻节点之间的一个或多个节点,但是全局搜索路径不包括相邻节点之间具有重复深度值的任何节点。在这种情况下,变异可以被描述为对应于局部搜索路径上的节点的插入。
[0078]
在图6中图示了这样的情况,其中,节点9(节点-id=9)和节点11(节点-id=11)沿着参考基因组路径的部分275彼此相邻,并且节点10沿着包括连接相邻节点的局部搜索路径270和299的变异路径在节点9和11之间。在节点9和节点11之间没有具有重复深度值的节点。局部搜索路径可以表示为9
→
10
→
11。在确定节点10是否构成插入时,必须确定节点9和节点11的深度值。节点9已被分配深度值4(d:4),并且节点11已被分配深度值5(d:5)。因此,节点9和节点11的深度值之间的差值因此≤1。因此,可以确定节点10(例如,节点10的内容,其可以是表型)是插入的变体或序列。
[0079]
图7图示了确定沿着连接参考基因组路径上的节点的变异(或局部搜索)路径缺失
的节点的示例。缺失的节点可以是也在参考基因组路径上的节点之间的内部节点。
[0080]
更具体地,确定基于图的人类基因组g中的缺失的存在最初可以涉及检查沿着参考基因组路径的节点。当内部节点位于参考基因组路径上的两个其他节点之间时,确定是否存在连接两个其他节点的变异(或局部搜索)路径。两个其他节点本身可以是内部节点,或两个其他节点中的一个可以是图g中的开始节点或结束节点。如果内部节点的深度值等于其他两个节点中的一个的深度值,则可以确定内部节点已经从连接其他两个节点的变异(或局部搜索)路径中缺失。
[0081]
参考图7,应用这些操作,确定内部节点1位于节点0和节点3之间,所有节点都沿着参考基因组路径定位,并且具体地是参考基因组路径的部分210和215。另外,确定变异或局部搜索路径225连接节点0和节点3。现在,检查深度值。节点0具有深度值1,节点1具有深度值2,并且节点3具有深度值2。因为内部节点1具有等于节点1或3中的一个的深度值的深度值(在这种情况下,节点1和节点3具有相同的深度值),所以可以确定沿着连接节点1和节点3的变异路径220已经缺失了节点1。
[0082]
图8图示了在基于图的基因组g中发生替换(或结构变异)的示例。节点的替换或结构变异可以对应于沿着变异或局部搜索路径(并且因此不沿着参考基因组路径)的尚未被确定为插入或缺失的所有那些节点。例如,如果变异路径的长度是单核苷酸,则沿着该路径的(一个或多个)节点可以被确定为单核苷酸替换。因此,例如,连接到包括局部搜索路径220和230(例如,0
→2→
4)的变异路径的图8中的节点2可以被确定为沿着参考基因组路径的节点1和节点3的替换。应用这些原理,可以确定节点8是节点9的替换,并且可以确定节点6和节点7中的每一个是节点4的替换。
[0083]
在图1中,在170处,可以处理变体以确定可以用作执行各种应用的基础的模式的存在。例如,变体的模式可以与某些疾病或对象在以后的生活中发展某种疾病的倾向相关。因此,本文公开的实施例可以用作预警检测器,其可以使对象改变他们的生活方式以便生活到更大的年龄。在另一示例中,变体的模式可以用作在新药的批准过程期间在临床试验期间开发指南或对象选择的基础。
[0084]
根据另一示例,基因组变体可以用于多种临床应用中。例如,在种系测试中,本文描述的实施例的实际应用是用于与癌症倾向相关联的变体,诸如乳腺癌的brca1/brca2变体和多种癌症类型的tp53变体。在特定癌症中识别的变体可以用作治疗靶标,例如,具有braf v600e突变的非小细胞肺癌患者可以受益于达拉非尼。
[0085]
在一个实际应用中,倾向于一起出现(例如,共遗传)的变体的集合可以被称为单倍型。在一些情况下,单倍型与特定病症或疾病易感性相关。对于复杂疾病如精神分裂症,存在若干这样的示例,其中,疾病风险与dlg4、comt和其他基因中的单倍型相关联。
[0086]
图9图示了用于从基因组数据确定变体的系统的实施例。该系统包括处理器910、接口920、数据库930、存储器940和显示器950。处理器可以是计算机、工作站、服务器或其他处理或计算设备。处理器可以通过接口920接收基因组数据并将该数据存储在数据库930中。该数据可以以原始形式接收,在此情况下,处理器910可以处理数据以生成如前所述的基于图的参考基因组。在一个实施例中,数据可以已经是图形形式。在这种情况下,处理器910可以将数据存储在数据库中,而无需涉及数据的图形格式的实质处理。
[0087]
存储器940可以存储由处理器执行以便执行包括在本文描述方法实施例中的操作
的指令。通过执行这些指令,处理器910可以确定接收到的基因组数据中变体的存在以及那些变体的类型,如前所述。处理器910可以执行另外的指令以定位可以用作预测例如具有一个或多个变体的个体是否可能在他或她的一生中发展疾病或其他病症的基础的趋势和/或模式。实施例还可以应用于执行其他应用,其非限制性示例在下面更详细地讨论。变体、全局(参考基因组)路径、局部(变异)路径、节点和其他数据的视觉表示可以从处理器910输出到显示器950。
[0088]
根据一个实施例,一种非瞬态计算机可读介质可以存储指令,所述指令在由处理器执行时执行本文描述的方法实施例的操作。计算机可读介质可以是只读存储器、随机存取存储器、闪存或另一类型的存储器。在一个实施例中,计算机可读介质可以对应于图7中的用于使处理器710执行本文描述的实施例的操作的存储器740。
[0089]
示例应用
[0090]
实施例的示例应用涉及群组研究的执行。在这样的应用中,开发一系列免疫治疗药物的制药公司希望识别可以预测对治疗的响应的新颖标志物。研究人员组装了一个由从接受免疫治疗的患者中提取的5000个肿瘤序列组成的大图基因组。每个序列与患者以及该患者的临床数据(包括人口统计和治疗响应)相关联。使用本文描述的实施例,针对每个患者跨图的hla区域(例如,与人类免疫响应相关联的关键区域)确定变体。可以分析或处理所得到的数据以识别特定单倍型(其中,某些变体跨hla-a、hla-b和hla-c基因共遗传)与对多种免疫疗法的阳性响应的强关联。然后,制药公司可以使用该知识作为用于针对新系列免疫治疗药物进行的临床试验的选择准则。
[0091]
尽管已经具体参考本发明的某些示范性方面详细描述了各种示例性实施例,但是应当理解,本发明能够具有其他示例实施例,并且其细节能够在各种明显的方面进行修改。对于本领域技术人员显而易见的是,能够在保持在本发明的精神和范围内的同时做出改变和修改。因此,前述公开内容、说明书和附图仅用于说明目的,而并不以任何方式限制本发明,本发明仅由权利要求来定义。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1