一种基于深度神经网络的融合表型和分子信息的中药处方人工智能评价方法与流程
1.本发明涉及一种基于深度神经网络的融合表型和分子信息的中药处方人工智能评价方法。
背景技术:
2.中药在临床上使用广泛,然而其不合理使用情况也比较严重。一项针对北京市第一中西医结合医院2013年门诊不规范中药饮片处方的分析[1]表明,2400张处方中不规范处方177张,占所抽处方的7.38%。另外一项针对北京积水潭医院2011年1月-2013年5月药师在调配前不合理处方的研究表明[2],663张不合理处方中共有709处不合理因素,其中以下3种情况居多:毒性饮片超量(73.76%),处方开具配伍禁忌未签字(12.13%),处方饮片输机错误(7.76%)。这些统计结果表明中药处方的不合理使用,除了常见的人工操作错误之外,主要是违法配伍禁忌、没有进行辨证论治、没有考虑不良反应等情况,而这些恰恰是比较严重的不合理使用情况。因此,准确地推荐中药处方、降低中药处方的不合理使用率是一个亟待解决的问题。
[0003]
随着人工智能、大数据时代的到来,越来越多的研究关注于使用人工智能的方法挖掘名医经验,从而实现中药处方的人工智能评价。实际上,人工智能技术已经在中医药领域具有了一定的应用,例如基于人工智能的中医“四诊”信息标准化采集、处理与分析,中医体质辨析,中医名医经验的挖掘等等。这些应用在一定程度上促进了中医的客观化和标准化。因此,人工智能技术应用于中药处方的人工智能评价即是中药临床精准使用所需,也是人工智能、大数据时代下多学科交叉发展所孕育的一大趋势。人工智能技术应用于中药处方人工智能评价的优势主要体现在:一方面人工智能技术的使用能够实现已经数据化的历代中医病案资料以及名师的临床诊疗记录的挖掘,从而更好地总结和传承名医的用药经验;另外一方面,能够促进临床中药处方的合理使用,降低中药处方不合理使用率,提高诊疗效率。
[0004]
目前,人工智能在医疗领域的应用较为广泛,包括医学影像处理、疾病诊断、药物推荐等。人工智能技术在中药处方人工智能评价方面的研究可以分为三类:第一类是基于中医药学积累的大量先验知识的数据挖掘,第二类是整合先验知识和临床信息的联合推荐;第三类是基于中药成分指纹图谱的中药活性鉴定和匹配。
[0005]
在中医药学先验知识的数据挖掘方面,liang yao[3]等提出了一个从方剂文献中挖掘方剂关系的系统,包括基于trie树建立的方剂组成成分的关系和以及基于主题模型建立的方剂功效关系。wei li[4]等人提出了一种具有覆盖机制和软损失函数的解码器。该研究从中药方剂数据库中抓取了85166张处方,获得82044个症状,验证得到准确率38.22%,召回率30.18%,f1值33.73%。根据中医专家的判断,生成的处方的正确率为73%。jinpeng chen等[5]提出了一种基于三部图的症状-证候-中药关系推断方法。该方法首先构建了一个承载丰富信息的异构三部信息网络,后从该信息网络中系统地提取出基于路径的拓扑特
征,最后用无监督的方法来学习与不同特征相关的最佳参数从而决定症状与中药的关系。在整合先验知识和临床信息的联合推荐方面,杨蕴[6]等使用基于高斯核的岭回归的算法,构建了中医药治疗肺癌处方系统。该处方系统利用2955例次中医肺癌门诊资料完成模型训练,使其能完成较高准确率的处方输出,并最终应用于临床治疗参考。通过专家评估实际的108病例来验证处方的准确率,发现出现频率次数高于300的药物,准确率达到62.9%,召回率80.2%,f1值为70.5%。kuo,yang等[7]提出一个多阶段的分析方法,整合倾向性病例配对、复杂网络分析和中药富集分析,以确定针对特定疾病(例如失眠)的有效药方。首先,应用倾向性病例匹配来匹配临床病例。然后,将核心网络提取和中药富集相结合,对核心有效中药处方进行检测。在基于中药成分指纹图谱的中药活性鉴定和匹配方面,chen h等[8]从中药成分的化学色谱指纹图谱角度出发,通过建立叠加多元线性回归(smlr)方法,从色谱指纹图谱预测中药的生物活性,相比于其他方法具有更好的通用性,能够为中药的精准使用提供支撑。
[0006]
这些方法大多集中在表型层次,其主要特点在于挖掘“症状”和“中药”在文本层次上的关系,缺少分子层次信息。中药处方治疗疾病分子层次上的机制对于精准地推荐中药处方至关重要。因此,提出一种融合表型信息和分子信息的中药处方评价方法是十分必须的。
技术实现要素:
[0007]
针对现有技术存在的不足,本发明提出一种基于深度神经网络的融合表型和分子信息的中药处方人工智能评价方法,从而在此基础上合理有效地建立实现中医处方的高精度人工智能评价。
[0008]
为实现上述目的,本发明提供了一种基于深度神经网络的融合表型和分子信息的中药处方人工智能评价方法,其特征在于包括如下步骤:
[0009]
步骤一、通过数学建模实现了诊断描述的数字化和向量化表示,其中诊断描述包括主诉、现病史、舌象和脉象信息,
[0010]
步骤二、通过构建融合分子信息的中药分子异构网络,使用网络嵌入表示实现对异构网络的特征智能提取,获得中药的低维向量特征,进而实现了中药处方的向量化表示,
[0011]
步骤三、进行训练集和测试集的划分,其中训练集和测试集的划分遵循疾病内部相似性原则,既保证诊断描述的相似性也保证中药处方的相似性,
[0012]
步骤四、训练集中药处方的分层采样,其中分层采样使得相似性较高尤其是排名靠前中药都相同但排名靠后中药略有差别的中药处方被纳入推荐范围,并以此在构建的深度神经网络模型中实现处方推荐的回归预测,
[0013]
步骤五、构建神经网络模型并训练该神经网络模型,其中神经网络模型包括3部分:基于卷积神经网络的诊断描述信息的深度特征提取、基于网络嵌入表示的中药处方信息的深度特征提取、以及基于卷积神经网络的中药处方人工智能评价,通过训练神经网络模型使其达到最优,实现给定诊断描述,能够批量、智能地推荐最优中药处方,
[0014]
步骤六、神经网络模型评价。模型评价包括:模型内部评价、和其他基线方法比较。评价指标包括但不限于hit ratio(hr)、auc和spearman相关性等。
[0015]
根据本发明的进一步的实施例的基于深度神经网络的融合表型和分子信息的中
药处方人工智能评价方法的特征包括下列中的至少一种:
[0016]
a)诊断描述主要包括主诉、现病史、舌象和脉象,使用长度不同的卷积核在文本上进行一维卷积,然后将不同长度的卷积核提取到的特征拼接在一起,作为一段文本的特征输入到神经网络模型中进行训练。
[0017]
b)中药处方信息的深度特征提取所使用的方法主要包括低维嵌入表示(networkembedding)的方式,包括:从公开的数据库搜集中药、化合物、靶点信息,构建中药-化合物-靶点异构网络;使用低维嵌入表示方法对异构网络进行低维嵌入表示,提取中药、化合物、靶点的特征;在通过低维嵌入表示的方式度量了中药的特征之后,进一步度量中药处方的特征,其中,中药处方的特征被定义为该处方所含中药各个维度上值的均值,即:假设中药处方中含有m个中药,每个中药特征的维度为d,则中药处方的特征表示为:
[0018][0019]
c)所述训练集和测试集的划分遵循疾病内部相似性原则。每种疾病训练集占比0.9,测试集占比0.1。针对每一条测试集数据,保证在当前疾病的训练集中至少有一条数目满足诊断描述相似性大于等于0.7并且中药处方相似性大于等于0.7。
[0020]
d)每一条训练集数据主要包含三方面信息:诊断描述,疾病和中药处方。我们将当前中药处方和当前疾病的其他中药处方计算jaccard相似性。然后根据jaccard值进行分层采样。jaccard的值分布在0-1之间,我们将0-1切分为20等长小区间。在每个小区间上进行采样并且采样量和当前疾病的样本量占总样本量的比例成正比:即:
[0021][0022]
具体的采样规则为:k=50,且设这个小区间上的中药处方量为x,如果x≥s,则不放回随机抽样s个中药处方;如果0《x《s,则x全部被采样,并且通过倒序依次删减当前中药处方尾部的中药产生新的s-x个中药处方。如果x=0,则通过倒序依次删减当前中药处方尾部的中药产生新的s个中药处方。采样完成之后的训练集为635120。通过该策略,即实现了大幅度扩充训练样本,也能够捕捉同一电子病历的“次优”推荐中药处方信息。
[0023]
e)所述神经网络模型包括3部分:基于卷积神经网络的诊断描述信息的深度特征提取,基于网络嵌入表示的中药处方信息的深度特征提取,以及基于卷积神经网络的中药处方人工智能评价。其中,根据本发明的一个实施例,(1)基于卷积神经网络的诊断描述信息的深度特征提取包括:诊断描述先经过一个嵌入层,嵌入层的维度为100,然后分别经过三个单元数各为16的一维卷积层,卷积核的长度分别为6,7,8,步长为10。每个卷积层后面连接一个一维maxpooling层,这三个maxpooling提取的特征拼接在一起作为诊断描述的特征;(2)基于网络嵌入表示的中药处方信息的深度特征提取包括:中药处方的特征经过网络嵌入方法提取特征后,长度归一化到256,依次经过两个长度分别为128,64的全连接层,激活函数都为relu;(3)基于卷积神经网络的中药处方人工智能评价包括:诊断描述的特征和中药处方的特征拼接在一起之后,依次经过两个单元数为32一维卷积层和maxpooling层,最后输出到两个单元数分别为32和16的全连接层,激活函数都为relu,输出层单元数为1,
[0024]
根据本发明的一个方面,提供了一种基于深度神经网络的融合表型与分子信息的
中药处方人工智能评价方法,其特征在于包括如下步骤:
[0025]
1)提取诊断描述的特征,其中:
[0026]
诊断描述包括主诉、现病史、舌象和脉象,
[0027]
诊断描述的特征提取采取是基于textcnn,包括使用长度不同的卷积核在文本上进行一维卷积,然后将不同长度的卷积核提取到的特征拼接在一起,作为一段文本的特征输入到网络中进行训练,
[0028]
2)提取中药处方信息的深度特征,包括:
[0029]
从公开的数据库搜集中药、化合物、靶点信息,构建中药-化合物-靶点异构网络;使用低维嵌入表示方法对异构网络进行低维嵌入表示,提取中药、化合物、靶点的特征;在通过低维嵌入表示的方式度量了中药的特征之后,进一步度量中药处方的特征,其中,中药处方的特征被定义为该处方所含中药各个维度上值的均值,即:假设中药处方中含有m个中药,每个中药特征的维度为d,则中药处方的特征表示为:
[0030][0031]
3)划分训练集和测试集,其中:
[0032]
训练集和测试集的划分遵循疾病内部相似性原则从而既保证诊断描述的相似性也保证中药处方的相似性,包括:首先使用doc2vec训练所有的诊断描述,从而能够度量任何两个诊断描述之间的相似性;然后,使用jaccard度量任意两个中药处方之间的相似性;最后,设定每种疾病训练集占比0.9,测试集占比0.1,针对每一条测试集数据,保证在当前疾病的训练集中至少有一条数目满足诊断描述相似性大于等于0.7并且中药处方相似性大于等于0.7,
[0033]
4)对训练集的中药处方进行分层采样,其中:
[0034]
每一个样本包含三方面信息:诊断描述,疾病和中药处方,包括:将当前中药处方和当前疾病的其他中药处方计算jaccard相似性;然后根据jaccard的值进行分层采样,其中jaccard的值分布在0-1之间;将该0-1切分为20等长小区间,在每个小区间上进行采样并且采样量和当前疾病的样本量占总样本量的比例成正比,即:
[0035][0036]
其中具体的采样规则为:k=50,且设这个小区间上的中药处方量为x,如果x≥s,则不放回随机抽样s个中药处方;如果0《x《s,则x全部被采样,并且通过倒序依次删减当前中药处方尾部的中药产生新的s-x个中药处方,如果x=0,则通过倒序依次删减当前中药处方尾部的中药产生新的s个中药处方;从而通过该策略即实现了大幅度扩充训练样本也能够捕捉同一诊断描述的“次优”中药处方信息,
[0037]
5)构建神经网络模型并进行训练,其中:
[0038]
神经网络模型分为3部分:
[0039]
基于卷积神经网络的诊断描述信息的深度特征提取,其中:诊断描述先经过一个嵌入层,嵌入层的维度为100,然后分别经过三个单元数为16的一维卷积层,卷积核的长度分别为6,7,8,步长为10,每个卷积层后面连接一个一维maxpooling层,三个maxpooling提
取的特征拼接在一起作为诊断描述的特征,
[0040]
基于网络嵌入表示的中药处方信息的深度特征提取,其中:中药处方的特征经过网络嵌入方法提取特征后,长度归一化到256,依次经过两个长度分别为128,64的全连接层,激活函数都为relu,
[0041]
基于卷积神经网络的中药处方人工智能评价,其中:诊断描述的特征和中药处方的特征拼接在一起之后,依次经过两个单元数为32一维卷积层和maxpooling层,最后输出到两个单元数分别为32和16的全连接层,激活函数都为relu,输出层单元数为1,
[0042]
6)确定神经网络模型评价指标并进行评价,其中:
[0043]
评价指标包括命中率hr和接收者操作特征曲线roc的曲线下方的面积大小auc,包括:
[0044]
按如下公式确定命中率hr:
[0045][0046]
其中,分母gt是所有的测试集合,分子numberofhits表示命中的样本个数,
[0047]
接收者操作特征曲线roc曲线的横轴为假正例率fpr,纵轴为真正例率tpr,其表达公式分别为:
[0048][0049][0050]
其中,fp为假阳性率,tp为真阳性率,tn为真阴性率,
[0051]
评价的方式为命中率hr和/或auc越高则模型越好,评价过程包括:
[0052]
按照上述的命中率hr公式直接计算命中率hr,
[0053]
auc的计算过程包括:
[0054]
把每个诊断描述和当前诊断描述对应的疾病的所有中药处方进行预测,从而每个诊断描述都有一个已知标签向量,一个预测的分数向量,以及当前中药处方和当前诊断描述对应的疾病的所有中药处方的jaccard相似性向量,按预测的分数对样本进行降序排序,
[0055]
对于不设定jaccard阈值的情况,直接依据已知标签向量和预测的分数向量计算tpr和fpr,对于设定jaccard阈值的情况,将jaccard相似性向量从上到下按照jaccard阈值进行划分,把jaccard相似性大于jaccard阈值的样本归为预测正确的样本,把jaccard相似性小于阈值的样本归为预测错误的样本,分别计算出此时的tpr和fpr,从而确定auc。
附图说明
[0056]
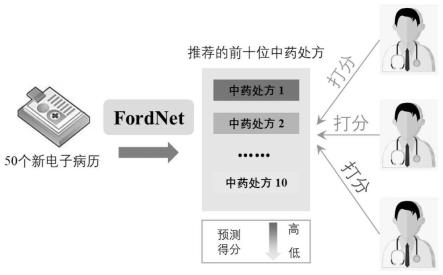
图1为中医专家评测的原理图;
[0057]
图2为对采用本发明的基于深度神经网络的融合表型与分子信息的中药处方人工智能评价方法获得的处方进行的中医专家打分的均值分布图;
[0058]
图3为对采用本发明的基于深度神经网络的融合表型与分子信息的中药处方人工智能评价方法获得的处方进行的中医专家打分的命中率图。
具体实施方式
[0059]
本发明实施例以国医大师中药处方的人工智能评价为例,设计并实现了融合表型信息和分子信息的中药处方人工智能评价方法。我们收集了安徽皖南医学院弋矶山医院2013-2020年3月的超过20000个国医大师电子病历。通过定义一系列规则,精选出了10种疾病6393个国医大师电子病历作为原始数据。10种疾病包括月经过少,虚劳,内科癌病,胃脘痛,乳痈病,风湿痹病,胃痞,咳嗽,痹病,不寐。每个样本由诊断描述、疾病和对应的中药处方构成。大部分样本的诊断结果50到200个中文字符,大部分中药处方包括10-25个中药。国医大师诊疗处方信息主要包括三部分:诊断描述、疾病和中药处方。实施步骤主要包括:诊断描述的特征提取,中药处方的特征提取,训练集和测试集的划分,训练集中药处方的分层采样,构建神经网络模型并训练,神经网络模型评价。具体实施例对本发明进行详细说明。
[0060]
实施例:
[0061]
根据本发明的一种基于深度神经网络的融合表型和分子信息的中药处方人工智能评价方法包括如下步骤:
[0062]
一、提取诊断描述的特征
[0063]
诊断描述主要包括主诉、现病史、舌象和脉象。诊断描述的特征提取采取的思想是基于textcnn[9]。主要是使用长度不同的卷积核在文本上进行一维卷积,然后将不同长度的卷积核提取到的特征拼接在一起,作为一段文本的特征输入到网络中进行训练。在输入到模型之前,需要提取诊断描述的特征,本实施例我们使用的是keras自带的tokenizer工具。tokenizer可以将文本转换为序列,即单词在字典中的下标构成的列表,从而实现诊断描述的数字化表示。除此之外,tokenizer还支持将不等长的多个诊断描述填充到等长,以便于模型的统一使用。本实施例中,诊断描述的最大长度为411,因此所有的诊断结果都在末尾填充0到411。
[0064]
二、提取中药处方的特征
[0065]
中药处方的特征提取使用的方法主要是低维嵌入表示(network embedding)的方式。中药处方由若干中药按照“君臣佐使”的配伍规律构成,并且每味中药都有特定含量。本实施例不考虑中药的用量信息,仅考虑中药处方包含了哪些中药,并重点纳入表型信息和分子信息。本实施例中,发明人从公开的数据库tcmid[10]、hit[11]、symmap[12]、组内自建数据库herbbiomap[13]搜集了中药、化合物、靶点信息,分别构建了中药-化合物,化合物-靶点网络。使用课题组自主开发的方法构建了中药-中药网络[14]。从pubchem数据库提取了化合物相似性数据,设定化合物相似性阈值为大于等于90,从而构建了化合物-化合物网络。从蛋白质相互作用数据库hprd(release 9)[15],biogrid(2019update)[16],intact[17],mint(2012update homosapiens)[18]和string(v10.5)[19]提取数据,构建靶点-靶点网络。在以上数据收集的基础上构建了中药-化合物-靶点异构网络。使用node2vec[20]对异构网络进行低维嵌入表示,提取中药、化合物、靶点的特征。在通过低维嵌入表示的方式度量了中药的特征之后,便可以度量中药处方的特征。中药处方的特征定义为该处方所含中药各个维度上值的均值。即:假设中药处方中含有m个中药,每个中药特征的维度为d,则中药处方的特征表示为:
[0066]
[0067]
三、划分训练集和测试集
[0068]
训练集和测试集的划分遵循疾病内部相似性原则,既保证诊断描述的相似性也保证中药处方的相似性。首先使用doc2vec[21]训练所有的诊断描述,从而能够度量任何两个诊断描述之间的相似性。然后,使用jaccard度量任意两个中药处方之间的相似性。最后,设定每种疾病训练集占比0.9,测试集占比0.1,针对每一条测试集数据,保证在当前疾病的训练集中至少有一条数目满足诊断描述相似性大于等于0.7并且中药处方相似性大于等于0.7。总的样本量为6393条,划分测试集和训练集后,训练集5757条,测试集636条。
[0069]
四、对训练集的中药处方进行分层采样
[0070]
我们认为国医大师开具的中药处方是最优的,但是当前疾病的其他中药处方也不能完全排除。中药处方的开具遵循“君臣佐使”的配伍规律,且在中药处方中排序约靠后的中药,一般来说重要性越低。因此,我们希望设计一种规则,使得相似性较高尤其是排名靠前中药都相同但排名靠后中药略有差别的中药处方也纳入推荐范围,并给与这种中药处方一种特定的分数来进行度量。我们设计的规则称为“中药处方的分层采样”,主要针对训练集进行。每一个样本主要包含三方面信息:诊断描述,疾病和中药处方。我们将当前中药处方和当前疾病的其他中药处方计算jaccard相似性。然后根据jaccard值进行分层采样。jaccard的值分布在0-1之间,我们将0-1切分为20等长小区间。在每个小区间上进行采样并且采样量和当前疾病的样本量占总样本量的比例成正比:即:
[0071][0072]
具体的采样规则为:k=50,且设这个小区间上的中药处方量为x,如果x≥s,则不放回随机抽样s个中药处方;如果0《x《s,则x全部被采样,并且通过倒序依次删减当前中药处方尾部的中药产生新的s-x个中药处方。如果x=0,则通过倒序依次删减当前中药处方尾部的中药产生新的s个中药处方。采样完成之后的训练集为635120。通过该策略,即实现了大幅度扩充训练样本,也能够捕捉同一诊断描述的“次优”中药处方信息。
[0073]
五、构建神经网络模型并进行训练
[0074]
神经网络模型主要分为3部分:基于卷积神经网络的诊断描述信息的深度特征提取,基于网络嵌入表示的中药处方信息的深度特征提取,以及基于卷积神经网络的中药处方人工智能评价。(1)基于卷积神经网络的诊断描述信息的深度特征提取:诊断描述先经过一个嵌入层,嵌入层的维度为100。然后分别经过三个单元数为16的一维卷积层,卷积核的长度分别为6,7,8,步长为10。每个卷积层后面连接一个一维maxpooling层。三个maxpooling提取的特征拼接在一起作为诊断描述的特征。(2)基于网络嵌入表示的中药处方信息的深度特征提取:中药处方的特征经过网络嵌入方法提取特征后,长度归一化到256,依次经过两个长度分别为128,64的全连接层,激活函数都为relu。(3)基于卷积神经网络的中药处方人工智能评价:诊断描述的特征和中药处方的特征拼接在一起之后,依次经过两个单元数为32一维卷积层和maxpooling层,最后输出到两个单元数分别为32和16的全连接层,激活函数都为relu,输出层单元数为1。具体而言:
[0075]
每个诊断描述都由若干个字符组成,经过embedding层之后,每个字符的维度为d=100,假设诊断描述包含的字符数为n,则每个诊断描述用一个随机初始化的d维向量表示:
[0076][0077]si:j
代表诊断描述中的第i个到第j个字符,即:
[0078][0079]
卷积层包括了不同尺寸的卷积核,每个尺寸都包含大量卷积核。卷积核的宽和s的宽相同,都为d=100。假设第k个卷积核的高为h,则卷积核可以表示为wk=rh×d,即:
[0080][0081]
卷积操作是对s的局部特征提取,我们举例说明卷积操作的过程。当和s
1,1
相遇,提取到的特征为:
[0082][0083]
上式中,s
i,j
是s的第i个字符的第j个维度上的值,是s
i,j
的权重,为偏差项。relu是非线性激活函数:
[0084]
f(x)=max(0,x)
[0085]
卷积操作是wk以一定的步长sc从s的顶部滑动到底部,产生的特征组合为:
[0086][0087]
池化操作和卷积操作类似,唯一的区别是池化操作计算mean或者max值。我们使用max类型的池化操作maxpooling。假设池化核的高度为h
p
,步长为s
p
,则池化操作的输出为:
[0088][0089]
其中,
[0090][0091][0092][0093]
诊断描述经过若干卷积-池化操作,所有卷积-池化操作结束之后,提取到的所有特征按照端到端的方式进行连接,得到:
[0094][0095]
其中,
[0096]
中药处方的特征经过网络嵌入方法提取特征后,长度归一化到256,依次经过两个长度分别为32,64的全连接层,激活函数都为relu。提取到的特征为:
[0097][0098]
将f
t
和c
t
进行拼接得到g,经过一系列卷积-池化操作之后,输入全连接层。全连接层的权复位义为wf,偏差项为bf,全连接层的输出为:
[0099]
y=wf×
g+bf[0100]
若干全连接层之后是最后的输出层,输出层的单元数为1,激活函数为sigmoid:
[0101][0102]
损失函数定义为:
[0103][0104]
损失函数包括两部分:误差项和正则项。λ为正则项系数。为样本的均方误差mse,定义为:
[0105][0106]
以上训练过程为一批次的样本,n为批次的大小。向量y
predicted
为模型的输出,即诊断描述和中药处方的预测分数,y
predictedi
为第i个诊断描述-中药处方组合的值。y
real
的结构和y
predicted
相同,表示诊断描述和中药处方的关系强弱。
[0107]
训练过程使用adam算法,权重的更新规则为:
[0108][0109][0110][0111][0112][0113]
其中,t是训练步数,η为学习率,∈=10e-8,β1和β2分别为梯度和二阶梯度的遗忘因子。全连接层的dropout设置为0.0005。训练的epoch为1500,学习率为1e-4,batch size为256。
[0114]
六、对神经网络模型进行评价
[0115]
神经网络模型评价包括三部分:模型内部评价、和其他方法比较、专家评审。评价指标主要是命中率hit ratio(hr)和接收者操作特征曲线receiver operatingcharacteristic curve(roc)曲线下方的面积大小area under curve(auc)。
[0116]
其中,hr是一种常用的衡量召回率的指标,计算公式为:
[0117][0118]
分母是所有的测试集合,分子表示测试集合的个数。
[0119]
roc曲线的横轴为假正例率fpr,纵轴为真正例率tpr。
[0120][0121][0122]
其中,fp为假阳性率,tp为真阳性率,tn为真阴性率,
[0123]
评价的方式为命中率hr和/或auc越高则模型越好,评价过程包括:
[0124]
按照上述的命中率hr公式直接计算命中率hr,
[0125]
auc的计算过程包括:
[0126]
把每个诊断描述和当前诊断描述对应的疾病的所有中药处方进行预测,从而每个诊断描述都有一个已知标签向量,一个预测的分数向量,以及当前中药处方和当前诊断描述对应的疾病的所有中药处方的jaccard相似性向量,按预测的分数对样本进行降序排序,
[0127]
对于不设定jaccard阈值的情况,直接依据已知标签向量和预测的分数向量计算tpr和fpr,对于设定jaccard阈值的情况,将jaccard相似性向量从上到下按照jaccard阈值进行划分,把jaccard相似性大于jaccard阈值的样本归为预测正确的样本,把jaccard相似性小于阈值的样本归为预测错误的样本,分别计算出此时的tpr和fpr,从而确定auc。
[0128]
在模型内部评价方面,除了node2vec来融入分子信息之外,我们还尝试了其他方法,主要包括line[22],sdne[23],以及不加分子信息。与不加分子信息的模型比,加入分子信息后,能够显著提升预测效果(表1)。通过比较fordnet
node2vec
、fordnet
line
、fordnet
sdne
以及fordnet
no molecule
四种模型的命中率和auc发现,通过fordnet line
添加分子信息之后能够最大幅度地提高中药处方推荐的top1,top5,top10和top50。相比于fordnet
no molecule
,fordnet
line top1提高24.24%,top5提高20.40%,top10提高17.28%,top50提高了9.24%。
[0129]
在和其他方法比较方面,我们比较了基线方法svm,random forest,linearregression(表1)。在不设定fst的情况下,fordnet
line
取得了最高的命中率,并且fordnet
line
和fordnet
no molecule
均要高于基线方法svm,random forest,linearregression。在不设定fst的情况下,fordnet
line
也取得了最高的auc(0.813),同样地,fordnet
line
和fordnet
no molecule
的auc也都要高于基线方法svm(auc=0.563),random forest(auc=0.751),linear regression(auc=0.513)。
[0130]
表1.不同方法性能对比
computing,f,2019[c].springer.
[0138]
[5]jinpeng,chen,josiah,et al.mining symptom-herb patterns from patient records using tripartite graph[j].evid-based compl alt,2015,2015:1-14.
[0139]
[6]杨蕴,阮春阳,裴朝翰,et al.引入人工智能构建肺癌中医处方系统探索[j].世界科学技术—中医药现代化,2019,21(5):977-982.
[0140]
[7]yang k,zhang r,he l,et al.multistage analysis method for detection of effective herb prescription from clinical data[j].front med,2018,12(2):206-217.
[0141]
[8]chen h,poon j,poon s k,et al.ensemble learning for prediction of the bioactivity capacity of herbal medicines from chromatographic fingerprints[j].bmc bioinformatics,2015,16(suppl 12):s4.
[0142]
[9]kim y.convolutional neural networks for sentence classification[j].arxiv preprint arxiv:14085882,2014:
[0143]
[10]lin h,xie d,yu y,et al.tcmid 2.0:a comprehensive resource for tcm[j].nucleic acids research,2018,(d1):d1117-d1120.
[0144]
[11]hao y,li y,hong k,et al.hit:linking herbal active ingredients to targets[j].nucleic acids research,2011,39(suppl_1):d1055
–
d1059.
[0145]
[12]wu y,zhang f,yang k,et al.symmap:an integrative database of traditional chinese medicine enhanced by symptom mapping[j].nucleic acids research,2018,47(d1):d1110
–
d1117.
[0146]
[13]欧阳子博.herbbiomap2.0数据库平台构建与挖掘[d];清华大学.
[0147]
[14]li s,zhang b,jiang d,et al.herb network construction and co-module analysis for uncovering the combination rule of traditional chinese herbal formulae[j].bmc bioinformatics,2010,11(suppl 11):s6.[15]keshava prasad t s,goel r,kandasamy k,et al.human protein reference database
‑‑
2009update[j].nucleic acids res,2009,37(suppl_1):d767-d772.
[0148]
[16]rose o,chris s,bobby-joe b,et al.the biogrid interaction database:2019update[j].nucleic acids res,2018,47(d1):d529
–
d541.
[0149]
[17]samuel k,bruno a,lionel b,et al.the intact molecular interaction database in 2012[j].nucleic acids res,2011,40(d1):d841
–
d846.
[0150]
[18]luana l,leonardo b,daniele p,et al.mint,the molecular interaction database:2012update[j].nucleic acids res,2012,40(d1):d857
–
d861.
[0151]
[19]damian s,morris j h,helen c,et al.the string database in 2017:quality-controlled protein
–
protein association networks,made broadly accessible[j].nucleic acids res,2016,45(d1):d362
–
d368.
[0152]
[20]grover a,leskovec j.node2vec:scalable feature learning for networks;proceedings of the the 22nd acm sigkdd international conference,f,2016[c].
[0153]
[21]le q,mikolov t.distributed representations of sentences and documents;proceedings of the international conference on machine learning,f,2014[c].
[0154]
[22]tang j,qu m,wang m,et al.line:large-scale information network embedding;proceedings of the proceedings of the 24th international conference on world wide web,f,2015[c].
[0155]
[23]wang d,peng c,zhu w.structural deep network embedding;proceedings of the acm sigkdd international conference on knowledge discovery&data mining,f,2016[c].
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1